刺参Apostichopus japonicus隶属于棘皮动物门Echinodermata海参纲Holothuroidea楯手目Aspidochirota,是无脊椎动物中与脊索动物门Chordata种类最为接近的后口类动物类群,其所处分类地位十分特殊[1]。刺参广泛分布于西北太平洋沿岸,是中国黄渤海重要的海产经济生物[2]。当受到敌害攻击或不良自然环境影响时,如水温骤变、水质恶化、海水中病原菌大量滋生等,刺参会表现出特殊的防御机制——“吐脏”,即将大部分消化道和与之相连的器官(呼吸树、性腺、血管系统等)通过泄殖腔破裂的方式排出体外,而刺参吐出消化道后,在食道与泄殖腔的残肢部位留下2个断面[3],经过一定时间的再生便可形成一套完整的内脏器官。一般认为,在海参内脏再生过程中既涉及变形再生,又涉及新建(发育)再生[4]。迄今为止,关于海参再生的研究主要集中在组织形态和分子水平。
多功能表皮生长因子(multiple epidermal growth factor,MEGF)是通过检测蛋白N-末端区域的EGF样结构域时而被发现的[5],其具有与表皮生长因子(EGF)相近的结构和功能特征[6],即都具有EGF样结构域。该结构域是进化上非常保守的模块化蛋白亚单位,一般由30~40个氨基酸残基构成[7]。已有研究证明,MEGF在胚胎发育、机体内环境稳定过程中具有重要作用,表现在调控细胞增殖、分化、凋亡、黏附、迁移等[8-9]。早期研究中已鉴定出9个megf基因家族成员(megf1~megf9),在后期高通量筛选中又发现了3个(megf10、megf11、megf12)[10-11]。MEGF蛋白家族具有相对分子量大(>100 000)的特点,家族中MEGF1~MEGF3和MEGF7~MEGF12是膜锚定蛋白,MEGF4~MEGF6为分泌蛋白[12]。截至目前,多种生物的megf基因家族的mRNA序列已经被得到,但关于刺参中megf的研究报道非常少。基于本实验室转录组数据分析结果发现,megf6在刺参肠再生的第5、10天时表达上调,结合megf6具有调控组织细胞生长和迁移功能的已有研究结果[13],推断megf6可能与刺参再生有一定关系。
MEGF6,又名EGFL3,是一种具有30余个egf样结构域的大分子量蛋白,megf6基因定位于人类1号染色体(1p36.32)[5]。已有研究发现,megf6基因敲除的小鼠是可正常发育的,但表达megf6的细胞可以参与皮肤表皮、大脑和肋骨的组分形成[14];megf6在成骨样细胞中表达,并能作为血管生成因子促进血管生成[8];也有研究证明,MEGF6与神经系统的紊乱发生有关[15]。本研究中,采用cDNA末端快速扩增技术(RACE)和实时定量PCR技术(Real-time quantitative PCR,qPCR)首次获得刺参megf6基因(简称为Aj-megf6)的cDNA全长序列,分析了基因的序列信息及其编码蛋白的结构特征,并探究了megf6在刺参肠再生过程中的表达变化,以期确定其生物学功能,为丰富megf6基因的研究提供基础资料。
1 材料与方法
1.1 材料
试验用刺参购于大连市新长兴水产品市场,体质量为(92.0±4.0)g,饲养于大连海洋大学实验室玻璃钢水槽(85 cm×45 cm×45 cm)中,水温为(16.0±0.5)℃,盐度为30.5±1.0,pH为8.10±0.05,24 h不间断充气,每天换水一次,换水量为总水体的1/3。试验前暂养10 d,暂养期间和吐脏再生10 d后,每天投喂有机底泥和藻粉混合物(体积比1∶1)一次,投喂量为刺参体质量的5%。
1.2 方法
1.2.1 样品的制备 暂养结束后取5头刺参解剖,取其肠组织,置于超低温冰箱(-80 ℃)中保存,用于基因克隆所需的总RNA提取。另取75头刺参通过体腔注射2 mL、0.35 mol/L KCl溶液,刺激排出内脏后用于再生试验。刺参吐脏后24 h为再生1 d,在再生3、6、9、12、15、18、21 d时,分别取5头刺参解剖,观察肠的形态变化后取出再生肠,置于超低温冰箱(-80 ℃)中保存,用于qPCR所需的总RNA提取。
1.2.2 总RNA的提取及cDNA的获得 采用Trizol 试剂盒(北京天根生化科技有限公司),按照产品说明书进行各时期肠样品的总RNA提取,并按照柳林等[16]的方法对总RNA的完整性进行检测,并将完整性好的总RNA样品在oligo(dT)引物和MLV反转酶(大连TaKaRa公司)参与下进行反转录反应,获得cDNA样品,并于冰箱(-20 ℃)中保存备用。
1.2.3 Aj-megf6基因EST序列的获得 从NCBI(National Center for Biotechnology Information)中下载刺参megf基因家族的EST序列,并利用Primer Premier 6.0软件设计上下游引物,交由上海捷瑞生物工程有限公司进行引物合成。本试验所用引物序列见表1。PCR反应体系(20 μL):ddH2O 12.8 μL,cDNA样品1 μL,10×Easy Taq Buffer(+Mg2+)2 μL,dNTP Mixture 2 μL,上、下游引物各1 μL,Easy Taq DNA Polymerase 0.2 μL。PCR反应条件为:94 ℃下预变性1 min;94 ℃下变性30 s,49 ℃下退火复性30 s,72 ℃下延伸1 min,共32个循环;最后在72 ℃下再延伸10 min。利用10 g/L琼脂糖凝胶电泳检测PCR产物的质量;利用DNA回收试剂盒(北京索莱宝科技有限公司)回收、纯化PCR产物中的目标片段,并与pEASY-T1载体(北京索莱宝科技有限公司)连接后,转入Trans5α感受态细胞(北京全式金生物技术有限公司)中,先后在液体、固体培养基中扩繁,挑取单个菌落,使用M13上、下游引物(表1)进行菌液PCR,产物经琼脂糖凝胶电泳检测后,将阳性克隆样品送至生工生物工程(上海)股份有限公司进行测序,将测序结果于NCBI在线工具中进行比对,得到Aj-megf6基因的EST序列。
1.2.4 Aj-megf6基因cDNA全长序列的获得 以获得的megf6基因EST序列为基础,利用Primer Premier 6.0软件设计5′、3′ RACE引物(表1),并由上海捷瑞生物工程有限公司合成。按照SMARTer® RACE 5′/3′试剂盒(Clontech实验室,美国)说明书进行5′、3′ RACE模板的制备。RACE反应体系(50 μL):5′或3′ RACE模板2.5 μL,UPM(10×)5.0 μL,5′或3′引物1.0 μL,ddH2O 34.5 μL,Advantage 2 PCR Buffer(10×)5 μL,dNTP(10 mmol/L)1 μL,Advantage 2 Polymerase Mi(50×)1 μL。反应条件:94 ℃下变性5 s,68 ℃下退火复性10 s,72 ℃下延伸3 min,共进行28个循环。PCR产物的检测、回收、纯化及测序方法同EST序列。利用DNAMAN软件对EST、5′端、3′端序列进行拼接,得到Aj-megf6基因的cDNA全长序列,并利用NCBI中的BLAST工具(http://blast.ncbi.nlm.nih.gov/Blast.cgi)进行比对分析。
表1 Aj-megf6基因克隆和qPCR用引物序列
Tab.1 Nucleotide sequences of primers used for Aj-megf6 genes cloning and for qPCR
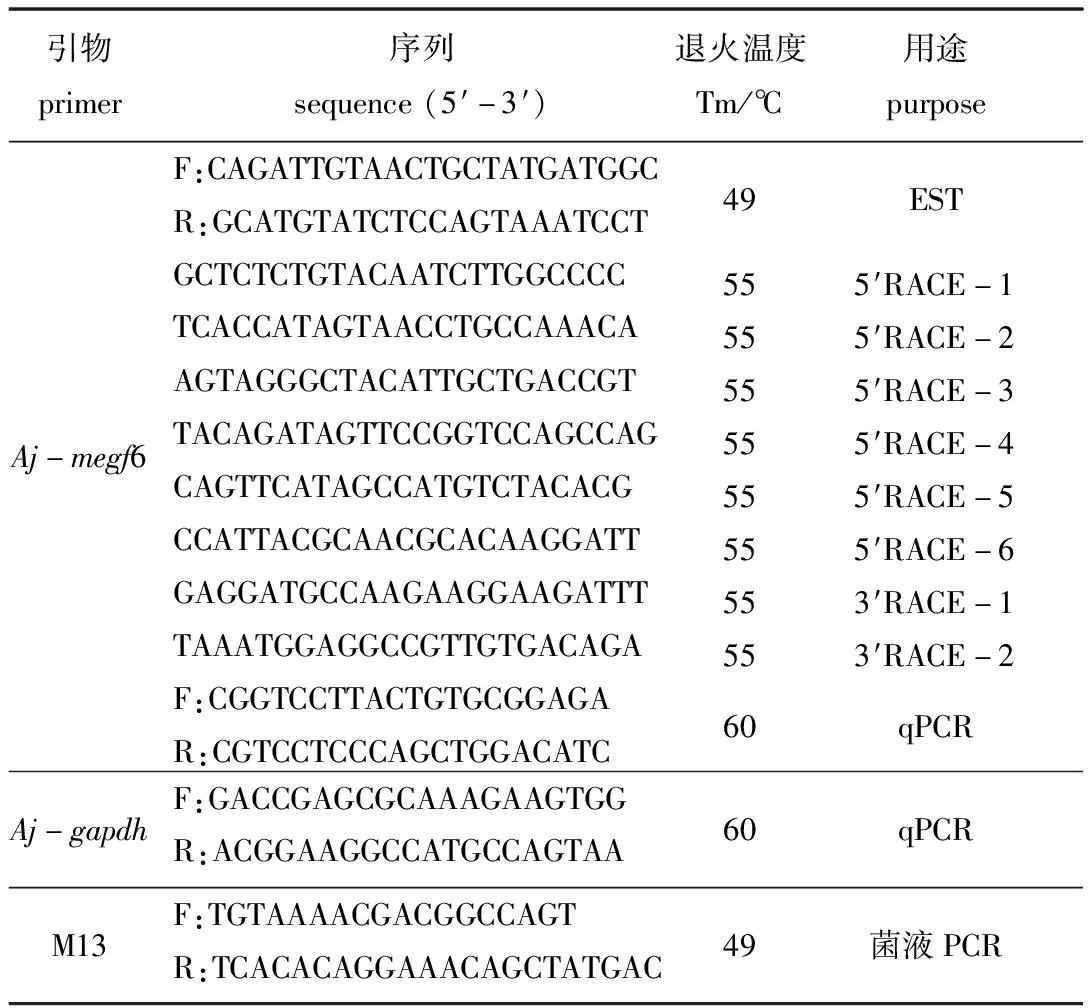
引物primer序列sequence(5′-3′)退火温度Tm/℃用途purposeAj-megf6F:CAGATTGTAACTGCTATGATGGCR:GCATGTATCTCCAGTAAATCCTGCTCTCTGTACAATCTTGGCCCCTCACCATAGTAACCTGCCAAACAAGTAGGGCTACATTGCTGACCGTTACAGATAGTTCCGGTCCAGCCAGCAGTTCATAGCCATGTCTACACGCCATTACGCAACGCACAAGGATTGAGGATGCCAAGAAGGAAGATTTTAAATGGAGGCCGTTGTGACAGAF:CGGTCCTTACTGTGCGGAGAR:CGTCCTCCCAGCTGGACATC49EST555′RACE-1555′RACE-2555′RACE-3555′RACE-4555′RACE-5555′RACE-6553′RACE-1553′RACE-260qPCRAj-gapdhF:GACCGAGCGCAAAGAAGTGGR:ACGGAAGGCCATGCCAGTAA60qPCRM13F:TGTAAAACGACGGCCAGTR:TCACACAGGAAACAGCTATGAC49菌液PCR
1.2.5 Aj-megf6基因的生物学信息分析 生物信息学分析按照孙冉冉等[17]对黄条鰤pten基因生物信息学分析时采用的方法进行。利用ClustalX和 DNAMAN 9.0软件对Aj-MEGF6和选取的其他18个物种MEGF6的氨基酸序列、MEGF家族中39条氨基酸序列进行多序列比对,并利用MEGA 4.1软件构建基于邻接法(Neighbor-Joining, NJ)的系统进化树(重复计算1000次)。
1.2.6 肠再生过程中Aj-megf6的表达分析 提取刺参再生过程中各时期(再生3、6、9、12、15、18、21 d)肠组织的总RNA,反转录获得相应的cDNA样品,并稀释浓度至100 ng/μL。按照SYBR Premix Ex Taq试剂盒(大连TaKaRa公司)说明书进行qPCR,检测Aj-megf6基因的表达水平。以正常未吐脏刺参的肠为对照组,选取刺参的gapdh基因为内参基因(表1),Aj-megf6基因的扩增片段长度为171 bp,gapdh基因的扩增片段长度为134 bp。反应体系(25 μL):SYBR® Premix Ex TaqTM(2×)12.5 μL,Forward Primer 0.5 μL,Reverse Primer 0.5 μL,cDNA样品2.0 μL,ddH2O 9.5 μL。使用Applied Biosystems StepOnePlus Real-time PCR System、两步法PCR扩增标准程序,第1步,95 ℃下预变性30 s;第2步,95 ℃下变性5 s,60 ℃下退火30 s,共进行40个循环。每个样品设3个重复。采用2-ΔΔCt法计算相对表达量。
1.3 数据处理
试验数据以平均值±标准差(mean±S.D.)表示,利用SPSS 19.0软件对数据进行单因素方差分析,用 LSD法进行多重比较,显著性、极显著性水平分别设为0.05、0.01。
2 结果与分析
2.1 Aj-megf6全长cDNA序列和生物信息学分析
刺参megf6基因cDNA全长序列为5665 bp,由389 bp的5′端非编码区(5′ Untranslated region, UTR)、4815 bp的开放阅读框序列和461 bp的3′端非编码区组成。开放阅读框序列共编码1604个氨基酸,具有37个EGF样结构域(图1)。利用ProtParam tool of ExPASy分析预测Aj-MEGF6蛋白的相对分子质量为172 500、等电点为4.74。对该蛋白的亲、疏水性预测分析得到,其平均值为-0.486(最大值3.389,最小值-2.700),表明该蛋白可能是亲水性蛋白。用NetPhos 3.1 Server分析预测该蛋白共有53个磷酸化位点,其中有23个Ser位点、12个Thr位点、18个Tyr位点。Aj-MEGF6蛋白具有信号肽,为分泌蛋白,但不具有跨膜结构,属于非跨膜蛋白。Aj-MEGF6蛋白的二级结构预测显示,有173个α螺旋、286个延伸链、197个β转角和948个无规则卷曲。
2.2 Aj-MEGF6序列同源性比对及系统进化树构建
通过多序列比对,刺参与其他18个物种的MEGF6氨基酸序列的一致性为38.59%~49.07%,其中与紫球海胆S.purpuratus的一致性最高,为49.07%,与长棘海星A.planci次之(48.71%)。利用ClustalX和MEGA 4.1软件,以19种生物的MEGF6氨基酸序列构建了NJ系统进化树(图2),结果显示,19种生物聚为两支,一支为脊椎动物,另一支为无脊椎动物。在无脊椎动物中,刺参与长棘海星、紫球海胆聚为一个分支,这符合刺参为棘皮动物的分类地位。利用Aj-MEGF6与MEGF家族中39条氨基酸序列构建的NJ系统进化树显示,同类MEGF的多个物种聚为一支,表明MEGF家族成员间具有一定的分化(图3)。
2.3 再生过程中肠的形态变化
刺参吐脏后,消化道前、后端分别在食道与胃部连接处、肠与泄殖腔连接处断裂,在体内留下食道及泄殖腔的残体。再生3~21 d时肠管逐渐增长;再生15 d时肠管前后贯通呈直线型,并有食物出现;再生18 d时肠形成绕环现象,外部形态已近似正常刺参状态(图4)。

注:实线方框指示起始密码子(ATG)和终止密码子(TAA);虚线方框指示信号肽;阴影部分为37个EGF样结构域
Note:Solid line boxes indicate the start codon(ATG)and terminal codon(TAA);Dotted line box indicates signal peptide;The 37 EGF domains are shadowed
图1 刺参megf6基因的核苷酸序列及其推测的氨基酸序列
Fig.1 Nucleotide and deduced amino acid sequences of megf6 gene in sea cucumber Apostichopus japonicus
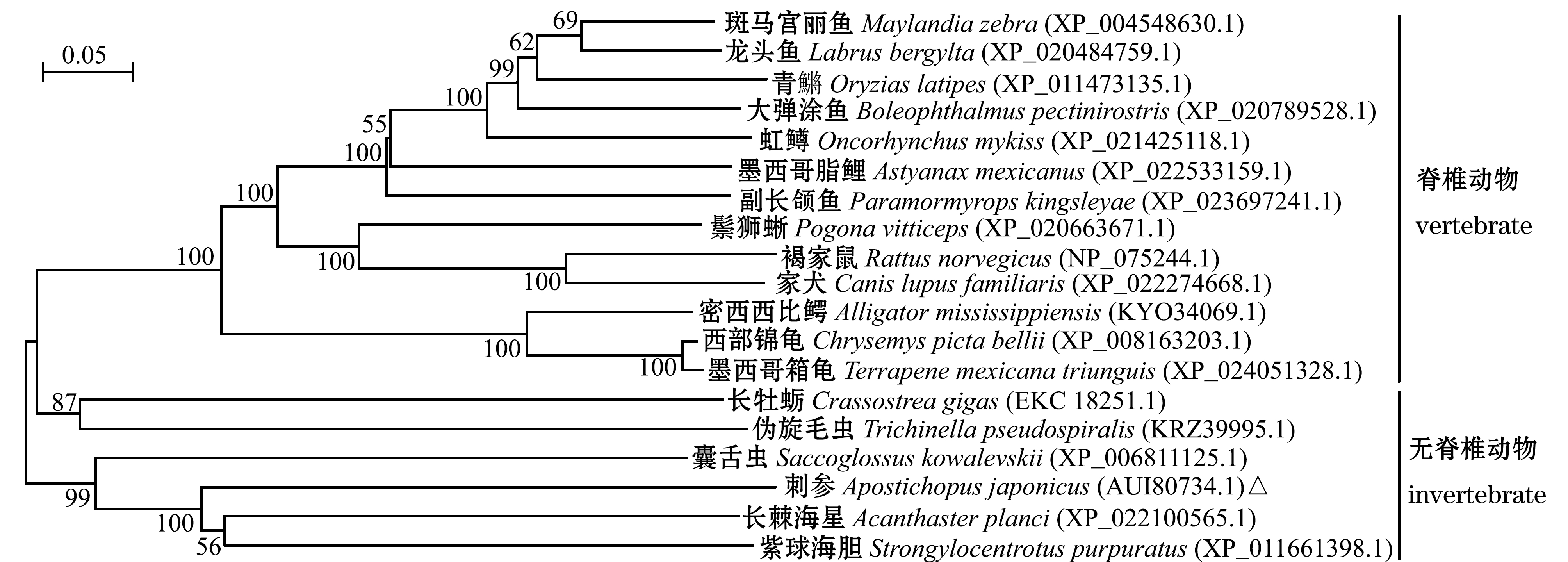
图2 基于19种生物MEGF6氨基酸序列构建的NJ系统进化树
Fig.2 NJ phylogenetic tree of MEGF6 amino acid sequences from 19 species

图3 基于40种生物MEGF氨基酸序列构建的NJ系统进化树
Fig.3 NJ phylogenetic tree of MEGF amino acid sequences from 40 species
2.4 肠再生过程中Aj-megf6基因的表达分析
在刺参肠再生过程中,megf6基因的相对表达量呈先升高后降低的趋势,其中在再生6 d时达到峰值,为对照组的126.47倍;自再生6 d后,表达量呈下降趋势;再生过程中,除了再生18、21 d时megf6基因表达水平与对照组无显著差异外,在其他时期均有显著或极显著性差异(P<0.05或P<0.01)(图5)。
3 讨论
3.1 刺参megf6基因的序列特征
本研究中,利用RACE技术首次克隆得到了成体刺参megf6基因的cDNA全长序列。生物信息学分析显示,该序列中开放阅读框为4815 bp,共编码1604个氨基酸。Aj-MEGF6包含37个EGF样结构域,这与Letunic and Bork利用SMART蛋白质注释工具对MEGF6结构域的分析结果相符合[12]。通过与18种其他生物的MEGF6氨基酸序列比较发现,19种生物的MEGF6氨基酸数量为1379~1717,刺参MEGF6的氨基酸序列与同源性最高的紫球海胆MEGF6相比,多出146个氨基酸,与长棘海星相比,多出225个氨基酸,与脊椎动物的MEGF6相比,氨基酸数量差值为-113~140,未表现出明显规律。通过多序列比对,刺参与其他18种生物的MEGF6氨基酸序列的一致性为38.59%~49.07%,其中与紫球海胆的一致性最高,为49.07%,与长棘海星次之(48.71%);构建的NJ系统进化树显示,刺参与紫球海胆、长棘海星聚为一个分支。这些结果表明,MEGF6在生物进化中具有一定的保守性,并且符合刺参与紫球海胆、长棘海星为棘皮动物的分类地位。MEGF家族成员间比对分析表明,MEGF1~MEGF12间具有比较明显的分化现象。

注:A正常刺参;B再生3 d;C再生6 d;D再生9 d;E再生12 d;F再生15 d;G再生18 d;H再生21 d
Note: A, showing normal individual; B, regeneration in 3 days; C, regeneration in 6 days; D, regeneration in 9 days ; E, regeneration in 12 days; F, regeneration in 15 days; G, regeneration in 18 days; H, regeneration in 21 days
图4 刺参肠再生过程中的形态变化
Fig.4 Morphological changes in intestine regeneration of sea cucumber Apostichopus japonicus
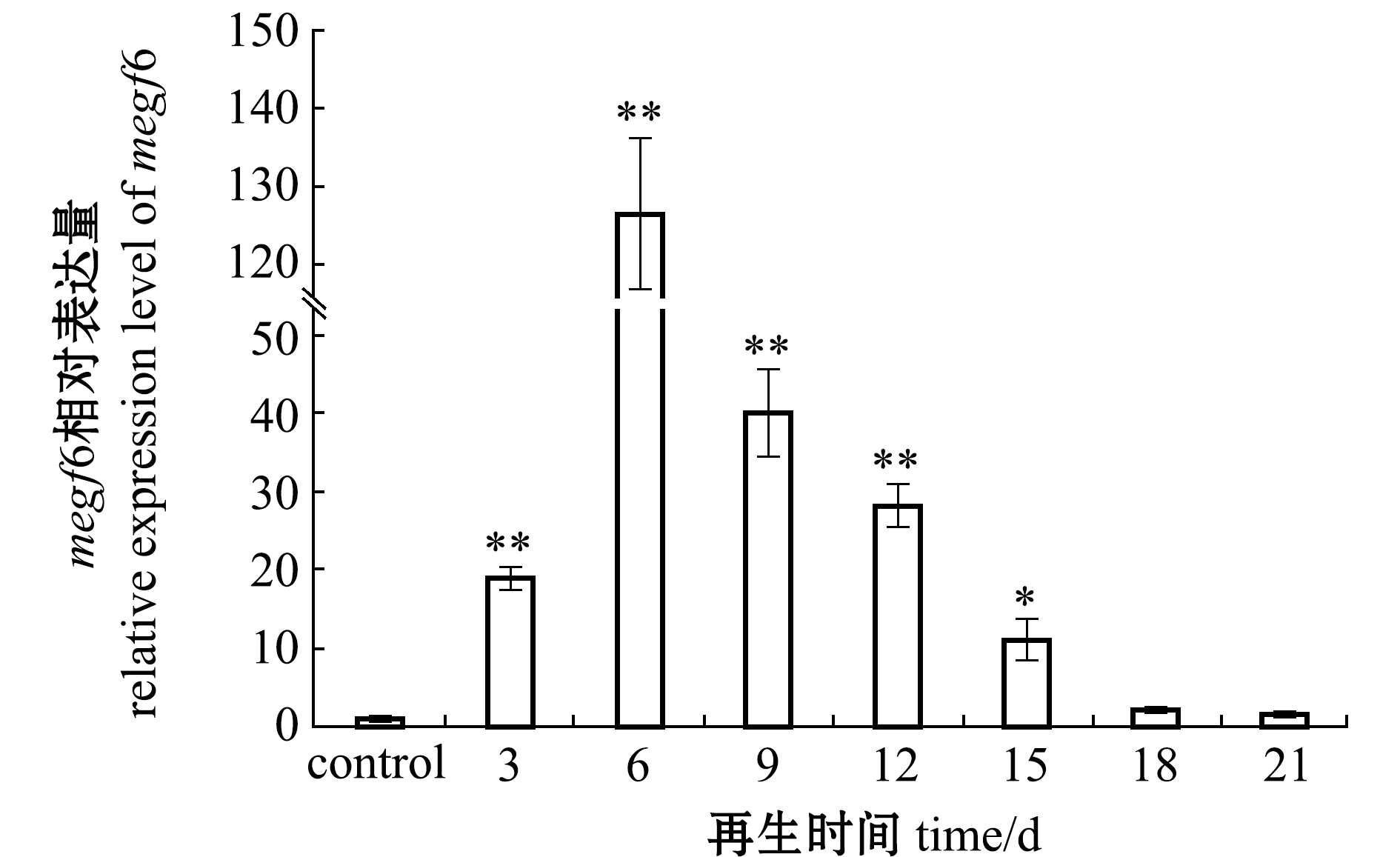
注:*表示与未吐脏的对照组有显著性差异(P<0.05);**表示与未吐脏的对照组有极显著性差异(P<0.01)
Note: *means significant difference compared with the without evisceration control group(P<0.05);**means very significant difference compared with the control group(P<0.01)
图5 肠再生过程中刺参megf6基因的表达水平
Fig.5 Expression levels of megf6 during intestine regeneration in sea cucumber Apostichopus japonicus
3.2 megf6在刺参肠再生过程中的表达模式
再生是一个与发育类似的复杂过程,也是一个多基因调控的生理现象。有关海参吐脏再生的机制目前认为有两种,即变形再生和新建(发育)再生。变形再生指海参排脏后的残留组织自身重建形成新的组织器官,而相应的细胞是通过分化、迁移完成的,不涉及细胞增殖和分裂活动。新建(发育)再生指再生过程中有细胞的分裂、增殖发生,新生细胞取代了原有的部位而形成一个新的胚基[18]。这两种再生机制会因海参种类的不同而表现出差异性,其中刺参的肠再生涉及的机制应是两种相融合的情况[4],而早期以变形再生(迁移)为主,后期以新建再生(增殖)为主[19]。
本研究中发现,megf6基因在刺参肠再生过程中显著上调,其中再生6 d时达到峰值,再生18 d时,其表达水平下降至正常水平。根据王霞等[20]对刺参消化道再生的研究结果,6 d时正是刺参吐脏后,食道与胃部断裂处、肠系膜断裂处的伤口愈合及原基形成阶段,有大量细胞分化和增殖现象发生。另有研究发现,在刺参肠再生第3天时,浆膜层、结缔组织层中增殖细胞比例不到3%,而再生第7天时,细胞增殖现象明显,约占细胞总数的29.5%,再生第14~21天时,细胞分裂活动减弱,肠腔上皮细胞分裂率下降至14.2%[19]。而在另一种海参Holothuria glaberrima的再生研究中也发现类似规律,再生6 d时增殖细胞所占比例最高,为12%,再生12 d时下降至6%,并在再生后期逐渐下降[21]。本研究中,通过形态观察发现,再生过程中的肠管随时间而逐渐延长,再生15 d时再生肠管前后贯通,刺参具有摄食和消化行为;再生18~21 d时肠管进一步生长及复杂化。Aj-megf6基因表达量的变化趋势与肠细胞增殖活动、肠管形态变化相吻合,推断Aj-megf6基因与刺参肠再生活动密切相关。
3.3 megf6在刺参肠再生过程中的作用机制
肠再生过程中,肠系膜的形态变化往往被认为是消化道再生的关键环节[20]。肠再生初期,肠系膜的游离端出现增厚现象,增厚过程涉及体腔上皮细胞的去分化、细胞增殖、再分化[20]。形成肠管的细胞主要是肠腔上皮细胞及肠系膜增厚区的间充质细胞[22]。已有研究表明,megf6具有调控组织细胞生长、迁移、黏附、变性的功能[13,15],其调控过程中涉及的CDH1(E-钙黏蛋白1,cadherin 1)可介导基底膜细胞间的连接,其异常表达会降低细胞间的黏附作用,使细胞易与周围组织分离[23]。CDH1缺失会促进细胞生长、增殖及转移[24],而诱导上皮间充质细胞转化(EMT)是其重要机制之一[25]。已有研究表明,SNAI家族(Snail and Slug)是CDH1的抑制因子之一[26-27]。megf6基因敲除可以显著抑制SNAI2的mRNA表达,而且癌症基因组图谱(TCGA)显示,megf6 mRNA的表达与SNAI2的表达呈正相关关系[28]。megf6可以通过转化生长因子β(TGFβ)/SMAD信号途径介导,下调Slug而抑制EMT;当megf6诱导Slug表达可以促进EMT,引起细胞的转移,而且间充质细胞是具有自我更新和多向分化潜能的,是正常发育、伤口愈合的基础[28]。刺参再生肠腔上皮层是来自肠系膜增厚处的间充质细胞[19],因此,推测在刺参肠再生过程中,megf6基因高表达时,上调CDH1的相关抑制因子表达,促进了上皮细胞向具有间质表型的细胞转化,为实现肠组织的再生重建提供了细胞来源。随着再生肠管的生长延伸,再生18 d时,刺参再生肠管已基本恢复至正常状态,megf6的表达下调时,肠管中细胞的增殖、分化活动减弱。
[1] 李霞,聂竹兰,魏杰.仿刺参体壁表皮再生组织学和超微结构观察[J].中国水产科学,2007,14(7):1-6.
[2] Hu Meiyan,Li Qi,Li Li.Effect of salinity and temperature on salinity tolerance of the sea cucumber Apostichopus japonicus[J].Fisheries Science,2010,76(2):267-273.
[3] 聂竹兰,李霞.海参再生的研究[J].海洋科学,2006,30(5):78-82.
[4] García-Arrarás J E,Estrada-Rodgers L,Santiago R,et al.Cellular mechanisms of intestine regeneration in the sea cucumber,Holothuria glaberrima Selenka(Holothuroidea:Echinodermata)[J].Journal of Experimental Zoology,1998,281(4):288-304.
[5] Nakayama M,Nakajima D,Nagase T,et al.Identification of high-molecular-weight proteins with multiple EGF-like motifs by motif-trap screening[J].Genomics,1998,51(1):27-34.
[6] Sunnerhagen M S,Persson E,Dahlqvist I,et al.The effect of aspartate hydroxylation on calcium binding to epidermal growth factor-like modules in coagulation factors IX and X[J].Journal of Biological Chemistry,1993,268(31):23339-23344.
[7] Wouters M A,Rigoutsos I,Chu C K,et al.Evolution of distinct EGF domains with specific functions[J].Protein Science,2005,14(4):1091-1103.
[8] Chim S M,Qin A,Tickner J,et al.EGFL6 promotes endothelial cell migration and angiogenesis through the activation of extracellular signal-regulated kinase[J].Journal of Biological Chemistry,2011,286(25):22035-22046.
[9] Singh A B,Harris R C.Autocrine,paracrine and juxtacrine signaling by EGFR ligands[J].Cellular Signalling,2005,17(10):1183-1193.
[10] Nagase T,Nakayama M,Nakajima D,et al.Prediction of the coding sequences of unidentified human genes.XX.The complete sequences of 100 new cDNA clones from brain which code for large proteins in vitro[J].DNA Research,2001,8(2):85-95.
[11] Scheib J L,Sullivan C S,Carter B D.Jedi-1 and MEGF10 signal engulfment of apoptotic neurons through the tyrosine kinase Syk[J].The Journal of Neuroscience,2012,32(38):13022-13031.
[12] Letunic I,Bork P.20 years of the SMART protein domain annotation resource[J].Nucleic Acids Research,2018,46(D1):D493-D496.
[13] Deribe Y L,Wild P,Chandrashaker A,et al.Regulation of epidermal growth factor receptor trafficking by lysine deacetylase HDAC6[J].Science Signaling,2009,2(102):ra84.
[14] Wang Ye,Song Hejie,Wang Wenfeng,et al.Generation and characterization of Megf6 null and Cre knock-in alleles[J].Genesis,2019,57(2):e23262.
[15] Lim J,Hao Tong,Shaw C,et al.A protein-protein interaction network for human inherited ataxias and disorders of Purkinje cell degeneration[J].Cell,2006,125(4):801-814.
[16] 柳林,张宝警,李莹莹,等.中间球海胆smad2/3基因克隆、组织表达及其脂多糖刺激响应[J].大连海洋大学学报,2019,34(3):345-354.
[17] 孙冉冉,史宝,柳学周,等.黄条鰤PTEN基因克隆、组织分布及早期发育阶段的表达分析[J].大连海洋大学学报,2019,34(1):47-55.
[18] Dolmatov I Y,Eliseikina M G,Ginanova T T,et al.Muscle regeneration in the holothurian Stichopus japonicus[J].Roux’s Archives of Developmental Biology,1996,205(7-8):486-493.
[19] 孙丽娜.仿刺参Apostichopus japonicas(Selenka)消化道再生的组织细胞特征与关键基因分析[D].青岛:中国科学院研究生院(海洋研究所),2013:28-31.
[20] 王霞,李霞.仿刺参消化道的再生形态学与组织学[J].大连水产学院学报,2007,22(5):340-346.
[21] San Miguel-Ruiz J E,García-Arrarás J E.Common cellular events occur during wound healing and organ regeneration in the sea cucumber Holothuria glaberrima[J].BMC Developmental Biology,2007,7(1):115.
[22] Shukalyuk A I,Dolmatov I Y.Regeneration of the digestive tube in the holothurian Apostichopus japonicus after evisceration[J].Russian Journal of Marine Biology,2001,27(3):168-173.
[23] Shen Zhisen,Zhou Chongchang,Li Jinyun,et al.The association,clinicopathological significance,and diagnostic value of CDH1 promoter methylation in head and neck squamous cell carcinoma:a meta-analysis of 23 studies[J].Onco Targets and Therapy,2016,9:6763-6773.
[24] Beavon I R G.The E-cadherin-catenin complex in tumour metastasis:structure,function and regulation[J].European Journal of Cancer,2000,36(13):1607-1620.
[25] de Fátima Senra Cardoso M,Castelletti C H M,de Lima-Filho J L,et al.Putative biomarkers for cervical cancer:SNVs,methylation and expression profiles[J].Mutation Research,2017,773:161-173.
[26] Barrallo-Gimeno A,Nieto M A.The snail genes as inducers of cell movement and survival:implications in development and cancer[J].Development,2005,132(14):3151-3161.
[27] Bolós V,Peinado H,Pérez-Moreno M A,et al.The transcription factor Slug represses E-cadherin expression and induces epithelial to mesenchymal transitions:a comparison with Snail and E47 repressors[J].Journal of Cell Science,2003,116(3):499-511.
[28] Hu Hanqing,Wang Meng,Wang Hongwei,et al.MEGF6 promotes the epithelial-to-mesenchymal transition via the TGFβ/SMAD signaling pathway in colorectal cancer metastasis[J].Cellular Physiology and Biochemistry,2018,46(5):1895-1906.