巨魾Bagarius yarrelli(Sykes)隶属于鲇形目Siluriformes鮡科Sisoridae魾属Bagarius[1-2],是云南省特有底栖肉食性鱼类,主要分布于红河、澜沧江、怒江流域[2]。巨魾全身无鳞,身体两侧各有一条明显的侧线,从头部鳃盖末端上缘贯穿全身至尾柄末端;皮肤上布满细密的微小颗粒物,使之皮肤较为粗糙,体表无黏液,体色灰黄,腹面较白,肌肉呈黄色,俗称为“黄鱼”[3]。田树魁等[4]比较了巨魾、叉尾鲇Wallago attu、斑点叉尾鮰Letalurus punetaus 3 种鱼肌肉中常规营养成分和氨基酸含量,发现巨魾肌肉中蛋白质含量比鲤、鲫、草鱼等鱼类高,表明巨魾是一种具有较高营养价值的有待驯养开发的野生鱼类。杜民等[5]研究表明,云南红河的河口江段野生巨魾具有较高的遗传多样性。但由于环境改变和人为影响,野生巨魾资源越来越少,巨魾现已被列入《云南省水生野生动物保护名录》[6]。因此,对巨魾的遗传多样性研究具有重要的理论意义。
线粒体DNA(mtDNA)是一种细胞核外(细胞质)遗传物质,与核DNA相比,具有分子量小、结构简单、进化速度快、母性遗传、无组织特异性及提取方便等特点[7],可以半保留复制方式进行自我复制、转录和翻译的共价闭合环状双链DNA。在脊椎动物中,mtDNA ND4 基因(脱氢酶第四亚基)是编码蛋白基因[8],位于L链上。研究表明,ND4基因与其他线粒体基因相比,在重建亲缘关系较远类群间的系统进化速率适中[9]。丁言伟等[10]利用ND4基因研究了黄颡鱼属两种黄颡鱼在不同水系中的遗传进化关系;刘思情等[11]在鳅超科鱼类系统进行ND4基因序列分析,表明鳅超科鱼类ND4基因全长为1380~1387 bp,以ATG为起始密码子,终止密码子为不完全终止信号,A+T含量高于C+G含量;王莉等[12]利用线粒体ND4基因对7种条纹光唇鱼的系统进化进行了研究。近年来,有关巨魾的研究主要集中在生理、繁殖、微卫星标记开发和线粒体基因方面[3-5],未见有巨魾ND4基因的研究报道。为此,本试验中以云南省境内特有的巨魾6个野生群体为研究对象,对其ND4基因碱基序列的变异情况进行分析,探讨6个群体的遗传多样性及其之间的亲缘关系,旨在为巨魾的种质资源保护和人工繁殖提供理论支持。
1 材料与方法
1.1 材料
试验以红河州蒙自市曼耗镇(Baya-M)、怒江州龙阳县三江口镇段即怒江下游(Baya-N)、怒江州保山市怒江坝段(Baya-B)、临沧市澜沧江大桥上游(Baya-L)、澜沧江景洪段(Baya-J)、红河州河口县段(Baya-H)6个河段为采样点,每个点采集12尾,取肌肉组织置于 1.5 mL EP 管中,加入无水乙醇,于冰箱(-20 ℃)中保存备用。采用酚/氯仿抽提法[13] 提取巨魾DNA,用10 g/L琼脂糖凝胶电泳确认后,贮存于冰箱(-20 ℃)中待用。
1.2 方法
1.2.1 引物设计 本试验中使用Du等[14]报道的两对引物序列,其中Baya10号引物设计的目的片段为1244 bp,引物序列Baya10-F为GATCAGCACGACTCCCCTT,Baya10-R为CTTCTACGTGGG CTTTDGGT;Baya11号引物设计的目的片段为1444 bp,引物序列Baya11-F为CCAACCATCATRCTATTYCCA, Baya11-R为AGGC CTCTTTCGGTGGAYAA。通过两对引物的扩增片段拼接后获得巨魾的ND4基因全长序列。
1.2.2 巨魾ND4基因的PCR扩增及检测 巨魾ND4基因的扩增在PCR扩增仪上进行。PCR反应体系(25 μL)包含:2×Taq PCR MasterMix 12.5 μL,正、反向引物(10 pmol/μL)各1.0 μL,DNA(80 ng/μL)1.0 μL,双蒸馏水 9.5 μL。PCR反应条件为:95 ℃预变性4 min;95 ℃变性30 s,利用梯度PCR进行引物退火温度的确定[14],72 ℃下延伸90 s, 共进行32个循环;最后在72 ℃下延伸6 min,4 ℃保存。利用10 g/L琼脂糖进行凝胶电泳,在凝胶成像系统中照相并保存。
1.2.3 目的片段回收、连接、转化 扩增出目的条带的PCR扩增产物用20 g/L琼脂糖凝胶电泳分离后在紫外仪中切下含有目的片段的凝胶。用DNA回收试剂盒回收,纯化后克隆至大肠杆菌中,具体操作步骤参照回收试剂盒说明书。
1.2.4 单克隆检测及测序 挑取单克隆,在含氨苄西林的LB培养基中,37 ℃下振荡培养14~16 h。利用M13通用引物即M13-F(-47)(5′CGCCAGGGTTTTCCCAGTCACGAC 3′)和M13-R primer NEW(5′GAGCGGATAACAATTTCACACAGG 3′)进行菌液PCR,筛选出阳性单克隆,送生工生物工程(上海)股份有限公司测序。
1.3 数据处理
将测序后获得的72条ND4序列进行编号,分别为Baya-B1~B12、Baya-H1~H12、Baya-J1~J12、Baya-L1~L12、Baya-M1~M12、Baya-N1~N12。将72条ND4序列与参照物种ND4基因[14]进行比对拼接,对拼接好的序列用DNAMAN 5.0软件进行比对,将比对的结果输入MEGA 5.02软件转换为meg格式,并计算碱基含量、遗传距离。采用 UPGMA中的Kimura 2-parameter 模型法构建系统进化树,利用DNASP 5.0软件分析ND4序列的单倍型、核苷酸多样性等。
2 结果与分析
2.1 巨魾ND4基因序列碱基组成及单倍型
经PCR扩增、大肠杆菌克隆、测序,表明所有个体均获得了碱基排列清晰、长度为1381 bp的ND4基因顺序。利用DNASP软件进行分析,在72条序列中有67种单倍型,并将序列递交到GenBank(登录号:MK529780~MK529844,MK546510~MK546511),结果显示,红河河口群体中,4尾鱼的单倍型相同,另外3尾鱼的单倍型相同,其他5尾鱼具有不同的单倍型;其他5个群体中60尾鱼的单倍型各不相同,单倍型平均多样性为0.967,核苷酸多样性平均为0.032 6(表1)。
表1 巨魾6个群体间单倍型及核苷酸多样性
Tab.1 Haploid and nucleotide diversities of six populations of Bagarius yarrelli
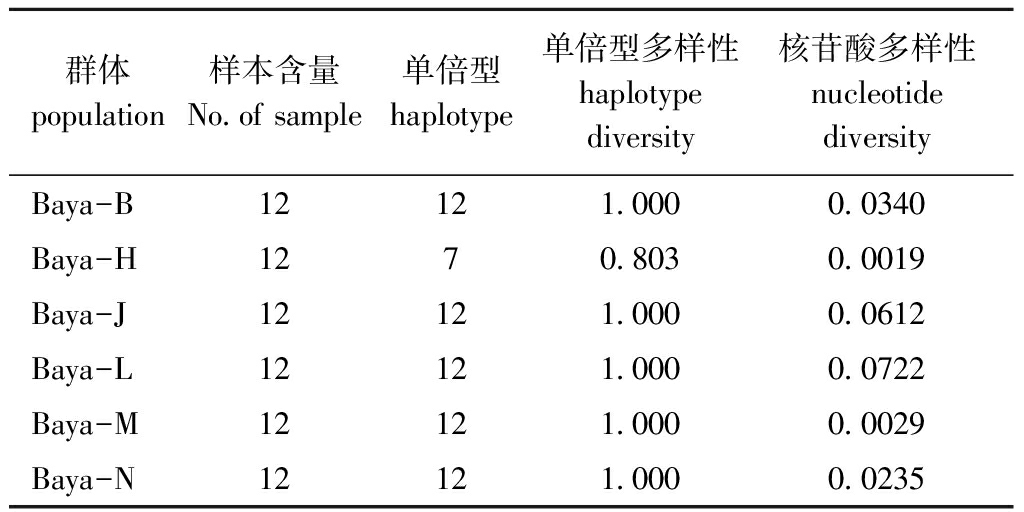
群体population样本含量No.of sample单倍型haplotype单倍型多样性haplotype diversity核苷酸多样性nucleotide diversityBaya-BBaya-HBaya-JBaya-LBaya-MBaya-N121212121212127121212121.0000.8031.0001.0001.0001.0000.03400.00190.06120.07220.00290.0235
采用MEGA 5.02 软件分析显示,共有501个变异位点。序列的碱基组成情况见表2,其中6个群体中4种碱基A、G、T、C的平均含量分别为30.29%、14.47%、27.31%、27.93%,G的含量低于其他3种碱基的含量,其中A+T的含量为58.22%,G+C的含量为42.40%,A+T含量明显高于G+C含量。
2.2 6个巨魾群体ND4基因的遗传距离
用MEGA 5.02软件中的双参数法,通过转换与颠换的比值计算6个群体之间的遗传距离 [15-16],结果如表3所示。基于所有单倍型的6个群体间转换/颠换 (Ts/Tv)及转换+颠换(Ts+Tv)的遗传距离平均值分别为6.769、0.106。基于单倍型转换+颠换(Ts+Tv)的群体间遗传距离为0.002~0.156,而基于单倍型转换/颠换(Ts/Tv)的群体间遗传距离为2.741~11.435。各单倍型的平均相对遗传距离为0.098。
表2 巨魾 6个群体ND4基因序列碱基含量
Tab.2 Basic contents of ND4 gene sequence of six populations of Bagarius yarrelli %
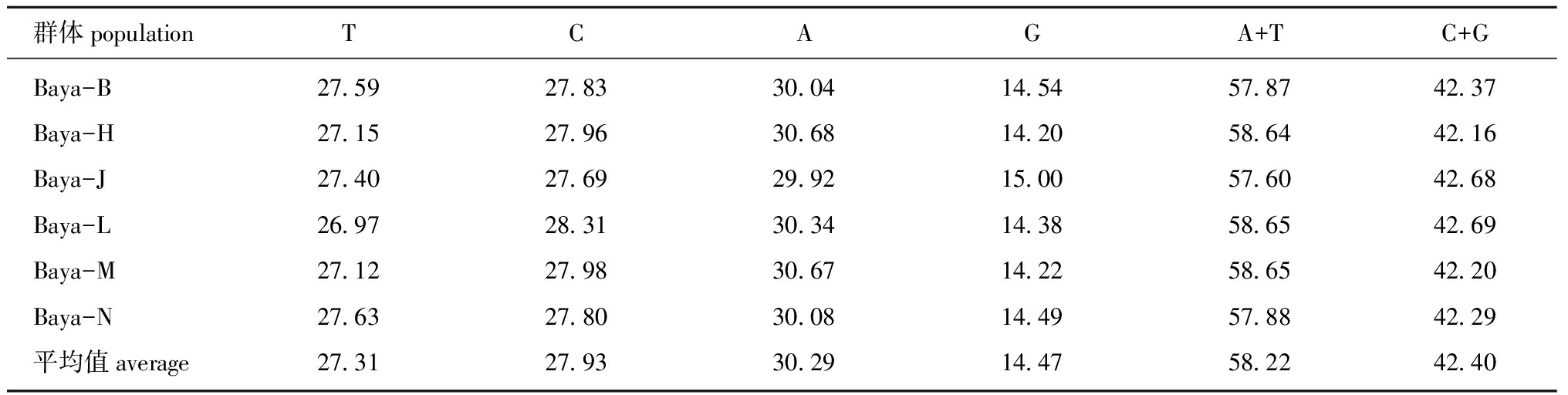
群体populationTCAGA+TC+GBaya-BBaya-HBaya-JBaya-LBaya-MBaya-N平均值average27.5927.1527.4026.9727.1227.6327.3127.8327.9627.6928.3127.9827.8027.9330.0430.6829.9230.3430.6730.0830.2914.5414.2015.0014.3814.2214.4914.4757.8758.6457.6058.6558.6557.8858.2242.3742.1642.6842.6942.2042.2942.40
表3 基于巨魾ND4基因单倍型的6个群体遗传距离
Tab.3 Genetic distance of six populations of Bagarius yarrelli based on ND4 gene haplotype
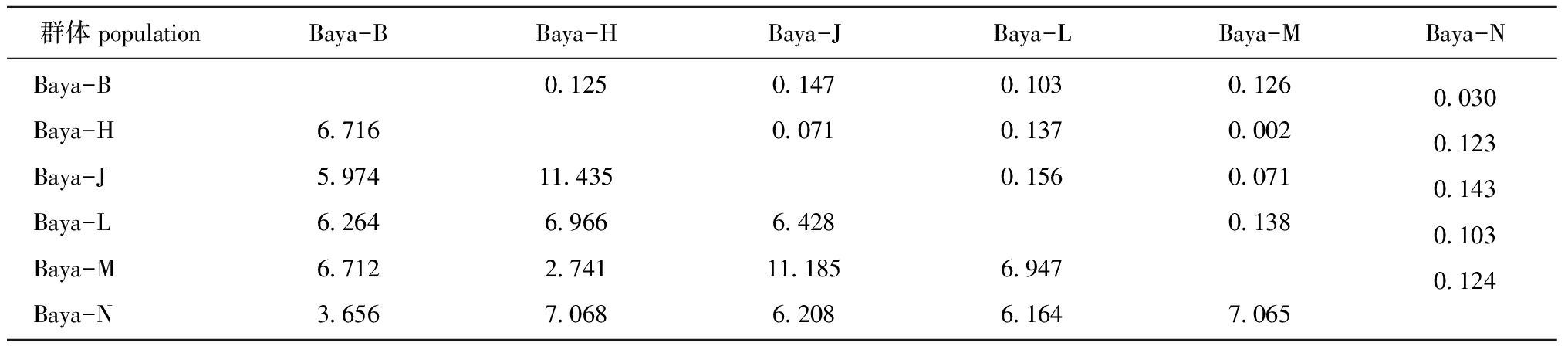
群体populationBaya-BBaya-HBaya-JBaya-LBaya-MBaya-NBaya-BBaya-HBaya-JBaya-LBaya-MBaya-N6.7165.9746.2646.7123.6560.12511.4356.9662.7417.0680.1470.0716.42811.1856.2080.1030.1370.1566.9476.1640.1260.0020.0710.1387.0650.0300.1230.1430.1030.124
注:下三角表示转换与颠换之比(Ts/Tv),上三角表示转换与颠换之和(Ts+Tv)
Note: The data in lower triangle represent the transformation of the transformation ratio(Ts/Tv),and the data in upper triangular represent the transformation+change(Ts+Tv)
2.3 6个巨魾群体ND4基因构建系统发育树
从图1可见,采用MEGA 5.02 软件中的UPGMA模型,以巨魾6个群体间的遗传距离(转换+颠换)构建系统发育树,其中,怒江坝群体(Baya-B)和怒江下游群体(Baya-N)巨魾先聚在一起后,再与澜沧江上游群体(Baya-L)聚在一起,而红河曼耗群体(Baya-M)、红河河口群体(Baya-H)巨魾先聚在一起后,再与景洪群体(Baya-J)聚在一起。
以三线纹胸鮡Glyptothorax trilineatus(简记为Gltr)(GenBank登录号:JQ026262)和长丝黑鮡Gagata dolichonema(简记为Gado)(GenBank登录号:NC_021596)两个物种为外群,对巨魾ND4基因中的67种单倍型进行聚类分析并构建系统发育树[17-18],从图2可以看出,系统发育树分为两大支,巨魾单独聚为一支。
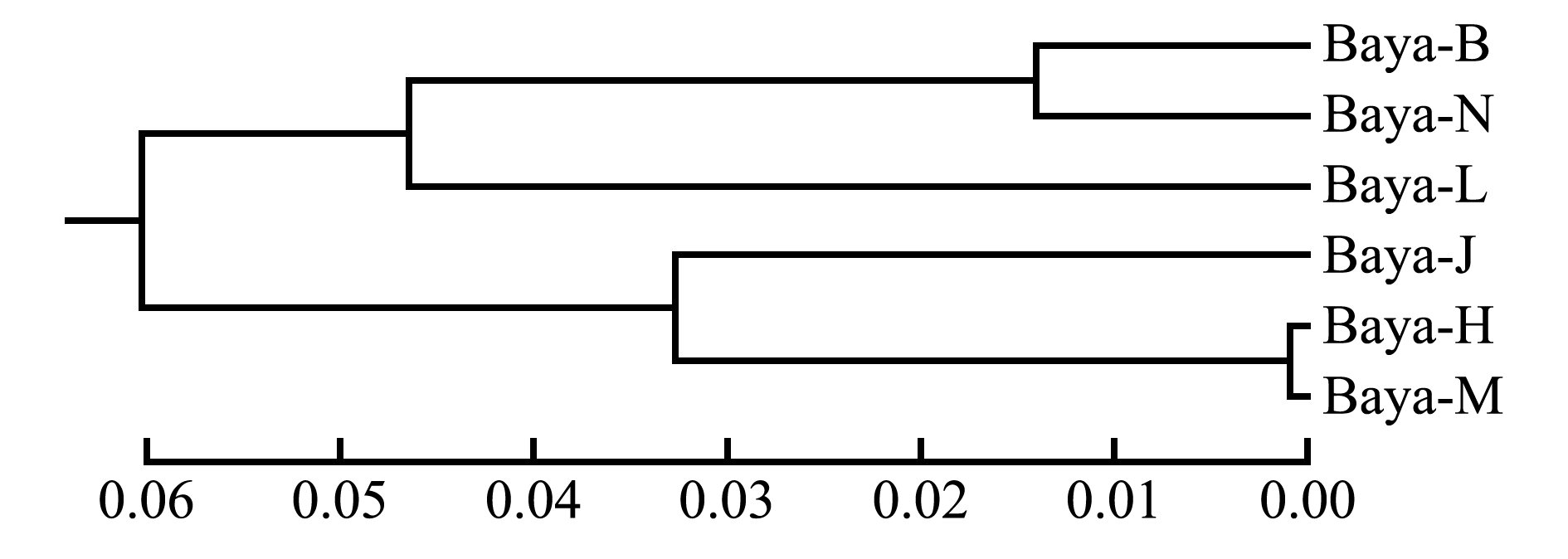
图1 6个巨魾群体ND4基因单倍型遗传关系聚类图
Fig.1 Dendrogram of genetic relationship of ND4 gene haploid among six populations of Bagarius yarrelli
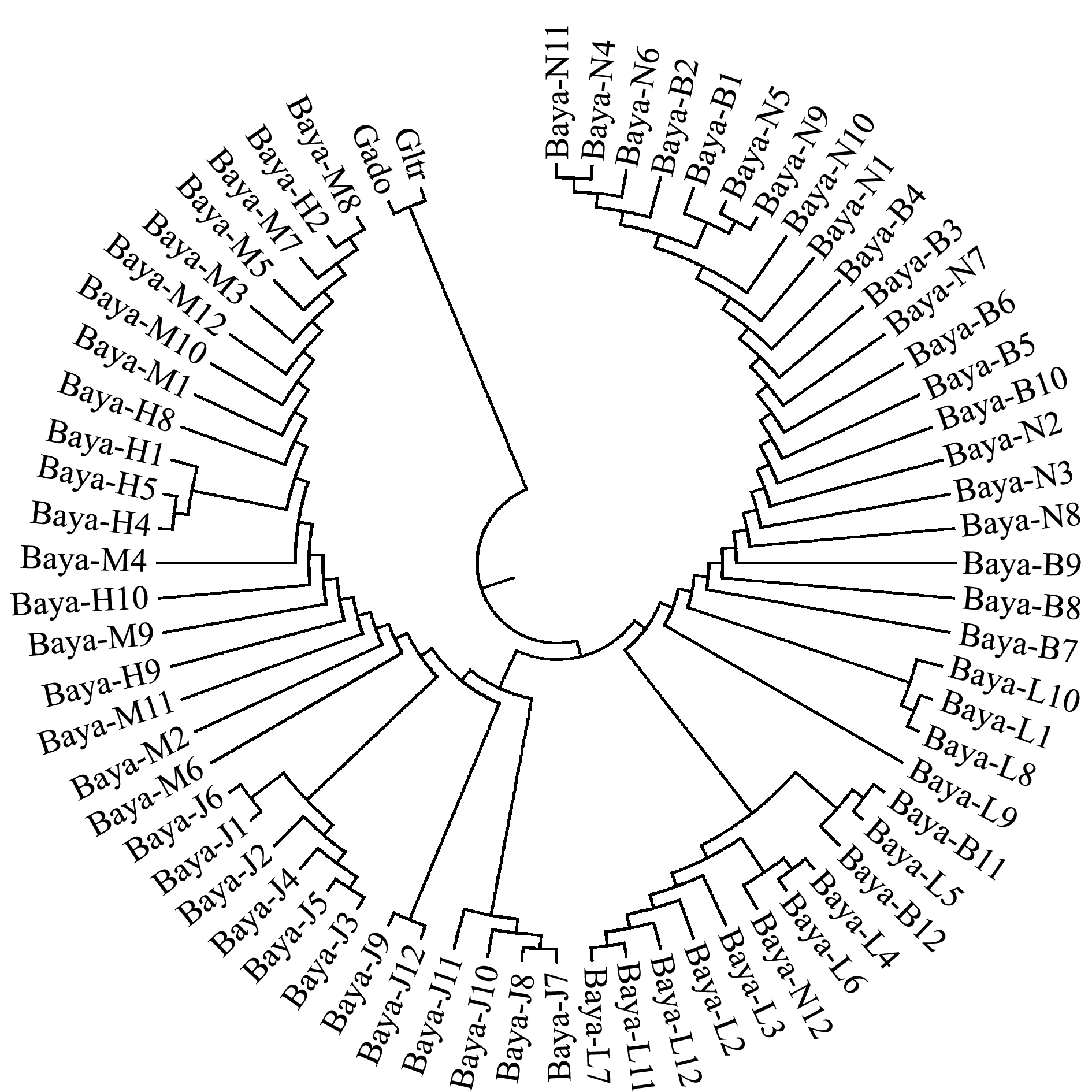
图2 巨魾67种ND4基因单倍型构建UPGMA聚类图
Fig.2 UPGMA dendrogram among 67 types of ND4 gene haploids of Bagarius yarrelli
3 讨论
3.1 巨魾ND4基因序列的碱基组成
本试验中得到巨魾6个群体的72条线粒体ND4基因序列,ND4基因全序列长度为1381 bp,共获得67个单倍型。对其碱基组成进行分析,结果显示,6个群体中ND4基因序列中A、T、G、C 4个碱基的平均含量分别为30.29%、14.47%、27.31%、27.93%,A+T含量明显高于C+G,同时可以看出,G 的含量明显低于其他3种碱基的含量,说明这部分基因表现出很强的碱基组成偏向性,与脊椎动物线粒体DNA 的特点是一致的[8]。
3.2 6个巨魾群体间的遗传距离
群体遗传多样性在遗传距离上可以反映出来,本研究中6个巨魾基于单倍型的转换+颠换(Ts+Tv)的不同群体间遗传距离为0.002~0.156(表3),其中红河河口群体与曼耗群体之间的遗传距离最小(0.002),说明红河河口群体与曼耗群体之间的分化程度较小,亲缘关系较近;而澜沧江群体与景洪群体之间的遗传距离最大(0.156),说明澜沧江群体与景洪群体之间的分化程度较大,亲缘关系较远。
3.3 巨魾与其他鱼的系统进化关系
在有外类群的情况下,所有巨魾鱼聚在一起,形成一支(图2),这种结果与传统的形态学分类基本相似。这一支又分为两部分,置信度分别为80%和71%,大体上,怒江坝群体(Baya-B)、怒江下游群体(Baya-N)、澜沧江上游群体(Baya-L)聚在一起,而景洪群体(Baya-J)、红河河口群体(Baya-H)、曼耗群体(Baya-M)聚在一起,同6个群体间遗传距离构建聚类关系图(图1)结果相同,说明他们之间存在地域差异。但是有些群体出现交叉现象,这可能是受人为和环境因素的影响。另一支置信度为100%,又分为2 支,说明巨纹胸鮡属和黑鮡属有较近的亲缘关系,与基于细胞色素氧化酶基因的巨魾系统进化研究结果一致[19]。
生物的遗传多样性水平是长期进化的产物,是评估生物资源现状的一个重要参数[20]。遗传多样性的高低是评价物种对环境适应能力和物种进化潜力的重要依据,遗传多样性越丰富,物种对环境变化的适应能力越强,遗传多样性的贫乏会导致物种低的进化适应性,使得物种易于遭受生态环境改变的影响[21]。单倍型多样性和核苷酸多样性(>0.05)是衡量一个物种群体遗传多样的重要指标[22]。本研究中,澜沧江上游群体、景洪群体单倍型多样性和核苷酸多样性都高,遗传多样性丰富;而怒江坝群体、红河河口群体、红河曼耗群体、怒江下游群体出现单倍型多样性高、核苷酸多样性低的模式(表1),可能是群体受到瓶颈效应后种群迅速扩张所导致的结果,因为核苷酸多样性的积累时间比单倍型多样性的积累时间要漫长得多[23]。
综上所述,3条河流巨魾群体的ND4基因序列具有丰富的多样性,但是基于线粒体其他基因序列的分析结果如何,以及3条不同河流中巨魾对疾病的抵抗能力又如何,还有待后续进行相关研究。
[1] 成庆泰,郑葆珊.中国鱼类系统检索(上册)[M].北京:科学出版社,1987:218-219.
[2] 褚新洛,陈银瑞.云南鱼类志:下册[M].北京:科学出版社,1990:194-195.
[3] 田树魁,薛晨江,冷云,等.巨魾的生物学特性初步研究[J].水生态学杂志,2009,2(3):115-117.
[4] 田树魁,易勇,薛晨江,等.野生巨魾肌肉营养成分测定和分析[J].淡水渔业,2009,39(3):73-76.
[5] 杜民,牛宝珍,罗彩艳,等.巨魾野生群体遗传多样性的RAPD分析[J].淡水渔业,2015,45(1):15-19,24.
[6] 云南省农业厅,水生野生动植物保护名录编辑委员会.云南省水生野生动植物保护名录[M].昆明:云南出版社,2003.
[7] 王存芳,曾勇庆,杜立新,等.线粒体DNA(mtDNA)的研究进展[J].动物科学与动物医学,2001,18(1):16-18.
[8] Meyer A.Evolution of mitochondrial DNA in fishes[M]//Hochachka P W,Mommsen T P.Biochemistry and Molecular Biology of Fishes.Amsterdam:Elsevier,1993:1-38.
[9] Zardoya R,Meyer A.Phylogenetic performance of mitochondrial protein-coding genes in resolving relationships among vertebrates[J].Molecular Biology and Evolution,1996,13(7):933-942.
[10] 丁言伟,彭作刚,张训蒲,等.黄颡鱼属两种鱼类的线粒体ND4基因序列变异性分析[J].水生生物学报,2006,30(4):413-419.
[11] 刘思情,张家波,唐琼英,等.基于ND4和ND5基因序列分析的鳅超科鱼类系统发育关系[J].动物学研究,2010,31(3):221-229.
[12] 王莉,王绪祯,何舜平.基于线粒体ND4基因序列的七种条纹光唇鱼系统发育及对半刺光唇鱼分类地位的质疑[J].水生生物学报,2010,34(6):1218-1222.
[13] Sambroo J,Fritsch E F,Maniatis T.Molecular Cloning:A Laboratory Manual[M].2nd ed.Cold Spring Harbor:Cold Spring Harbor Laboratory Press,1989:1024-1031.
[14] Du M,Zhou C J,Niu B Z,et al.The complete mitochondrial genome of the Bagarius yarrelli from Honghe River[J].IOP Conference Series:Earth and Environmental Science,2016,41(1):012031.
[15] Kimura M.A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J].Journal of Molecular Evolution,1980,16(2):111-120.
[16] Sneath P H A,Sokal R R.Numerical Taxonomy[M].San Francisco:W.H.Freeman,1973.
[17] Tamura K,Dudley J,Nei M,et al.MEGA4:molecular evolutionary genetics analysis (MEGA) software version 4.0[J].Molecular Biology and Evolution,2007,24:1596-1599.
[18] Felsenstein J.Confidence limits on phylogenies:an approach using the bootstrap[J].Evolution,1985,39(4):783-791.
[19] 杜民,牛宝珍,王婷婷,等.巨魾鱼细胞色素氧化酶基因多态性及系统进化研究[J].上海海洋大学学报,2016,25(3):337-343.
[20] 王瑾瑾,童金苟,张耀光,等.厚颌鲂两个野生群体遗传多样性分析[J].水生生物学报,2014,38(5):975-979.
[21] 陈灵芝.中国的生物多样性现状及其保护对策[M].北京:科学出版社,1993:100-113.
[22] Vrijenhoek R C.Genetic diversity and fitness in small populations[M]//Loeschcke V,Jain S K,Tomiuk J.Conservation Genetics.Basel:Birkhäuser,1994:37-53.
[23] 范启,何舜平.长江流域![]() 种群遗传多样性和遗传结构分析[J].水生生物学报,2014,38(4):627-635.
种群遗传多样性和遗传结构分析[J].水生生物学报,2014,38(4):627-635.