金乌贼Sepia esculenta隶属于头足纲Cephalopoda、鞘亚纲Coleoidea、乌贼目Sepioidea、乌贼科Sepiidae、乌贼属Sepia,俗称墨鱼、乌鱼等,属一年生洄游性头足类,生活在浅海大陆架中下层水域,日本、菲律宾、越南等国家近海均有分布,金乌贼曾是中国重要捕捞种类,与大黄鱼Pseudosciaena crocea、小黄鱼Pseudosciaena polyatis、带鱼Trichiurus lepturus并列为20世纪60年代中国四大渔业[1-2]。然而,20世纪80年代,中国近海的过度捕捞及产卵场环境破坏,导致了金乌贼资源量锐减[3]。
为了有效恢复金乌贼自然种群数量,自1991年起,山东省将其列入地方资源增殖计划[4-5],并开展了政府主导的规模性增殖放流。截至2016年,全省投入专项资金1896.92万元,累计投放金乌贼苗种386.46万尾、受精卵33.5万粒[6]。经过20余年的不断摸索与实践,目前金乌贼放流取得了较好的经济、社会和生态效益。尤其2010年中国水产科学研究院黄海水产研究所金乌贼苗种人工繁育技术的突破,为其增殖放流计划顺利开展奠定了坚实基础,至此,金乌贼增殖放流由投放附卵乌贼笼改为直接放流幼乌,放流群体贡献率显著提高。由于人工繁育亲体为自然海域野生个体,故研究野生群体的遗传多样性水平具有重要意义,一方面,可为原始群体遗传学特征评价提供基础数据;另一方面,野生种群的遗传多样性水平对于放流效果评价具有重要参考价值。
迄今为止,有关金乌贼的研究多侧重于繁殖生物学[3,7-9]、生理生化特性[10-13]、人工繁育技术[14-16]等方面,而种群遗传学相关研究报道较少。Zheng等[17]应用同工酶技术研究了日照及青岛两个金乌贼种群的遗传差异,通过对外套膜、口球、眼等4个组织的39个同工酶位点杂合度比对,发现二者无显著的遗传差异。Wei等[18]采用水平淀粉凝胶电泳技术对日照近海金乌贼群体遗传多样性进行了研究,计算同工酶特异性表达组织的平均观测杂合度及期望杂合度分别为0.015 9、0.014 3,进而得出该区域金乌贼遗传多样性水平较低的结论。2009年Zheng等[19]应用9个微卫星标记位点[20]进行了中日共5个金乌贼群体的遗传结构分析,结果表明,微卫星标记的平均观测杂合度及期望杂合度高于同工酶分析数据,9个位点中平均等位基因频率为15.8~21.4,平均杂合度为0.80~0.90,5个群体间存在弱但显著的遗传差异。后经COI基因片段分析[21],亦得出日本4个地理种群间遗传分化较小,中日群体间遗传差异较大的结论。
微卫星标记(Microsatellite),又称简单重复序列(Simple sequence repeats,SSR),是基因组中由2~6个碱基组成核心串联排列的重复DNA序列,具有中性、量大、高度变异性、共显性遗传等特性,是比较理想的遗传标记[22],可广泛应用于种群遗传与进化、种质鉴定与亲权分析、分子标记辅助育种、遗传图谱构建和数量性状连锁定位等方面[23-25],在头足类的种群结构和遗传多样性研究中亦有广泛应用。本研究中应用磁珠富集法开发了金乌贼的微卫星标记,并用开发的引物扩增了青岛和长江口两个金乌贼野生群体的DNA样本,旨在扩充金乌贼微卫星标记信息的同时,为探讨近海金乌贼野生群体遗传多样性、开发养护近海渔业资源提供基础数据。
1 材料与方法
1.1 材料
试验用金乌贼野生群体共2个,具体信息详见表1。其中用于开发微卫星标记位点的群体是青岛群体,取腕足小段用75%酒精固定后4 ℃下保存待用。
表1 金乌贼群体样品采集信息
Tab.1 Information on population samples of golden cuttlefish Sepia esculenta

群体population采样位置location海域sea area样本数/ind.sample number采样日期sampling date青岛Qingdao(QD)120°E, 36°N黄海Yellow Sea1382009-10长江口Changjiangkou(CJK)125°E, 30°N黄东海Yellow Sea and East China Sea502009-10
1.2 方法
1.2.1 基因组DNA提取 参考《分子克隆实验指南》中酚—氯仿抽提法[26],稍加修改提取金乌贼基因组DNA,用ddH2O稀释至100 μg/mL左右,于-20 ℃下保存待用。
1.2.2 微卫星富集文库构建 采用磁珠富集法构建微卫星富集文库(GT)n,具体方法如下:
取约1000 ng基因组DNA进行Mse Ⅰ酶切,将检测酶切效果后的产物与接头MA-A(5′TACTCAGGACTCAT 3′)和MA-B(5′GACCGATGAGTCCTGAG 3′)连接。以连接好的产物为模板,以MA(5′GATGAGTCCTGAGTAA 3′)为引物进行PCR预扩增。反应体系(25 μL)包含: 10×Buffer 2.5 μL,MgC12 (25 mmol/L) 1.5 μL,dNTPs (10 mmol/L)2.5 μL,MA 1 μL,DNA模板1 μL,Taq 酶0.2 μL,用ddH2O补足至25 μL。反应条件为:94 ℃下预变性5 min;94 ℃下变性30 s,53 ℃下退火1 min,72 ℃下延伸l min,共进行20个循环;最后在72 ℃下再延伸10 min。反应产物使用TaKaTa公司的DNA核酸共沉剂回收。设计合成探针Biotin(GT)13与PCR预扩增产物杂交,杂交条件为95 ℃下变性5 min,58 ℃下杂交15 min。取100 mL磁珠与杂交体系混合,去除非特异性DNA片段后,进行两次目的片段洗脱并回收洗脱产物。以两次洗脱产物为底物,MA为引物进行PCR扩增,条件同预扩增,循环数增至30。用15 g/L琼脂糖凝胶电泳检测产物分布范围及产物量,选取长度为500~1000 bp的目的片段切胶回收。富集片段与载体连接后,转化于Top10感受态细胞中恒温培养,以纯化后的菌液为模板进行PCR扩增,反应体系为15 μL,扩增条件同上。筛选产物后由北京华大基因科技有限公司完成测序工作,构建富集片段克隆文库。
1.2.3 重复序列的筛选及微卫星序列引物设计 手工剪去阳性克隆序列上下接头片段,并用SSRHunter软件查找序列中的微卫星片段。基于含微卫星的重复单元,使用Primer Premier 5软件设计引物并送北京华大基因科技有限公司合成。随机挑选青岛群体的6个个体,作为DNA模板筛选多态性微卫星引物。反应体系(共15 μL):10×PCR Buffer 0.6 μL,MgCl2(25 mmol/L)1.5 μL,dNTPs(10 mmol/L)0.9 μL,双向引物(10 μmol/L)各1.5 μL,DNA模板1 μL,Taq 酶(5 U/μL)0.12 μL,用ddH2O补足至15 μL。PCR扩增程序为:94 ℃下预变性5 min;94 ℃下变性 45 s,在每对引物的退火温度下退火45 s,72 ℃下延伸45 s,共进行35个循环;最后在72 ℃下再延伸7 min。
1.2.4 聚丙烯酰胺凝胶电泳 在PCR产物中加入等体积变性剂,于95 ℃下变性5 min后,立即冷却。在电泳槽(北京市六一仪器厂,DYCZ-20C)中用聚丙烯酰胺凝胶电泳检测微卫星位点等位基因的多态性。
选取经6个个体预筛选呈多态性的引物,分别用48个个体的青岛和长江口群体做聚丙烯酰胺凝胶电泳检测。
1.3 数据处理
用Photoshop CS软件处理PAGE扫描后得到的分型图像,校正电泳偏差后识别各等位基因,等位基因的大小通过pBR322/Msp I Marker分子量标准比对获得,按照带型的大小记录各等位基因。使用Genepop 4.0软件[27]统计每个微卫星位点等位基因数量,计算等位基因数(A)、观测杂合度(HO)、期望杂合度(HE)、多态信息含量(PIC)、零等位基因(null)等遗传多样性参数,并根据哈迪—温伯格平衡假设(Hardy-Weinberg equilibrium)和连锁不平衡(Linkage disequilibrium)检验,验证是否存在零等位基因(null)。
2 结果与分析
2.1 金乌贼微卫星位点的开发
基因组DNA酶切及微卫星重复序列检测结果如图1所示。本试验中使用质粒检测方法,共获得269个克隆,其中192个序列含有微卫星,占所有克隆子比例的71.38%。基于含有微卫星的重复序列共设计85对引物,其中20对引物通过筛选,其余经优化退火温度亦无稳定产物或表现的为非多态。
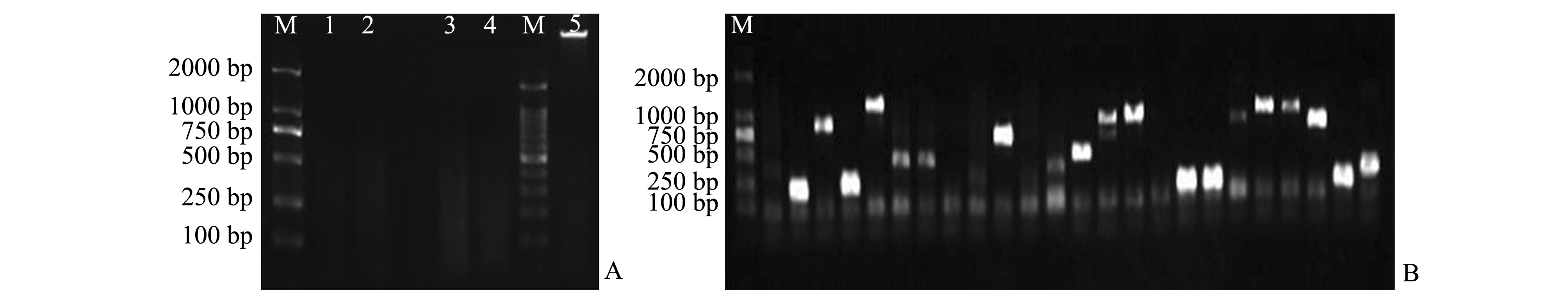
注:M为2000 bp DNA标准片段(DL2000);A为酶切结果,其中1、2、3、4为加Mse Ⅰ的DNA结果,5为不加Mse Ⅰ的DNA结果;B为微卫星重复序列PCR检测结果
Note: M,DNA Ladder 2000;A, Mse Ⅰ digestion result, 1-4 signs DNA samples added Mse Ⅰ, 5 signs DNA samples without Mse Ⅰ; B, PCR test result
图1 酶切与PCR结果
Fig.1 Results of enzyme digestion and PCR
根据Weber[28]提出的微卫星分类标准,开发的20个微卫星序列中完美型(Perfect)12个(占60%),非完美型(Imperfect)2个(占10%),复合型(Compound)6个(占30%)。完美型中二核苷酸微卫星10个(占83.33%),四核苷酸微卫星2个(占16.67%);非完美型中二核苷酸微卫星和四核苷酸微卫星各1个;复合型全部为二核苷酸微卫星。对所获得的20个多态性微卫星位点进行48个个体的青岛群体验证分析,每个位点的等位基因数在2~17个不等,平均等位基因数8.15个。其中观测杂合度分布范围为0.146~0.936,平均为0.630;期望杂合度分布范围为0.172~0.930,平均为0.702(表2)。经Bonferroni检验,成功扩增的20对多态性位点中,有5个位点(Secu 8、Secu 16、Secu 53、Secu 63、Secu 72)的等位基因频率不符合哈迪—温伯格平衡假设。在5个偏离平衡的位点中,4个位点(Secu 8、Secu 16、Secu 53、Secu 63)存在零等位基因。经Bonferroni检验,在任意两个位点间均无明显的连锁现象。
表2 金乌贼的微卫星位点信息
Tab.2 Characteristics of microsatellite loci in golden cuttlefish Sepia esculenta
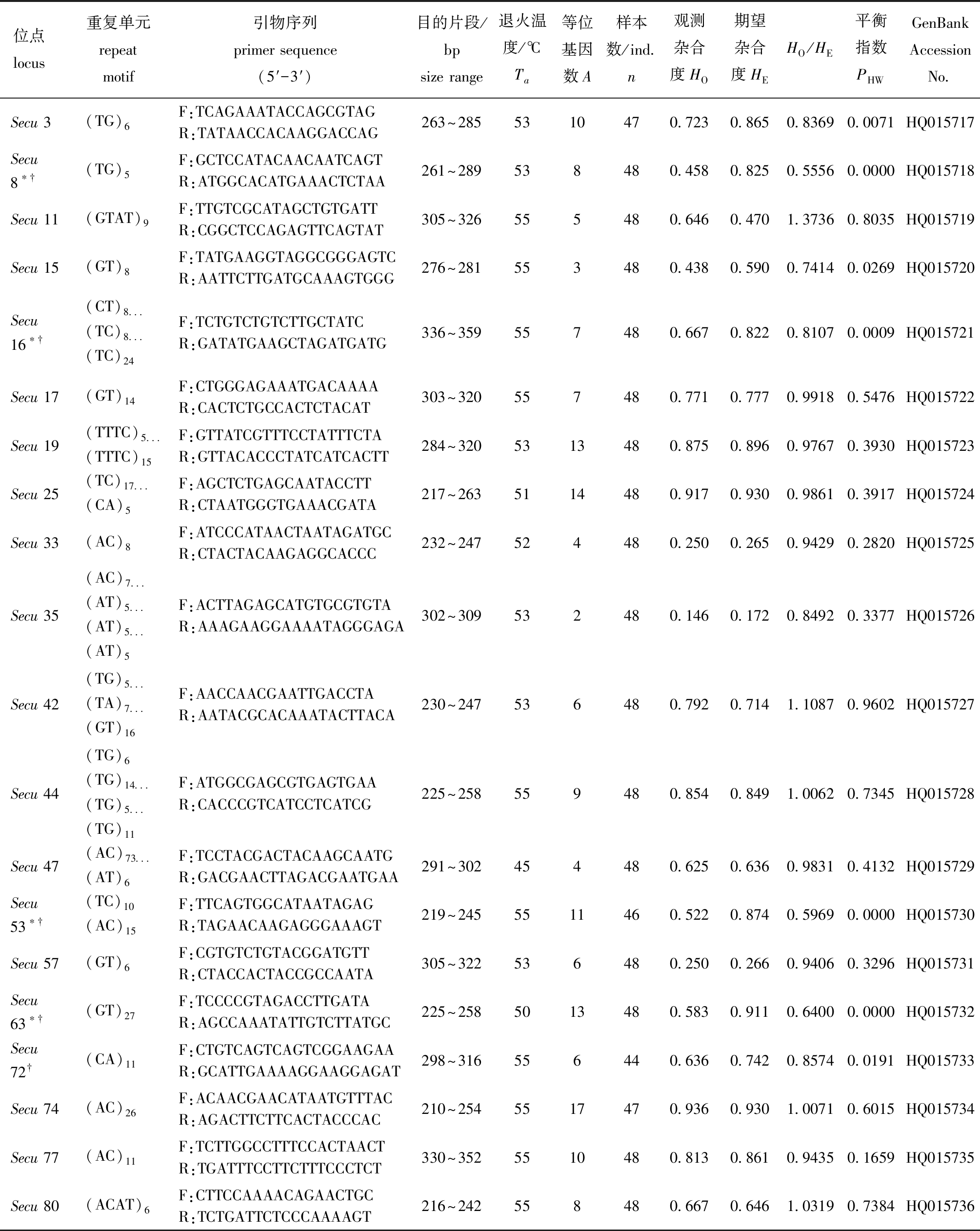
位点 locus 重复单元 repeat motif 引物序列 primer sequence (5′-3′) 目的片段/bpsize range退火温度/℃Ta等位基因数A样本数/ind.n观测杂合度HO期望杂合度HEHO/HE平衡指数PHWGenBank Accession No.Secu 3(TG)6F:TCAGAAATACCAGCGTAGR:TATAACCACAAGGACCAG263~2855310470.7230.8650.83690.0071HQ015717Secu 8∗†(TG)5F:GCTCCATACAACAATCAGTR:ATGGCACATGAAACTCTAA261~289538480.4580.8250.55560.0000HQ015718Secu 11(GTAT)9F:TTGTCGCATAGCTGTGATTR:CGGCTCCAGAGTTCAGTAT305~326555480.6460.4701.37360.8035HQ015719Secu 15(GT)8F:TATGAAGGTAGGCGGGAGTCR:AATTCTTGATGCAAAGTGGG276~281553480.4380.5900.74140.0269HQ015720Secu 16∗†(CT)8...(TC)8...(TC)24F:TCTGTCTGTCTTGCTATCR:GATATGAAGCTAGATGATG336~359557480.6670.8220.81070.0009HQ015721Secu 17(GT)14F:CTGGGAGAAATGACAAAAR:CACTCTGCCACTCTACAT303~320557480.7710.7770.99180.5476HQ015722Secu 19(TTTC)5...(TTTC)15F:GTTATCGTTTCCTATTTCTAR:GTTACACCCTATCATCACTT284~3205313480.8750.8960.97670.3930HQ015723Secu 25(TC)17...(CA)5F:AGCTCTGAGCAATACCTTR:CTAATGGGTGAAACGATA217~2635114480.9170.9300.98610.3917HQ015724Secu 33(AC)8F:ATCCCATAACTAATAGATGCR:CTACTACAAGAGGCACCC232~247524480.2500.2650.94290.2820HQ015725Secu 35(AC)7...(AT)5...(AT)5...(AT)5F:ACTTAGAGCATGTGCGTGTAR:AAAGAAGGAAAATAGGGAGA302~309532480.1460.1720.84920.3377HQ015726Secu 42(TG)5...(TA)7...(GT)16F:AACCAACGAATTGACCTAR:AATACGCACAAATACTTACA230~247536480.7920.7141.10870.9602HQ015727Secu 44(TG)6(TG)14...(TG)5...(TG)11F:ATGGCGAGCGTGAGTGAAR:CACCCGTCATCCTCATCG225~258559480.8540.8491.00620.7345HQ015728Secu 47(AC)73...(AT)6F:TCCTACGACTACAAGCAATGR:GACGAACTTAGACGAATGAA291~302454480.6250.6360.98310.4132HQ015729Secu 53∗†(TC)10(AC)15F:TTCAGTGGCATAATAGAGR:TAGAACAAGAGGGAAAGT219~2455511460.5220.8740.59690.0000HQ015730Secu 57(GT)6F:CGTGTCTGTACGGATGTTR:CTACCACTACCGCCAATA305~322536480.2500.2660.94060.3296HQ015731Secu 63∗†(GT)27F:TCCCCGTAGACCTTGATAR:AGCCAAATATTGTCTTATGC225~2585013480.5830.9110.64000.0000HQ015732Secu 72†(CA)11F:CTGTCAGTCAGTCGGAAGAAR:GCATTGAAAAGGAAGGAGAT298~316556440.6360.7420.85740.0191HQ015733Secu 74(AC)26F:ACAACGAACATAATGTTTACR:AGACTTCTTCACTACCCAC210~2545517470.9360.9301.00710.6015HQ015734Secu 77(AC)11F:TCTTGGCCTTTCCACTAACTR:TGATTTCCTTCTTTCCCTCT330~3525510480.8130.8610.94350.1659HQ015735Secu 80(ACAT)6F:CTTCCAAAACAGAACTGCR:TCTGATTCTCCCAAAAGT216~242558480.6670.6461.03190.7384HQ015736
注:†为不符合哈迪-温伯格平衡预期的位点(矫正P值<0.0025);*为可能存在零等位基因的位点(零等位基因频率>5%);下标…表示非完美型微卫星重复核心序列中的非重复碱基。
Note:† indicates that locus is not in Hardy-Weinberg equilibrium (adjusted P-value<0.0025); * indicates that locus may harbor null alleles (null alleles frequency >5%);… indicates non-repetitive bases between imperfect SSR
2.2 近源种通用性检测
在跨种扩增中,分别用12个个体的小群体验证金乌贼多态性位点在同属其他种及近源属中的跨种扩增效率,结果表明,在同属的针乌贼Sepia andreana中有6个位点表现为多态,在近源属曼氏无针乌贼属的曼氏无针乌贼Sepiella maindroni中有4个位点表现为多态(表3)。
表3 金乌贼微卫星位点在近源种属的跨种扩增效率
Tab.3 Cross-amplification test of the microsatellite loci developed in golden cuttlefish Sepiella esculenta in two closely related species

种类speciesSecu3Secu8Secu11Secu15Secu16Secu17Secu19Secu25Secu33Secu35Secu42Secu44Secu47Secu53Secu57Secu63Secu72Secu74Secu77Secu80曼氏无针乌贼Sepiella maindroni----------+++-+-----针乌贼Sepia andreana+--+-+-+---++-------
注:+为成功扩增且表现为多态;-为无稳定扩增产物或非多态
Note:+ shows successful amplifying and polymorphic; - shows unsuccessful amplifying or monpolymorphic
2.3 两个野生群体的遗传差异比较
2.3.1 群体遗传多样性比较 应用符合哈迪—温伯格平衡假设且多态性较好的11个微卫星标记,分别对青岛(QD)和长江口(CJK)金乌贼产卵群体的各48个个体进行种群遗传多样性检测,各产卵群体遗传多样性指标的计算和统计值见表4。在所有位点中,青岛群体共检测出163个等位基因,长江口群体共检测出152个等位基因,两个群体的平均等位基因数为157.5个。两个种群的平均等位基因数分别为14.818和13.818,不同位点的等位基因数分布范围为5~28和6~26。对每个标记的等位基因数在各群体中的分布进行观察和比较,所有标记的等位基因数在两个群体中都呈现近似均匀的分布,未发现任何显著的分布不均。共检测出群体独有的等位基因27个,其中青岛群体19个特有等位基因,长江口群体8个特有等位基因。两个群体的观测杂合度分布范围为0.250~0.936和0.500~0.896;期望杂合度分布范围为0.265~0.930和0.623~0.960。两个群体的平均观测杂合度为0.731(QD)、0.731(CJK),平均期望杂合度为0.771(QD)、0.857(CJK),青岛群体的多态信息含量为0.223~0.946,平均为0.746,除J33、J57外,各标记的PIC值均大于0.5;长江口群体多态信息含量为0.614~0.948,平均为0.845,各标记PIC值均大于0.5,且平均值高于青岛群体。这表明,各群体内部均具有高度的遗传多样性,且各群体内部遗传多样性无明显差异。
在48个群体—标记组合中,各组合在经过Sequential Bonferroni校正后,均符合哈迪—温伯格平衡假设。两个群体在Secu 3位点的聚丙烯酰胺凝胶电泳图谱见图2。
2.3.2 群体间的遗传分化(Fst)及分配分析 检测结果显示,两个群体间存在较弱的遗传分化(Fst<0.05),遗传分化指数为0.032 5。用STRUCTRE(K=2)[29]对两个群体进行分配分析,结果显示:两个采样点样品可明显分为两个不同的群体,其中青岛群体的所有个体可正确分配到该种群的概率为84.0%,分配到长江口种群的概率为16.0%;长江口群体中的所有个体可正确分配到该种群的概率为86.4%,分配到青岛群体的概率为13.6%(详见表5、图3)。
3 讨论
3.1 微卫星标记开发方法比较
序列的分离和富集是开发微卫星标记最关键的一步,最经典的常用方法是基因组文库法和微卫星富集法[30]。基于限制位点相关DNA(Restriction-site Associated DNA,RAD)构建酶切标签序列文库的高通量测序新技术(RAD-seq)是基于二代测序技术发展起来的新方法[31],其具有试验数据准确性高、利用率高、性价比高等特点,可通过RAD测序快速获得微卫星标记[32-33],并有效避免筛选出过多低多态性标记位点[34],亦在遗传图谱构建、群体遗传结构研究等领域有广泛应用[35-36]。
表4 金乌贼各地理群体遗传多样性信息
Tab.4 Informations on genetic diversity in two wild populations of golden cuttlefish Sepia seculenta
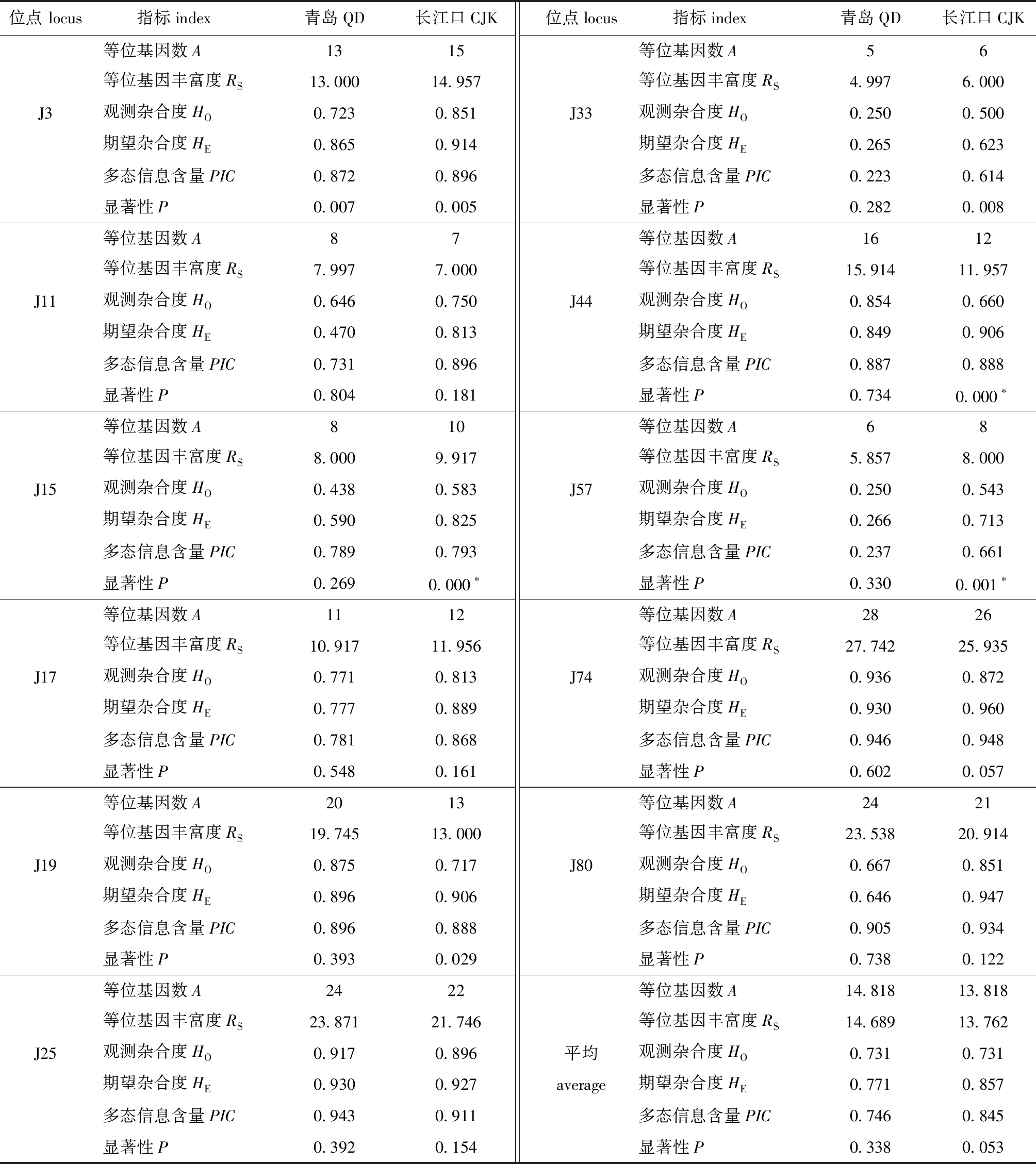
位点 locus 指标index青岛QD长江口CJK位点locus 指标index青岛QD长江口CJK等位基因数A1315等位基因数A56等位基因丰富度RS13.00014.957等位基因丰富度RS4.9976.000J3观测杂合度HO0.7230.851J33观测杂合度HO0.2500.500期望杂合度HE0.8650.914期望杂合度HE0.2650.623多态信息含量PIC0.8720.896多态信息含量PIC0.2230.614显著性P0.0070.005显著性P0.2820.008等位基因数A87等位基因数A1612等位基因丰富度RS7.9977.000等位基因丰富度RS15.91411.957J11观测杂合度HO0.6460.750J44观测杂合度HO0.8540.660期望杂合度HE0.4700.813期望杂合度HE0.8490.906多态信息含量PIC0.7310.896多态信息含量PIC0.8870.888显著性P0.8040.181显著性P0.7340.000∗等位基因数A810等位基因数A68等位基因丰富度RS8.0009.917等位基因丰富度RS5.8578.000J15观测杂合度HO0.4380.583J57观测杂合度HO0.2500.543期望杂合度HE0.5900.825期望杂合度HE0.2660.713多态信息含量PIC0.7890.793多态信息含量PIC0.2370.661显著性P0.2690.000∗显著性P0.3300.001∗等位基因数A1112等位基因数A2826等位基因丰富度RS10.91711.956等位基因丰富度RS27.74225.935J17观测杂合度HO0.7710.813J74观测杂合度HO0.9360.872期望杂合度HE0.7770.889期望杂合度HE0.9300.960多态信息含量PIC0.7810.868多态信息含量PIC0.9460.948显著性P0.5480.161显著性P0.6020.057等位基因数A2013等位基因数A2421等位基因丰富度RS19.74513.000等位基因丰富度RS23.53820.914J19观测杂合度HO0.8750.717J80观测杂合度HO0.6670.851期望杂合度HE0.8960.906期望杂合度HE0.6460.947多态信息含量PIC0.8960.888多态信息含量PIC0.9050.934显著性P0.3930.029显著性P0.7380.122等位基因数A2422等位基因数A14.81813.818等位基因丰富度RS23.87121.746等位基因丰富度RS14.68913.762J25观测杂合度HO0.9170.896平均观测杂合度HO0.7310.731期望杂合度HE0.9300.927average期望杂合度HE0.7710.857多态信息含量PIC0.9430.911多态信息含量PIC0.7460.845显著性P0.3920.154显著性P0.3380.053
注:*表示经过Sequential Bonferroni校正后,仍然背离哈迪-温伯格平衡(校正P值≤0.005)
Note: * indicates significant deviation from genetic Hardy-Weinberg equilibrium (adjusted P-value≤0.005)
表5 金乌贼野生群体间分配分析矩阵
Tab.5 Population structure in two wild populations of golden cuttlefish Sepia esculenta
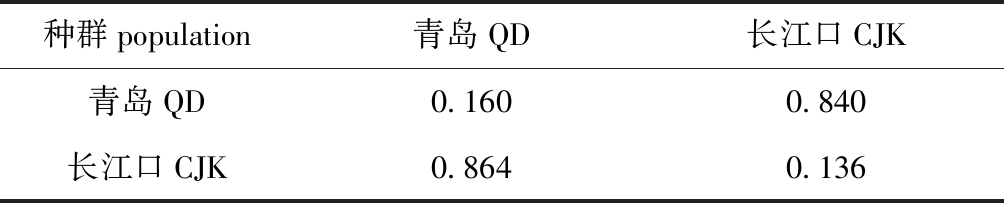
种群population青岛QD长江口CJK青岛QD0.1600.840长江口CJK0.8640.136
尽管RAD测序具有传统磁珠富集法无可比拟的优势,但开发的微卫星序列类型单一,酶切对微卫星结构造成的破坏,使其获得的微卫星标记均为完美型[37-38],这可能在一定程度上会对评价遗传多样性产生局限。本研究中应用磁珠富集法开发的20对具有多态性金乌贼微卫星标记,各种类型均有,其中,二核苷酸微卫星占开发总数的85%,比例较高,此现象在花鲈Lateolabrax maculates、长薄鳅Leptobotia elongata、缢蛏Sinonovacula constricta等物种微卫星开发中也较为常见[38-40]。核苷酸类型与多态性的关系并无统一说法,高焕等[41]认为,中国明对虾基因组中二核苷酸序列微卫星标记多态性高于三、四核苷酸,且多态性与核心序列重复拷贝数有关;而廖小林[42]则认为,四核苷酸重复的微卫星位点具有较高的遗传多样性。本研究中开发的二核苷酸微卫星序列多态性低于四核苷酸微卫星序列,而二核苷酸序列核心单元平均重复次数高于四核苷酸序列,故认为位点多态性与核苷酸数量或重复次数并无直接线性关系,这与蔡磊等[43]的结论一致。
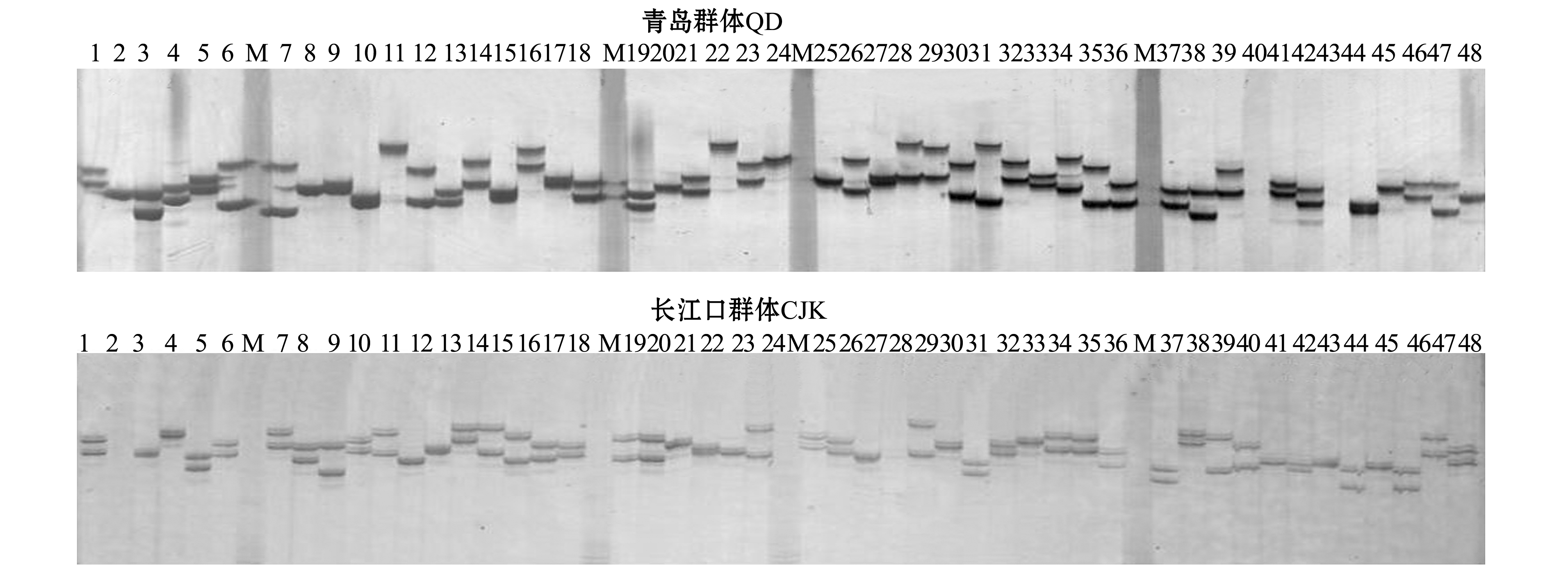
注:M为2000 bp的DNA标准片段(DL2000);1~48为青岛或长江口每个群体里的48个个体
Note: M,DNA Ladder 2000;1-48 shows 48 individuals in the two populations
图2 青岛和长江口群体在位点Secu 3的聚丙烯酰胺凝胶电泳图谱
Fig.2 Polyacrylamide gel electrophoresis of locus Secu 3 in two wild populations QD and CJK
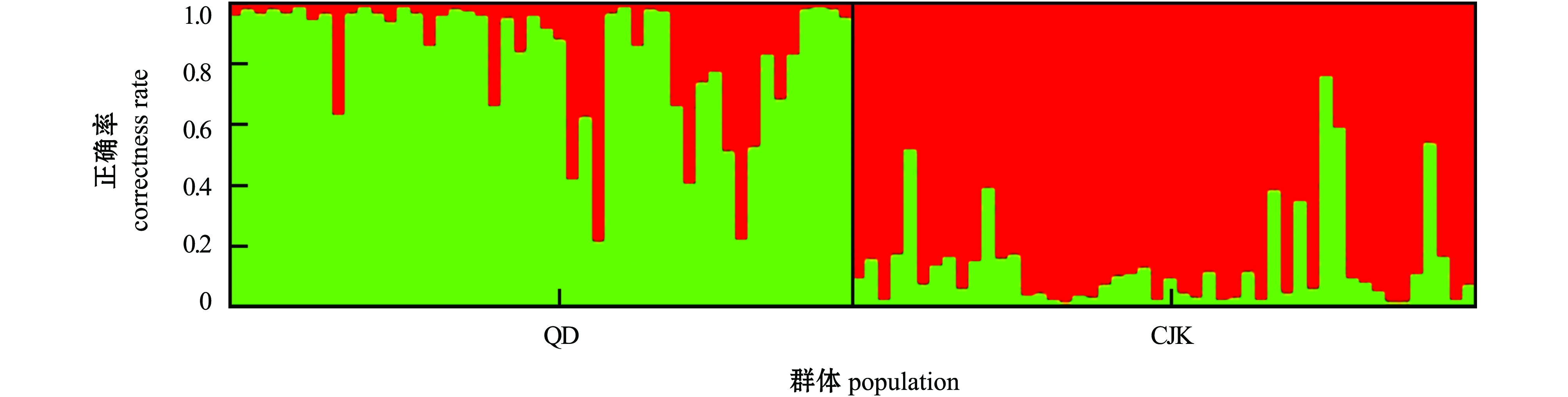
注:绿色表示青岛群体,红色表示长江口群体;纵坐标表示每个群体的个体被正确分配到该种群的概率
Note:Red indicates Qingdao group, green indicates Changjiangkou group; ordinate signs the rate of each individuals classified to the correct population
图3 金乌贼野生群体间分配分析图
Fig.3 Population structure in two wild populations of golden cuttlefish Sepia esculenta
3.2 金乌贼微卫星位点开发
本试验中采用的是生物素结合磁珠富集法获取微卫星标记位点,在水产动物微卫星标记的开发中有广泛应用[37-43]。目前,关于金乌贼微卫星标记开发的报道仅有2007年Zheng等[20]报道的11个位点,其中10个位点符合哈迪—温伯格平衡。其研究中用45~50个日本爱媛(Ehime)金乌贼野生个体评价引物,得到结果为:11个位点都具有多态性,每个位点的等位基因数为7~27,平均等位基因数为17;观测杂合度范围为0.380~0.980,平均为0.792;期望杂合度范围为0.645~0.940,平均为0.856;微卫星标记得到的种群遗传多样性水平明显高于同工酶标记所得到的结果(HE=0.017~0.025,HO=0.016~0.023)。鉴于金乌贼的经济性,本试验中为增加金乌贼种群多样性研究的基础数据,开发了该物种的微卫星标记,与已有报道相比,本研究中开发的微卫星位点平均等位基因数、观测杂合度和期望杂合度都略低,说明日本爱媛(Ehime)种群遗传多样性水平高于青岛群体。
与已有报道的其他头足类相比,中国青岛金乌贼种群的平均等位基因数为14.818,平均期望杂合度为0.771,其遗传多样性略低于枪乌贼Loligo forbesi(平均等位基因数为10.6,平均期望杂合度为0.79;同工酶检测的期望杂合度为0.08)[44]、皮氏枪鱿鱼Loligo pealeii(平均等位基因数为10.5,平均期望杂合度为0.84)[45]、真蛸Octopus vulgris(平均期望杂合度为0.91)[46];阿根廷滑柔鱼Illex argentines(平均等位基因数为24.1,平均期望杂合度为0.84)[47]、秘鲁小鱿鱼Loligo gahi(平均等位基因数14.7,平均期望杂合度为0.87)[48]、埃布短柔鱼Todaropsis eblanae(平均等位基因数为17.6,平均期望杂合度为0.90)[49],但高于前人研究的英吉利海峡商乌贼Sepia officinalis(平均等位基因数为7.0,平均期望杂合度0.575)[50]和比斯开湾商乌贼(平均等位基因数9.6,平均期望杂合度0.725)[51]时得到的遗传多样性数据。
3.3 野生群体遗传多样性比较
期望杂合度、观测杂合度、多态信息含量等遗传学参数可衡量个体在微卫星位点上的遗传与变异水平,是检测种群遗传多样性的重要评价指标,其中,期望杂合度较观测杂合度更能准确评价种群遗传多样性水平[52-53]。本研究中两个野生金乌贼群体均有较高的观测杂合度(平均值为0.731)、期望杂合度(平均值为0.814)。根据Botstein的标准,除青岛群体有两个位点的多态信息含量处于中度多态水平外,其余均处于高度多态水平(0.614~0.946>0.5)。这说明中国海域的金乌贼野生群体具有较高的遗传多样性。比较两个群体,尽管平均等位基因数和群体特有等位基因数均为青岛群体高于长江口群体,但长江口群体具有更高的多态信息含量和平均期望杂合度,综合认为,长江口群体较青岛群体有更丰富的遗传信息,多样性水平略高。
此外,单彬彬等[5]基于线粒体COI基因序列比较分析了乳山、青岛、日照、舟山4个金乌贼地理群体的遗传多样性,结果表明,黄海金乌贼3个群体(乳山、青岛、日照)间遗传分化较小(-0.007~0.039), 其与东海舟山群体间遗传分化较大(0.186~0.292),由此提示,山东的金乌贼增殖放流活动可能对其遗传多样性水平产生了一定影响。本研究中两个群体的遗传分化值为0.032 5,根据Wtight[54]提出的种群间遗传分化指数(Fst)介于0~0.05为较低水平的遗传分化,0.05~0.15为中等分化,0.15~0.25为高水平遗传分化,大于0.25为极大遗传分化的理论,本研究中青岛群体和长江口群体的遗传分化水平较低。长江口群体采样位置与单彬彬等[5]的舟山群体相近,得到的遗传分化水平却存在差异,推测与研究方法不同有关。Zheng等[19,21]用微卫星和COI基因两种分子标记法分析日本福井、长崎、爱媛、爱知及中国日照5个群体的遗传结构,因方法不同计算得到的遗传分化指数(Fst)相差较大。以遗传分化较大的福井与日照群体Fst值为例,COI分析得到Fst为0.725 9,而用微卫星标记获得的Fst仅为0.024,这也较好地解释了本研究与单彬彬等[5]研究结果的差异。综合比较,本研究与Zheng等[19]用微卫星标记研究金乌贼不同地理群体间的遗传分化指数(0.037)较为接近。
从分配分析结果来看,本研究中两个采样地点的种群明显被分为两个不同群体,与单彬彬等[5]用线粒体COI基因研究发现黄海金乌贼与东海金乌贼不存在随机交配,基因流较弱的结果相吻合。
[1] 李嘉泳.金乌贼Sepia esculenta Hoyle在黄渤海的结群生殖和洄游[J].山东海洋学院学报,1963(2):69-108.
[2] Okutani T.Cuttlefish and Squids of the World in Color[M].Tokyo:National Cooperative Association of Squid Processors,1995:43.
[3] 郝振林,张秀梅,张沛东.金乌贼的生物学特性及增殖技术[J].生物学杂志,2007,26(4):601-606.
[4] 刘莉莉,万荣,段媛媛,等.山东省海洋渔业资源增殖放流及其渔业效益[J].海洋湖沼通报,2008(4):91-98.
[5] 单彬彬,宋娜,刘淑德,等.基于线粒体COI基因序列的金乌贼群体遗传学研究[J].中国海洋大学学报:自然科学版,2017,47(5):50-56.
[6] 卢晓,董天威,刘沛栋,等.山东省金乌贼增殖放流回顾与思考[J].齐鲁渔业,2018,35(1):42-46.
[7] 汪金海,韩松,郑小东,等.金乌贼(Sepia esculenta)繁殖模式的分子学鉴定[J].海洋与湖沼,2017,48(1):184-189.
[8] 韦柳枝,高天翔,韩志强,等.日照近海金乌贼生物学的初步研究[J].中国海洋大学学报:自然科学版,2005,35(6):923-928.
[9] 雷舒涵.金乌贼胚胎与幼体发育生物学研究[D].青岛:中国海洋大学,2013.
[10] 金洋,薛张芝,张洪超,等.金乌贼肌肉中三甲胺脱甲基酶的分离纯化及酶学性质[J].水产学报,2017,41(6):845-853.
[11] 刘长琳,赵法箴,陈四清,等.金乌贼胚胎发育阶段主要生化成分的变化[J].中国海洋大学学报:自然科学版,2016,46(11):62-72.
[12] 刘长琳,阮飞腾,秦搏,等.野生金乌贼成体肌肉的营养成分分析及评价[J].海洋科学,2016,40(8):42-48.
[13] 刘长琳,刘思玮,赵法箴,等.金乌贼(Sepia esculenta)早期发育阶段相关酶活性的变化[J].渔业科学进展,2016,37(6):105-109.
[14] 周维武.金乌贼(Sepia esculenta Hoyle)人工孵化与培育技术[J].现代渔业信息,2007,22(11):27-29.
[15] 陈四清,刘长琳,庄志猛,等.饵料、盐度对金乌贼幼体生长的影响[J].渔业现代化,2008,35(6):23-25,32.
[16] 刘长琳,庄志猛,陈四清,等.金乌贼亲体驯养与繁殖特性研究[J].渔业现代化,2009,36(2):34-42.
[17] Zheng Xiaodong,Zhao Jiaming,Xiao Shu,et al.Isozymes analysis of the golden cuttlefish Sepia esculenta (Cephalopoda:Sepiidea)[J].Journal of Ocean University of China,2004,3(1):48-52.
[18] Wei Liuzhi,Gao Tianxiang,Zhang Xiumei.Isozymes analysis of Sepia esculenta (Cephalopoda:Sepiidea)[J].Journal of Fishery Sciences of China,2005,12(5):449-555.
[19] Zheng X D,Ikeda M,Kong L F,et al.Genetic diversity and population structure of the golden cuttlefish,Sepia esculenta (Cephalopoda:Sepiidea) indicated by microsatellite DNA variations[J].Marine Ecology,2009,30,448-454.
[20] Zheng Xiaodong,Ikeda M,Barinova A,et al.Isolation and characterization of microsatellite DNA loci from the golden cuttlefish, Sepia esculenta Hoyle (Cephalopoda)[J].Molecular Ecology Notes,2007,7(1):40-42.
[21] 郑小东,许然,池田実,等.基于COI基因的西北太平洋金乌贼种群遗传学研究[J].中国海洋大学学报:自然科学版,2017,47(9):55-61.
[22] 孙孝文,张晓锋,赵莹莹,等.水产生物微卫星标记技术研究进展及其应用[J].中国水产科学,2008,15(4):689-703.
[23] 徐浩,鲁翠云,孙孝文.利用164个微卫星标记分析镜鲤家系的遗传多样性和经济性状[J].大连海洋大学学报,2013,28(3):247-253.
[24] 董颖,杨瑞,姜志强,等.微卫星标记在人工养殖小体鲟种群的数据分析方法比较和遗传多样性分析[J].大连海洋大学学报,2016,31(5):516-521.
[25] 李云霞,李娇,丁君.等.基于微卫星标记的刺参群体遗传结构分析及与经济性状的相关性研究[J].大连海洋大学学报,2013,28(5):438-444.
[26] Sambroo J,Fitch E F,Maniatis T.Molecular Cloning:A Laboratory Manual[M].2nd ed.Cold Spring Harbor,N.Y.:Cold Spring Harbor Laboratory Press,1989:463-470.
[27] Raymond M,Rousset F.Genepop (version 1.2):population genetics software for exact tests and ecumenicism[J].Journal of Heredity,1995, 86(3):248-249.
[28] Weber J L.Informativeness of human (dC-dA)n·(dG-dT)n polymorphisms[J].Genomics,1990,7(4):524-530.
[29] Beadell J S,Hyseni C,Abila P P,et al.Phytogeography and population structure of Glossina fuscipes fuscipes in Uganda:implications for control of testes[J].PLoS Neglected Tropical Diseases,2010,4(3)e636.
[30] 林婷婷.大刺鳅(Mastacembelus armatus)微卫星标记开发及野生群体遗传多样性分析[D].广州:广州大学,2017.
[31] 王洋坤,胡艳,张天真.RAD—seq技术在基因组研究中的现状及展望[J].遗传,2014,36(1):41-49.
[32] Barchi L,Lanteri S,Portis E,et al.Identification of SNP and SSR markers in eggplant using RAD tag sequencing[J].BMC Genomics,2011,12:304.
[33] Hohenlohe P A,Bassham S,Etter P D,et al.Population genomics of parallel adaptation in three spine stickleback using sequenced RAD tags[J].Plos Genetics,2010,6(2):e1000862.
[34] 韩承慧,马海涛,姜海滨,等.许氏平鲉(Sebastes schlegeli)微卫星标记开发及野生、养殖群体遗传多样性分析[J].海洋与湖沼,2016,47(1):213-220.
[35] Andrews K R,Luikart G.Recent novel approaches for population genomics data analysis[J].Molecular Ecology,2014,23(7):1661-1667.
[36] Hand B K,Lowe W H,Kovach R P,et al.Landscape community genomics:understanding eco-evolutionary processes in complex environments[J].Trends in Ecology & Evolution,2015,30(3):161-168.
[37] 常诚,韩慧宗,王腾腾,等.单环刺螠(Urechis unicinctus)微卫星标记开发及5个地理种群遗传结构分析[J].海洋与湖沼,2017,48(3):498-507.
[38] 翟云,吴仁协,牛素芳,等.采用高通量技术开发花鲈二碱基重复微卫星标记[J/OL].基因组学与应用生物学, 2018[2018-09-28].http://www.cnki.net/KCMS/detail/45.1369.Q.2018 0928.0959.002.html
[39] 熊飞,刘红艳,段辛斌,等.长薄鳅基因组四碱基重复微卫星的分离及序列特征分析[J].华中师范大学学报:自然科学版,2013,47(6):824-829.
[40] 吴雪萍,马海涛,冯艳微,等.缢蛏(Sinonovacula constricta)微卫星标记的分离及近缘物种通用性[J].海洋与湖沼,2014,45(6):1330-1337.
[41] 高焕,于飞,栾生,等.中国明对虾基因组微卫星重复单元类型与其多态性关系[J].水生生物学报,2009,33(1):94-102.
[42] 廖小林.长江流域几种重要鱼类的分子标记筛选开发及群体遗传分析[D].武汉:中国科学院水生生物研究所,2006.
[43] 蔡磊,陈小曲,郑伟强,等.诸氏鲻虾虎鱼多态性微卫星标记的开发及评价[J].中国实验动物学报,2015,23(1):57-62.
[44] Shaw P W,Pierce G J,Boyle P R.Subtle population structuring within a highly vagile marine invertebrate, the veined squid Loligo forbesi, demonstrated with microsatellite DNA markers[J].Molecular Ecology,1999,8(3):407-417.
[45] Maxwell M R,Buresch K,Hanlon R T.Pattern of inheritance of microsatellite loci in the squid Loligo pealeii (Mollusca:Cephalopoda)[J].Marine Biotechnology,2002,2(6):517-521.
[46] Murphy J M, Balguerías E,Key L N,et al.Microsatellite DNA markers discriminate between two Octopus vulgaris (Cephalopoda:Octopoda) fisheries along the northwest African coast[J].Bulletin of Marine Science,2002,71(1):545-553.
[47] Adcock G J,Shaw P W,Rodhouse P G,et al.Microsatellite analysis of genetic diversity in the squid Illex argentinus during a period of intensive fishing[J].Marine Ecology,1999,187:171-178.
[48] Shaw P W,Arkhipkin A I,Adcock G J,et al.DNA markers indicate that distinct spawning cohorts and aggregations of Patagonian squid,Loligo gahi,do not represent genetically discrete subpopulations[J].Marine Biology,2004,144(5):961-970.
[49] Dillane E,Galvin P,Coughlan J,et al.Genetic variation in the lesser flying squid Todaropsis eblanae (Cephalopoda,Ommastrephidae) in east Atlantic and Mediterranean waters[J].Marine Ecology Progress Series,2005,292:225-232.
[50] Wolfram K,Mark F C,John U,et al.Microsatellite DNA variation indicates low levels of genetic differentiation among cuttlefish (Sepia officinalis L.) populations in the English Channel and the Bay of Biscay[J].Comparative Biochemistry and Physiology, 2006,1(3):375-383.
[51] Pérez-Losada M,Guerra A,Carvalho G R,et al.Extensive population subdivision of the cuttlefish Sepia officinalis (Mollusca:Cephalopoda) around the Iberian Peninsula indicated by microsatellite DNA variation[J].Heredity,2002,89(6):417-424.
[52] Nei M.Molecular Evolutionary Genetics[M].New York:Columbia University Press,1987.
[53] Leberg P L.Estimating allelic richness:effects of sample size and bottlenecks[J].Mol Ecol,2002,11(11):2445-2449.
[54] Wtight S.Ecolution and the genetics of populations:variability within and among natural populations[M].Chicago:University of Chicago Press,1978.