近年来,随着深度测序技术的飞速发展,生物组学在水产动物生长发育、生物代谢、组织进化等诸多方面起着重要的作用[1-2]。组学技术主要包括基因组学、蛋白质组学和转录组学,其中,转录组学是后基因组学的象征,是组学数据获取的最常用方法之一[3]。转录组是在RNA水平上所有转录本的总和,包括编码的mRNA和非编码的RNA,可充分反映出某一时期或者某一特定条件下组织细胞基因表达和生物代谢通路的调节情况,具有高灵敏、高分辨和易重复的特性[4]。目前,该技术在斑马鱼Danio rerio[5]、大竹蛏Solen grandis[6]、花斑裸鲤Gymnocypris eckloni[7]、大菱鲆Scophthalmus maximu[8]、鲤Cyprinus carpio[9]、白鲑Coregonus spp.Salmonidae[10]、大鲵Andrias davidianus[11]和达氏鳇Huso dauricus[12]等水产动物中已被广泛应用,均获得了较多的高通量测序数据。
锦鲤Cyprinus carpio是中国养殖的主要观赏鱼类之一,红白锦鲤Cyprinus carpio haematopterus、大正三色锦鲤Taisho sanshoku、昭和三色锦鲤Taisho sanke是锦鲤十三大品系中的3个主流品系,其中红白锦鲤是由红色和白色两种体色组成,其他两个品系则由红色、白色和墨色3种体色组成。锦鲤是鲤鱼的变种,经过几百年来的杂交选育而来,其体色变幻多端,如人的指纹一般,无相同者,即使是同一尾鱼在不同生长时期、不同生长环境中,其体表花纹的颜色和形状也截然不同,因此,锦鲤被认为是研究鱼类体色变化过程和机理的重要模型。锦鲤的价值取决于体型、体色和斑纹的分布,因锦鲤遗传机制较为模糊,目前选育优质锦鲤仍应用较为原始的苗种挑选方法,即从数以万计的锦鲤苗种中挑选出几十尾或者几尾价值较高的个体,人力、物力和财力耗费较大,且要求操作者具有极为丰富的养殖经验[13]。因此,亟待需要利用先进的生物技术手段,通过相关组学技术揭示锦鲤体色遗传规律,从而攻克优质锦鲤选育难题。本研究中,以红白锦鲤为研究对象,在应用转录组测序基础上,通过基因结构、基因表达和肤色差异基因功能注释分析,综合阐述红白锦鲤转录组信息特点,以期为红白锦鲤的体色遗传机制研究提供基础数据,同时也为多体色水产动物基因调控和新品种选育提供基础资料。
1 材料与方法
1.1 材料
试验用红白锦鲤取自北京通州鑫淼水产总公司,体长为(28.34±3.12)cm。试验开始前,将试验用鱼在温室暂养池暂养7 d,以减少环境对基因转录表达的影响。暂养时,水温为(25±1)℃,曝气自来水,每日光照8~10 h,每日9:00、17:00分别投喂颗粒饲料,投喂总量为鱼体质量的2%~3%。
1.2 方法
1.2.1 试验设计 试验开始时,随机选取生长状态良好的健康红白锦鲤10尾,采用MS-222(Sigma,USA)将试验鱼麻醉,分别取红色皮肤、白色皮肤等2个组织混合样品,每个混样200 mg,分别用Trizol Reagent法提取总RNA,各样品设3个生物学重复。各混样总RNA用琼脂糖凝胶电泳、Nanodrop、Qubit、Qsep 100等方法对降解与污染程度、浓度、纯度、完整性等进行检测。随后,对检测合格的混样进行文库构建和拼接组装,用Qubit 3.0和Q-PCR方法对文库有效浓度、插入片段等进行定量。进行基因功能注释的数据库包括Nr、GO、KEGG、KOG、Pfam、Swiss-Prot和eggNOG,利用COG和KEGG数据库进行Unigene的功能注释和代谢通路分类,并进行GO分类统计,得到注释信息。
1.2.2 转录组数据分析 将过滤得到高质量Clean Data进行拼接组装,获得转录组序列,将此序列与鲤鱼基因组(ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCF/000/951/615/GCF_000951615.1_common_carp_genome/GCF_000951615.1_common_carp_genome_genomic.fna.gz)进行比对,获得比对效率。用BLAST软件分别与Nr、GO、KEGG、KOG、Pfam、Swiss-Prot、eggNOG等7个数据库进行比对,并进行基因功能、差异基因筛选等数据分析。
1.2.3 qRT-PCR验证分析 从转录组数据库随机选取9个差异基因进行qRT-PCR验证。基于测序获得mRNA序列,使用Primer3(http://bioinfo.ut.ee/primer3-0.4.0/)软件在线设计扩增引物序列(表1),送交北京擎科生物科技有限公司合成引物序列。根据文献[14]中的方法选择鲤β-actin基因为内参,以SYBR GREEN(TaKaRa,DRR041A)为荧光染料,采用荧光定量PCR仪(ABI,Stepone)进行实时荧光定量分析。反应体系(共20 μL):上、下游引物各1 μL,SYBR green 10 μL,cDNA模板1 μL,ddH2O 7 μL。反应程序为:95 ℃下预变性30 s;95 ℃变性5 s,60 ℃下退火并延伸30 s,共进行40个循环。相对表达量按照 2-ΔΔCT 方法计算。所有样本设置3个生物学重复。
表1 qPCR引物序列信息
Tab.1 Primer sequence information required for qPCR

基因 IDgene ID引物 primer序列 sequence(5′-3′)产物长度/bp product length1001510015-F10015-RTTACGCCAGTTACGTACGTAGACTGAAGACTAGCCGTGCTGGAG1961002410024-F10024-RATCGATCGGATCTGAACGTCAGCCATCGGTACTACGGTGAGCTAG1681008810088-F10088-RTGGCACGTGTACCAGAGTCGATACTGTCTCTACTCAGAGTTCGATCA1801009210092-F10092-RGAGCTATACTCTGCGATATCGGTAACGCGAATGCTGTGAGCC1751012510125-F10125-RGACAGCACTCTACGGACCTAGCTTGTAGTCATGAACCTTCGAC2031012810128-F10128-RCTATATAGCATGTGTCATACCAAGTCTAGCCATGAAGCATCG1521022510225-F10225-RGTCTAGTCCGACTGTAGCGTGATGTCTGAACTGATGTGCAT1881027310273-F10273-RTGGTCTATGTGAGCTCGACGCGTGCGGATAGCGAGGCTATG1711028710287-F10287-RTCGATGGCGCTCTGTATGTAGTCCGTGAGTGTGACAGTGCGAGG18510193(β-actin)10193-F10193-RGAAGAGTTACGAGCTGCCTGACATGGATACCGCAAGATTCC106
2 结果与分析
2.1 转录组测序与组装
经Illumina Hiseq 2500测序,获得116.26 Gb Clean Data,各混养数据量均达到8.24 Gb。从表2可知,Q20值为95.74%以上,Q30值为89.82%以上,GC含量为45.97%~57.59%,测序错误率为0.03%。上述测序评估表明,测序质量较高,可用于试验后续数据分析。选用TopHat2软件,将红白锦鲤皮肤各混样转录组测序Clean Reads与指定参考基因组进行比对,比对效率为61.15%~70.91%。对转录组测序所获得的reads进行组装和拼接,获得转录本Unigene总长度为355 100 379 bp,平均长度为2180 bp。
表2 样品测序数据质量评估表
Tab.2 Sequencing data quality assessment
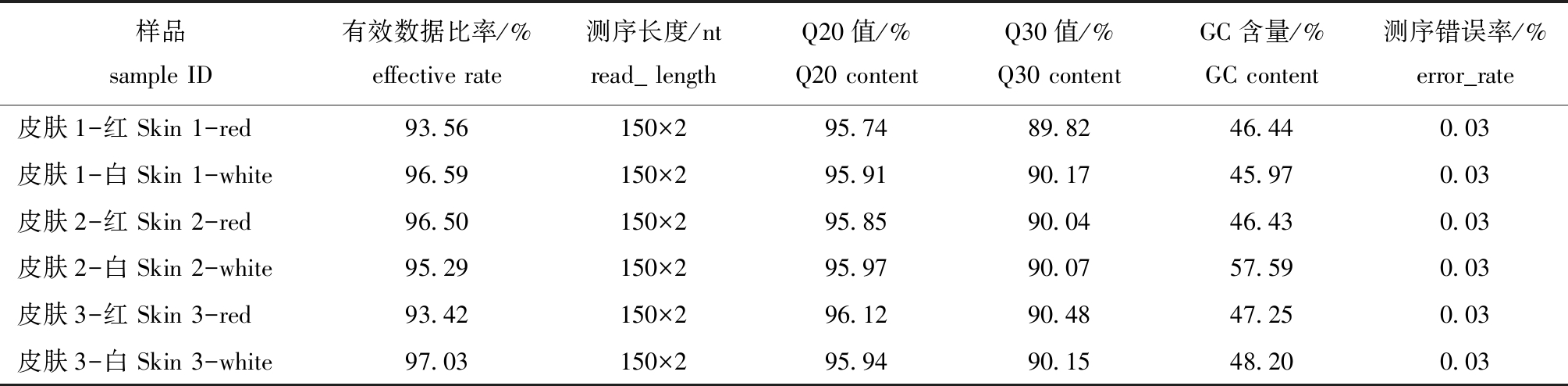
样品sample ID有效数据比率/%effective rate测序长度/ntread_ lengthQ20值/%Q20 contentQ30值/%Q30 contentGC含量/%GC content测序错误率/%error_rate皮肤1-红 Skin 1-red93.56150×295.7489.8246.440.03皮肤1-白 Skin 1-white96.59150×295.9190.1745.970.03皮肤2-红 Skin 2-red96.50150×295.8590.0446.430.03皮肤2-白 Skin 2-white95.29150×295.9790.0757.590.03皮肤3-红 Skin 3-red93.42150×296.1290.4847.250.03皮肤3-白 Skin 3-white97.03150×295.9490.1548.200.03
2.2 基因功能注释和新转录本预测
选用7个数据库进行基因数量的BLAST比对,如表3所示,不同数据库因筛选条件不同,注释成功后的基因数量有较大差别。7个数据库中Nr数据库注释成功45 432个基因,注释比例最高为35.62%,KOG注释成功基因数量最少,注释比例最低。所有转录组在所有数据库(All)注释成功为10 476个基因。将所有测序reads数据的基因组定位结果放到一起,用Cufflinks进行组装,然后用Cuffcompare和已知基因模型进行比较,以发现新的未知基因、已知基因新的外显子区域,并对已知基因的起止位置进行优化,优化后的转录本与7个数据库进行比对,有9040个新转录本得到功能注释。
表3 红白锦鲤基因功能注释统计
Tab.3 New gene function anotation
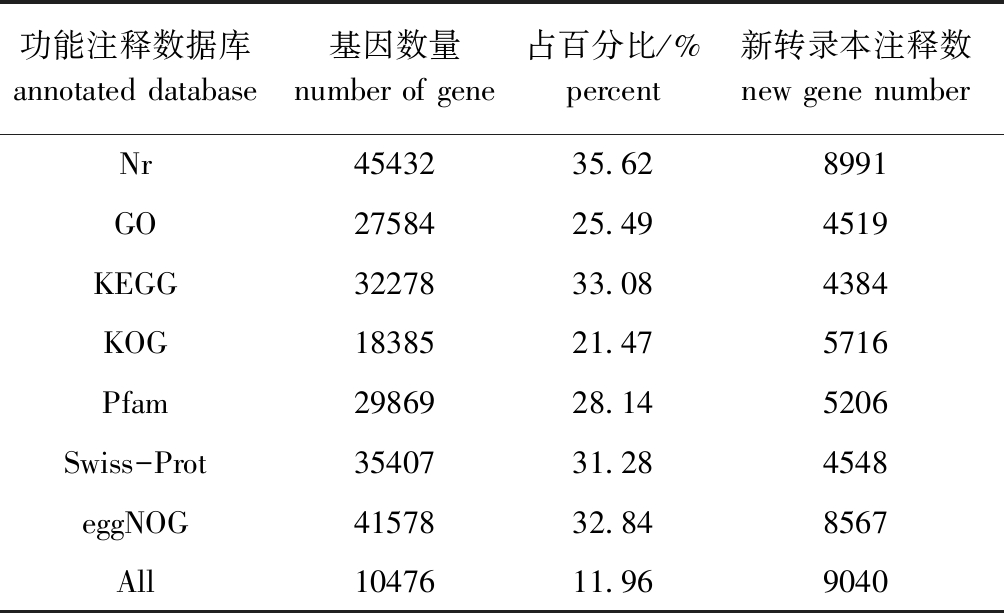
功能注释数据库annotated database基因数量number of gene占百分比/%percent新转录本注释数new gene numberNr4543235.628991GO2758425.494519KEGG3227833.084384KOG1838521.475716Pfam2986928.145206Swiss-Prot3540731.284548eggNOG4157832.848567All1047611.969040
2.3 差异表达分析
为了分析红白锦鲤成鱼肤色调控基因,将红色部位皮肤样品与白色部位皮肤样品测序结果进行相关筛选分析,即将fold change≥2或fold change≤1/2且FDR<0.01作为筛选标准,筛选差异表达基因,统计结果如图1所示,其中Skin 1、Skin 2、Skin 3三个组共同表达的差异基因有532个,Skin 1与Skin 2组、Skin 2与Skin 3组、Skin 1与Skin 3组共表达差异基因分别为276、648、690个。
2.4 差异表达基因的富集和分类分析
2.4.1 KEGG富集分析 如图2所示,红、白肤色样品对比差异表达基因KEGG分类后,主要注释到细胞进程类目下肌动蛋白细胞骨架调节通路(注释基因数为75)、黏着斑通路(82)、紧密连接通路(58),环境信息处理类目下钙信号通路(84)、MAPK信号通路(70),以及有机系统类目下心肌细胞中的肾上腺素能信号转导通路(66)。此外,注释基因数较高的通路还有:内吞作用通路(75)、细胞黏附分子通路(49)、刺激神经组织的配体受体交互作用通路(70)、嘌呤代谢(47)、胰岛素信号通路/黑素生成/Wnt信号通路/碳代谢通路(44/35/42/27)、细胞因子受体相互作用通路(38)。
2.4.2 GO富集分析 皮肤样品差异表达基因归类到分子功能的基因最多,归类到生物过程的基因最少,经对比发现,具有明显比例差异的二级功能主要有细胞组成类目下胶原蛋白三聚物、细胞外基质,分子功能类目下核酸结合转录因子活性、脒基核苷酸交换因子活性、电子载体活性,以及生物过程类目下的生物黏附、多细胞生物历程、发展过程和生长。
为更直观地反映出在生物过程、细胞组成和分子功能富集的GO term上差异基因的个数分布情况,挑选了富集最显著的GO term构建了差异基因GO富集柱状图,如图3所示。差异表达基因从生物学功能角度分析,主要富集到细胞过程、单组织过程、代谢过程;从细胞组成角度分析,主要富集到细胞组分、细胞、细胞器;从分子功能角度分析,主要富集到黏合、催化活性、转运活性。
2.4.3 COG分类 利用COG数据库对基因产物进行直系同源分类,差异表达基因COG分类统计结果如图4所示。差异基因聚集最多的是通用功能(基因数为180,所占比例为28.12%),其次为信号转导机制(86,13.44%)、复制、重组、修复(78,12.19%)、转录(85,13.28%)、细胞骨架(29,4.53%)。与细胞运动、细胞外结构、核结构相关的差异基因最少,均为0。
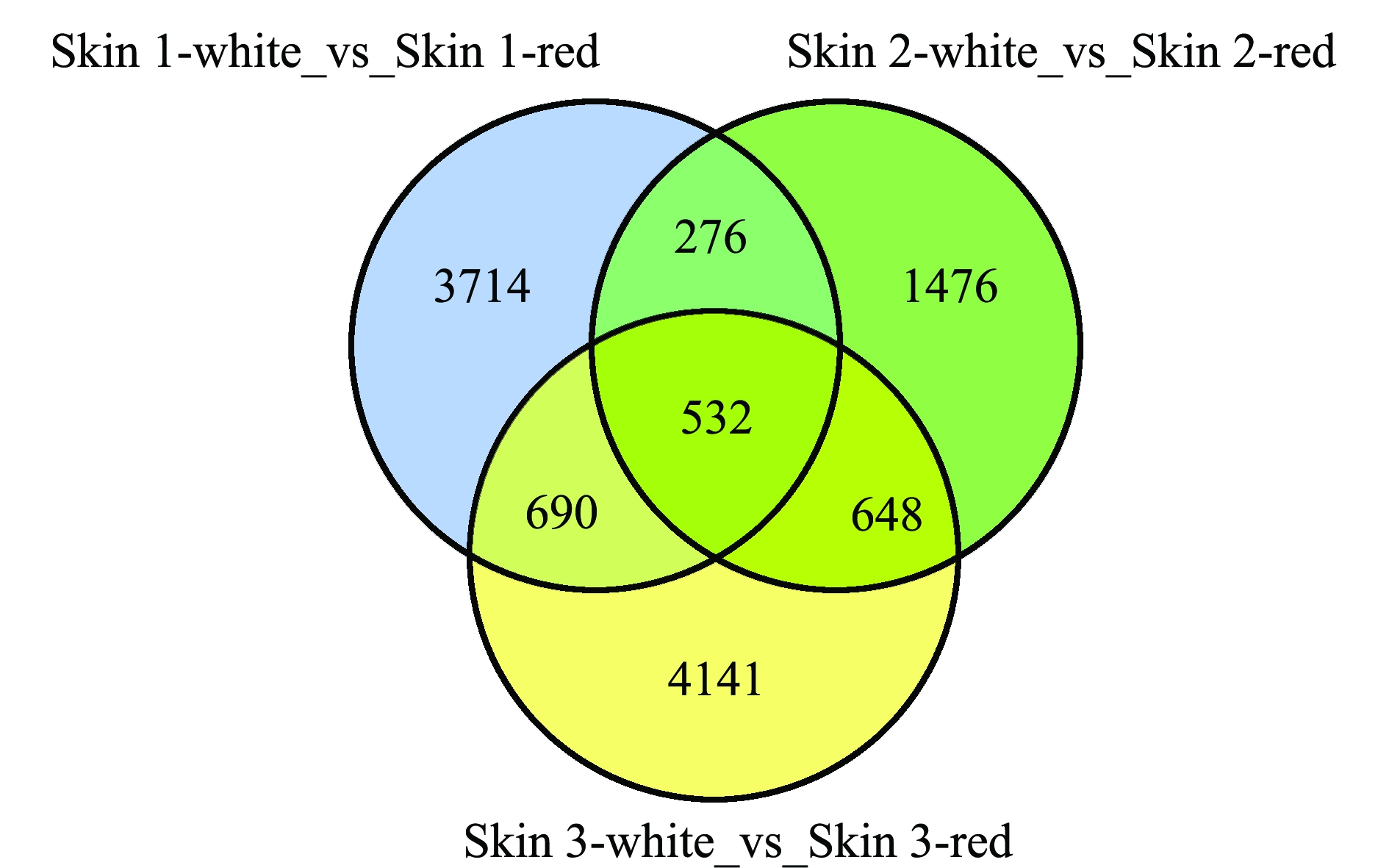
图1 差异基因维恩图
Fig.1 Venn diagram of DEGs
2.5 qRT-PCR验证
从转录组数据库随机选取9个差异基因(基因ID分别为10015、10024、10088、10092、10125、10128、10225、10273、10287)进行qRT-PCR验证,运用2-△△Ct方法对红白锦鲤皮肤中差异表达基因进行相对表达水平定量分析,结果发现,红白锦鲤皮肤差异表达基因的表达趋势与RNA-seq定量结果一致(图5),表明转录组测序结果是可靠的。

注:纵坐标为KEGG代谢通路的名称,横坐标为注释到该通路下的基因个数及其个数占该通路差异基因的比例
Note:The ordinate is the name of the KEGG metabolic pathway, and the abscissa is the number of genes annotated to the pathway, and the proportion of the number of genes in the pathway differential gene
图2 KEGG富集分析
Fig.2 KEGG enrichment analysis

注:图中右侧纵坐标蓝色字体表示差异基因(DE gene)数量,黑色字体表示总基因(All gene)数量
Note:The blue font on the right side of the figure represents the number of differential gene(DE gene), while the black font represents the number of all genes
图3 GO富集分析
Fig.3 GO enrichment analysis

图4 差异表达基因COG注释分类统计图
Fig.4 COG classification of DEGs
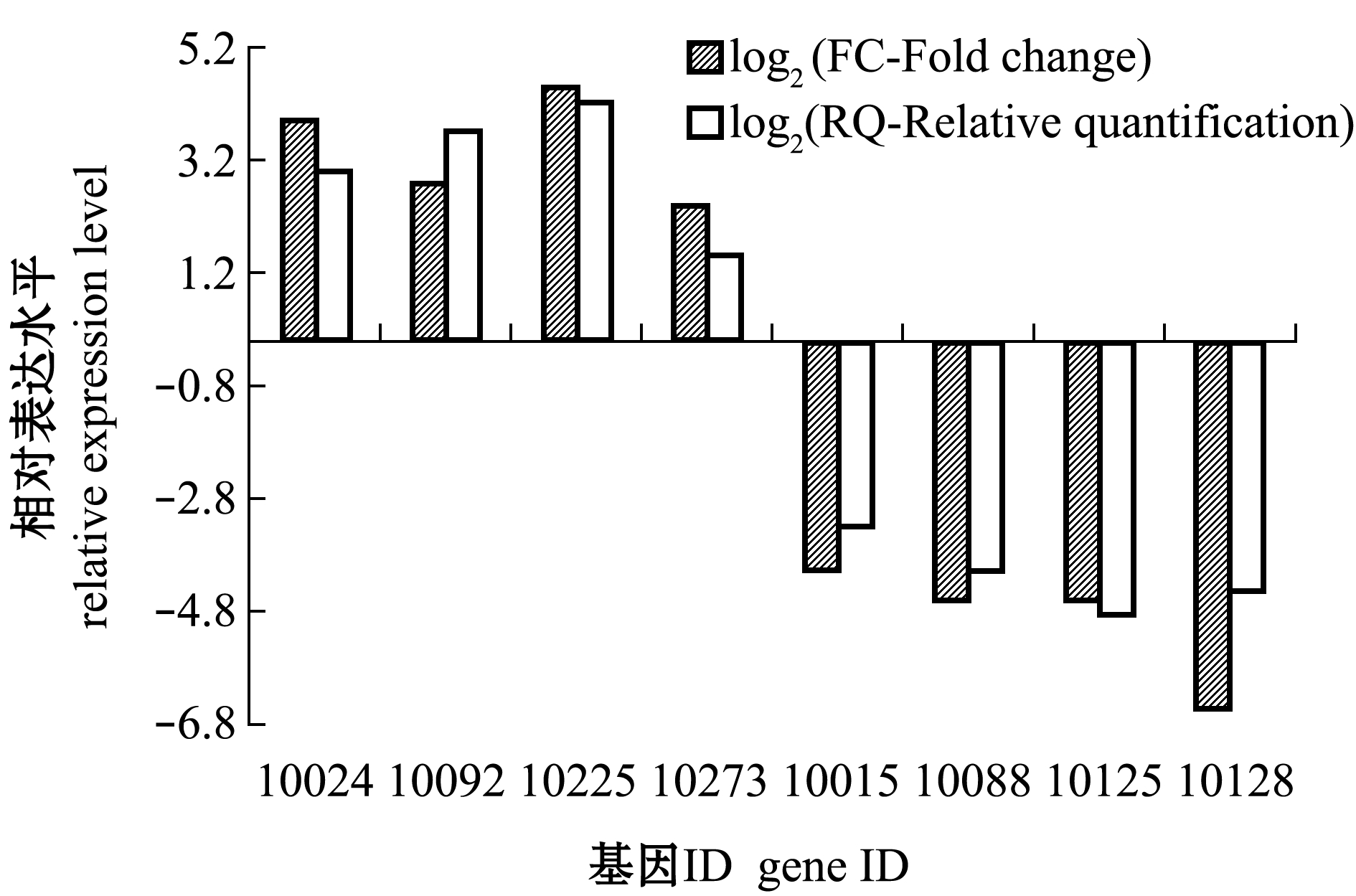
图5 qRT-PCR定量结果
Fig.5 Quantitative results of qRT-PCR
3 讨论
3.1 红白锦鲤转录组组装与注释分析
近年来,关于锦鲤体色的研究也多有文献报道,绝大多数集中于体表色素的形成机制,着色剂饲料增色效果、理化因子对体色的影响及单色锦鲤体色基因功能分析等[15-21],而从组学角度研究锦鲤体色调控机理则尚缺乏,可能是因为锦鲤色素调控机制复杂多变的缘故。因此,亟需利用先进的生物技术手段,通过相关组学技术揭示锦鲤体色遗传规律,从而破解生物难题。本研究中在参考鲤鱼基因组的情况下,采用Illumina Hiseq PE150测序技术对红白锦鲤皮肤转录组进行测序分析,初步得到了红白锦鲤白色和红色皮肤的转录组特点,可为今后观赏鱼肤色关键基因开发及功能分析等研究提供基础数据。本研究中,对红白锦鲤转录组文库序列分别在7个生物学常用数据库中进行比对,Nr数据库注释成功45 432个基因,注释比例最高为35.62%,此结果高于花斑裸鲤转录组31.32%的注释比例[7],但低于岩原鲤44.63%的注释比例[22]。虽然均为鲤科鱼类,且均为总RNA转录组数据在同一个数据库比对,但结果却相差明显。这可能与不同比对时期数据库收录的基因注释信息数量不同有关,也可能与取样部位不同相关。本试验中所用的总RNA来源于红白锦鲤的体表皮肤组织,而花斑裸鲤取样部位为脑、肌肉、肾组织和肝胰脏等4个部位,岩原鲤则为全组织转录测序。同时,在整体数据库注释中,未见有超过50%比例的序列与数据库基因无同源性。通过Nr数据库的比对,有64.38%皮肤基因未被注释成功,推测这些基因可能是调控红白锦鲤体色的基因,但这些基因可能不完全是调控红色或白色皮肤的基因,因为经过多年的锦鲤繁养殖经验,两尾亲本均为红白锦鲤的个体其后代也会出现三色锦鲤,即体色中也会出现墨色,同时,这些未被注释的基因也可能是非编码RNA,也可能不是功能基因,这些基因将是后期深入研究的重点。
3.2 红白锦鲤转录组文库功能分析
为分析红白锦鲤成鱼肤色调控基因,本研究中将红色与白色部位皮肤样品测序结果进行相关筛选,分析筛选差异表达基因,并对差异表达基因分别进行KEGG代谢通路分析、GO功能分类和COG基因产物同源分类。经KEGG分析,差异表达基因主要注释到肌动蛋白细胞骨架调节通路、黏着斑通路、紧密连接通路、钙信号通路、MAPK信号通路和心肌细胞中的肾上腺素能信号转导通路。GO富集分析显示,差异表达基因和全部基因具有明显比例差异的二级功能主要有胶原蛋白三聚物、细胞外基质、核酸结合转录因子活性、脒基核苷酸交换因子活性、电子载体活性、生物黏附、多细胞生物历程、发展过程和生长等。差异表达基因富集最显著的GO term主要分布在膜功能和ATP结合功能,钙离子结合,胞外区和肌动蛋白结合。通过COG分析,三个对比组差异基因最多聚集在通用功能,而与细胞运动、细胞外结构、核结构相关的差异基因最少。
4 结语
本研究中对红白锦鲤皮肤转录组进行了高通量测序,并通过对获得的大量转录组信息进行基因注释分类和对比分析,整体概述了各差异功能基因在众数据库中的归属,初步了解了差异基因参与的生物学过程和代谢通路,为后期红白锦鲤肤色调控基因的克隆、肤色调控关键基因的筛选等提供了有价值的数据。同时,这些差异基因归类分析也为后期研究锦鲤肤色调控关键基因提供了基础数据,有利于进一步对锦鲤肤色调控关键基因的研究。
[1] Cui Jun,Xu Jian,Zhang Songhao,et al.Transcriptional profiling reveals differential gene expression of Amur ide (Leuciscus waleckii) during spawning migration[J].International Journal of Molecular Sciences,2015,16(6):13959-13972.
[2] Mininni A N,Milan M,Ferraresso S,et al.Liver transcriptome analysis in gilthead sea bream upon exposure to low temperature[J].BMC Genomics,2014,15:765.
[3] Lockhart D J,Winzeler E A.Genomics,gene expression and DNA arrays[J].Nature,2000,405(6788):827-836.
[4] Garg R,Patel R K,Tyagi A K,et al.De novo assembly of chickpea transcriptome using short reads for gene discovery and marker identification[J].DNA Research,2011,18(1):53-63.
[5] Hussainzada N,Lewis J A,Baer C E,et al.Whole adult organism transcriptional profiling of acute metal exposures in male zebrafish[J].BMC Pharmacology and Toxicology,2014(15):15-29.
[6] 聂洪涛,姜力文,郑梦鸽,等.大竹蛏高通量转录组测序数据组装和分析[J].大连海洋大学学报,2017,32(6):658-663.
[7] 赵永利,夏明哲,吴蓉蓉,等.基于高通量测序的花斑裸鲤转录组及功能分析[J].青海大学学报,2018,36(1):1-8.
[8] Pereiro P,Balseiro P,Romero A,et al.High-throughput sequence analysis of turbot (Scophthalmus maximus) transcriptome using 454-pyrosequencing for the discovery of antiviral immune genes[J].PLoS One,2012,7(5):e35369.
[9] 刘思嘉,田菲,张存芳,等.鲤在低温胁迫下肝胰腺转录组测序分析[J].生物技术通报,2018,34(11):168-178.
[10] Renaut S,Nolte A W,Bernatchez L.Mining transcriptome sequences towards identifying adaptive single nucleotide polymorphisms in lake whitefish species pairs (Coregonus spp.,Salmonidae)[J].Molecular Ecology,2010,19(S1):115-131.
[11] 黄勇,高小婵,孙西红.基于RNA-Seq高通量测序技术的大鲵转录组分析[J].基因组学与应用生物学,2018,37(9):3759-3768.
[12] 赵文,高峰英,石振广.达氏鳇肌肉组织转录组测序和功能分析[J].水产学报,2014,38(9):1255-1262.
[13] 史东杰,梁拥军,孙砚胜,等.锦鲤“御三家”新品系的选育[J].水产科技情报,2014,41(3):152-154.
[14] Murakami M,Ohi M,Ishikawa S,et al.Adaptive expression of uncoupling protein 1 in the carp liver and kidney in response to changes in ambient temperature[J].Comparative Biochemistry and Physiology Part A:Molecular & Integrative Physiology,2015,185:142-149.
[15] 张春兰,赵文,徐立蒲,等.微生态制剂和着色剂对锦鲤体色的影响[J].大连海洋大学学报,2011,26(4):328-332.
[16] 史东杰,梁拥军,许金华,等.微生态制剂对锦鲤生长性能、肌肉组成、体色及非特异性免疫的影响[J].河南农业科学,2017,46(1):144-148.
[17] 崔培,姜志强,王雪,等.不同蛋白水平的虾青素饲料对锦鲤体色、生长及免疫的影响[J].上海海洋大学学报,2012,21(3):382-388.
[18] 何志刚,王冬武,邓时铭,等.不同饲料对锦鲤体色和着色成本的影响[J].中国饲料添加剂,2015(8):23-25.
[19] 何志刚,王冬武,曾国清,等.不同饲料对红白锦鲤生长、体色和血液生化指标的影响[J].广东农业科学,2015,42(20):107-111.
[20] 孙向军,罗琳,姜志强,等.饲料脂肪水平对锦鲤体色和几项免疫指标的影响[J].大连海洋大学学报,2011,26(5):397-401.
[21] 王巍,胡红霞,孙向军,等.锦鲤酪氨酸酶基因序列分析及其在不同锦鲤品系不同组织中的表达[J].水产学报,2012,36(11):1658-1666.
[22] 赵刚,龚全,刘亚,等.基于Illumina高通量测序的岩原鲤转录组分析[J].西南农业学报,2016,29(7):1743-1749.