尖裸鲤Oxygymnocypris stewarti隶属于鲤形目Cypriniformes、鲤科Cyprinidae、裂腹鱼亚科Schizothoracinae、尖裸鲤属Oxygymnocypris,亦称斯氏裸鲤或拉萨白鱼,是中国特有的高原冷水性鱼类,仅在海拔3600 m以上的西藏雅鲁藏布江中游及其支流处分布[1]。尖裸鲤主要以小型鱼类及水生昆虫为食,其肉质鲜美、营养价值高、开发前景广阔,是西藏地区主要经济鱼类。20世纪50年代后,由于捕捞过度、环境改变、生物入侵等因素,使尖裸鲤种群数量急剧减少,2006年尖裸鲤被西藏自治区列为一级重点保护水生野生动物,同时被列入《中国濒危动物红皮书》[2]。尖裸鲤种群资源若不能及时保护,其种群丰度将会快速变小,甚至有灭绝的可能,这对于栖息于雅鲁藏布江水生生物群落结构将造成严重的影响。目前,有关尖裸鲤的研究主要集中在生物学、种群动态学、胚胎与仔稚鱼发育、食性组成和肌肉营养成分分析等方面[3-5]。为了更有效地开展尖裸鲤的种质资源保护与人工繁殖,并为雅鲁藏布江的特有鱼类资源保护利用提供科学依据,开展尖裸鲤遗传学及分子生物学等相关基础研究十分必要。
GAPDH(3-磷酸甘油醛脱氢酶)是糖异生、糖酵解和光合作用碳固定循环过程中的关键酶;ACTB(β-肌动蛋白)是细胞骨架的主要构成成分肌动蛋白的一种,参与微丝的形成[6];RPL13(核糖体蛋白L13)、RPL19(核糖体蛋白L19)、RPL13a(核糖体蛋白L13a)属于核糖体蛋白,是构成细胞内核糖体的主要成分之一;SDHA(琥珀酸脱氢酶亚基A)属于黄素酶类,是线粒体内膜的结合酶,是连接氧化磷酸化与电子传递的枢纽之一[7];HPRT1(次黄嘌呤鸟嘌呤磷酸核糖转移酶1)为一种细胞质酶,是鸟嘌呤或次黄嘌呤磷酸核糖化的一种非必需酶,参与细胞内嘌呤核苷酸的补救合成途径[8]。以上蛋白的基因表达均比较稳定[6-8],是RT-PCR中最为常用的内参基因。
在理想的情况下,内参基因应在所研究的各种试验因素条件下均恒定表达,而研究发现大多数内参基因在不同物种间的表达不尽相同。一个合适的内参基因能够减小试验误差,使试验结果更准确。因此,要针对研究对象筛选出适合该物种的内参基因,并对目的基因进行校正。为此,选择合适的内参基因是开展尖裸鲤基因表达研究的前提条件。
本研究中,以尖裸鲤的血液、心脏、肝、等18个组织为材料,利用RT-PCR方法检测了7个候选内参基因在尖裸鲤不同组织中的表达量,并运用GeNorm、NormFinder和BestKeeper 3个软件对7个内参基因的稳定性表达进行分析[8-11],筛选出适于尖裸鲤RT-PCR分析的内参基因,以期为尖裸鲤的后续基因表达研究提供参考依据。
1 材料与方法
1.1 材料
试验用尖裸鲤于2018年4月采捕于西藏雅鲁藏布江,选择6龄以上的鱼10尾(6尾雄鱼,4尾雌鱼),体质量为300~400 g,体长为34~40 cm,饲养于中国水产科学研究院黑龙江水产研究所,利用鱼缸冷水机(中国广河隆公司)将养殖水温控制在最适范围内(9~12 ℃)[4]。
荧光定量PCR 仪为美国 ABI 公司的 Step One Plus Real-Time PCR System。
1.2 方法
1.2.1 样品的采集 试验时将试验鱼进行麻醉,采集血液、心脏、肝、脾、鳃、头肾、中肾、后肾、前肠、中肠、后肠、性腺、眼、垂体、脑、红肌、白肌和皮肤18个组织样品,立即置于干冰中冷冻后储藏于冰箱(-80 ℃)中,用于提取RNA。
1.2.2 总RNA提取及逆转录 用Trizol法进行各个组织总RNA的提取,经15 g/L琼脂糖凝胶电泳和紫外分光光度计(美国Waters公司)检测RNA的质量和浓度。取1.0 μg的RNA,逆转录为cDNA,并按照cDNA第一链合成试剂盒(德国Contech公司)说明书进行逆转录。反应体系(10 μL):5×gDNA Eraser Buffer 2.0 μL,gDNA Eraser 1.0 μL,Total RNA 1.0 μg,用ddH2O 补足至10 μL。进行反应条件1:42 ℃下反应2 min,反应结束后加入5×Prime Script Buffer 2 4.0 μL、Prime Script RT Enzyme Mix 1.0 μL、RT Primer Mix 1.0 μL,用ddH2O 补足至20 μL。进行反应条件2:37 ℃下反应15 min,85 ℃下反应5 s。进行实时荧光定量PCR(RT-PCR)反应前,将cDNA用无酶的水稀释10倍。
1.2.3 引物设计合成和荧光定量PCR 对GAPDH、HPRT1、RPL13、RPL19、RPL13a、SDHA和ACTB7个常用内参基因进行基因表达的稳定性分析[6-8](表1)。
表1 7个候选内参基因信息
Tab.1 Information on 7 candidate reference genes

基因gene引物序列(5'-3')primer sequence产物长度/bpamplicon length GenBankaccession No.GAPDHF:AGGGGCTCAGTATGTTGTGG R:AGGAGGCATTGCTGACAACT185AJ870982HPRT1F:TCATGGACAGGAACTGAACG R:CTCTTGAGGCGGATGAAGTC188 LHQP01004735RPL13F:CTGGCAAAGGCCTTGAAGT R:GCGTAACCGATCCACAGAGT180LHQP01000153RPL19F:ATTCGCAAGTTGGTGAAGGA R:TACGTCTCATCCAGCACAGC175LHQP01011721RPL13aF:ATTGTGGTGGTGAGGTGTGA R:TCAGACGCACAATCTTGAGG190LHQP01006903SDHAF:TCCAAACACAACCAGATCCA R:AGCCCATCCCAGTATTACCC200LHQP01067112ACTBF:GTTGCACACTTGATGGATGG R:GCACACTGCGCTATTTTTCA211LHQP01041979
采用SYBR GreenⅠ荧光染料法扩增目的基因片段,采用 LunaUniveral qPCR Master Mix (2X)试剂盒(New England Biolabs)扩增。反应体系(10 μL):Luna Univeral qPCR Master Mix 5 μL,cDNA 1.0 μL,上、下游引物(10 μmol/L)各 0.5 μL,用ddH2O 补足至10 μL。反应条件为:95 ℃下预变性30 s;95 ℃下变性 15 s,61 ℃下退火30 s,72 ℃下延伸40 s,共进行40个循环。每个样品进行3个生物学重复操作。
1.3 数据处理
根据RT-PCR得到的Ct值(Cycle threshold)[10](3个生物学重复的平均值),采用GeNorm、NormFinder和BestKeeper软件对7个内参基因的稳定性进行统计学分析[12-15]。Ct值与基因的表达量成反比,即Ct值越小,表达量越大,Ct值越大,则表达量越小[9]。根据Ct值计算7个候选内参基因在尖裸鲤不同组织中的相对表达量(Q),公式[13]为
Q=2-ΔCt,ΔCt=Ct样品-Ct最小。
将Q值分别输入GeNorm软件和NormFinder 软件,分析后得出各候选内参基因在尖裸鲤不同组织的平均表达稳定值(M)[11];直接将 Ct值输入BestKeeper软件,分析后得出各候选内参基因在不同组织中表达变异系数(CV)和标准差(S.D.)[15]。
2 结果与分析
2.1 样品RNA检测
从图1可见,28S、18S条带清晰,28S条带的亮度约为18S的2.0倍,说明RNA无降解,完整性较好。用紫外分光光度计检测RNA浓度和纯度,各个组织RNA的A260 nm/A280 nm≈1.9~2.0,A260 nm/A230 nm≈2.0,说明纯度较好,浓度在1000~2000 ng/μL,RNA质量符合标准,可用于后续操作。
注:M为DL 2000 DNA Marker;1为肝;2为脾;3为头肾;4为后肾; 5为鳃; 6为前肠;7为中肾; 8为皮肤;9为中肠;10为血液; 11为脑 ;12为红肌;13为后肠;14为心脏;15为眼;16为垂体;17为白肌;18为性腺
Note: M,DL 2000 DNA Marker;1,liver;2,spleen;3,head kidney;4,metanephros;5,gill;6,foregut;7,midkidney;8,skin;9,midgut;10,blood;11,brain;12,red muscle;13,hindgut;14,heart;15,eye;16,pituitary;17,white muscle;18,gonad
图1 尖裸鲤18种组织的总RNA电泳图谱
Fig.1 Electrophoretogram of total RNA from samples of 18 tissues in Stewart’s naked high-Asian-carpOxygymnocypris stewarti
2.2 RT-PCR反应产物的特异性检测
经过制作RPL13基因的溶解曲线,得到7个内参基因的溶解曲线均呈现单一峰,且阴性对照无荧光信号,说明产物特异性较好。
2.3 Ct值比较
从图2可见:RPL13a的平均Ct值最大,为22.33;其次是HPRT1,平均Ct值为21.91;RPL19和SDHA基因的平均Ct值相近,分别为20.20和20.55;GAPDH和RPL13a基因的平均Ct值相近,分别为18.76和18.79;ACTB在各个组织中的平均Ct值最小,为16.64,其Ct值范围为12.68~20.35。这表明,7个内参基因在尖裸鲤不同组织中的表达量存在差异。
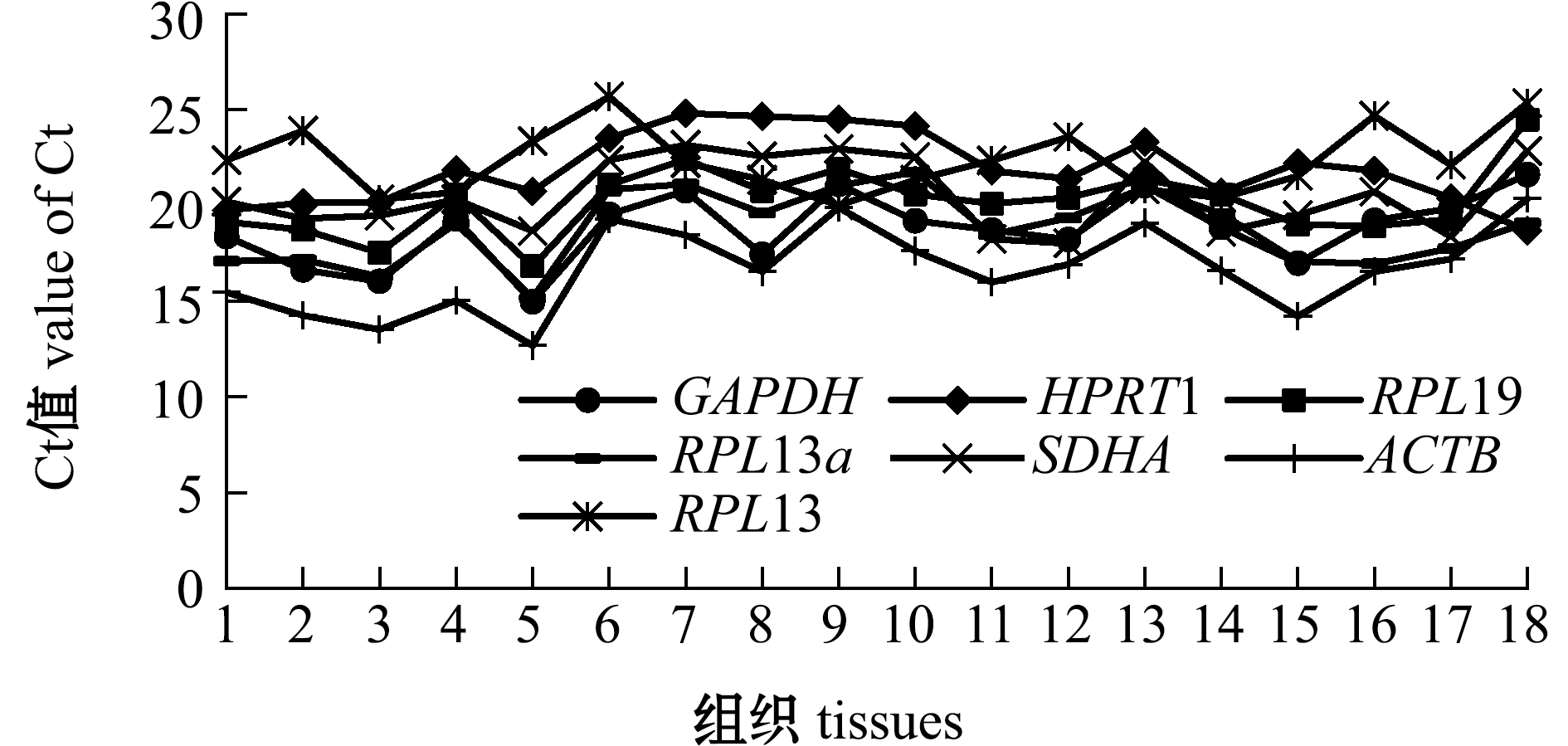
注: 1为肝;2为脾;3为头肾;4为后肾; 5为鳃; 6为前肠;7为中肾; 8为皮肤;9为中肠;10为血液; 11为脑 ;12为红肌;13为后肠;14为心脏;15为眼;16为垂体;17为白肌;18为性腺
Note: 1,liver;2,spleen;3,head kidney;4,metanephros;5,gill;6,foregut;7,midkidney;8,skin;9,midgut;10,blood;11,brain;12,red muscle;13,hindgut; 14,heart;15,eye;16,pituitary;17,white muscle;18,gonad
图2 7个候选内参基因在尖裸鲤不同组织中的Ct值
Fig.2 Ct values of seven candidate reference genes in samples of different tissues in Stewart’s naked high-Asian-carpOxygymnocypris stewarti
2.4 GeNorm软件分析结果
采用GeNorm软件计算候选内参基因在尖裸鲤不同组织中的平均表达稳定值M(图3)和配对差异值V(图4),7个候选内参基因的M值依次为RPL13=RPL13a<RPL19<GAPDH<ACTB<SDHA<HPRT1,M值越小,基因表达越稳定。该软件以0.15为默认取舍值,当Vn/V(n+1)<0.15时,最适内参基因数为n,由图4可知,V2/V3<0.15,说明最适内参基因数目为2个。综合M和V值,RPL13和RPL13a是表达最稳定的内参基因。
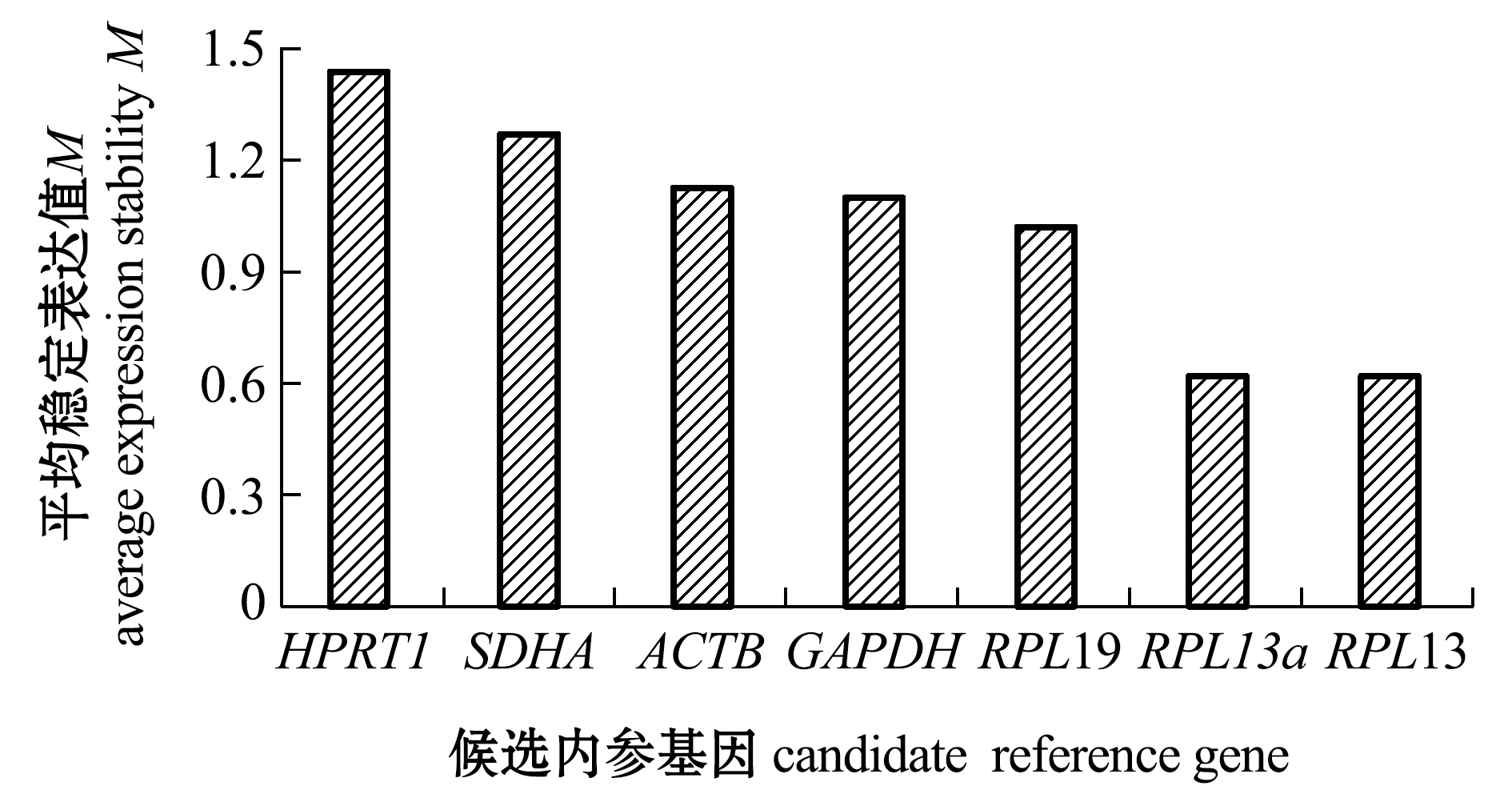
图3 GeNorm软件计算的7个候选内参基因的平均表达稳定值
Fig.3 Average expressed stable values of 7 candidate reference genes by GeNorm
2.5 NormFinder软件分析结果
采用NormFinder软件计算候选内参基因在尖裸鲤不同组织的平均表达稳定值M(图5),7个候选内参基因的M值依次为RPL13<RPL13a<RPL19<GAPDH<ACTB<SDHA<HPRT1,其中RPL13的M值最小(0.30),是最稳定的内参基因。
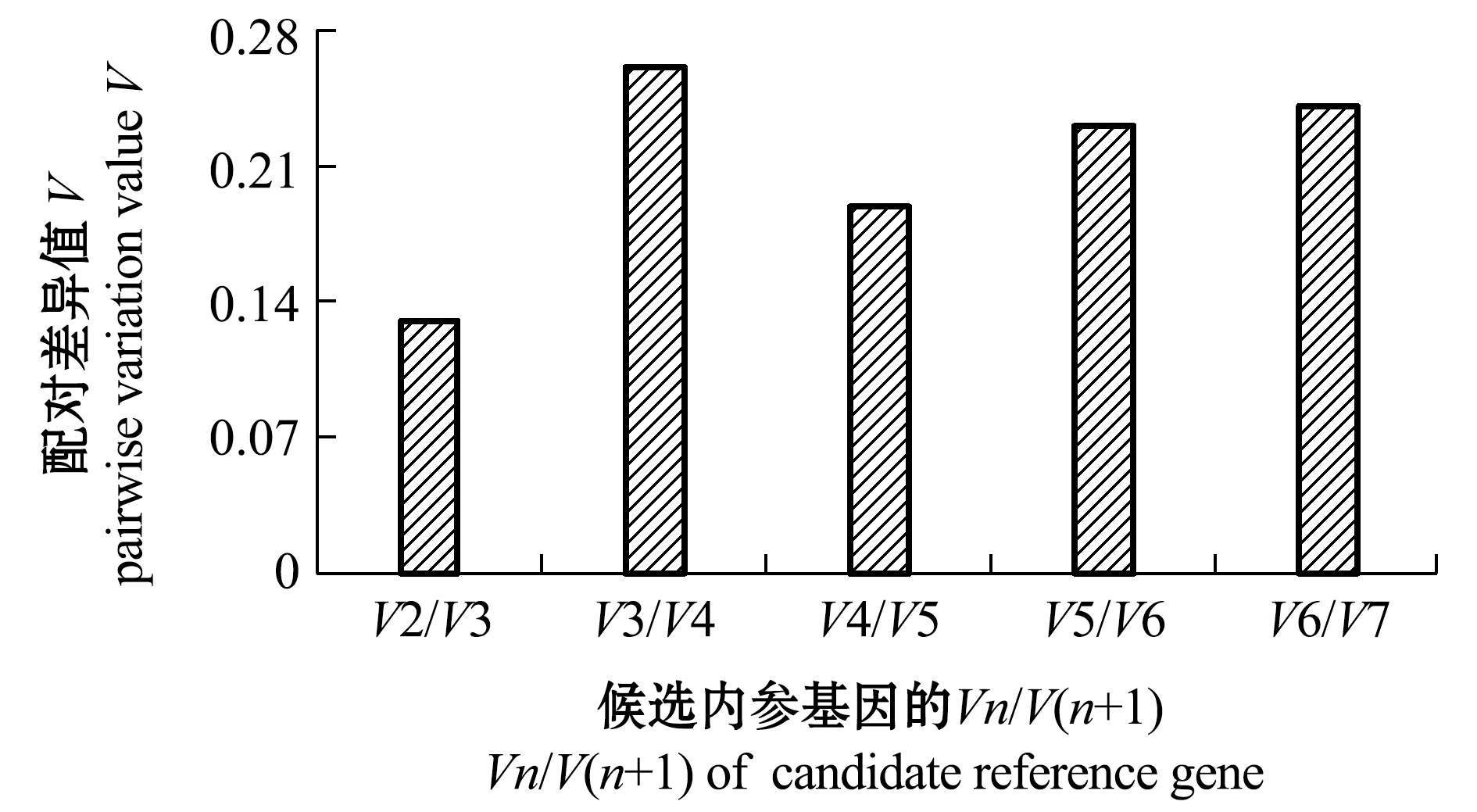
图4 GeNorm软件计算的7个候选内参基因的配对差异值
Fig.4 Pairwise variation values of 7 candidate reference genes by GeNorm
2.6 BestKeeper软件分析结果
从表2可见:7个候选内参基因的S.D.值依次为RPL13<RPL19<GAPDH<HPRT1<RPL13a<SDHA<ACTB,其中RPL13的S.D.值最小(0.84);各基因的CV值依次为RPL13<RPL19<HPRT1<GAPDH<SDHA<RPL13a<ACTB,其中RPL13的CV值最小(4.54%)。而S.D.的默认值为1,内参基因的S.D.小于1则稳定性好,反之则稳定性差。对S.D.和CV值综合分析表明,RPL13是最适内参基因。
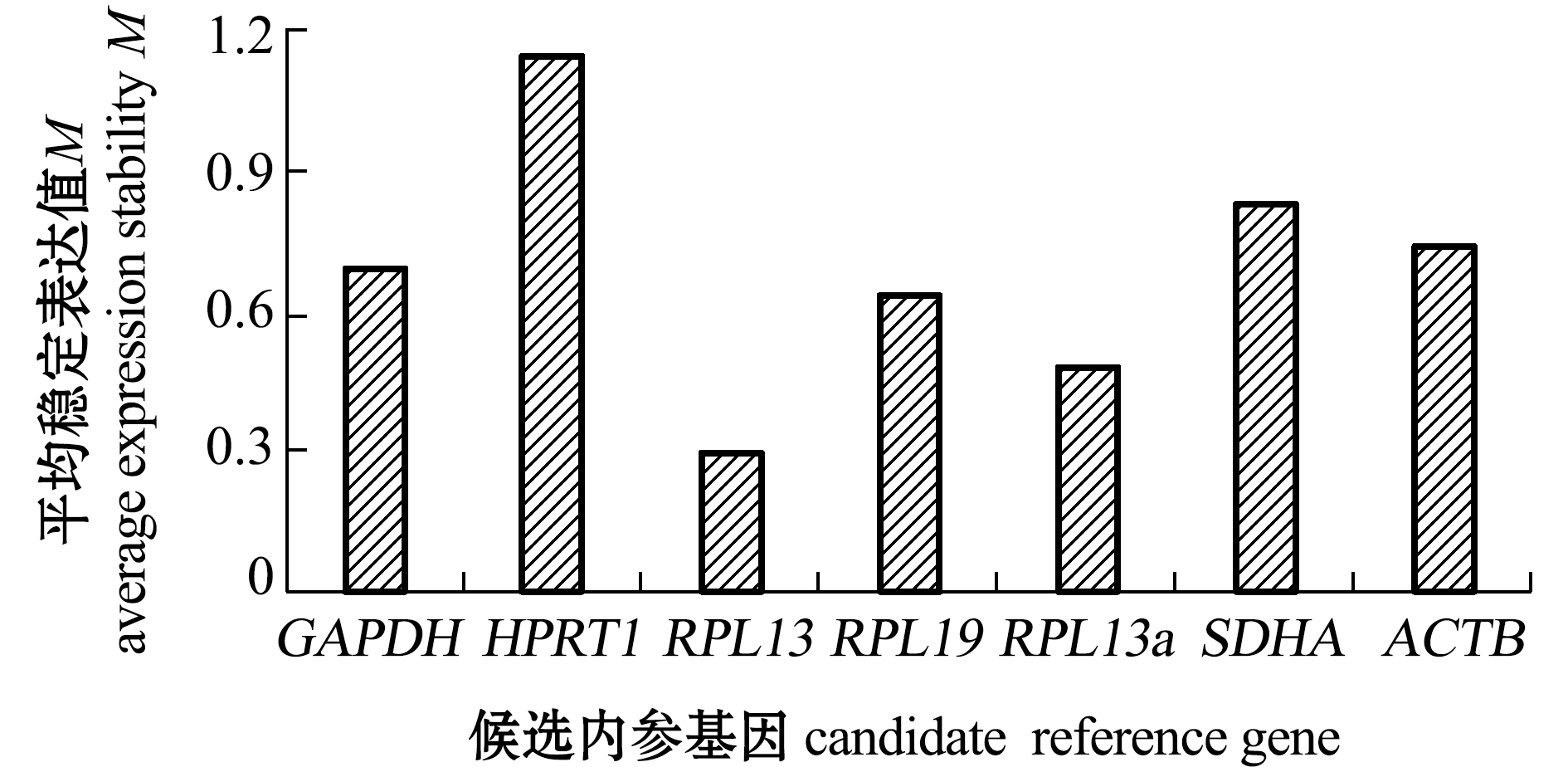
图5 NormFinder软件计算的7个候选内参基因的平均表达稳定值
Fig.5 Average expressed stable values of 7 candidate reference genes by NormFinder
表2 BestKeeper软件对7个候选内参基因Ct值的分析结果
Tab.2 Analysis of 7 candidate reference genes by BestKeeper
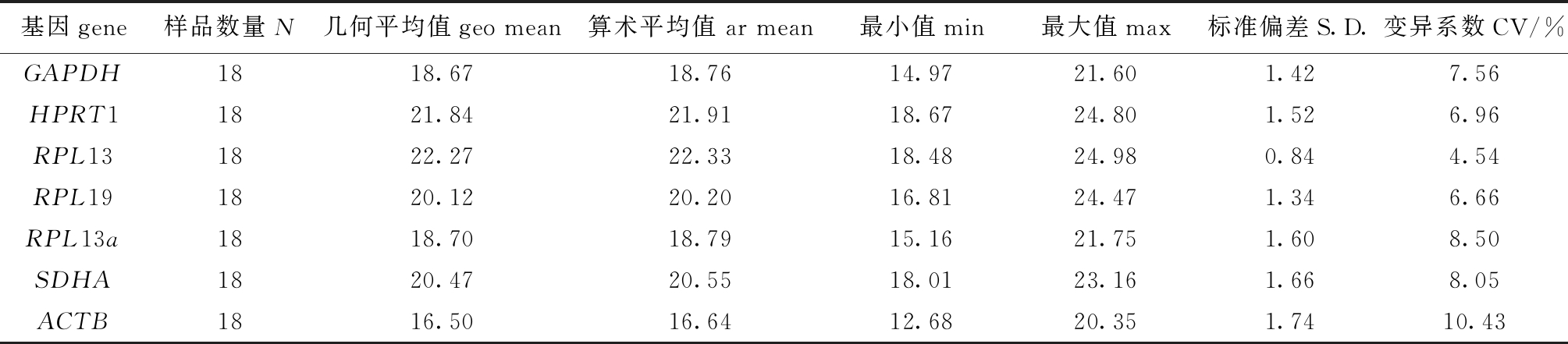
基因gene样品数量N几何平均值geo mean 算术平均值 ar mean 最小值min 最大值max 标准偏差S.D.变异系数CV/%GAPDH1818.6718.7614.9721.601.427.56HPRT11821.8421.9118.6724.801.526.96RPL131822.2722.3318.4824.980.844.54RPL191820.1220.2016.8124.471.346.66RPL13a1818.7018.7915.1621.751.608.50SDHA1820.4720.5518.0123.161.668.05ACTB1816.5016.6412.6820.351.7410.43
3 讨论
3.1 实时荧光定量PCR中内参基因的重要性
在实时荧光定量PCR(RT-PCR)技术中,用内参基因作为参照来衡量目的基因的表达,而且内参基因和目的基因应该具有相似的转录水平[15],但由于基因表达的复杂性及试验操作过程中造成的误差,会导致荧光定量PCR结果与基因的真实表达量存在误差[16-17],故内参基因的选择对RT-PCR的结果可信度具有重要影响。
3.2 GeNorm、NormFinder和BestKeeper软件对内参基因的选择比较
本研究中利用实时荧光定量PCR对GAPDH、HPRT1、RPL13、RPL19、RPL13a、SDHA和ACTB7个候选内参基因在不同组织内的表达情况进行分析,并经GeNorm、NormFinder和BestKeeper软件对这7个候选内参基因进行了稳定性分析。
GeNorm、NormFinder软件都是通过计算平均表达稳定值M来评价内参基因的稳定性。判定准则为M值越小,内参基因稳定性就越好,反之,M值越大则稳定性越差。GeNorm软件还可以计算出引入1个新的内参基因后的配对变异V值,并按照Vn/V(n+1)值来确定所需最适内参基因的数目[8]。n值为最佳内参基因数目,默认的V值为0.15,分析结果显示,Vn/V(n+1)值<0.15,说明最适内参基因的数量为n个;而如果Vn/V(n+1)值>0.15,说明最适内参基因的数量是n+1个[8]。在一些对表达定量精确度要求较高的试验中,通常一个内参基因不能达到较好的校正作用,常需要同时使用两个或两个以上稳定表达的内参基因来对目的基因表达量进行校正[17-18]。本试验中,根据GeNorm分析结果,确认最适内参基因数为2个,分别是RPL13和RPL13a。再根据NormFinder程序的分析结果,确认最适内参基因是RPL13。
BestKeeper软件是由标准差值和变异系数值的大小体现内参基因的表达稳定性,此软件把标准差的默认值定为1,如果内参基因的标准差值大于1,说明稳定性较差,如果内参基因的标准差值小于1,说明稳定性较好。标准差值和变异系数值越小,说明内参基因表达越稳定[19]。本研究中显示,7个候选内参基因中,RPL13的标准差和变异系数最小,说明该基因表达稳定,且分析结果同GeNorm和NormFinder软件分析结果一致。用3个软件分析得出的结果基本相同,在某种程度上验证了试验筛选出的内参基因相对可靠。
RPL13作为核糖体大亚基蛋白,在真核生物中高度保守,其不仅参与蛋白质合成,还参与RNA的加工,DNA修复、复制、转录、翻译调控[20]。与其他内参基因相比,RPL13在尖裸鲤各个组织中具有相对更稳定的表达,适合RT-PCR的校正和标准化,因此,RPL13是尖裸鲤的最适内参基因。
3个软件各有其特点:GeNorm软件能够筛选出多个样品的多个内参基因,并确定合适的内参基因及最适内参基因数目;NormFinder软件能够比较内参基因的表达差异及样品组间的变异程度,最终选出1个最适内参基因;BestKeeper软件最多能够比较100个样品中10个内参基因的表达差异[8]。建议做内参基因选择时,要综合3个软件分析的结果,最终确定最适内参基因。
3.3 不同物种间内参基因的表达差异
在RT-PCR技术中,内参基因的选择、RNA的提取、逆转录和PCR步骤中的试验误差等,均会影响试验结果的准确性,而内参基因是对目的基因进行定量表达分析的关键因素,因此,内参基因的筛选十分重要。研究发现,不同物种间内参基因的表达也存在差异,如在金鲳鱼Trachinotus ovatus中稳定表达的内参基因是B2M和18S[21],在大西洋鳕Gadus morhua中ARP和Ubi则是表达最稳定的内参基因[22]。同一物种在不同试验条件下内参基因的表达也存在差异[23],如大菱鲆受到细菌感染前,RPSD在各个组织中稳定表达,但细菌感染后,发现ACTB是最稳定表达的基因[24]。在条石鲷Oplegnathus fasciatus中MRPt、ACTB可作为RT-PCR分析的内参基因,然而在细菌或病毒感染之后,未发现合适的内参基因[25]。牙鲆在受到细菌感染前,ACTB、UBCE是稳定表达的内参基因,但受到细菌感染后,发现TUBA在脾脏、心脏、肌肉和鳃中稳定表达,适用于这4个组织[26]。
综上所述,确定RPL13为最适于尖裸鲤RT-PCR分析的内参基因,使用最适的内参基因能够提高尖裸鲤RT-PCR结果的准确度。并为其基因表达分析提供数据资料。
[1] 赵亚辉,张洁,张春光.青藏高原鱼类的多样性[J].生物学通报,2008,43(7):8-10.
[2] 乐佩琪,陈宜瑜.中国濒危动物红皮书[M].北京:科学出版社,1998:158-159.
[3] 霍斌.尖裸鲤个体生物学和种群动态学研究[D].武汉:华中农业大学,2014.
[4] 许静,谢从新,邵俭,等.雅鲁藏布江尖裸鲤胚胎和仔稚鱼发育研究[J].水生态学杂志,2011,32(2):86-95.
[5] 洛桑,张强英,丹增达瓦,等.拉萨河尖裸鲤(Oxygymnocypris stewartii)肌肉营养组成与分析评价[J].西藏大学学报:自然科学版,2014,29(1):8-12,53.
[6] Mori R,Wang Qingcai,Danenberg K D,et al.Both β-actin and GAPDH are useful reference genes for normalization of quantitative RT-PCR in human FFPE tissue samples of prostate cancer[J].Prostate,2008,68(14):1555-1560.
[7] Casadei R,Pelleri M C,Vitale L,et al.Identification of housekeeping genes suitable for gene expression analysis in the zebrafish[J].Gene Expr Patterns,2011,11(3-4):271-276.
[8] Hüttner E,Speit G,Lambert B,et al.European HPRT workshop in collaboration with GUM Gatersleben-Quedlinburg,4-7 May,1995[J].Mutat Res,1996,359(1):71-76.
[9] Pfaffl M W,Tichopad A,Prgomet C,et al.Determination of stable housekeeping genes,differentially regulated target genes and sample integrity:BestKeeper-Excel-based tool using pair-wise correlations[J].Biotechnol Lett,2004,26(6):509-515.
[10] 吴建阳,何冰,杜玉洁,等.利用GeNorm、NormFinder和BestKeeper软件进行内参基因稳定性分析的方法[J].现代农业科技,2017(5):278-281.
[11] Silveira É D,Alves-Ferreira M,Guimarães L A,et al.Selection of reference genes for quantitative real-time PCR expression studies in the apomictic and sexual grassBrachiaria brizantha[J].BMC Plant Biol,2009,9:84.
[12] Hellemans J,Mortier G,De Paepe A,et al.qBase relative quantification framework and software for management and automated analysis of real-time quantitative PCR data[J].Genome Biol,2007,8(2):R19.
[13] Vandesompele J,De Preter K,Pattyn F,et al.Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J].Genome Biol,2002,3(7):research0034.
[14] Andersen C L,Jensen J L,Ørntoft T F.Normalization of real-time quantitative reverse transcription-PCR data:a model-based variance estimation approach to identify genes suited for normalization,applied to bladder and colon cancer data sets[J].Cancer Res,2004,64(15):5245-5250.
[15] Jung M,Ramankulov A,Roigas J,et al.In search of suitable reference genes for gene expression studies of human renal cell carcinoma by real-time PCR[J].BMC Mol Biol,2007,8:47.
[16] Imbeaud S,Graudens E,Boulanger V,et al.Towards standardization of RNA quality assessment using user-independent classifiers of microcapillary electrophoresis traces[J].Nucleic Acids Res,2005,33(6):e56.
[17] Fleige S,Pfaffl M W.RNA integrity and the effect on the real-time qRT-PCR performance[J].Mol Aspects Med,2006,27(2-3):126-139.
[18] Johnson G,Nour A A,Nolan T,et al.Minimum information necessary for quantitative real-time PCR experiments[M]//Quantitative Real-Time PCR.New York:Humana Press,2014:5-17.
[19] Gantasala N P,Papolu P K,Thakur P K,et al.Selection and validation of reference genes for quantitative gene expression studies by real-time PCR in eggplant (Solanum melongenaL.)[J].BMC Res Notes,2013,6:312.
[20] Wool I G.Extraribosomal functions of ribosomal proteins[J].Trends Biochem Sci,1996,21(5):164-165.
[21] Chen X J,Zhang X Q,Huang S,et al.Selection of reference genes for quantitative real-time RT-PCR on gene expression in golden pompano (Trachinotus ovatus)[J].Pol J Vet Sci,2017,20(3):583-594.
[22] Olsvik P A,Softeland L,Lie K K.Correction:selection of reference genes for qRT-PCR examination of wild populations of Atlantic codGadus morhua[J].BMC Res Notes,2011,4:456.
[23] Zhang Qilin,Zhu Qianhua,Liao Xin,et al.Selection of reliable reference genes for normalization of quantitative RT-PCR from different developmental stages and tissues in amphioxus[J].Sci Rep,2016,6:37549.
[24] Dang Wei,Sun Li.Determination of internal controls for quantitative real time RT-PCR analysis of the effect ofEdwardsiella tardainfection on gene expression in turbot (Scophthalmus maximus)[J].Fish and Shellfish Immunol,2011,30(2):720-728.
[25] Zhang Baocun,Sun Li,Xiao Zhizhong,et al.Quantitative real time RT-PCR study of pathogen-induced gene expression in rock bream (Oplegnathus fasciatus):internal controls for data normalization[J].Mar Genom,2014,15:75-84.
[26] Zheng Wenjiang,Sun Li.Evaluation of housekeeping genes as references for quantitative real time RT-PCR analysis of gene expression in Japanese flounder (Paralichthys olivaceus)[J].Fish and Shellfish Immunol,2011,30(2):638-645.