口虾蛄Oratosquilla oratoria隶属于节肢动物门Arthropoda、甲壳动物亚门Crustacea、软甲纲Malacostraca、口足目Stomatopoda、虾蛄科Squilidae、口虾蛄属Oratosquilla,广泛分布于俄罗斯东南部、日本、中国、越南、澳大利亚东部近海[1]。在中国近海区和沿岸,口虾蛄一般在50~60 m水深均有分布[2]。随着中国近海渔业作业结构的调整及渔具捕捞性能的改善,口虾蛄已成为近海渔业主要捕捞对象之一[3-4],目前已开展对其生物量的时空变化及渔业生物学特征研究[5-8]。有关口虾蛄种群遗传多样性及遗传结构研究已有大量报道,主要采用线粒体DNA(mtDNA)序列分析方法。研究表明,口虾蛄黄渤海群体与南海群体存在显著的遗传分化,而黄渤海群体与东海群体是否存在显著的遗传分化因采用不同的分子标记而异[9-13]。
杜欣蔚[9]采用16S rRNA和细胞色素氧化酶Ⅰ(COⅠ)分子标记均检测到口虾蛄黄海群体与东海群体存在显著的遗传分化,Zhang等[10]采用NADH脱氢酶亚基5(ND5)分子标记检测出中国沿海口虾蛄分为2个分支,一支包括渤海群体、黄海群体和东海群体,另一支包括南海群体。目前,细胞色素b(Cytb)分子标记尚未应用于口虾蛄群体遗传学研究中。
16S rRNA和ND5分子标记由于进化速率较慢,常用于种以上水平遗传变异的检测,而COⅠ和Cytb分子标记进化速率较快,常用于种群水平遗传变异的检测[14]。由于Cytb分子标记能够检测种群历史动态,判定种群是否发生群体扩张,因而被广泛应用于种群遗传结构模式和分子系统地理学格局研究[15-16]中。本研究中,采用Cytb分子标记检测了口虾蛄黄海群体、东海群体和南海群体间的遗传变异水平,并探讨了其种群遗传结构模式及系统发育地理格局模式成因,以期为口虾蛄遗传多样性的保护及资源的可持续利用提供科学依据。
1 材料与方法
1.1 材料
试验样本于2014年11月—2016年3月采自江苏省、浙江省、福建省和广东省沿海,其中,连云港群体(LYG,23尾,34°52.15′N、119°15.75′E)和珠江口群体(ZJK,17尾,22°18.90′N、113°40.22′E)分别属于黄海群体和南海群体,南韭山群体(NJS,21尾,29°25.66′N、122°16.65′E)、南麂岛群体(NJD,22尾,28°28.28′N、121°8.92′E)和福州群体(FZ,13尾,26°8.48′N、120°1.03′E)均属于东海群体。
1.2 方法
1.2.1 基因组DNA提取 试验中剪取各样本肌肉组织固定于95%乙醇中,保存于冰箱(-20 ℃)中备用。采用EasyPureGenomic DNA Kit(北京全式金生物技术有限公司)提取基因组DNA,并于-20 ℃下保存备用。
1.2.2 PCR扩增与测序 PCR引物为自行设计,参照NCBI(National Center for Biotechnology Information)上传的口虾蛄mtDNA全序列。引物序列:
上游引物F:GTAGCCCACATTTGCCGAGA;
下游引物R:GACGAGCGCCAATTCATGTC。
PCR反应体系(50 μL):10 × PCR Buffer 5 μL,dNTP(各2.5 mmol/L)4 μL,TaqDNA polymerase(5 U/μL)0.4 μL,上、下游引物(10 μmol/L)各1.2 μL,DNA模板40 ng,用ddH2O补足至50 μL。PCR扩增反应退火温度为55 ℃,以ddH2O代替模板DNA做阴性对照。PCR产物用EasyPure PCR Purification Kit纯化后,送至生工生物工程(上海)股份有限公司进行双向测序。
1.3 数据分析
通过BioEdit软件对测序结果进行人工校对,处理后序列的单倍型数、单倍型多样性指数(h)、核苷酸多样性指数(π)由DnaSP 4.10软件计算。构建单倍型NJ(Neighbor-Joining)系统树和最小跨度树,反映不同单倍型间的连接关系及不同单倍型类群间的谱系结构。采用AMOVA(Analysis of Molecular Variance)分析检测口虾蛄种群遗传结构、群体间遗传分化系数(F-statistics,Fst),采用Tajima’sD与Fu’sFs中性检验和核苷酸不配对分布检测群体历史动态。以上数据分析涉及的参数及其运算方法参照刘连为等[17]的研究。
2 结果与分析
2.1 单倍型多样性与核苷酸多样性
测序结果经过校对、排序后获得96条长度为829 bp的Cytb基因片段,共包含168个变异位点。从表1可见:所有分析序列共定义72个单倍型,其中,单倍型H7为珠江口群体与东海所有地理群体共有,单倍型H10为东海福州群体和南韭山群体共有,未发现连云港群体、东海群体与珠江口群体共有单倍型;各群体特有单倍型除H34、H44、H48、H51、H57、H65、H67外出现的频率均较低,仅为群体中单个个体具有。
所有群体总的单倍型多样性指数与核苷酸多样性指数分别为0.976±0.010、0.039 25±0.019 13;福州群体的单倍型多样性指数最高,为0.987±0.035,南麂岛群体最低,为0.931±0.046;珠江口群体的核苷酸多样性指数最高,为0.064 84±0.033 02,连云港群体最低,为0.003 59±0.00217(表2)。
2.2 种群遗传结构
AMOVA结果显示,遗传变异主要来自于组群间,且遗传分化极显著(变异系数为73.88%,P<0.01),东海群体内、连云港群体内及珠江口群体内遗传分化均不显著(P>0.05)(表3)。
两两群体间Fst分析表明,连云港群体、珠江口群体与其他地理群体间遗传分化均显著(P<0.05),东海各地理群体间遗传分化不显著(表4)。
单倍型NJ系统树和最小跨度树显示(图1、图2),存在明显的系统发育谱系结构,即谱系A、B、C存在于口虾蛄群体中。其中,谱系A仅包括珠江口群体单倍型类群;谱系B仅包括连云港群体单倍型类群;谱系C是东海群体的主体单倍型类群,也包括珠江口群体单倍型类群。珠江口群体在谱系A内的分布频率为29.4%,连云港群体在谱系B内的分布频率为100%,东海群体和珠江口群体在谱系C内的分布频率分别为100%和70.6%(表2)。口虾蛄3个谱系单倍型类群在空间上的分布频率存在显著差异,且遗传分化极显著(Fst=0.695~0.842,P<0.01)。
表1 口虾蛄Cytb序列单倍型在群体中的分布
Tab.1 Haplotype distribution of Cytbsequences in various populations of mantis shrimpOratosquilla oratoria

单倍型haplotype单倍型分布 haplotype distribution连云港LYG南韭山NJS南麂岛NJD福州FZ珠江口ZJK合计total单倍型haplotype单倍型分布haplotype distribution连云港LYG南韭山NJS南麂岛NJD福州FZ珠江口ZJK合计totalH111H3811H211H3911H311H4011H411H4111H511H4211H611H4311H7462214H4422H811H4511H911H4611H10112H4711H1111H4855H1211H4911H1311H5011H1411H5122H1511H5211H1611H5311H1711H5411H1811H5511H1911H5611H2011H5722H2111H5811H2211H5911H2311H6011H2411H6111H2511H6211H2611H6311H2711H6411H2811H6522H2911H6611H3011H6722H3111H6811H3211H6911H3311H7011H3422H7111H3511H7211H3611H3711合计total232122131796
表2 口虾蛄群体遗传多样性及其在系统发育谱系中的百分比
Tab.2 Genetic diversity and its percentage in the phylogenetic lineages in various populations of mantis shrimpOratosquilla oratoria
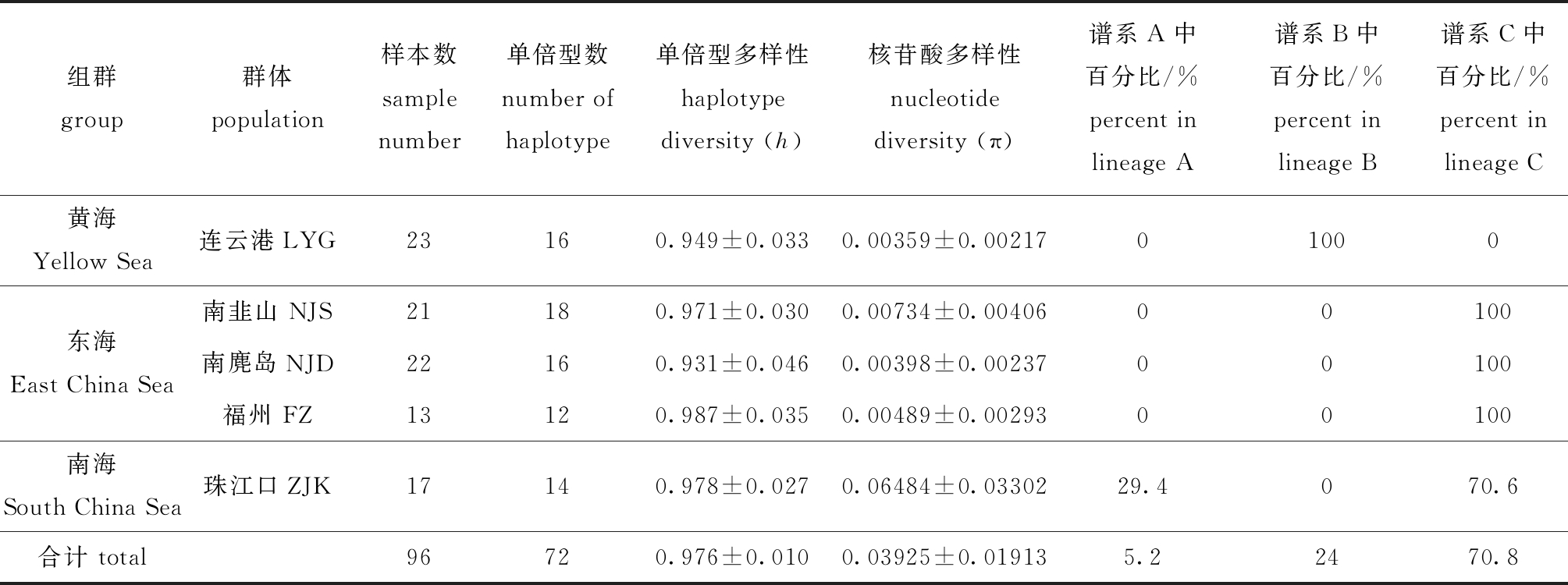
组群group群体population样本数sample number单倍型数number of haplotype单倍型多样性haplotype diversity (h)核苷酸多样性nucleotide diversity (π)谱系A中百分比/%percent in lineage A谱系B中百分比/%percent in lineage B谱系C中百分比/%percent in lineage C黄海 Yellow Sea连云港LYG23160.949±0.0330.00359±0.0021701000南韭山 NJS21180.971±0.0300.00734±0.0040600100东海 East China Sea南麂岛NJD22160.931±0.0460.00398±0.0023700100福州 FZ13120.987±0.0350.00489±0.0029300100南海 South China Sea珠江口ZJK17140.978±0.0270.06484±0.0330229.4070.6合计 total96720.976±0.0100.03925±0.019135.22470.8
表3 基于Cytb序列的口虾蛄种群遗传结构
Tab.3 Population genetic structure of mantis shrimpOratosquilla oratoriabased on Cytbsequences
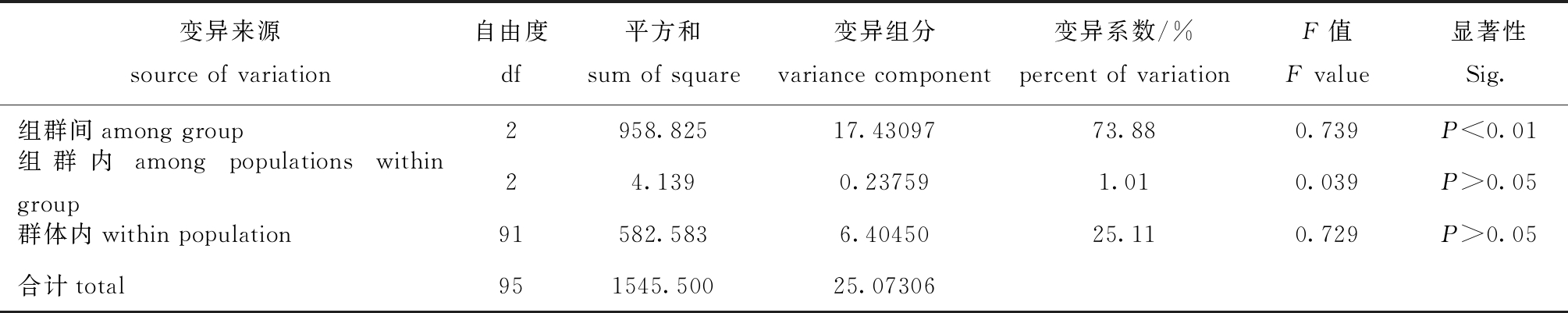
变异来源source of variation自由度df平方和sum of square变异组分variance component变异系数/%percent of variationF值F value显著性Sig.组群间among group2958.82517.4309773.880.739P<0.01组群内among populations within group24.1390.237591.010.039P>0.05群体内within population91582.5836.4045025.110.729P>0.05合计total951545.50025.07306
表4 基于Cytb序列的口虾蛄群体间遗传分化系数(Fst)
Tab.4 F-statistics (Fst) among populations of mantis shrimpOratosquilla oratoriabased on Cytbsequences
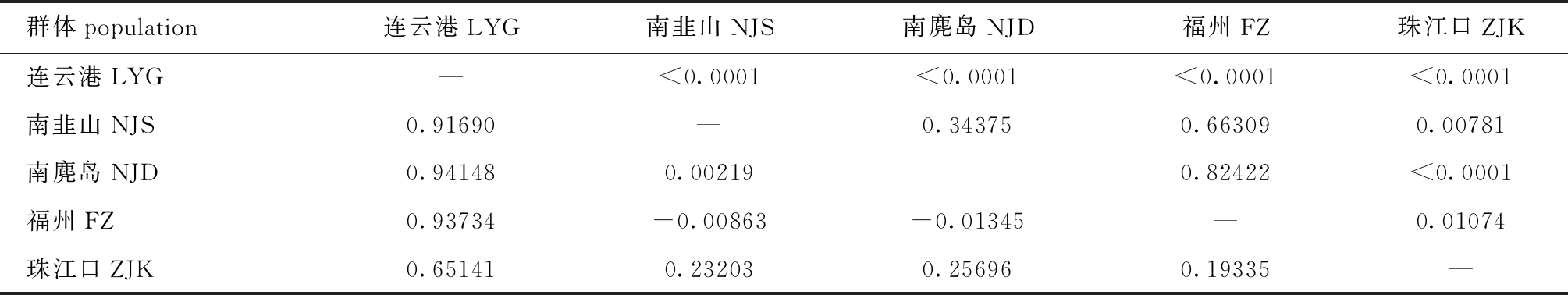
群体population连云港LYG南韭山NJS南麂岛NJD福州FZ珠江口ZJK连云港LYG—<0.0001<0.0001<0.0001<0.0001南韭山NJS0.91690—0.343750.663090.00781南麂岛NJD0.941480.00219—0.82422<0.0001福州FZ0.93734-0.00863-0.01345—0.01074珠江口ZJK0.651410.232030.256960.19335—
注:对角线以下为Fst值,对角线以上为P值
Note: values ofFst(below diagonal) andP(above diagonal) based on Cytbsequences from mantis shrimpOratosquilla oratoriapopulations
2.3 群体历史动态
判断群体是否经历过近期历史扩张事件须满足以下2个条件:
(1) Tajima’sD中性检验D值与Fu’sFs中性检验Fs值均为显著性负值;
(2) 核苷酸不配对分布图呈单峰类型,且观测值未明显偏离模拟值。
本研究结果显示(表5、图3),仅谱系C满足以上条件,口虾蛄谱系C发生群体扩张的时间大约在11.0万年前(核苷酸分歧速率采用2%/百万年)。
表5 口虾蛄Cytb序列的中性检验
Tab.5 Neutral test for natural selection on Cytbsequences of mantis shrimpOratosquilla oratoria
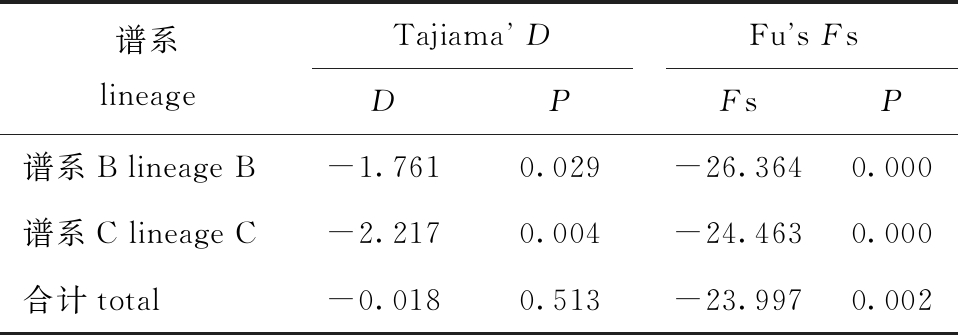
谱系lineageTajiama DFus FsDPFsP谱系B lineage B-1.7610.029-26.3640.000谱系C lineage C-2.2170.004-24.4630.000合计total-0.0180.513-23.9970.002
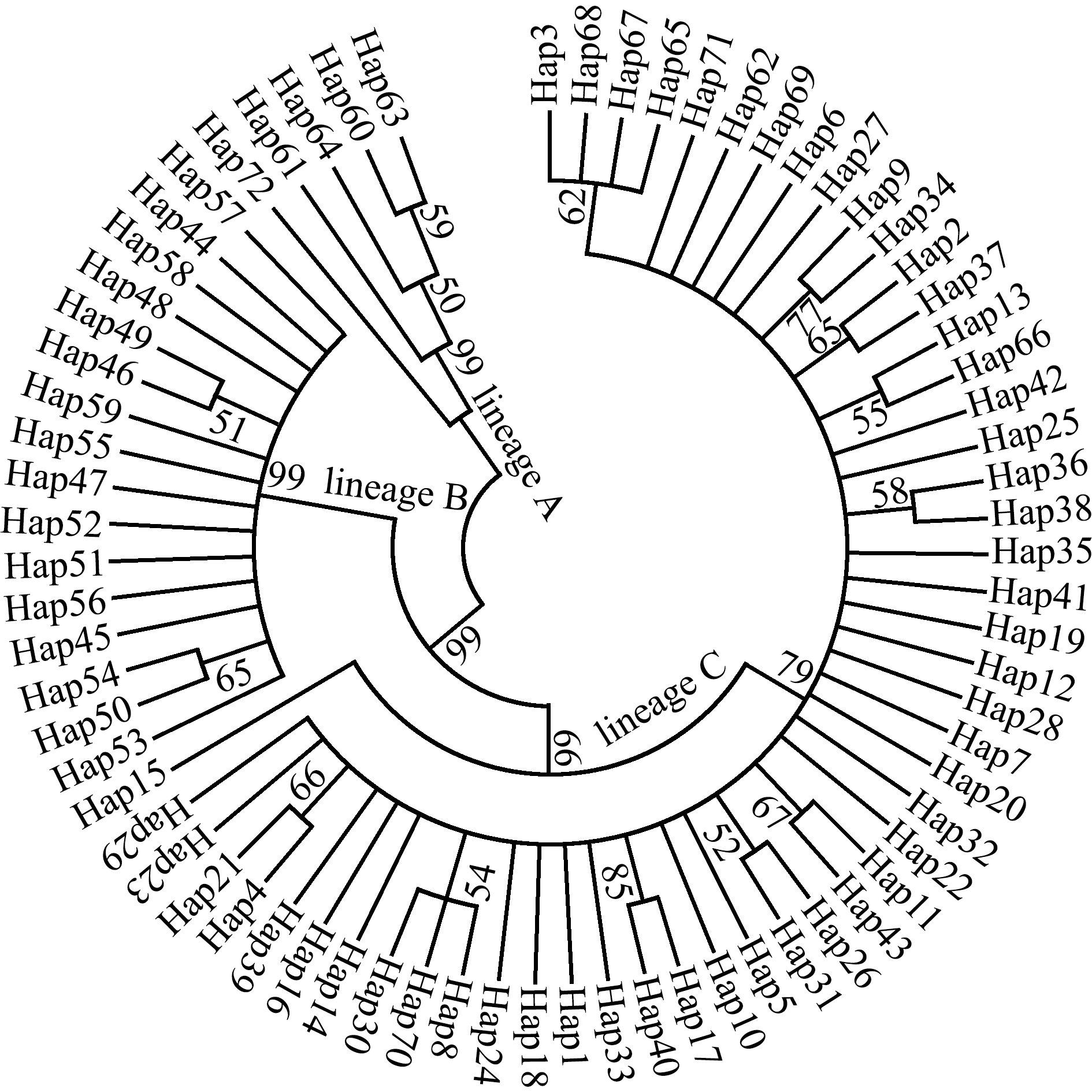
图1 基于Cytb序列单倍型构建的口虾蛄分子系统树
Fig.1 Molecular phylogenetic tree of mantis shrimpOratosquilla oratoriabased on haplotypes of Cytbsequences
3 讨论
3.1 口虾蛄种群遗传结构
Cytb分子标记具有较高的进化速率,适用于种群内及种群间水平遗传变异检测[18]。本研究中,基于该分子标记检测到口虾蛄存在显著的遗传结构,其中,黄海群体与东海群体间遗传分化显著,这与基于16S rRNA和COⅠ分子标记得出的结论一致,而与ND5分子标记得出的结论相反[9-10];南海群体与东海群体间遗传分化显著,这与基于ND5分子标记得出的结论一致。ND5分子标记也应用于其他甲壳动物群体遗传学分析[19],但在种内检测到的遗传变异水平较低,基于该分子标记检测出口虾蛄北部群体(黄海群体、渤海群体、东海群体)遗传分化不显著,这可能与选择的分子标记有关,在今后的研究中可采用核基因标记(微卫星标记)做进一步分析。口虾蛄的种群遗传结构模式可能与其栖息地海洋环境条件及自身的生活史特征相关。Du等[13]分析认为,口虾蛄成体营底栖生活,不同群体间发生基因交流依赖于浮游阶段幼体分布范围的连续性。分布于黄海和东海的口虾蛄产卵期为5—8月,这段时期长江径流量大,长江淡水的注入导致长江口海域盐度骤降,从而形成天然屏障阻碍了口虾蛄幼体连续性分布。而东海与南海的口虾蛄则可能由于全年受到海流(黑潮流系和赤道流系)影响,幼体被隔离在海流两侧[20]。基于现有的研究结果,可以认为,口虾蛄存在黄渤海群体、东海群体、南海群体3个显著分化的群体,在渔业管理中应将它们看作3个独立的管理单元。
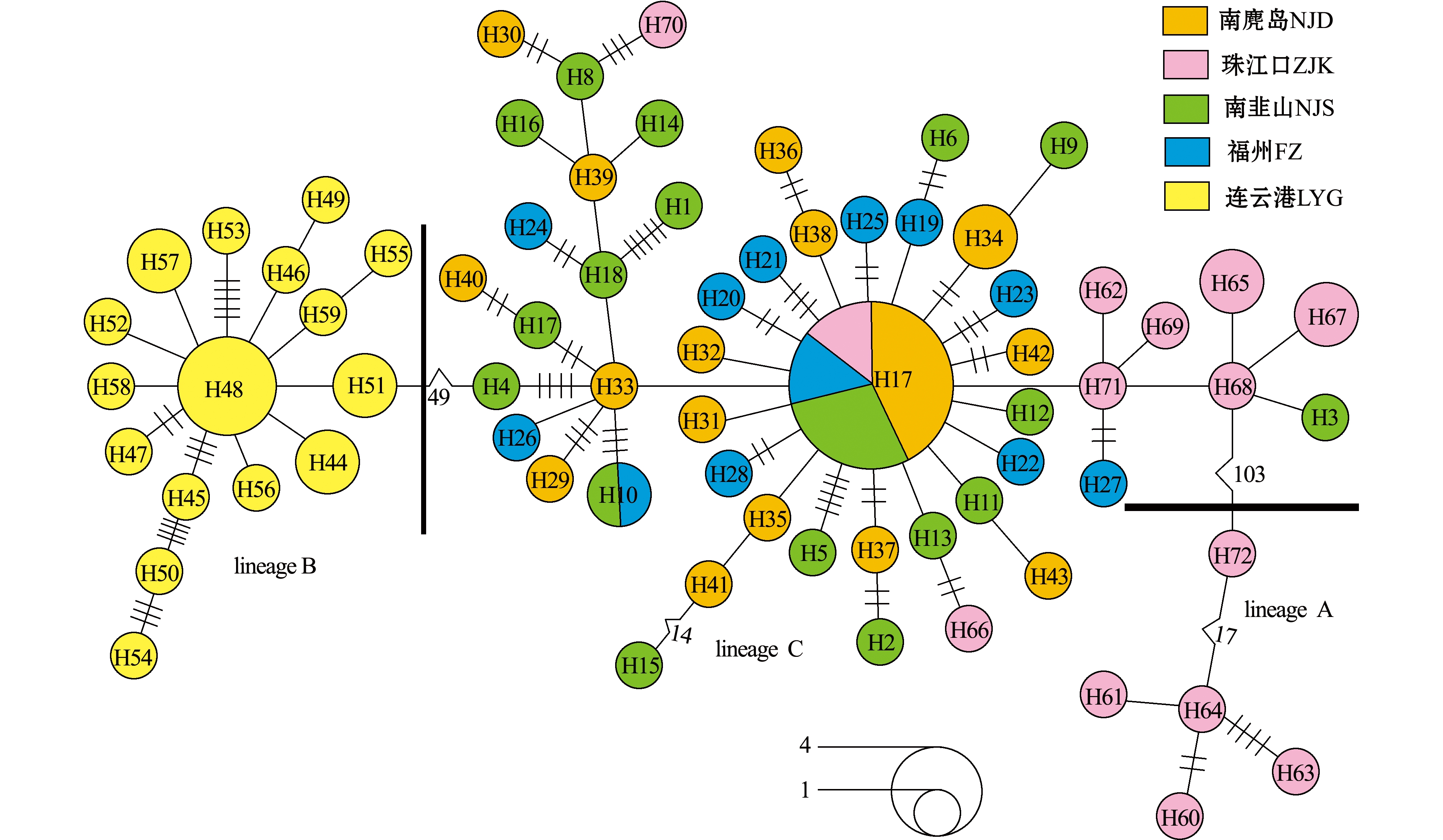
图2 基于Cytb序列单倍型构建的口虾蛄最小跨度树
Fig.2 Reduced median network in mantis shrimpOratosquilla oratoriabased on haplotypes of Cytbsequences
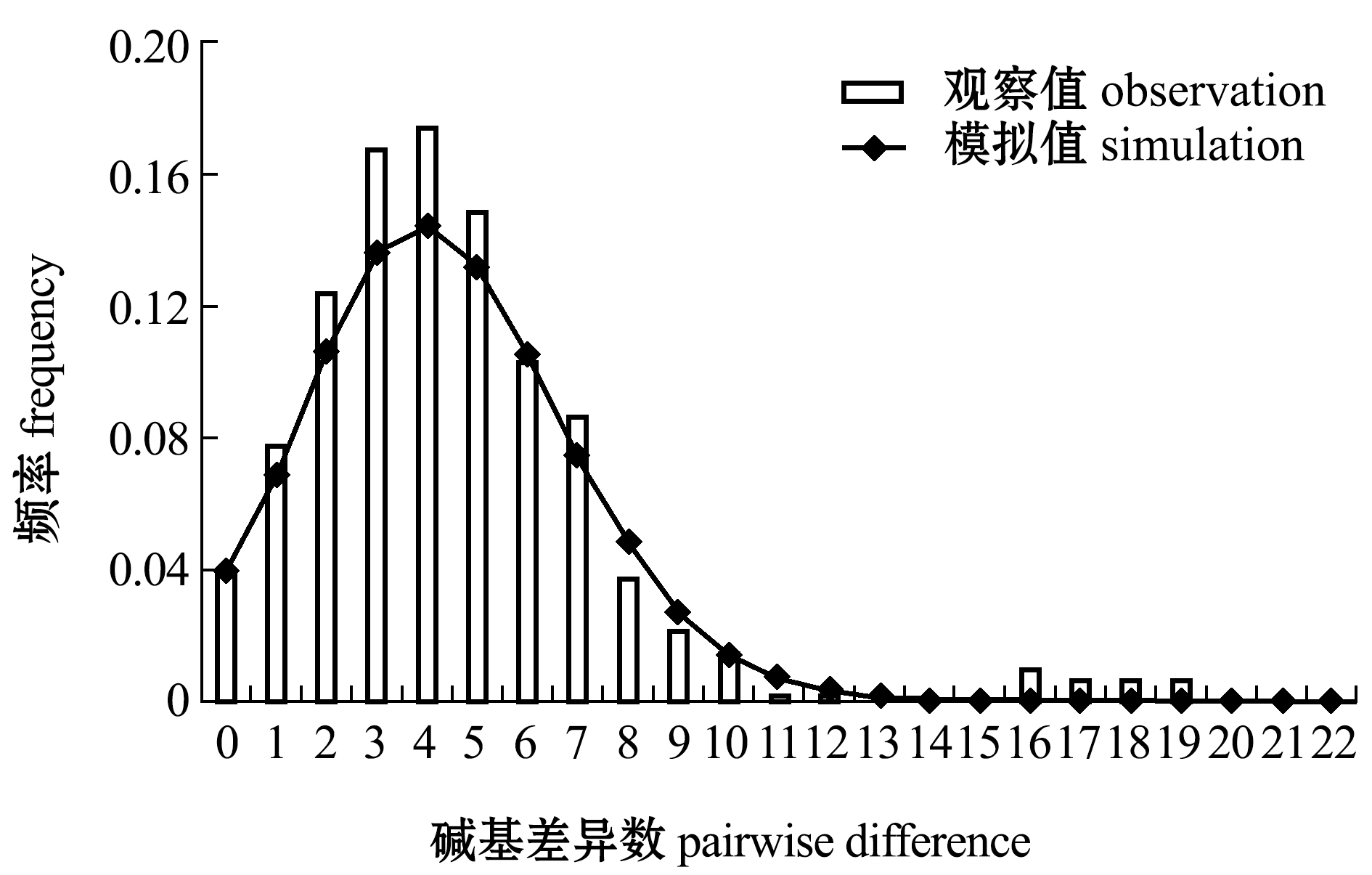
图3 口虾蛄谱系C Cytb序列核苷酸不配对分布图
Fig.3 Mismatch distributions of nucleotides of Cytbsequences in the lineage C of mantis shrimpOratosquilla oratoria
3.2 系统发育地理格局与群体历史动态
Cytb分子标记的核苷酸分歧速率常被用来估算种群扩张时间及种间分化年代,从而追溯种群的历史动态[18]。中国沿海口虾蛄群体存在明显的系统发育谱系结构,3个谱系单倍型类群组成在空间上的分布频率存在显著差异。根据谱系单倍型类群间净遗传距离及Cytb核苷酸分歧速率,估算谱系C与谱系A分化时间大约为745万年前,谱系C与谱系B分化时间大约为320万年前,谱系A与谱系B分化时间大约为785万年前。谱系A仅包括珠江口群体单倍型类群,谱系B仅包括连云港群体单倍型类群,提示谱系A、谱系B分别在南海和黄海被隔离分化;谱系C包括东海3个地理群体与珠江口群体的单倍型类群,东海群体与珠江口群体间遗传分化极显著(Fst=0.242,P<0.001),提示东海群体与珠江口群体由于隔离导致分化,在更新世间冰期发生群体扩张事件后(约11.0万年前)重新混合。西北太平洋许多生物在冰期-间冰期循环期间经历了明显的扩张和收缩,从而形成当今的系统发育地理格局[21]。Zhang等[10]认为,台湾岛的形成(约4000万年前)限制了口虾蛄南北群体发生基因交流,而末次冰期台湾海峡的波动进一步加快了口虾蛄的遗传分化,通过化石校准检测到口虾蛄谱系分化的时间大致为1090万年前。
[1] Ahyong S T,Chan T Y,Liao Y Z.A Catalog of the Mantis Shrimps (Stomatopoda) of Taiwan[M].Keelung,Taiwan,China:National Taiwan Ocean University,2008:147.
[2] 浙江动物志编辑委员会.浙江动物志甲壳类[M].杭州:浙江科学技术出版社,1991:84-85.
[3] 卢占晖,薛利建,张亚洲.东海口足类(Stomatopod)种类组成和数量分布[J].自然资源学报,2013,28(12):2159-2168.
[4] 李显森,吴亚飞,尤宗博,等.渤海口虾蛄三重刺网渔获组成及其捕捞性能分析[J].海洋渔业,2016,38(5):516-524.
[5] 潘国良,张龙,朱增军,等.浙江南部近岸海域春季口虾蛄(Oratosquilla oratoria)生物量的时空分布[J].海洋与湖沼,2013,44(2):366-370.
[6] 刘修泽,郭栋,王爱勇,等.辽东湾海域口虾蛄的资源特征及变化[J].水生生物学报,2014,38(3):602-608.
[7] 吴强,陈瑞盛,黄经献,等.莱州湾口虾蛄的生物学特征与时空分布[J].水产学报,2015,39(8):1166-1177.
[8] 盛福利.青岛近海口虾蛄(Oratosquilla oratoria)渔业生物学的初步研究[D].青岛:中国海洋大学,2009.
[9] 杜欣蔚.中国东部近海口虾蛄群体遗传多样性研究[D].舟山:浙江海洋大学,2016.
[10] Zhang Daizhen,Ding Ge,Ge Baoming,et al.Geographical distribution,dispersal and genetic divergence of the mantis shrimpOratosquilla oratoria(Stomatopoda:Squillidae) in China Sea[J].Biochemical Systematics and Ecology,2016,65:1-8.
[11] Zhang Daizhen,Ding Ge,Ge Baoming,et al.Comparative phylogeography of two marine species of crustacean:recent divergence and expansion due to environmental changes[J].Gene,2014,550(1):141-147.
[12] 董鑫,邢坤,隋宥珍,等.基于线粒体COI基因序列的4个海域口虾蛄群体的遗传多样性研究[J].海洋科学,2015,39(7):29-36.
[13] Du Xinwei,Cai Shanshan,Yu Cungen,et al.Population genetic structure of mantis shrimpsOratosquilla oratoria:testing the barrier effect of the Yangtze Riveroutflow[J].Biochemical Systematics and Ecology,2016,66:12-18.
[14] 于旭蓉,仇雪梅,柳晓瑜,等.线粒体DNA多态性在海洋动物群体遗传结构研究中的应用[J].生物技术通报,2011(10):49-54.
[15] 徐志进,章霞,柳敏海,等.利用Cytb和16S rRNA序列研究多鳞四指马鲅和四指马鲅的种群遗传结构[J].大连海洋大学学报,2015,30(3):266-270.
[16] 刘连为.三种大洋性柔鱼类种群遗传结构及分子系统地理学研究[D].上海:上海海洋大学,2014.
[17] 刘连为,周永东,陆化杰,等.西北太平洋鸢乌贼种群遗传结构[J].水产学报,2017,41(9):1355-1364.
[18] 蓝昭军,范明君,黄小林,等.基于线粒体Cytb基因的中国南方唇鱼骨(Hermibarbus labeo)和间鱼骨(Hermibarbus medius)种群分化及亲缘地理研究[J].生态学报,2016,36(19):6091-6102.
[19] 孙钦艳,王敏强,苏培,等.三疣梭子蟹不同地理群体线粒体DNAND5基因序列的变异与分化[J].大连海洋大学学报,2010,25(6):518-522.
[20] 张弦,俞慕耕,江伟,等.菲律宾海及其邻近海区的水文特征[J].海洋通报,2004,23(1):8-14.
[21] Hewitt G.The genetic legacy of the quaternary iceages[J].Nature,2000,405(6789):907-913.