Smads蛋白(drosophila mothers against decapentaplegic protein)家族是一类存在于真核生物的转录因子,在调控细胞的增殖分化,以及修复损伤、胚胎发生等方面发挥重要作用[1-3]。按照生物功能的不同,smad蛋白家族可分为受体活化型(R-smad)、共同通路型(Co-smad)和抑制型(I-smad)3种类型,其中,R-smads能被Ⅰ型受体激活并与受体形成短暂的复合物,主要包括由TGF-β激活的AR-smads(如smad2/3)和由BMP(bone morphogenetic protein,BMP)激活的BR-smads(如smad1、smad5、smad8、smad9等)[4]。Co-smad是TGF-β家族各类信号传导过程中共同需要的介质(如smad4)[5]。I-smads可与Ⅰ型受体结合进而调节TGF-β家族的信号传导(如smad6和smad7)[6-8]。大量研究显示,smad家族成员可结合目的基因的增强子或启动子区域,对细胞的生长发育起到增强或抑制的作用[9-10],在转录调控、动物生理学和疾病免疫过程中均发挥重要作用[10-17]。目前,关于smad基因家族成员的研究主要集中在基因序列、结构,以及胚胎发生过程中的表达模式和作用机制等方面[17-21]。有研究证实,斑马鱼Danio rerio smad2/3可参与斑马鱼胚胎的内胚层、外胚层和胚轴的形成过程[22-23]。还有研究发现,在细胞癌变和衰老过程中,人体内的smad2/3可以被TGF-β信号激活,起到调控细胞癌变和抗衰老的作用[24-27]。目前,针对海洋生物smad基因的研究尚处于起步阶段,截至2019年1月,在美国国立生物信息中心(National Center for Biotechnology Information,NCBI)登录的smad基因家族序列共17 519条,其中,脊索动物smad家族基因共有14 038条,节肢动物2524条,软体动物191条,棘皮动物39条,其他物种727条。在棘皮动物中,登录的海胆Echinoidea纲 smad家族基因序列为20条,海星Asteroidea纲 14条,海参Apostichopus japonicus5条。
中间球海胆Strongylocentrotus intermedius原产于日本北海道和俄罗斯远东等地沿海,1989年由大连海洋大学(原大连水产学院)自日本引进[28],现已在辽宁、山东等地形成了一定的人工养殖规模,是中国重要的经济海胆养殖种类之一。目前,围绕中间球海胆的基础研究主要集中在遗传力评估[28]、营养需求和病原体鉴定[29-30]等方面,但对中间球海胆smad基因家族的序列信息、组织表达规律和生物学功能的研究尚未见报道。脂多糖(lipopolysaccharides,LPS)作为一种免疫刺激剂,可通过免疫刺激观察动物组织中基因的表达情况来验证该基因的免疫功能。本研究中,采用cDNA末端快速扩增技术(Rapid-amplification of cDNA ends,RACE)和实时荧光定量PCR技术(qRT-PCR),首次获得中间球海胆smad2/3基因(命名为Si-smad2/3)的cDNA全长,并利用生物信息学技术分析了Si-smad2/3基因的序列信息及其编码蛋白的结构特征、组织表达规律及脂多糖刺激对其组织表达的影响,以期初步明确Si-smad2/3基因在中间球海胆免疫应答中的生物学作用,为水产动物smad基因家族的研究提供基础资料。
1 材料与方法
1.1 材料
试验用中间球海胆为大连海洋大学农业农村部北方海水增养殖重点实验室养殖群体,其湿体质量为(56±15)g,壳高为(31.9±5.5)mm,壳径为(54.7±7.7)mm。试验前,暂养于实验室循环水槽(60 L)中,水温为16~17 ℃,盐度为31.22~31.36,pH 为8.15~8.25,充足溶氧,暂养期间投喂海带Laminaria japonica,换水周期为2 d。
1.2 方法
1.2.1 试验设计及样品的制备
(1)Si-smad2/3基因cDNA序列克隆及组织表达模式分析。取健康中间球海胆3只,分别取每只海胆的肠、体腔细胞(抽取海胆体腔液,超低温离心机以3000 r/min离心10 min,弃上清液取沉淀)、性腺(不区分雌雄)、围口膜、齿间肌和管足,做好标记后置于液氮中保存备用。
(2)LPS免疫刺激试验。取42只健康中间球海胆,随机平均分成两组,给试验组每只海胆分别注入100 mL脂多糖(0.1 mg/mL)溶液,对照组注入100 L无菌海水。由于体腔细胞、管足和齿间肌是海胆与外界环境能够发生直接接触的3个重要器官,也是海胆对外界环境变化或刺激产生快速响应的重要组织,在海胆免疫防御和敌害防御过程中均发挥重要作用,因此,本研究中以中间球海胆体腔细胞、管足和围口膜为目标组织,探讨LPS刺激后不同时间点Si-smad2/3基因的表达变化情况。在分别注入脂多糖溶液和无菌海水后的3、6、9、12、24、48、72 h时,从每组中随机选取3只海胆,低温条件下,提取每只海胆的体腔细胞、管足和围口膜,做好标记后置于冰箱(-80 ℃)中保存备用。
1.2.2 总RNA的提取 采用RNAprep Pure Tissue Kit(目录号:DP431)(北京天根公司)试剂盒,按推荐方法提取中间球海胆各组织样品的总RNA。采用10 g/L琼脂糖凝胶电泳检测RNA的完整性,并使用Implen Nano Photometer核酸蛋白分析仪(德国)检测其纯度和浓度,将获得的RNA样品置于冰箱(-80 ℃)中保存备用。
1.2.3 中间球海胆Si-smad2/3基因核心片段的获得 从NCBI中选择下载海胆及其他近缘物种smad2/3基因的核酸序列,运用DNAMan 6.0软件进行多重序列比对,得到其核心保守区域,并在核心区域设计引物smad3′和smad5′(表1),并将引物送至生工生物工程(上海)股份有限公司进行引物合成。
以RACE cDN为模板进行核心片段PCR扩增,反应体系(10 μL):10×LA PCR Buffer 1 μL, dNTPs 0.8 μL, cDNA模板 0.5 μL,10 μmol/L的smad3′引物 0.4 μL,10 μmol/L的smad5′引物 0.4 μL,LA Taq 0.2 μL,ddH2O 6.7 μL。PCR反应程序:94 ℃下预变性10 min;94 ℃下循环变性30 s,60 ℃下退火复性30 s,72 ℃下延伸1 min,共进行35个循环;最后在72 ℃下再延伸10 min。PCR扩增后使用10 g/L琼脂糖凝胶电泳检测PCR目的片段产物条带的大小,采用EasyPure©Quick Gel Extraction Kit胶回收试剂盒(北京全式金生物技术有限公司)对RACE扩增获得的PCR产物切胶回收纯化。将目的片段与pEASY-T1 载体连接,转入到Trans1-T1感受态细胞中,摇菌涂板后,放置培养箱(37 ℃)中恒温过夜。翌日挑取单菌落接种于1 mL LB液体培养基(Amp终浓度为100 μg/mL)中振荡培养,通过菌落PCR使用M13通用引物(表1)初步鉴定阳性克隆,对重组子进行鉴定,将阳性克隆送至生工生物(上海)工程股份有限公司进行测序。将测序结果于NCBI中进行比对,获得中间球海胆Si-smad2/3基因核心片段。
1.2.4 中间球海胆Si-smad2/3基因cDNA全长的获得 以获得的Si-smad2/3基因核心片段为模板,运用Primer Premier 5.0软件设计Si-smad2/3基因的RACE引物(表1),并送至生工生物工程(上海)股份有限公司进行引物合成。
表1 用于smad2/3基因cDNA RACE和实时定量PCR的引物序列
Tab.1 Primer sequences used forsmad2/3 gene cDNA RACE and qRT-PCR

引物 primer 序列 sequence (5'-3')smad3'TCTTCATTTGT-GACGAGTGGAsmad5'AGGCTCACAG-TATGACACGGGsmad2/3-3'-outTTCACATCGT-TCTACAGCCTTGsmad2/3-5'-outGCCCGTGT-CATACTGT-GAGCCTsmad2/3-3'-inACGGCAGTAG-TACAGCCCCACAsmad2/3-5'-inCTTTGGGAT-GGGGGTGTGGTUPM-longTAATACGACT-CACTAT-AGGGCAAG-CAGTGGTAT-CAACGCAGAGTUPM-shortTAATACGACT-CACTATAGGGCSi-smad2/3-FGTGAAGAGGTT-GCTTGGATG-GAAGASi-smad2/3-RGATGGGGGTGT-GGTATAAATT-GTGCβ-actin-FAGAGGCG-TAGAGGGAAAG-CACβ-actin-RACAGG-GAAAAGATG-GCACAGA
3′RACE和5′RACE第一条链的合成按照SMARTERTMRACE5′/3′cDNA扩增试剂盒(Clontech公司,美国)操作步骤进行,获得RACE的cDNA模板。以RACE cDNA为模板进行5′/3′ PCR扩增。反应体系(10 μL):10×LA PCR Buffer 1 μL,dNTPs 0.8 μL,3′ cDNA模板/5′ cDNA模板 0.5 μL,10×UPM 0.4 μL,10 μmol/L的上、下游引物各 0.4 μL,LA Taq 0.2 μL,ddH2O 6.7 μL。PCR反应程序:94 ℃下预变性10 min;94 ℃下循环变性 30 s,60 ℃ 下退火复性30 s,72 ℃ 下延伸2 min,共进行35个循环;最后在72 ℃下再延伸10 min。对得到的PCR产物进行回收和纯化,与pEASY-T1载体连接后转至刚解冻的Trans-T1感受态细胞中,扩大培养并筛选阳性重组子(方法同Si-smad2/3基因核心片段克隆),并送至生工生物工程(上海)股份有限公司测序。
1.2.5 中间球海胆Si-smad2/3基因的生物信息学分析 利用DNAMan 6.0软件对测序得到的序列进行组装和拼接,得到Si-smad2/3基因完整的全长cDNA序列,采用BLAST(http://www.ncbi. nlm. nih.gov/blast)对得到的Si-smad2/3基因的全长cDNA序列进行比对分析。利用ORF Finder软件(http://www.ncbi.nlm.nih.gov/gorf/gorf)确定Si-smad2/3基因的开放阅读框(open reading frame,ORF);利用SMART(http://www.smart. emblheidelbergde)预测Si-smad2/3基因编码蛋白的结构域;使用ProtParam(http://web.expasy. org/ protparam)在线软件预测Si-smad2/3基因编码蛋白的相对分子质量;使用ExPasy(https://web.expasy.org/protscale/)在线软件分析Si-smad2/3基因编码蛋白的亲疏水性;使用PSIPRED(http://bioinf.cs.ucl.ac.uk/psipred/)在线软件分析Si-smad2/3全长cDNA编码蛋白序列的二级结构;使用Swiss-model(http://swissmodel. expasy.org/)在线软件分析Si-smad2/3全长cDNA编码蛋白序列的三维结构;利用DNAMan 6.0软件进行中间球海胆与其他物种smad2/3蛋白氨基酸序列的多重比对;利用MEGA 5.0软件构建基于邻接法(Neighbor-Joining,NJ)的系统进化树。
1.2.6 中间球海胆Si-smad2/3基因的组织表达分析 利用PrimeScriptTMRT reagent Kit(TaKaRa,大连)试剂盒分别将样品RNA反转录成cDNA,使用ABI公司的7500 (Life Technologies,USA)对Si-smad2/3基因表达情况进行荧光定量PCR(qRT-PCR)分析。选取β-actin作为内参基因[31],qRT-PCR所用引物见表1。为减小试验误差,每组设3个平行,所有cDNA稀释5倍,于-20 ℃下保存备用。试验采用二步法进行PCR反应,试剂盒为SYBR©Premix Ex TaqTMⅡ(Tli RNAseH Plus,TaKaRa)。反应体系(20 μL):2×SYBR Green Master mix 10 μL,上、下游引物各0.8 μL,ROX Reference DyeⅡ 0.4 μL,cDNA模板 2 μL,最后用RNase-Free ddH2O补足至20 μL。反应程序:95 ℃下预变性30 s;95 ℃下循环变性5 s,60 ℃下退火复性 32 s,共进行40个循环;溶解曲线阶段为95 ℃下变性15 s,60 ℃下退火复性1 min,95 ℃下变性15 s,60 ℃下退火复性15 s。采用2-ΔΔCt法分析Si-smad2/3基因的相对表达量。
1.3 数据处理
试验数据用平均值±标准差(mean±S.D.)表示,采用SPSS 16.0软件进行单因素方差分析(One-way ANOVA),用t检验法进行两组间的差异分析,显著性水平设为0.05,极显著性水平设为0.01。
2 结果与分析
2.1 Si-smad2/3基因全长cDNA序列及生物信息分析
从图1可见:中间球海胆Si-smad2/3基因cDNA序列全长为2146 bp,包含一个1341 bp编码446个氨基酸的开放阅读框,一个197 bp 5′ 端非编码区(5′ Untranslated Region)和一个608 bp 3′ 端非编码区。
生物信息学分析发现,中间球海胆Si-smad2/3基因编码的Si-smad2/3蛋白相对分子质量为50 300,等电点为6.93,该蛋白包含有两个显著的保守结构域,分别是位于第23~151位氨基酸的MH1(MAD homology 1)结构域和位于第251~422位氨基酸的MH2(MAD homology 2)结构域(图1阴影部分)。疏水性分析显示,中间球海胆Si-smad2/3蛋白序列中疏水性氨基酸残基所占面积小于亲水性氨基酸残基,其中疏水性最大值为1.80,亲水性最大值为3.04,提示该蛋白属于亲水性蛋白(图2)。蛋白二级结构预测结果显示,中间球海胆Si-smad2/3蛋白的二级结构中,包含6个α螺旋(α-helix)、14个β转角(β-strand)和20个无规则卷曲(coil)(图3)。利用Swiss-model软件进行中间球海胆Si-smad2/3蛋白的三维结构同源建模预测,结果显示,Si-smad2/3蛋白为多聚蛋白体,其Ile12~Leu152区域结构与人Homo sapienssmad3蛋白的Ile4~Leu128区域结构(GenBank登录号:5od6.1.A)高度相似(一致性为84.43%)(图4-A、B),而Si-smad2/3蛋白序列的Ser237~Ser446区域结构则与人smad2蛋白的Asn18~Ser227区域结构(GenBank登录号:1khx.1.A)高度相似(一致性为88.57%)(图4-C、D)。

注:方框代表起始密码子ATG;*为终止密码子TAA;阴影部分为smad2/3的两个结构域,分别为MH1结构域和MH2结构域
Note:The letters in box indicate the start codon (ATG);The asterisk (*) represents a terminal codon(TAA); The two domains with shadowedsmad2/3 are MH1 domain and MH2 domain,respectively
图1 中间球海胆Si-smad2/3基因核苷酸序列及其编码的氨基酸序列
Fig.1 Nucleotide and deduced amino acid sequences ofSi-smad2/3 in sea urchinStrongylocentrotus intermedius
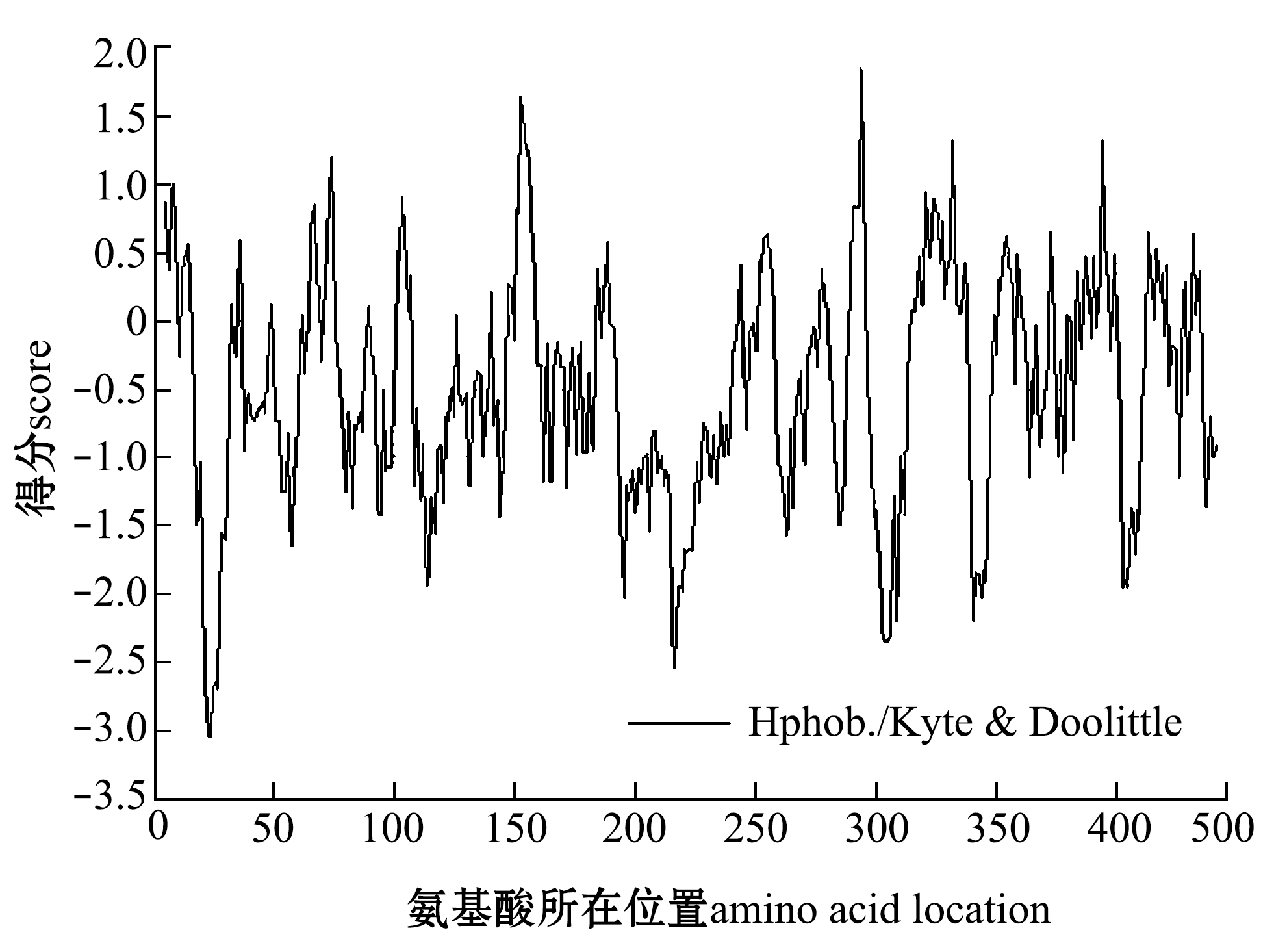
图2 中间球海胆Si-smad2/3蛋白疏水性/亲水性分析结果
Fig.2 Hydrophobicity/hydrophilicity analysis of Si-smad2/3 in sea urchinStrongylocentrotus intermedius

图3 中间球海胆Si-smad2/3蛋白二级结构
Fig.3 Predicted secondary structure of the Si-smad2/3 protein in sea urchinStrongylocentrotus intermedius
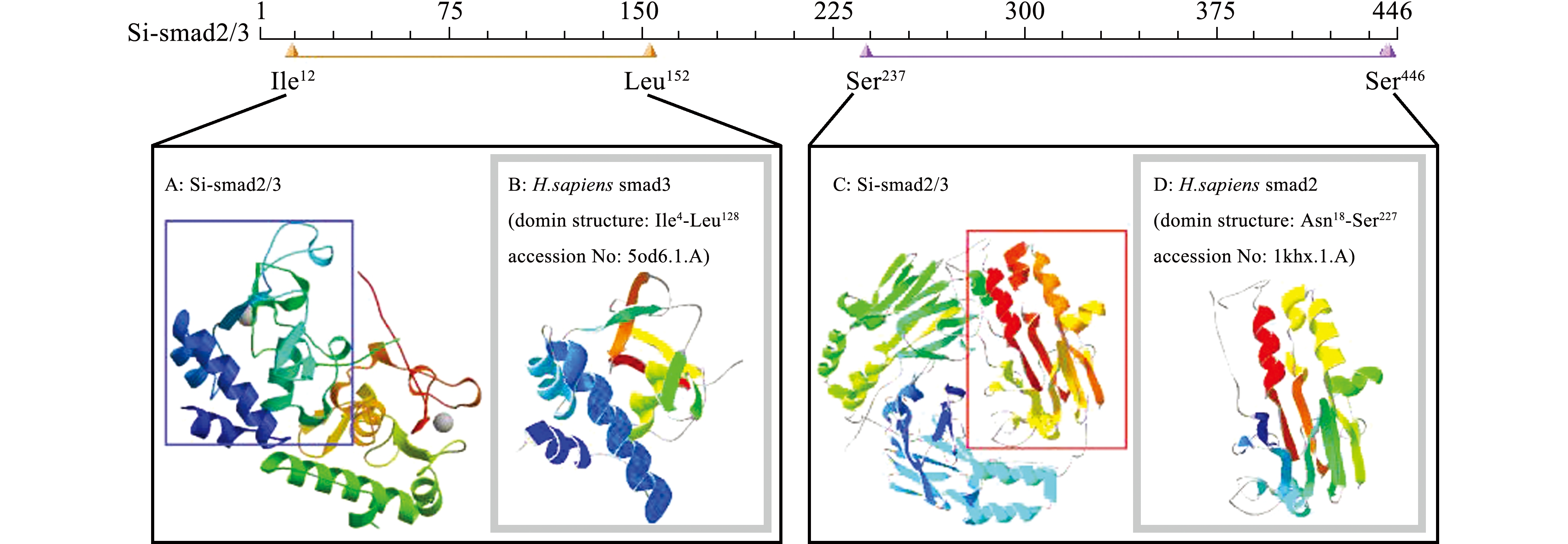
注:A为以人smad3为模板预测的中间球海胆Si-smad2/3蛋白三维结构,蓝框表示预测的Si-smad2/3蛋白的Ile12~Leu152区域;B为人smad3蛋白的Ile4~Leu128区域结构;C为以人smad2为模板预测的中间球海胆Si-smad2/3蛋白三维结构,红框表示预测的Si-smad2/3蛋白的Ser237~Ser446区域;D为人smad2蛋白的Asn18~Ser227区域结构
Note:A, 3D structure of Si-smad2/3 protein predicted by human smad3 as template and blue frame as Ile12-Leu152region of predicted Si-smad2/3 protein; B, Ile4-Leu128domain structure of smad3 in human beingsHomo sapiens; C, 3D structure of Si-smad2/3 protein predicted by human smad2 as template and red frame indicating the predicted Ser237-Ser446region of Si-smad2/3 protein; D, Asn18-Ser227domain structure of smad2 in human beingsHomo sapiens
图4 中间球海胆Si-smad2/3蛋白三维结构预测
Fig.4 The 3D structure prediction of Si-smad2/3 in sea urchinStrongylocentrotus intermedius
2.2 Si-smad2/3序列同源性比较及系统进化分析
多序列比对结果(图5)显示:中间球海胆Si-smad2/3氨基酸序列与其他13种生物smad氨基酸序列(表2)的平均一致性为74%,其中与紫球海胆S.purpuratussmad2/3氨基酸序列一致性高达96%;与脊椎动物人Homo sapienssmad2、人smad3、小鼠Mus musculussmad2、小鼠smad3、原鸡Gallus gallussmad2、原鸡smad3、斑马鱼Danio reriosmad2、非洲爪蟾Xenopus laevissmad2和非洲爪蟾smad3的一致性分别为71%、73%、71%、72%、71%、70%、74%、73%、73%;与无脊椎动物长棘海星Acanthaster plancismad2/3、虾夷扇贝Mizuhopecten yessoensissmad3、长牡蛎Crassostrea gigassmad3的一致性分别为79%、70%和71%。

图5 14种生物smad基因编码的氨基酸多序列比对
Fig.5 Multiple sequence alignment of thesmadencoded amino acid sequences from sea urchinStrongylocentrotus intermediusand other species
表2 多重序列比对和系统进化树所用的smad序列信息
Tab.2 Sequence information of smad2/3 for multiple sequence alignment and phylogenetic analysis
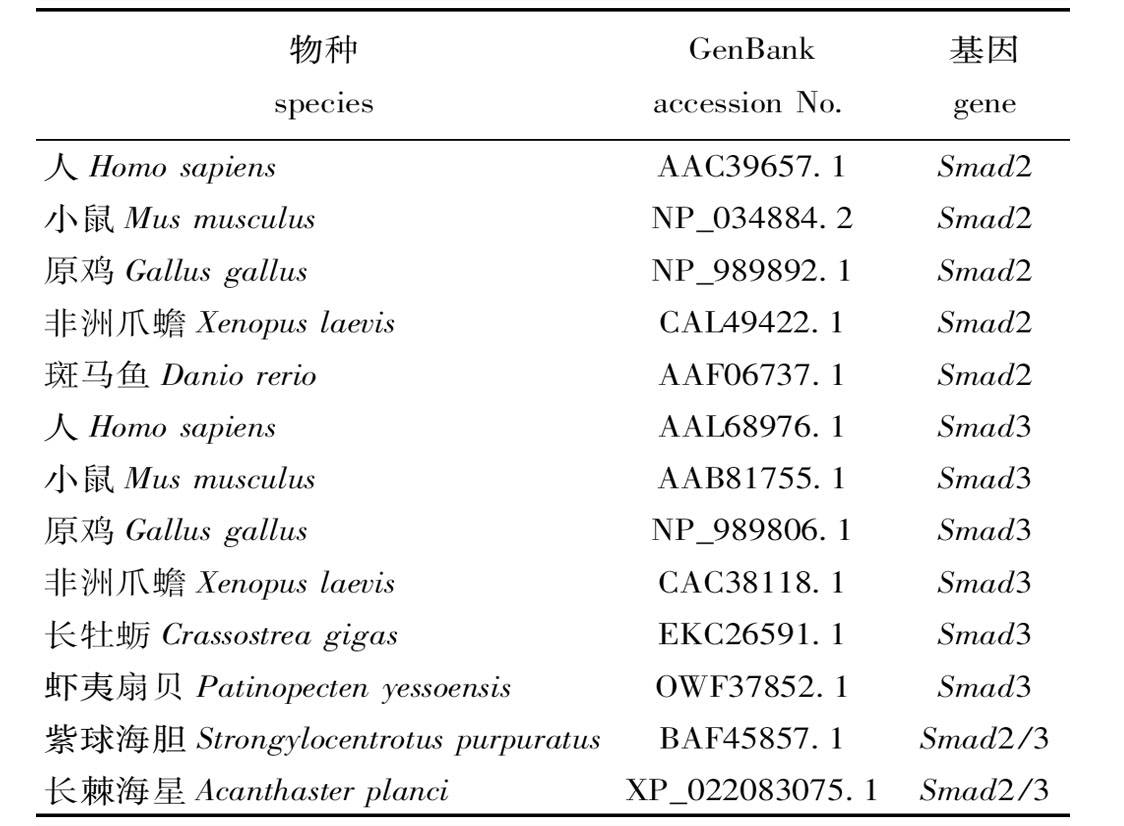
物种 speciesGenBankaccession No.基因gene 人Homo sapiensAAC39657.1Smad2小鼠Mus musculusNP_034884.2Smad2原鸡Gallus gallusNP_989892.1Smad2非洲爪蟾Xenopus laevisCAL49422.1Smad2斑马鱼Danio rerioAAF06737.1Smad2人Homo sapiensAAL68976.1Smad3小鼠Mus musculusAAB81755.1Smad3原鸡Gallus gallusNP_989806.1Smad3非洲爪蟾Xenopus laevisCAC38118.1Smad3长牡蛎Crassostrea gigasEKC26591.1Smad3虾夷扇贝Patinopecten yessoensisOWF37852.1Smad3紫球海胆Strongylocentrotus purpu-ratusBAF45857.1Smad2/3长棘海星Acanthaster planciXP_022083075.1Smad2/3
利用MEGA 5.0软件以邻接法(Neighbor-Joining,NJ)构建基于smad2/3氨基酸序列的分子系统进化树(图6),结果显示,脊椎动物和无脊椎动物的smad各自聚为一支,其中,中间球海胆smad2/3氨基酸序列与紫球海胆smad2/3、长棘海星smad2/3聚为一支,且与紫球海胆亲缘关系最近,符合中间球海胆的进化地位和分类地位。
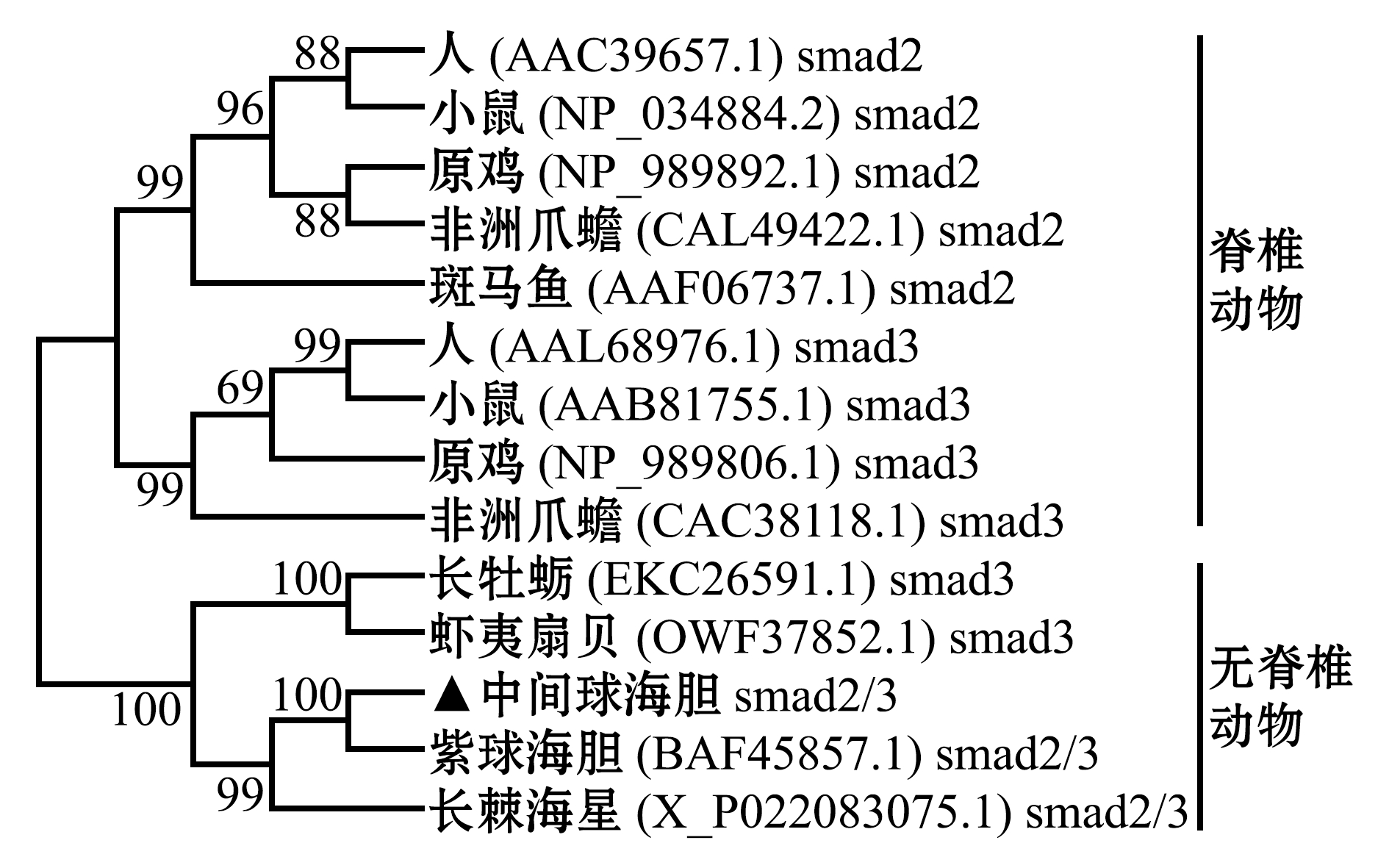
图6 基于14种生物smad2/3氨基酸序列的系统进化树
Fig.6 Phylogenetic tree of smad2/3 amino acid sequences from 14 species animals
2.3 中间球海胆smad2/3基因的组织表达特征
从图7可见:Si-smad2/3基因在中间球海胆齿间肌、管足、围口膜、体腔细胞、性腺和肠6种组织中均有表达,且呈现一定的组织表达规律,其相对表达量依次为体腔细胞>管足>性腺>围口膜>肠>齿间肌;仅体腔细胞中的表达量显著高于其他组织(P<0.05)。
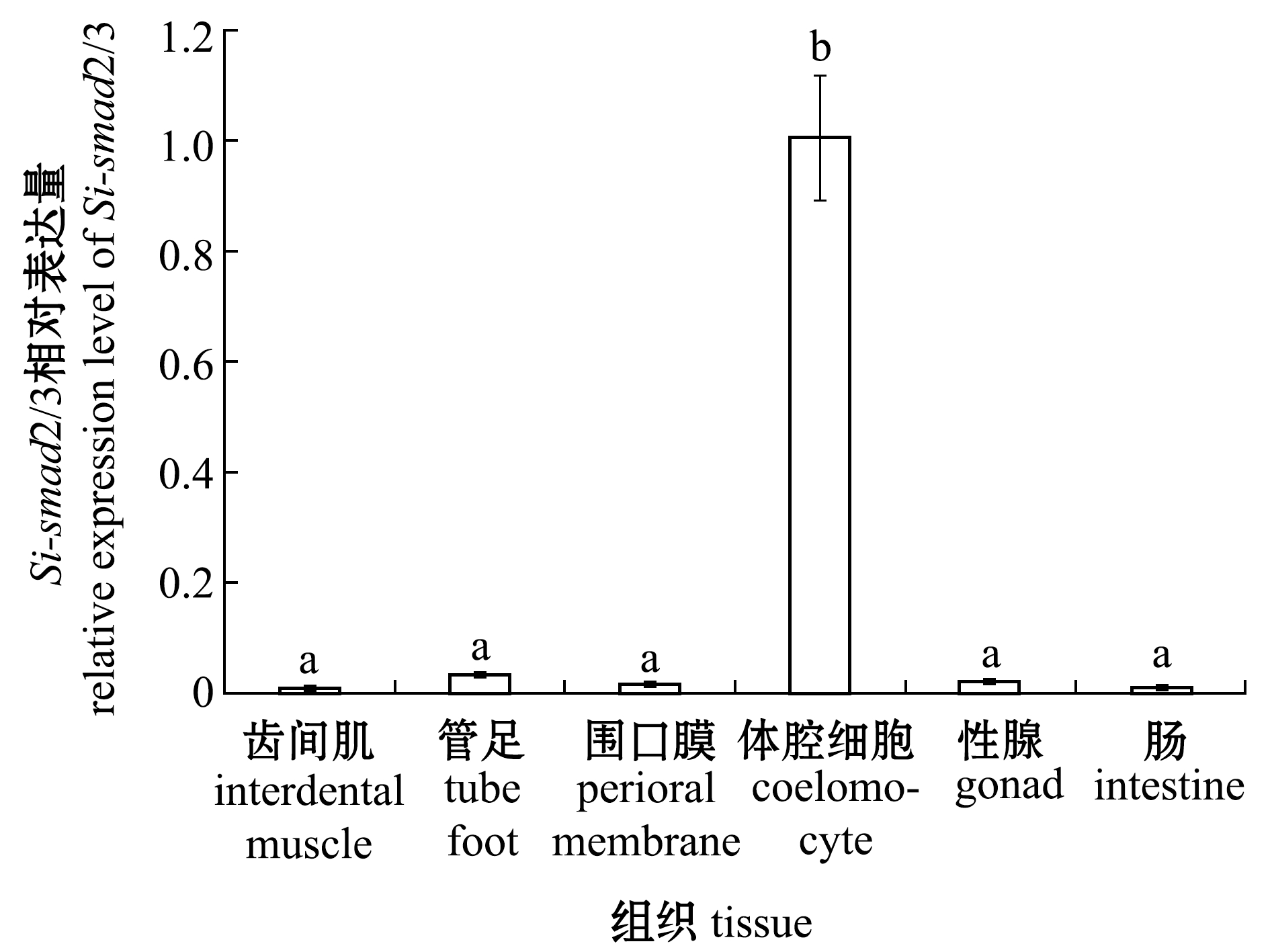
注:标有不同字母者表示组间有显著性差异(P<0.05),标有相同字母者表示组间无显著性差异(P>0.05)
Note:The means with different letters within are significantly different in the groups at the 0.05 probability level, and the means with the same letters are not significant differences
图7 中间球海胆Si-smad2/3基因的组织表达分析
Fig.7 Relative expression level analysis ofSi-smad2/3 in different tissues ofStrongylocentrotus intermedius
2.4 LPS刺激后中间球海胆Si-smad2/3基因的时空表达分析
从图8-A可见:经LPS刺激后,试验组中间球海胆Si-smad2/3基因在体腔细胞中的相对表达量呈现先升高后降低再升高再降低的变化趋势,两个表达高峰分别出现在LPS刺激后的6 h和9 h(P<0.05),其中,9 h时的相对表达量最高;在LPS刺激后的各个时间点,试验组的表达量与未经LPS刺激的对照组相比,均存在显著或极显著性差异(P<0.05或P<0.01)。
从图8-B可见:经LPS刺激后,试验组中间球海胆Si-smad2/3基因在管足中的相对表达量呈现先升高后降低再升高再降低的变化趋势,两个表达高峰分别出现在LPS刺激后的6 h和24 h(P<0.05),其中,6 h时相对表达量最高;在LPS刺激后的各个时间点,试验组的表达量与未经LPS刺激的对照组相比,均存在显著或极显著性差异(P<0.05或P<0.01)。
从图8-C可见:经LPS刺激后,试验组中间球海胆Si-smad2/3在围口膜中的相对表达量呈现先升高后降低再升高的趋势,两个表达高峰分别出现在LPS刺激后的9 h和72 h(P<0.05),其中,72 h时的相对表达量最高;除6 h外,其余时间点试验组的表达量与未经LPS刺激的对照组相比,均存在显著或极显著性差异(P<0.05或P<0.01)。
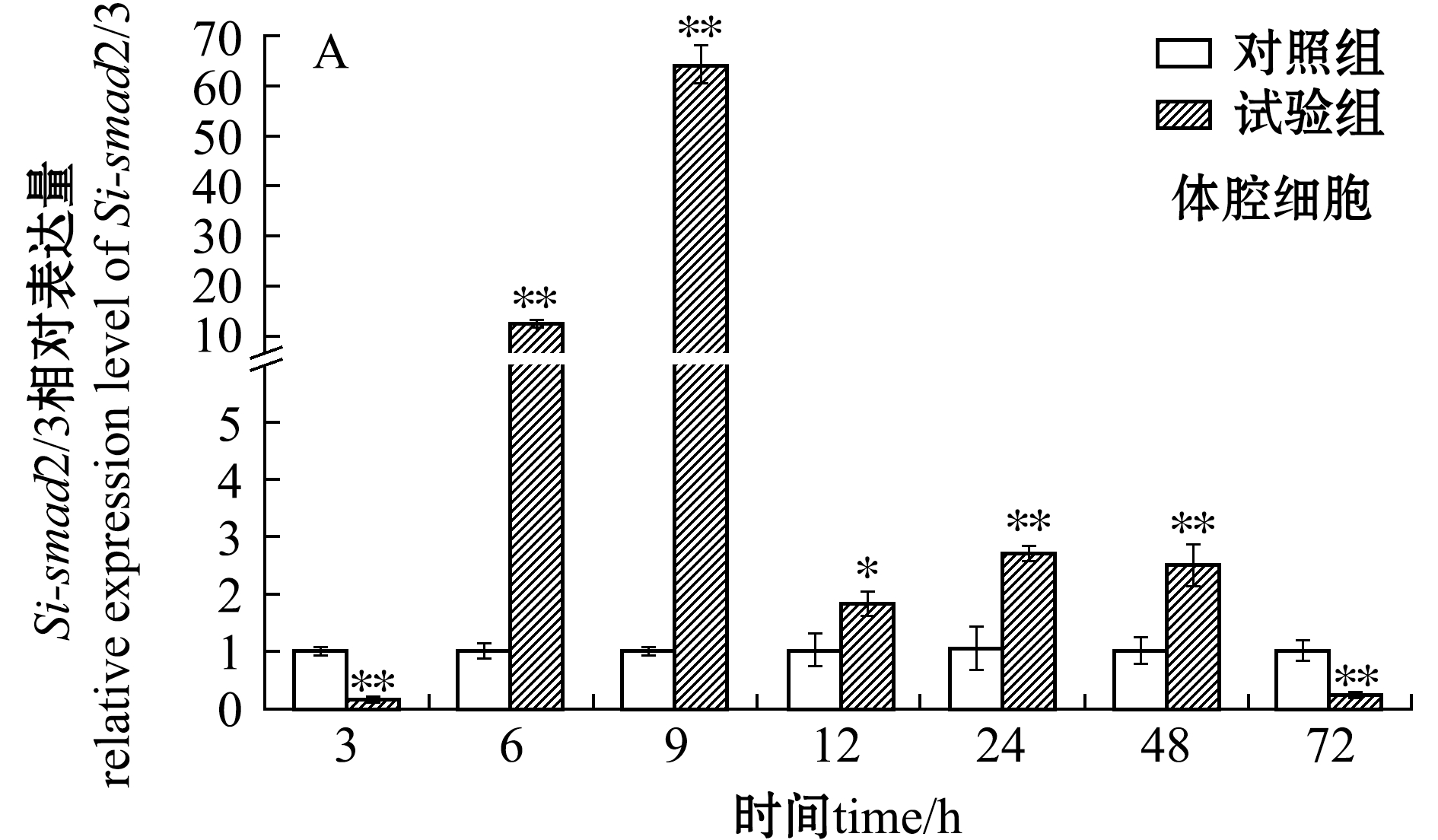
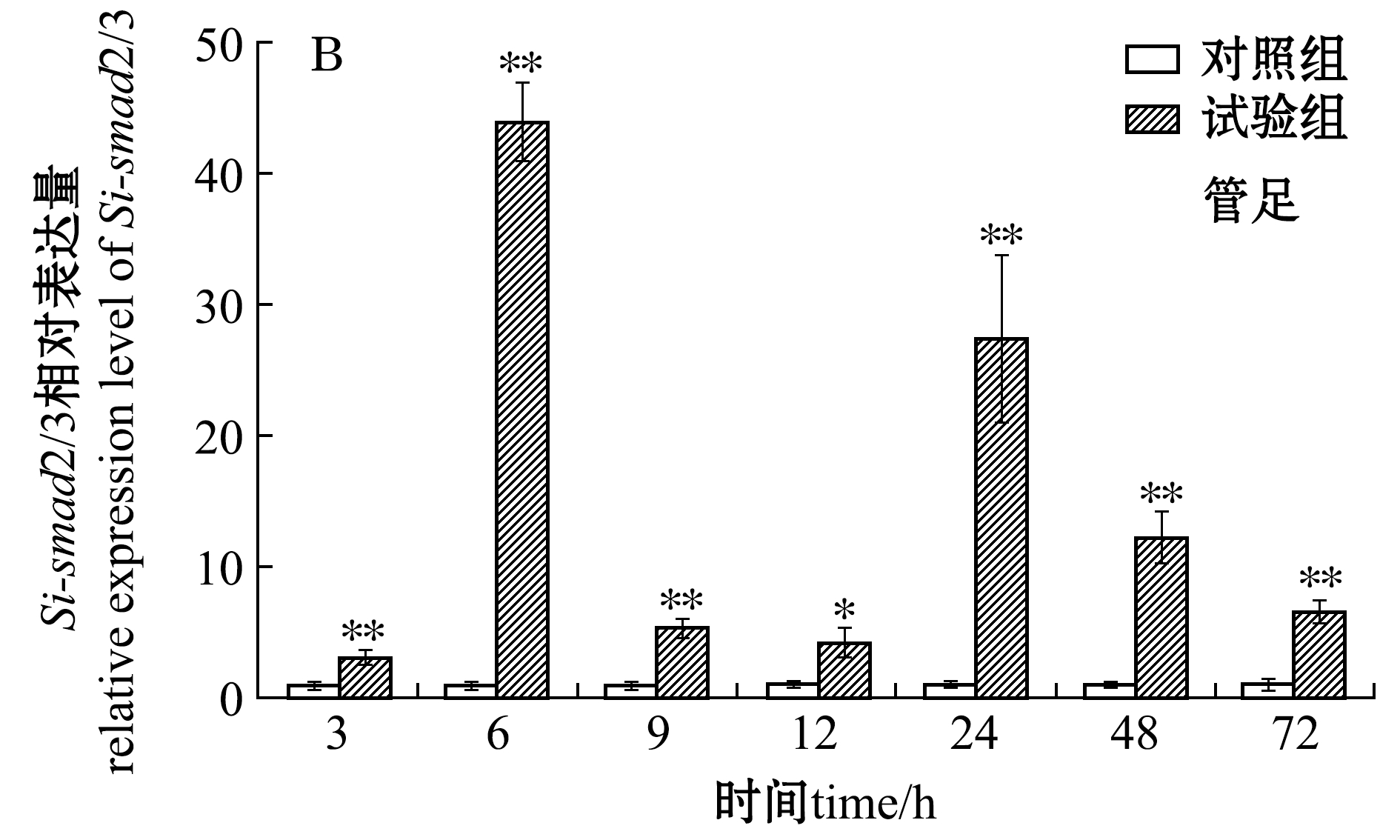
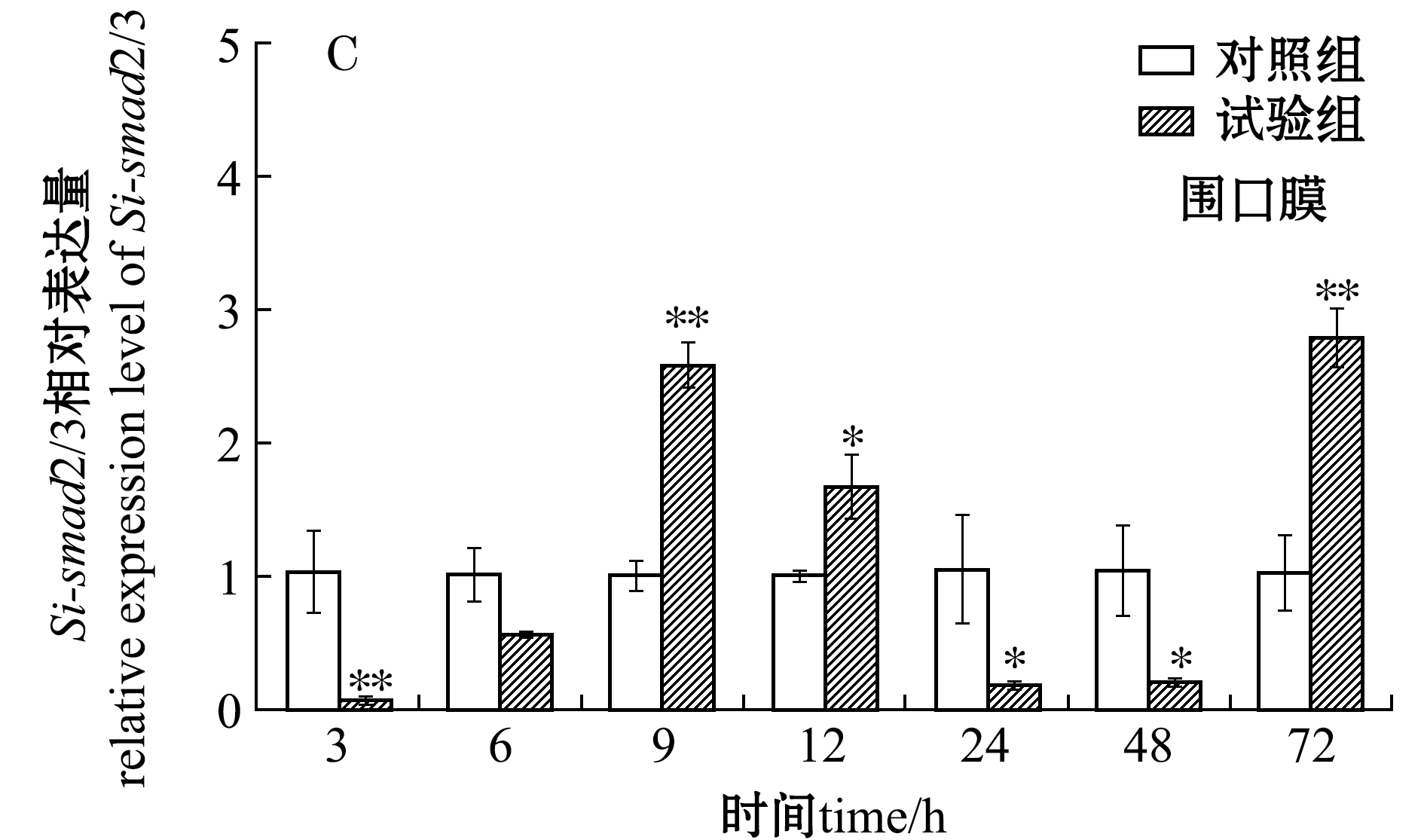
注:*表示与对照组有显著性差异(P<0.05);**表示与对照组有极显著性差异(P<0.01)
Note: *means significant difference compared with the control(P<0.05);**means very significant difference compared with the control(P<0.01)
图8 LPS刺激后中间球海胆体腔细胞、管足和围口膜中Si-smad2/3基因的变化表达
Fig.8 Relative expression levels ofSi-smad2/3 in coelomocytes, tube feet and peristomial membranes of sea urchinStrongylocentrotus intermediusexposed to LPS stimulation
3 讨论
3.1 Si-smad2/3的序列特征
本研究中利用同源克隆和RACE技术首次扩增得到了中间球海胆Si-smad2/3 cDNA全长序列。通过生物信息学分析发现,中间球海胆Si-smad2/3基因与已知9种生物的smad2/3基因具有高度的保守性,该序列编码446个氨基酸,与同源性最高的紫球海胆smad2/3氨基酸序列相比多了19个氨基酸,分析其原因可能是在物种进化过程中,该基因在转录时发生了种属特异性可变剪接。蛋白二级结构预测显示,中间球海胆Si-smad2/3蛋白二级结构与紫球海胆smad2/3蛋白二级结构均具有超过50%的无规则卷曲(coil),与斑马鱼smad2/3蛋白二级结构中的α螺旋(helix)和无规则卷曲的数量一致。此外,本研究中Si-smad2/3蛋白是由N端的一个MH1结构域、中间富含脯氨酸的连接区和C端的MH2结构域组成,与Shi等[32]的研究结果具有一定的一致性。由于目前结构数据库中尚无完整的smad蛋白结构预测模型,本研究团队仅在Swiss-model数据库中挖掘到了人smad2蛋白和smad3蛋白的部分空间结构模型,所以采用同源建模方法仅预测到了Si-smad蛋白的部分三维结构,由于Si-smad蛋白与人的smad2和smad3蛋白都具有相似的空间结构,因此,本研究中将其命名为Si-smad2/3,如果进一步调整算法,进行Si-smad2/3蛋白空间结构的从头预测,则可以进一步明确Si-smad2/3蛋白的空间结构,为Si-smad2/3蛋白的亚型分类提供更为明确的证据支持。对14种生物smad2/3蛋白的多序列比对结果表明,中间球海胆Si-smad2/3蛋白序列与人、小鼠等脊椎动物平均一致性达到72%;与海星、紫球海胆和贝类等无脊椎动物平均一致性达到79%,这一结果提示smad2/3基因在真核生物中是高度保守的。系统进化分析结果显示,中间球海胆Si-smad2/3与紫球海胆smad2/3聚为最近的一支,表明海胆的smad2/3在进化过程中高度保守,这一关系也符合中间球海胆的进化和分类地位。
3.2 Si-smad2/3基因的免疫防御机制
qRT-PCR检测结果显示,中间球海胆Si-smad2/3基因在成体中间球海胆齿间肌、管足、围口膜、体腔细胞、性腺和肠等6个组织中均有表达,其中,Si-smad2/3基因在中间球海胆体腔细胞中的相对表达量最高,其次是管足、性腺、围口膜和肠,齿间肌中的相对表达量最低,这一结果表明,中间球海胆Si-smad2/3基因的表达不仅具有鲜明的组织特异性,且在不同组织中执行的生物功能也可能有所不同。棘皮动物具有真体腔,其免疫机制属于先天免疫,体腔中的体腔液含有参与免疫反应的体腔细胞及多种体液免疫因子,可直接作用于入侵病原体,是棘皮动物先天性免疫防御的重要组织[33]。LPS是革兰氏阴性细菌细胞壁的主要组成成分,对LPS的识别与信号转导是机体自身防御反应的重要环节,同时也是导致内毒素性休克、全身炎症反应综合征和多器官功能衰竭等疾病的重要机制[34]。本研究中,正常的中间球海胆Si-smad2/3基因在体腔细胞中的相对表达量最高,表明Si-smad2/3可能在中间球海胆的免疫防御中发挥作用。进一步的免疫刺激试验结果显示,经LPS刺激之后,中间球海胆Si-smad2/3基因在体腔细胞中的相对表达量呈现先升高后降低再升高再降低的变化趋势,这与姬南京等[31]报道的经LPS(0.1 mg/mL)刺激后中间球海胆溶菌酶(lysozyme,LYZ)在体腔液中的相对表达量显著增加,且呈现先上升后下降的趋势具有一定的一致性,这一结果从一定角度证实了Si-smad2/3基因在中间球海胆免疫防御中发挥作用的可能性。管足是一种多功能的器官,具有感觉、运动、吸附和呼吸作用,在海水中与底质接触的过程中可与病原菌直接接触[34],中间球海胆Si-smad2/3基因在管足中的相对表达量仅次于体腔液,也进一步表明,Si-smad2/3基因可能在中间球海胆免疫防御中发挥一定的作用。海胆的围口膜长期附着在底泥和藻类等食物上面,易受病原体侵染[31],李太武等[35]研究显示,中间球海胆受病原侵染的一个主要病理特征为围口膜变黑。本研究中,中间球海胆Si-smad2/3基因虽然在围口膜中呈现较低的相对表达量,但值得注意的是,经LPS刺激后,中间球海胆Si-smad2/3基因的相对表达量却呈现出先升高后降低再升高再降低的趋势,表明中间球海胆Si-smad2/3基因可能参与围口膜组织的免疫应答过程。
[1] Lapraz F,Röttinger E,Duboc V,et al.RTK and TGF-β signaling pathways genes in the sea urchin genome[J].Developmental Biology,2006,300(1):132-152.
[2] Range R,Lapraz F,Quirin M,et al.Cis-regulatory analysis of nodal and maternal control of dorsal-ventral axis formation byunivin,a TGF-β related to Vg1[J].Development,2007,134(20):3649-3664.
[3] Yaguchi S,Yaguchi J,Burke R D.Sp-Smad2/3 mediates patterning of neurogenic ectoderm by nodal in the sea urchin embryo[J].Developmental Biology,2007,302(2):494-503.
[4] Zito F,Costa C,Sciarrino S,et al.Expression ofunivin,a TGF-β growth factor,requires ectoderm-ECM interaction and promotes skeletal growth in the sea urchin embryo[J].Developmental Biology,2003,264(1):217-227.
[5] Zhao Jingsong,Crowe D L,Castillo C,et al.Smad7 is a TGF-β-inducible attenuator of Smad2/3-mediated inhibition of embryonic lung morphogenesis[J].Mechanisms of Development,2000,93(1-2):71-81.
[6] Datto M,Wang X F.The Smads:transcriptional regulation and mouse models[J].Cytokine & Growth Factor Reviews,2000,11(1-2):37-48.
[7] Yoshida K,Matsuzaki K,Mori S,et al.Transforming growth factor-β and platelet-derived growth factor signal via c-Jun N-terminal kinase-dependent Smad2/3 phosphorylation in rat hepatic stellate cells after acute liver injury[J].The American Journal of Pathology,2005,166(4):1029-1039.
[8] Gao Sheng,Alarcón C,Sapkota G,et al.Ubiquitin ligase Nedd4L targets activated Smad2/3 to limit TGF-β signaling[J].Molecular Cell,2009,36(3):457-468.
[9] Jia Shunji,Wu Di,Xing Cencan,et al.Smad2/3 activities are required for induction and patterning of the neuroectoderm in zebrafish[J].Developmental Biology,2009,333(2):273-284.
[10] Preisser F,Giehl K,Rehm M,et al.Inhibitors of oxygen sensing prolyl hydroxylases regulate nuclear localization of the transcription factors Smad2 and YAP/TAZ involved in CTGF synthesis[J].Biochimica et Biophysica Acta (BBA) - Molecular Cell Research,2016,1863(8):2027-2036.
[11] García R,Nistal J F,Merino D,et al.p-SMAD2/3 and DICER promote pre-miR-21 processing during pressure overload-associated myocardial remodeling[J].Biochimica et Biophysica Acta (BBA) - Molecular Cell Research,2015,1852(7):1520-1530.
[12] Jung K J,Kim J,Park Y K,et al.Wen-pi-tang-Hab-Wu-ling-san reduces ureteral obstructive renal fibrosis by the reduction of oxidative stress,inflammation,and TGF-β/Smad2/3 signaling[J].Food and Chemical Toxicology,2010,48(2):522-529.
[13] Ruetz T,Pfisterer U,Di Stefano B,et al.Constitutively active SMAD2/3 are broad-scope potentiators of transcription-factor-mediated cellular reprogramming[J].Cell Stem Cell,2017,21(6):791-805.
[14] Singh A M,Reynolds D,Cliff T,et al.Signaling network crosstalk in human pluripotent cells:a Smad2/3-regulated switch that controls the balance between self-renewal and differentiation[J].Cell Stem Cell,2012,10(3):312-326.
[15] Coutts S M,Childs A J,Fulton N,et al.Activin signals via SMAD2/3 between germ and somatic cells in the human fetal ovary and regulates kit ligand expression[J].Developmental Biology,2008,314(1):189-199.
[16] Will A E,Baker J C.E2a is necessary for Smad2/3-dependent transcription and the direct repression ofleftyduring gastrulation[J].Developmental Cell,2015,32(3):345-357.
[17] Lan Debin,Lu Min,Sharma S,et al.Trans-resveratrol inhibits phosphorylation of Smad2/3 and represses FSHβ gene expression by a SirT1-independent pathway in LβT2 gonadotrope cells[J].Reproductive Toxicology,2011,32(1):85-92.
[18] Lapraz F,Duboc V,Lepage T.A genomic view of TGF-β signal transduction in an invertebrate deuterostome organism and lessons from the functional analyses of Nodal and BMP2/4 during sea urchin development[J].Signal Transduction,2007,7(2):187-206.
[19] Kumar S,Pan C C,Shah N,et al.Activation of mitofusin2 by Smad2-RIN1 complex during mitochondrial fusion[J].Molecular Cell,2016,62(4):520-531.
[20] Finnson K W,Parker W L,Chi Y,et al.Endoglin differentially regulates TGF-β-induced Smad2/3 and Smad1/5 signalling and its expression correlates with extracellular matrix production and cellular differentiation state in human chondrocytes[J].Osteoarthritis and Cartilage,2010,18(11):1518-1527.
[21] Blaney Davidson E N,Van Caam A P M,Vitters E L,et al.TGF-β is a potent inducer of nerve growth factor in articular cartilage via the ALK5-Smad2/3 pathway.Potential role in OA related pain?[J].Osteoarthritis and Cartilage,2015,23(3):478-486.
[22] Miosge N.Chondrogenic progenitor cells are key players in the regeneration of cartilage tissue[J].Osteoarthritis and Cartilage,2015,23 (Suppl 2):A381-A382.
[23] 贾顺姬.Smad2/3在斑马鱼胚胎早期发育中的作用[D].北京:清华大学,2008.
[24] Thien A,Prentzell M T,Holzwarth B,et al.TSC1 activates TGF-β-Smad2/3 signaling in growth arrest and epithelial-to-mesenchymal transition[J].Developmental Cell,2015,32(5):617-630.
[25] Murai M,Tsuji G,Hashimoto-Hachiya A,et al.An endogenous tryptophan photo-product,FICZ,is potentially involved in photo-aging by reducing TGF-β-regulated collagen homeostasis[J].Journal of Dermatological Science,2018,89(1):19-26.
[26] Jung H Y,Shin J C,Park S M,et al.Pinus densifloraextract protects human skin fibroblasts against UVB-induced photoaging by inhibiting the expression of MMPs and increasing type I procollagen expression[J].Toxicology Reports,2014,1:658-666.
[27] 程志芬,孙凤丹,张金子,等.胃癌组织中TGF-β1、TGF-βRⅡ、Smad2/3和CDC25蛋白的表达及其临床意义[J].临床与实验病理学杂志,2011,27(10):1044-1047.
[28] 韩奋杰,张伟杰,秦宇博,等.中间球海胆幼体及稚海胆生长性状的遗传参数估计[J].大连海洋大学学报,2017,32(2):145-149.
[29] 常亚青,封妮莎,王轶南,等.虾夷马粪海胆致病菌强壮弧菌的PCR检测方法[J].大连海洋大学学报,2012,27(4):289-293.
[30] 李磊,杨丹,齐守冰,等.中间球海胆繁殖前后性腺中脂类和脂肪酸含量变化[J].大连海洋大学学报,2018,33(4):423-429.
[31] 姬南京,杨芸菲,丁君,等.虾夷马粪海胆溶菌酶基因全长cDNA的克隆与表达分析[J].中国水产科学,2013,20(5):950-957.
[32] Shi Yigong,Massagué J.Mechanisms of TGF-β signaling from cell membrane to the nucleus[J].Cell,2003,113(6):685-700.
[33] 孟繁伊,麦康森,马洪明,等.棘皮动物免疫学研究进展[J].生物化学与生物物理进展,2009,36(7):803-809.
[34] 常亚青,丁君,宋坚,等.海参、海胆生物学研究与养殖[M].北京:海洋出版社,2004.
[35] 李太武,徐善良,王仁波,等.虾夷马粪海胆黑嘴病的初步研究[J].海洋科学,2000,24(3):41-44.