过氧化物酶体增殖物激活受体(Peroxisome proliferator-activated receptors,PPAR)是一类可以被脂肪酸样化合物过氧化物酶体增殖剂PP(Peroxisome proliferator,PP)激活的甾体激素受体。PPARα、PPARβ和PPARγ是其超家族成员,是调节碳水化合物和脂质代谢的关键转录因子,能够适应能量需求和营养状况的变化[1-2]。PPARα主要作用是控制脂肪酸摄取及活化;PPARβ主要分布于脂肪组织、骨骼和肌肉中,控制脂肪酸氧化;PPARγ则控制脂类储存和脂肪形成组织[3-4]。从结构来看,PPARα/β/γ的3种亚型都分别存在6个区域(A~F),并可分为4个功能结构域,A、B区是非配体依赖的转录活化区,C区是DNA结合区(DNA-banding-domain,DBD),E、F区则是配基结合区(LBD),D区是链接DBD和LBD的区域;PPARα/β/γ首先通过配体激活,然后与类视黄醇X受体(RXR)结合形成异二聚体,并与靶基因启动子区域中的特定PPAR(Peroxisome proliferator response element)反应元件结合,特异性地调节基因表达[5-7]。
菲律宾蛤仔Ruditapes philippinarum(以下简称蛤仔)常栖息于潮间带,适应性强,能在无水条件下长时间存活[8-10]。近年来研究发现,双壳类生物在干露胁迫下的生理表现出一定规律:干露时间和干露温度均与存活率成反比[11-13],并且随干露时间的延长,蛤仔体内的活性氧(ROS)含量升高,并超过蛤仔本身的缓解能力,从而引起氧化应激并产生脂质过氧化,造成机体氧化性损伤[8]。研究发现,有PPARα缺陷的小鼠,会显著增加氧化应激并且导致脂质过氧化损伤,这表明PPARα在维持ROS与抗氧化稳态中起重要作用[14]。Ali等[15]对日本群体蛤仔研究表明,干露期间蛤仔主要能量来源是由脂质分解而来的葡萄糖,干露胁迫下,蛤仔停止外源性能量供给,只能通过消耗自身脂肪获取葡萄糖,进而维持生命活动。而Giordano Attianese等[16]对PPAR持激动剂的研究表明,葡萄糖分泌直接受到PPARβ表达的调控。PPARγ则作为脂肪细胞分化及储存的主要调控基因,保持机体内部能量稳态[4]。因此,作为脂质代谢过程中其主要调控作用的基因,PPARα/β/γ在蛤仔机体抗干露胁迫过程中维持机体抗氧化稳态及能量供给至关重要[17]。目前对哺乳动物及鱼类PPARα/β/γ基因表达的研究较为常见[18-21]。本研究中首次在菲律宾蛤仔中鉴定了PPARα、PPARβ和PPARγ 3种PPAR基因,并分析了蛤仔不同组织中PPARα/β/γ的分布情况及两种温度条件下短期干露胁迫后基因的表达变化,探究PPARα/β/γ在抗干露胁迫中的作用,旨在为进一步研究蛤仔在干露条件下脂质代谢调节及脂肪酸合成提供参考依据。
1 材料与方法
1.1 材料
于2017年7月,从大连獐子岛收集一批3龄健康野生菲律宾蛤仔(壳长2.5~3.0 cm),共15 kg,运到大连海洋大学贝类遗传育种重点实验室暂养7 d。暂养期间,持续充氧,保持恒定水温20 ℃,每日投饵换水,待无个体死亡后开始正式试验。
共挑选180枚蛤仔等分为3组:空白对照组、常温(20 ℃)干露组和低温(5 ℃)干露组,每组设3个平行,每个平行30枚蛤仔。空白组为干露0 h的样品,干露组则先擦干蛤仔外壳水分,分别在5 ℃和20 ℃下进行干露胁迫试验,低温组在实验室冰箱内进行,常温组则置于空调房中,调至20 ℃且保证室内温度恒定。在干露处理的0、3、6、12、24、48 h 时,从每个平行随机取3枚蛤仔。取其外套膜、鳃、性腺、消化腺和闭壳肌5个组织样品,所有样品均于超低温冰箱(-80 ℃)中保存待用。
1.2 方法
1.2.1 数据库挖掘、基因鉴定和序列分析 鉴定PPAR基因家族特征,通过NCBI(http://www. ncbi.nlm.nih.gov)选取无脊椎动物PPARα/β/γ蛋白序列,包括太平洋牡蛎Crassostrea gigas、虾夷扇贝Patinopecten yessoensis、美洲牡蛎Crassostrea virginica、福寿螺Pomacea canaliculata、海蜗牛Aplysia californica、紫色球海胆Strongylocentrotus purpuratus、刺参Apostichopus japonicus;选取脊椎动物蛋白序列,包括人Homo sapiens、褐家鼠Rattus norvegicus、非洲爪蟾Xenopus laevis、腔棘鱼Latimeria chalumnae、斑马鱼Danio rerio、虹鳟Oncorhynchus mykiss和大西洋鲑Salmo salar。从蛤仔全基因组中得到蛤仔PPARα/β/γ的编码基因(CDS),使用TBLASTN获得PPARα/β/γ转录组序列集,然后通过比较转录组序列和全基因组序列来进行BLASTN以验证CDS序列[22];通过ORFfinder(https://www.ncbi.nlm.nih.gov/orffinder/)确定PPARα/β/γ的开放阅读框范围,并预测编码的氨基酸序列,预测的氨基酸序列由NCBI的BLASTP非冗余蛋白序列数据库确认;使用多序列比对工具BoxShade(https://embnet.vital-it.ch/software/BOX_form.html)进行序列比对及结构分析;使用Compute pI /Mw工具(http://web.expasy.org/compute_pi/)计算PPARα/β/γ等电点(PI)和分子量;使用Psipred(https://bioinf.cs.uc/ac.uk/psipred/)预测蛤仔PPAR蛋白的二级结构。
1.2.2 系统发育分析 用上述物种与蛤仔的PPARα/β/γ蛋白序列共同建立系统进化树,通过MEGA 7中邻近法Maximum Likelihood构建,重复1000次评估系统进化树。用于同源性比对及系统发育树构建的其他物种PPARα/β/γ氨基酸序列如表1所示。
1.2.3 RNA提取和实时荧光定量PCR分析 用Trizol法从30 mg组织样品中提取总RNA。使用NanoDrop ND-2000C分光光度计(Thermo,美国)测定RNA浓度和纯度,通过琼脂糖凝胶电泳评估RNA的完整性。使用带有gDNA Eraser(TaKaRa,日本)的Prime ScriptTM RT反转录试剂盒合成首链cDNA。将所有cDNA产物稀释至100 ng/μL以用作qPCR模板,置于冰箱(-20 ℃)中长期保存。
使用SsoFastTM EvaGreen©Supermix荧光染料,通过LightCycler 480实时PCR(Roche Diagnostics,Mannheim,Germany)进行PPAR基因家族的相对表达量测定。反应程序如下:94 ℃下预变性5 min;94 ℃下变性30 s,55 ℃下退火30 s,72 ℃下延伸1 min,共进行35个循环。根据实验室前期内参基因筛选,选用β-actin作为内参基因[23]。实时PCR中使用的所有引物均使用Primer Premier 5.0设计,结果如表2所示。
1.2.4 基因表达量分析 根据公式2-ΔΔCt计算得到基因相对表达量。不同组织相对表达量测定选取闭壳肌Ct值作为参照,然后计算各个组别中各组织基因的相对表达量。试验组选取性腺、消化腺和鳃组织,进行不同时间点相对表达量测定,以0 h各组织Ct值作为参照。使用Origin 2017软件制作基因相对表达量柱状图。
表1 用于构建NJ系统进化树的物种
Tab.1 Species in volved in NJ phylogenetic tree

蛋白 protein种类speciesGenBank accession No.人 Homo sapiensNP_001001928.1褐家鼠 Rattus norvegicusNP_037328.1非洲爪蟾 Xenopus laevisNP_001083282.1斑马鱼 Danio rerioNP_001096037.1PPARα大西洋鲑 Salmo salarNP_001117032.1长牡蛎 Crassostrea gigasXP_011412920.1虾夷扇贝 Patinopecten yessoensisXP_021370914.1紫色球海胆 Strongylocentrotus purpuratusXP_781750.2刺参 Apostichopus japonicusPIK48830.1人 Homo sapiensNP_001165289.1褐家鼠 Rattus norvegicusNP_037273.2非洲爪蟾 Xenopus laevisNP_001081310.1斑马鱼 Danio rerioNP_571543.1大西洋鲑 Salmo salarXP_013987341.1PPARβ腔棘鱼 Latimeria chalumnaeXP_005988478.1虾夷扇贝 Patinopecten yessoensisXP_021371614.1长牡蛎 Crassostrea gigasXP_011420515.1美洲牡蛎 Crassostrea virginicaXP_022301760.1福寿螺 Pomacea canaliculataXP_025110669.1人 Homo sapiensNP_619726.2褐家鼠 Rattus norvegicusNP_037256.1斑马鱼 Danio rerioNP_571542.1大西洋鲑 Salmo salarNP_001117018.1虹鳟 Oncorhynchus mykissXP_021462299.1PPARγ海蜗牛 Aplysia californicaXP_012946006.1紫色球海胆 Strongylocentrotus purpuratusXP_011673603.1虾夷扇贝 Patinopecten yessoensisXP_021355978.1长牡蛎 Crassostrea gigasXP_011455071.1美洲牡蛎 Crassostrea virginicaXP_022288354.1福寿螺 Pomacea canaliculataXP_025108555.1)
1.3 数据处理
试验数据以平均值±标准差(mean±S.D.)表示,采用SPSS 17.0软件进行单因素方差分析(One-way ANOVA),用Duncan法进行组间多重比较,显著性水平设为0.05。
表2 试验中所用的引物序列
Tab.2 Summary of primers used in the experiment

基因 gene序列 sequence (5′-3′)PPARα-FCCTTTGCCTCATACTGCGTCTCPPARα-RTTGGTCGTCTGCCCATCTTTACPPARβ-FGGGTTTCTTCAGACGAACATTAPPARβ-RAACTCTTCTTTGTCCGCCATTAPPARγ-FACCATGACAGAGCCGGTGAATAPPARγ-RGCTCGCTTCTTGTCCTCGGTATβ-actin-FCTCCCTTGAGAAGAGCTACGAβ-actin-RGATACCAGCAGATTCCATACCC
2 结果与分析
2.1 PPARα/β/γ序列鉴定分析
根据无脊椎动物PPAR基因家族命名方式,将菲律宾蛤仔转录组数据库中鉴定的PPAR基因分别命名为PPARα、PPARβ和PPARγ。PPARα、PPARβ和PPARγ的开放阅读框长度分别为912、1644、2124 bp,分别编码239、547和707个氨基酸,预测相对分子量范围为26 740~80 620,等电点为5.68~9.46(表3)。PPARα有5个外显子,PPARβ有9个外显子,PPARγ有11个外显子(图1)。
2.2 PPARα/β/γ蛋白结构域及二级结构分析
对菲律宾蛤仔PPARα/β/γ蛋白多序列比对发现,PPARα/β/γ配基结合区(LBD)较为保守,DNA结合区(DBD)保守性弱于LBD,而A、B区(调节区)保守性较差(图2)。预测PPARα/β/γ蛋白的二级结构中,PPARα、PPARβ和PPARγ分别有128、201和206个α螺旋,分别有10、15和27个β折叠,分别有108、331和474线圈数(图3)。
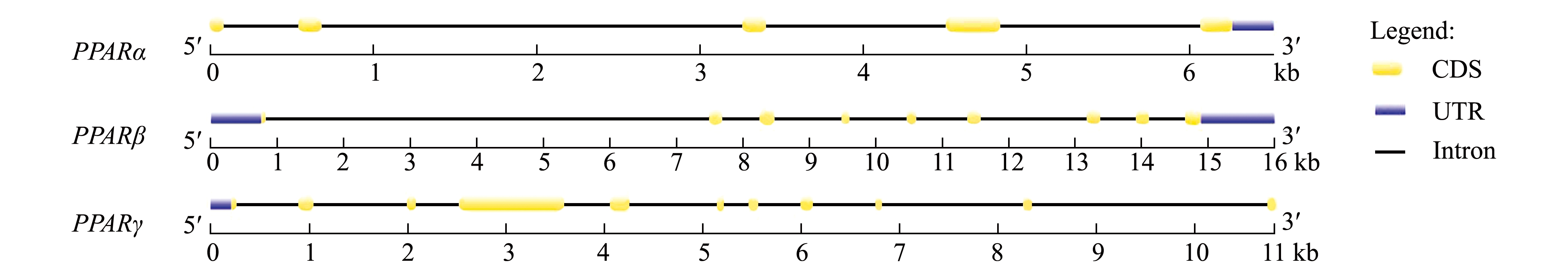
注:深色区域表示3′端和5′端;浅色区域表示外显子,横线表示内含子
Note: The dark color boxes indicate the 3′UTRs and the 5′UTRs. The light color indicate the exons. The horizontal bars indicate the introns
图1 菲律宾蛤仔PPARα/β/γ基因结构图
Fig.1 Structure of PPAR genes in Manila clam Ruditapes philippinarum
表3 菲律宾蛤仔PPARα/β/γ基因特征
Tab.3 Sequence features of PPARα/β/γ from Manila clam Ruditapes philippinarum
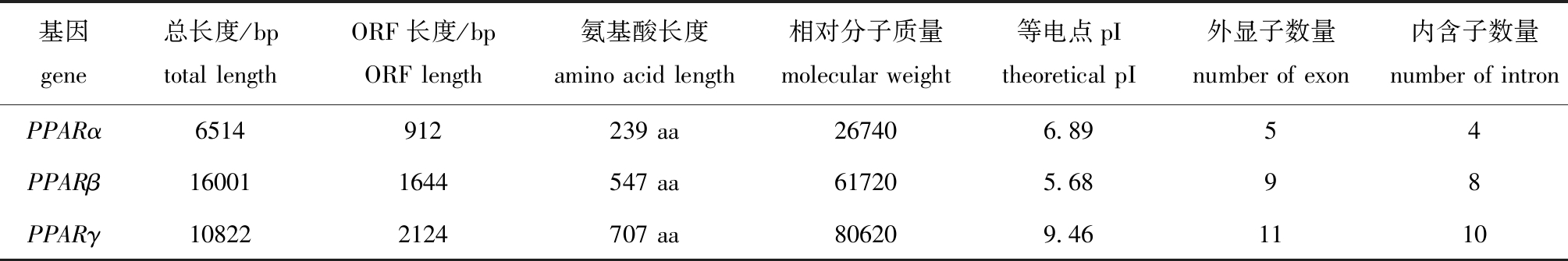
基因gene 总长度/bptotal lengthORF长度/bpORF length氨基酸长度amino acid length相对分子质量molecular weight等电点pItheoretical pI外显子数量number of exon内含子数量number of intronPPARα6514912239 aa267406.8954PPARβ160011644547 aa617205.6898PPARγ108222124707 aa806209.461110

图2 菲律宾蛤仔与其他物种PPARα/β/γ蛋白多序列比对及功能域
Fig.2 Multiplesequence alignments and functional domains of PPARα/β/γ protein between Manila clam Ruditapes philippinarum and other species

注:黑色区域代表保守氨基酸残基,灰色阴影区代表相似的氨基酸残基,不同结构域之间用星号隔开;第一段结构域为DNA结合区(DBD),第二段结构域为配基结合区(LBD)
Note:Conserved amino acid residues are shaded in black.The gray-shaded regions represent similar amino acid residues.The different structure domains are separated by asterisks; the first domain is DNA binding domain (DBD), and the second domain is ligand binding domain (LBD)
续图2 菲律宾蛤仔与其他物种PPARα/β/γ蛋白多序列比对及功能域
Cont.Fig.2 Multiplesequence alignments and functional domains of PPARα/β/γ protein between Manila clam Ruditapes philippinarum and other species
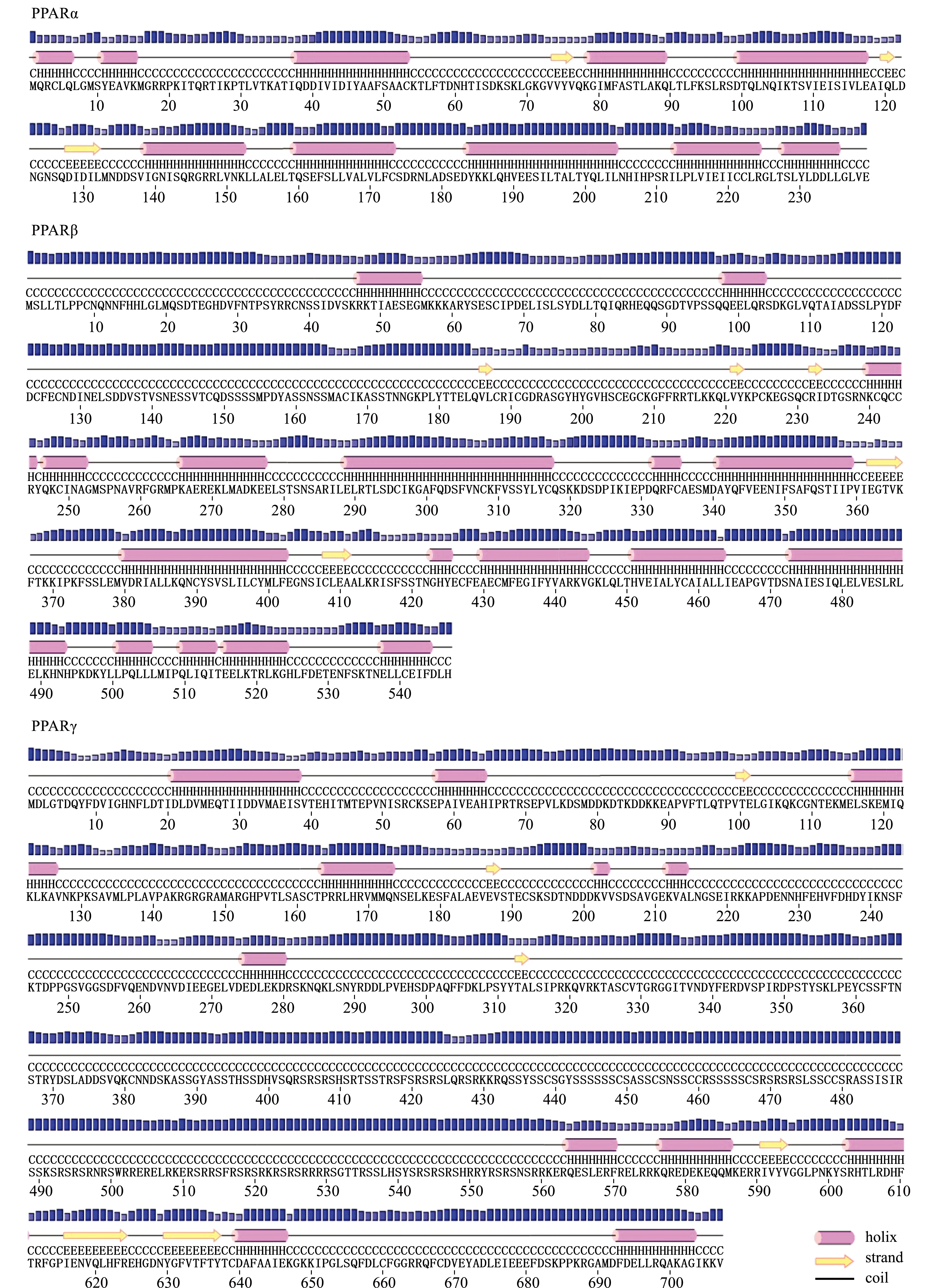
图3 菲律宾蛤仔PPARα/β/γ蛋白二级结构预测
Fig.3 Predicted secondary structure of PPARα/β/γ in Manila clam Ruditapes philippinarum
2.3 菲律宾蛤仔PPARα/β/γ系统进化树分析
根据获得的蛤仔PPARα/β/γ氨基酸序列,利用MEGA 7构建系统发生树。从图4可以看出,脊椎动物与无脊椎动物的PPARα/β/γ蛋白聚成两类;在无脊椎动物中,蛤仔PPARα和PPARγ都与虾夷扇贝亲缘关系较近,蛤仔PPARβ与福寿螺亲缘关系较近,首先聚类成一支,美洲牡蛎和太平洋牡蛎聚为一类,最后再与虾夷扇贝聚类。
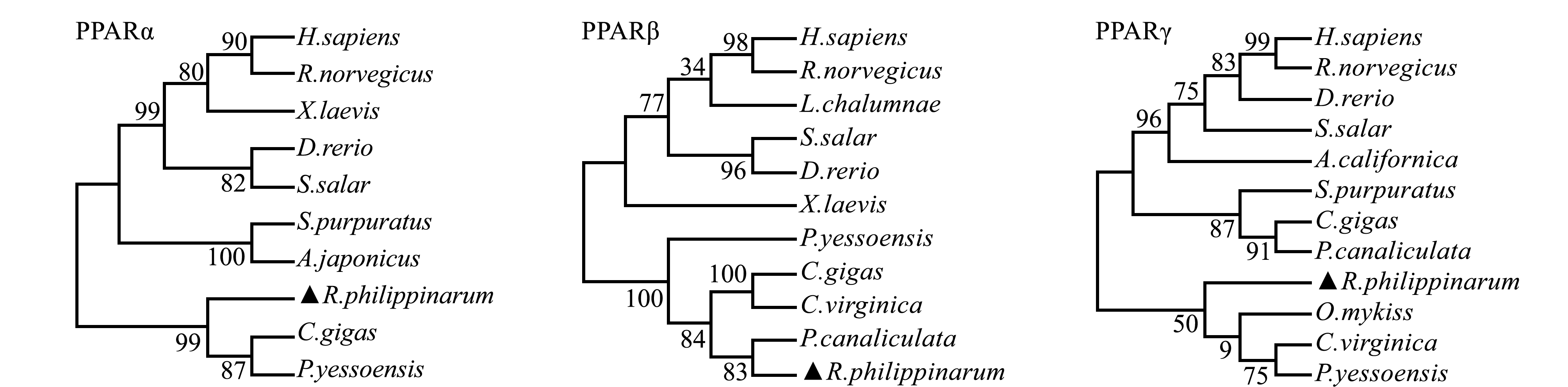
图4 菲律宾蛤仔与其他物种PPARα/β/γ氨基酸序列系统发育树聚类分析
Fig.4 Cluster analysis of phylogenetic tree of PPARα/β/γ amino acid sequence between Manila clam Ruditapes philippinarum and other species
2.4 菲律宾蛤仔PPARα/β/γ荧光定量PCR组织表达
对菲律宾蛤仔PPARα、PPARβ和PPARγ基因进行5种组织(性腺、消化腺、鳃、外套膜、闭壳肌)荧光定量PCR,组织表达结果如图5所示。以闭壳肌中PPARα/β/γ相对表达量为参照,可以看出3种基因在各个组织中均有不同程度的表达。其中,性腺及外套膜中PPARα表达量很低,仅为闭壳肌的0.05、0.009倍,鳃中PPARα表达量最高,为闭壳肌的1.7倍;消化腺和鳃中PPARβ表达量较高,分别为闭壳肌的4.30、2.53倍;PPARγ在消化腺中表达量最高,为闭壳肌的5.52倍,在性腺中的表达量略低于消化腺;PPARα、PPARβ和PPARγ在外套膜中表达量都很低(图5)。
2.5 干露胁迫下菲律宾蛤仔PPARα组织表达变化
在性腺组织中,与空白组(0 h)相比,3 h时低温干露组和常温干露组PPARα表达量均显著升高(P<0.05),分别为 0 h的4.9倍和3.2倍,但两组间无显著性差异(P>0.05);6 h时常温组表达量显著高于低温组(P<0.05),24 h时低温组显著高于常温组(P<0.05),但这两个时间点的表达量与空白组相比并无显著性差异(P>0.05);48 h时常温组相对表达量达到峰值,与组内和组间相比均有显著性差异(P<0.05),为0 h的10.3倍,相比之下,48 h时低温组仅为0 h 的0.8倍(图6-A)。整体来看,低温抑制PPARα在性腺中的表达。
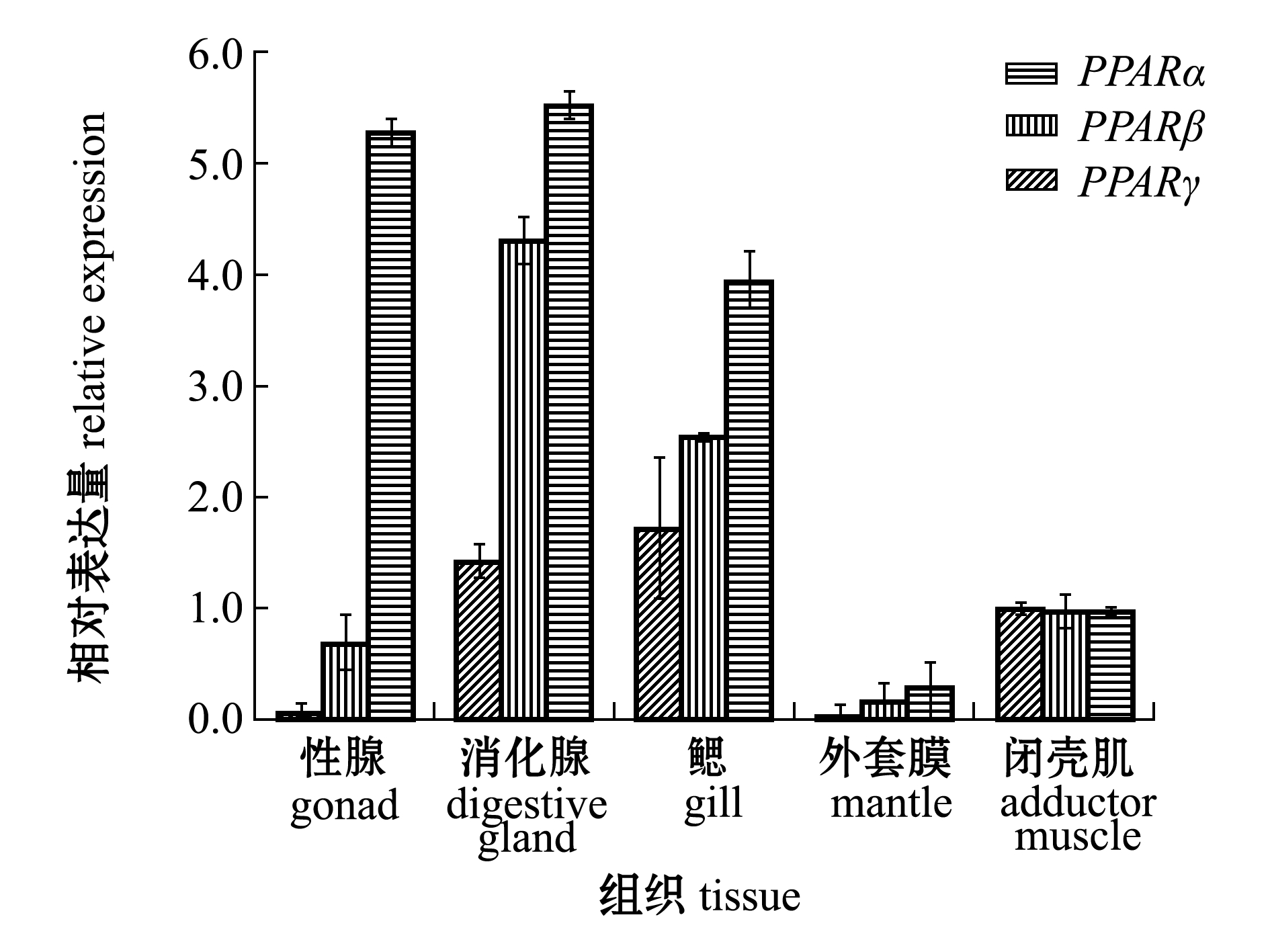
图5 菲律宾蛤仔健康组织中PPARα/β/γ相对表达量
Fig.5 Relative expression levels of PPARα/β/γ in health tissues of Manila clam Ruditapes philippinarum
在消化腺组织中,3 h时低温组和常温组PPARα表达均显著低于空白组(0 h)(P<0.05),分别为0 h的0.1倍和0.6倍,且低温组显著高于常温组 (P<0.05);6、24、48 h时,低温组表达量均显著低于常温组(P<0.05)(图6-B)。整体来看,干露对PPARα在消化腺组织中的表达有抑制作用,其中低温对其表达的抑制作用更明显。
在鳃组织中,低温组除在6 h时PPARα表达量显著高于0 h(P<0.05)之外并达到峰值,在其余时间点均显著低于0 h(P<0.05);常温组PPARα表达量则呈逐渐下降趋势;12 h时低温组表达量显著低于常温组(P<0.05),24 h时低温组显著高于常温组(P<0.05)(图6-C)。整体来看,干露对PPARα在鳃组织中表达表现出抑制作用,但温度变化对其影响不大。
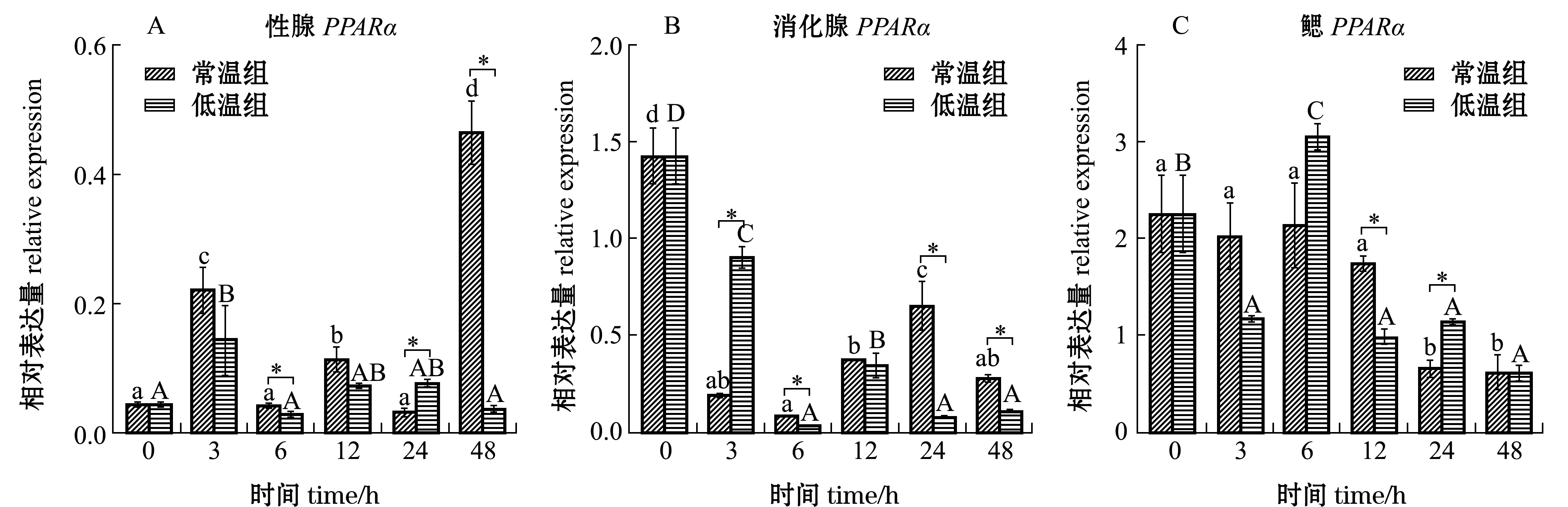
注:标有不同小写字母者表示常温组内有显著差异性(P<0.05);标有不同大写字母者表示低温组内有显著差异性(P<0.05); * 表示低温组与常温组间有显著差异性(P<0.05),下同
Note: The means with different letters are significant differences in normal temperature group; the means with different capital letters are significant differences in low temperature group; * denotes significant difference between low temperature group and control group,et sequentia
图6 干露胁迫下菲律宾蛤仔性腺、消化腺和鳃组织中PPARα的相对表达水平
Fig.6 Relative expression levels of PPARα in gonad,intestine and gill tissues of Manila clam Ruditapes philippinarum under air exposure
2.6 干露胁迫下菲律宾蛤仔PPARβ组织表达变化
在性腺组织中,3 h时低温组和常温组表达量均显著高于空白组(0 h)(P<0.05),且低温组PPARβ的表达量显著低于常温组(P<0.05),常温组表达量在此时达到峰值,为0 h的4.2倍;24 h时低温组表达量较0 h显著升高,达2.9倍(P<0.05)且达到峰值,并显著高于常温组(P<0.05);48 h时低温组表达量骤降为0 h的0.08倍,且显著低于常温组(P<0.05)(图7-A)。整体来看,常温干露会诱导性腺中PPARβ表达升高,低温组表达不规律。
在消化腺组织中,3 h时低温组PPARβ表达显著低于常温组(P<0.05),但两者与0 h相比无显著差别(P>0.05);24 h时常温组PPARβ表达较0 h显著升高10.5倍(P<0.05),并显著高于低温组(P<0.05)(图7-B)。整体来看,常温干露对消化腺组织PPARβ表达有一定诱导作用,低温对其表达无明显抑制作用。
在鳃组织中,低温组PPARβ在3、12、48 h时均显著低于常温组(P<0.05),而在6、24 h时较常温组显著升高(P<0.05);低温组在6 h时表达量为0 h的3.7倍,且达到峰值,常温组在48 h时表达量为0 h时的7.4倍,且达到峰值(图7-C)。整体来看,干露对鳃组织中PPARβ表达起诱导作用,常温干露对其诱导作用更加明显。
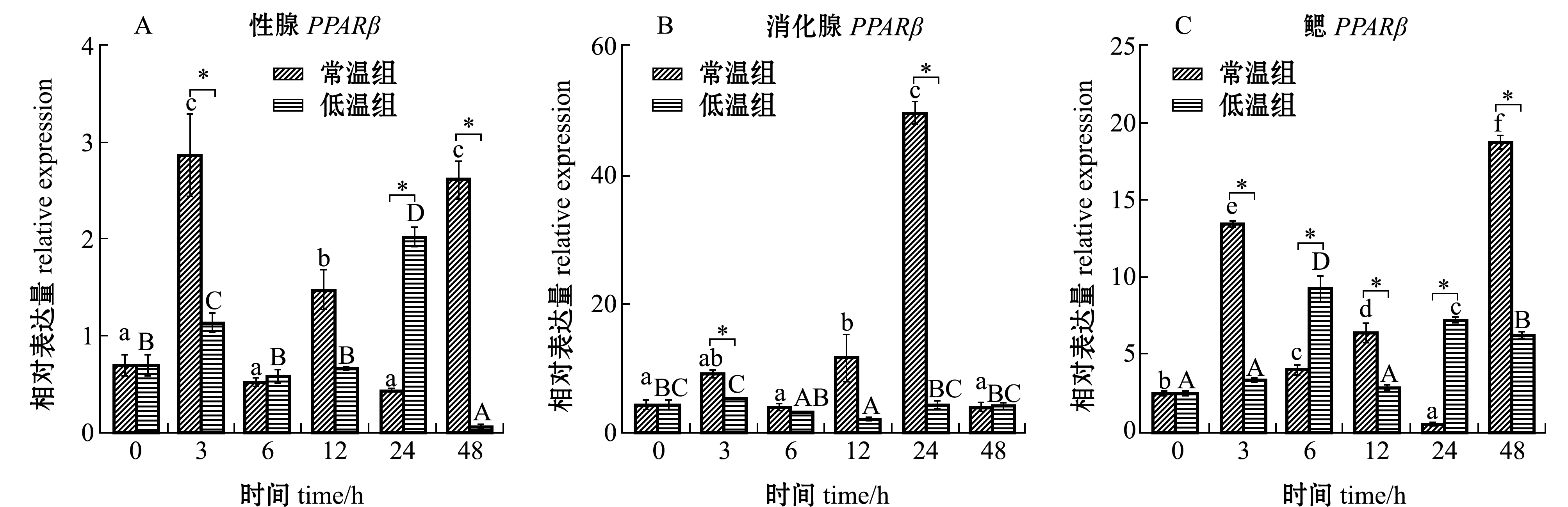
图7 干露胁迫下菲律宾蛤仔性腺、消化腺和鳃组织中PPARβ的相对表达水平
Fig.7 Relative expression levels of PPARβ in gonad,intestine and gill tissues of Manila clam Ruditapes philippinarum under air exposure
2.7 干露胁迫下菲律宾蛤仔PPARγ组织表达变化
在性腺组织中,除24 h外,低温组PPARγ表达量均显著低于常温组(P<0.05);低温组表达量呈显著下降趋势(P<0.05),常温组呈先下降后上升趋势;在48 h时,低温组PPARγ表达量仅为0 h的0.04倍,此时常温组表达量达峰值,较0 h显著升高5.3倍(P<0.05)(图8-A)。整体来看,常温干露在前24 h对PPARγ有一定抑制作用,低温干露则对其表达抑制更明显,并超过48 h。
消化腺组织中,除3 h外,低温组PPARγ表达均显著低于常温组(P<0.05);在24 h时常温组表达量达峰值,为0 h 的5.1倍,低温组呈逐渐下降趋势(图8-B)。整体来看,低温对消化腺组织中PPARγ表达抑制作用明显。
在鳃组织中,3 h时低温组和常温组PPARγ表达量均较空白组(0 h)显著下降(P<0.05),并且低温组表达量显著低于常温组(P<0.05),分别为0 h的0.1倍和0.7倍;常温组表达量在前24 h呈先下降后升高趋势,并在24 h时达到峰值;48 h时低温组表达量显著高于常温组(P<0.05)(图8-C)。整体来看,干露对鳃组织中PPARγ在前12 h有一定抑制作用,超过12 h后抑制作用减弱。

图8 干露胁迫下菲律宾蛤仔性腺、消化腺和鳃组织中PPARγ的相对表达水平
Fig.8 Relative expression levels of PPARγ in gonad,intestine and gill tissues of Manila clam Ruditapes philippinarum under air exposure
3 讨论
3.1 菲律宾蛤仔PPARα/β/γ序列鉴定及进化分析
本研究中通过对菲律宾蛤仔全基因组测序数据库获得的过氧化物酶体增殖物激活受体基因家族(PPARα/β/γ)序列分析表明,菲律宾蛤仔PPARα/β/γ编码的蛋白的LBD结构域进化上高度保守,激素受体配体结合域对配体结合起反应,导致受体的构象改变以诱导响应,从而作为开启转录活性的分子开关,此结构域占据氨基酸数量最多,含有7个螺旋结构,共同参与配体结合。通过进化树分析表明,菲律宾蛤仔PPARα/β/γ都与软体动物聚到一类,尤其是与虾夷扇贝亲缘关系较近。
3.2 菲律宾蛤仔PPARα/β/γ的组织表达分析
PPARα/β/γ在菲律宾蛤仔性腺、消化腺、鳃、外套膜和闭壳肌组织中均有表达,其中PPARα在性腺中表达水平很低,这与对斑马鱼成鱼[18]的研究结果一致,但与对虹鳟[19]的研究结果不同;PPARβ和PPARγ主要在性腺、消化腺和鳃中高表达,这与已报道的长鳍蓝子鱼[20]、草鱼[21]类似,其中PPARγ相对表达量是3种基因中最高的,这可能是由于PPARγ具有高度脂肪专一性[3]。
关于干露胁迫对贝类的影响,近年来已有不少报道,但多数集中在研究稚贝生存指标,如太平洋牡蛎[12]、海湾扇贝[13]等,或是成贝在干露胁迫下的各项生理指标和一些免疫相关基因表达如虾夷扇贝[24]、菲律宾蛤仔[8,11]。王文文[11]研究表明,干露胁迫会导致蛤仔体内产生脂质过氧化反应。脂质由脂肪酸和甘油三酸组成,生物摄取的脂肪酸当中一部分能量转化为ATP,其余通过热量损失掉。脂肪酸被摄取之后,首先转化为甘油三酯的形式,储存在一种叫作脂滴(LD)的细胞器中,作为备用能量以防外界脂肪酸供应的不稳定。过氧化物酶体增殖物激活受体(PPARα/β/γ)是一组在LD相关蛋白的转录调节及LD重塑过程中参与脂质生成和脂解酶的调节中起关键作用的转录因子。干露胁迫所引起的蛤仔机体脂质过氧化反应,直接导致了机体活性氧(ROS)含量升高,使蛤仔机体受到氧化性损伤,而PPARα/β/γ是抗氧化及脂质代谢调控的主要基因[14,16]。本研究中将菲律宾蛤仔置于两种温度干露条件下,发现其体内的脂质代谢相关基因PPARα/β/γ均可以不同程度地响应干露胁迫。干露胁迫阻断了蛤仔外源性脂肪酸的摄取,同时有证据表明[4],受PPARα调节的抗氧化酶如SOD和CAT等表达下降,导致蛤仔机体PPARα整体表达降低;低温条件下,PPARα表达更低,也说明了低温对抗氧化酶表达抑制作用更强,这与王文文[11]的研究结果一致。
由于PPARβ在大多数细胞和组织中表达,导致鉴定PPARβ的组织特异性功能比较困难,目前了解到PPARβ的主要功能主要是刺激脂肪酸氧化分解[16,25-26],产生葡萄糖,以维持蛤仔干露期间生命所需[16]。在常温干露胁迫下,3种组织中的PPARβ均上调明显,这也表明蛤仔各组织通过脂肪酸的氧化,以维持机体能量代谢平衡,这与日本蛤仔干露后体内葡萄糖含量明显升高的结果一致[15],同时可以看出,低温对PPARβ表达有一定抑制作用,这也说明低温条件下蛤仔维持机体代谢平衡所需的能量较少,这可能是由于低温降低蛤仔耗氧率[27],使机体代谢水平下降。
PPARγ是葡萄糖稳态和脂肪生成的关键调节因子[28-29],在脂肪细胞分化过程中,PPARγ可调控多种有关脂类代谢的基因表达[30-31]。根据本试验结果,发现干露胁迫下3种组织中PPARγ表达,在前12 h均有不同程度下调,这可能是因为在切断外源脂肪供给条件下,PPARγ无法指导细胞内酯类合成,而12 h后,为了维持机体能量代谢平衡,则会首先从消化腺获取脂肪酸,也或许能解释常温干露条件下,PPARβ和PPARγ显著上调的原因。而低温条件下,即使超过12 h,PPARγ依旧保持相对稳定的表达,这也说明低温有助于维持蛤仔机体内部脂质代谢平衡。此外,常温干露胁迫使性腺组织中3种PPAR基因在48 h时均显著上调,并显著高于低温组,其原因还有待进一步研究。
[1] Tyagi S,Gupta P,Saini A S,et al.The peroxisome proliferator-activated receptor:a family of nuclear receptors role in various diseases[J].Journal of Advanced Pharmaceutical Technology & Research,2011,2(4):236-240.
[2] 张乾勇.PPAR的结构与功能及其生物学作用[J].国外医学卫生学分册,2000,27(5):284-288.
[3] 杨谷良,潘敏雄,向福,等.PPARγ调控脂肪细胞增殖和分化机理研究进展[J].食品科学,2017,38(3):254-260.
[4] 杨亚维,袁杰.PPARα抗氧化应激作用的研究进展[J].东南大学学报:医学版,2016,35(1):147-150.
[5] He Shan,Liang Xufang,Qu Chunmei,et al.Identification,organ expression and ligand-dependent expression levels of peroxisome proliferator activated receptors in grass carp(Ctenopharyngodon idella)[J].Comparative Biochemistry and Physiology Part C:Toxicology & Pharmacology,2012,155(2):381-388.
[6] 陈娜,吴英良.PPARδ的结构及其生物学功能与疾病[J].中国药理学通报,2006,22(9):1035-1038.
[7] 陈永熙,王伟铭,周同,等.PPAR-γ作用及其相关信号转导途径[J].细胞生物学杂志,2006,28(3):382-386.
[8] Yin Xuwang,Chen Peng,Chen Hai,et al.Physiological performance of the intertidal Manila clam (Ruditapes philippinarum) to long-term daily rhythms of air exposure[J].Scientific Reports,2017,DOI:https://doi.org/10.1038/srep41648.
[9] 闫喜武.菲律宾蛤仔养殖生物学、养殖技术与品种选育[D].青岛:中国科学院研究生院(海洋研究所),2005.
[10] 刘慧慧,周晏琳,张晴,等.菲律宾蛤仔捕后干露处置对其复水湿藏稳定性的影响[J].大连海洋大学学报,2018,33(2):244-250.
[11] 王文文.干露胁迫对菲律宾蛤仔免疫指标和基因表达的影响[D].大连:大连海洋大学,2016.
[12] 于瑞海,王昭萍,孔令锋,等.不同发育期的太平洋牡蛎在不同干露状态下的成活率研究[J].中国海洋大学学报,2006,36(4):617-620.
[13] 于瑞海,辛荣,赵强,等.海湾扇贝不同发育阶段耐干露的研究[J].海洋科学,2007,31(6):6-9.
[14] Guellich A,Damy T,Lecarpentier Y,et al.Role of oxidative stress in cardiac dysfunction of PPAR -/- mice[J].American Journal of Physiology-Heart and Circulatory Physiology,2007,293(1):H93-H102.
[15] Ali F,Nakamura K.Metabolic characteristics of the Japanese clam Ruditapes philippinarum (Adams & Reeve) during aerial exposure[J].Aquaculture Research,2000,31(2):157-165.
[16] Giordano Attianese G M P,Desvergne B.Integrative and systemic approaches for evaluating PPARβ/δ (PPARD) function[J].Nuclear Receptor Signaling,2015,13:1-32.
[17] Grimaldi P A.The roles of PPARs in adipocyte differentiation[J].Progress in Lipid Research,2001,40(4):269-281.
[18] Ibabe A,Bilbao E,Cajaraville M P.Expression of peroxisome proliferator-activated receptors in zebrafish (Danio rerio) depending on gender and developmental stage[J].Histochemistry and Cell Biology,2005,123(1):75-87.
[19] 贾成霞,张照斌,张清靖,等.虹鳟PPARα基因克隆、序列分析及其组织表达分布[J].中国水产科学,2012,19(4):707-714.
[20] You Cuihong,Jiang Danli,Zhang Qinghao,et al.Cloning and expression characterization of peroxisome proliferator-activated receptors (PPARs) with their agonists,dietary lipids,and ambient salinity in rabbitfish Siganus canaliculatus[J].Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology,2017,206:54-64.
[21] 林亚秋,吉红,郑玉才.草鱼PPARα和PPARγ基因的克隆与组织表达差异[J].水产科学,2011,30(2):94-97.
[22] Altschul S F,Gish W,Miller W,et al.Basic local alignment search tool[J].Journal of Molecular Biology,1990,215(3):403-410.
[23] 牟政强,闫路路,王化敏,等.菲律宾蛤仔不同发育时期及成体不同组织中内参基因筛选[J].水产学报,2018,42(5):663-672.
[24] 刘忠颖,刘卫东,李军,等.温度和规格对虾夷扇贝干露耐受性的影响[J].水产科学,2018,37(4):464-468.
[25] Gilde A J,van der Lee K A J M,Willemsen P H M,et al.Peroxisome proliferator-activated receptor (PPAR) α and PPARβ/δ,but not PPARγ,modulate the expression of genes involved in cardiac lipid metabolism[J].Circulation Research,2003,92(5):518-524.
[26] Luquet S,Lopez-Soriano J,Holst D,et al.Peroxisome proliferator-activated receptor δ controls muscle development and oxidative capability[J].FASEB Journal:Official Publication of the Federation of American Societies for Experimental Biology,2003,17(15):2299-2301.
[27] 林元烧,沈国英,张华.菲律宾蛤仔耗氧率的研究[J].厦门大学学报:自然科学版,1996,35(3):407-411.
[28] Marx N,Hombach V.Peroxisome proliferator-activated receptors (PPARs) in der Gefäßw and-neue Regulatoren der Genexpression in vaskulären Zellen[J].Zeitschriftfü Kardiologie,2001,90(7):470-477.
[29] Barak Y,Nelson M C,Ong E S,et al.PPARγ is required for placental,cardiac,and adipose tissue development[J].Molecular Cell,1999,4(4):585-595.
[30] Momose H,Kaminuma T,Tanaka Y,et al.Analysis of gene regulation network by nuclear receptor PPAR[J].Genome Informatics,2003,14:364-365.
[31] 徐世文,樱桃,李术.PPAR-γ功能的研究进展[J].东北农业大学学报,2011,42(9):1-6.