近年来,研究人员发现,人类基因组中有超过2/3是重复DNA序列,其中大部分是转座元件[1]。长散在核重复元件(Long interspersed nuclear elements,LINEs)是可以自主转座的一类逆转录转座子,来源于RNA聚合酶Ⅱ的转录产物,是散分布在哺乳动物基因组中的一类重复序列。LINE1(以下简称为L1)是唯一活跃的自主移动的DNA,占人类基因组的17%,包括两个主要开放阅读框(ORF1、ORF2),其中,ORF1编码核酸结合蛋白,ORF2编码核酸内切酶和逆转录酶[2]。最近有研究发现,在L1的5′UTR包括一个反向的ORF0,可以增强L1的流动性[3]。L1的扩增通过“复制和粘贴”的方式,包括了几个主要的过程,即转录、转运至细胞质、翻译、形成核酸蛋白复合体,返回至细胞核内、靶位点逆转录转座和插入新的位点[4-5]。
转座子常被称为“基因组寄生虫” 或者“自私的基因”,其能够导致基因组的插入、删除[6]。由于L1逆转座产生的基因组插入占哺乳动物DNA的50%,曾有研究表明,L1对哺乳动物的基因组结构和功能有持续性的影响。研究发现,L1与疾病发生相关,多见于肿瘤组织中,如前列腺癌、胃癌等肿瘤组织L1的去甲基化[7-10]。更多关于L1功能活性的调控、L1复制的生物化学过程和L1与宿主间的相互作用均是未知的,这对于理解哺乳动物的基因功能有较大阻碍。
RNA-seq技术是通过转录组测序建库的试验方法,在单核苷酸水平对所有物种的转录活动进行检测,可为试验提供更为全面的转录信息,并提供准确的数字化表达谱检测[11]。本研究中,基于RNA-seq技术分析了L1在细胞中转录水平的变化,探讨了L1在哺乳动物基因组中的功能,以期为进一步研究L1的基因功能提供理论基础。
1 材料与方法
1.1 材料
Hela细胞购自ATCC公司,质粒pc-L1-1FH购自Invitrogen公司(含FLAG标签),空载质粒为pc-1FH(无L1)(图1-A)。
高糖培养基(DMEM/High Glucose)购自Hyclone公司;Penicillin-Streptomycin Solution (ps)购自生物工程(上海)股份有限公司;胎牛血清(fetal bovis serum,FBS)购自Gibco公司;胰酶(不含EDTA)购自Life Technology公司;转染试剂(TurbofectTM Transfection Reagent)购自Thermo Scientific公司;嘌呤霉素(puromycin)购自生物工程(上海)股份有限公司;Trizol购自Life Technology公司;anti-Flag抗体和RIPA buffer蛋白裂解液均购自Sigma公司;PrimeScriptTM RT reagent Kit with gDNA Eraser购自TaKaRa公司。
1.2 方法
1.2.1 Hela细胞培养 Hela细胞培养基为89% High-Glucose、10% FBS和1%的ps,混合后使用。将细胞置于37 ℃、湿度95%、CO2浓度为5%的环境中,在无菌条件下进行培养。
1.2.2 细胞转染 将状态较好的Hela细胞进行传代,接种于100 mm的培养皿中。加入6 mL高糖完全培养基进行培养,并保证在12 h后细胞的混合度达到60%(此时细胞生长呈现指数形式)。分别加入6 μg的pc-L1-1FH和pc-1FH空载质粒于600 μL的DMEM高糖空白培养基中,混匀后加入15 μL的TurboFect转染试剂,快速、小心地混匀,室温下反应15~30 min,将混合物加入细胞中,手动混匀培养基,再置于 37 ℃条件下培养24 h。两组试验分别命名为37-L1、37-NC。
1.2.3 转染L1后的蛋白表达和定位 清洗转染3 d的细胞,用RIPA buffer裂解细胞后,提取蛋白,用anti-FLAG抗体进行Western blot 检测L1-ORF1蛋白的表达(ORF1p)。
利用anti-FLAG抗体,对转染3 d的Hela细胞进行免疫荧光表达试验,检测ORF1p的表达位置。
1.2.4 转录组测序 用5 μg/mL的嘌呤霉素(puromycin)筛选30 d获得稳定遗传的细胞系。每组细胞分别提取RNA,进行转录组测序。简要步骤为:分别提取分选的37-L1、37-NC细胞总RNA,使用DNase消化DNA 后富集mRNA,使用VAHTSTM Stranded mRNA-seq Library Prep Kit for Illumina试剂盒建库,在Illumina HiSeqTM 2000测序仪进行测序,上样量为50 ng。
1.2.5 生物信息学分析 使用Fastqc检查原始数据即raw data的质量,用Trimmomatic去除接头,过滤raw data后得到clean data。
利用HISAT软件将clean data比对到人基因组GRCh38.p12上面,获取在参考基因组上的位置信息。使用StringTie软件对基因组进行组装(merge),通过FPKM(Reads Per Kilobase of exon model per Million mapped reads)法对基因进行均一化处理。
1.3 数据处理
使用DEseq2软件包对结果进行基因差异表达分析。在本试验中,将37-NC组作为空载对照,与37-L1组(37 ℃常温处理试验组)进行比较,差异倍数大于2且P<0.05时,说明有显著性差异。使用Cluster Profile软件包对差异表达基因进行GO注释和KEGG通路富集分析,P<0.05时表示富集显著。使用Ggplot 2和Gheatmp软件作图。
2 结果与分析
2.1 L1在Hela细胞中的表达与定位
首先,对构建的pc-L1-1FH表达载体进行检测。pc-L1-1FH表达载体包含ORF1和ORF2序列及8个氨基酸的FLAG标签序列(图1-A),ORF1和ORF2分别转录翻译表达对应的蛋白ORF1p和ORF2p,通过FLAG标签的表达检测ORF1p的表达。本研究中,将L1表达载体转染至Hela细胞中,收集转染3 d的细胞,进行Western blot 和免疫荧光验证。预测ORF1p蛋白的相对分子质量为40 000,本试验中通过Western blot检测得到的条带分子质量也为41 000(含Flag)(图1-B),符合预期的表达结果。这说明构建的pc-L1-1FH质粒可以在细胞中正确表达。
本试验中利用免疫荧光试验,对L1在细胞中的表达位置进行定位,对照组转染的为空载质粒。图1中蓝色荧光是DAPI染的细胞核,结果显示,在细胞核周围的细胞质中存在ORF1p蛋白,且细胞核中的表达量较高,这可能是由于部分ORF1p可以结合到自己的 mRNA上,与 ORF2p形成核糖核酸复合物(ribonucleic acid particles)后一起转运回到细胞核中(图1-C、图1-D)。这说明L1可以在Hela细胞中正确表达,这为后面的试验提供了依据。
2.2 转录组测序及质量评估
为了探究L1在Hela细胞中的作用机制,取转染L1的Hela细胞稳定细胞系和对照组细胞,分别提取RNA。
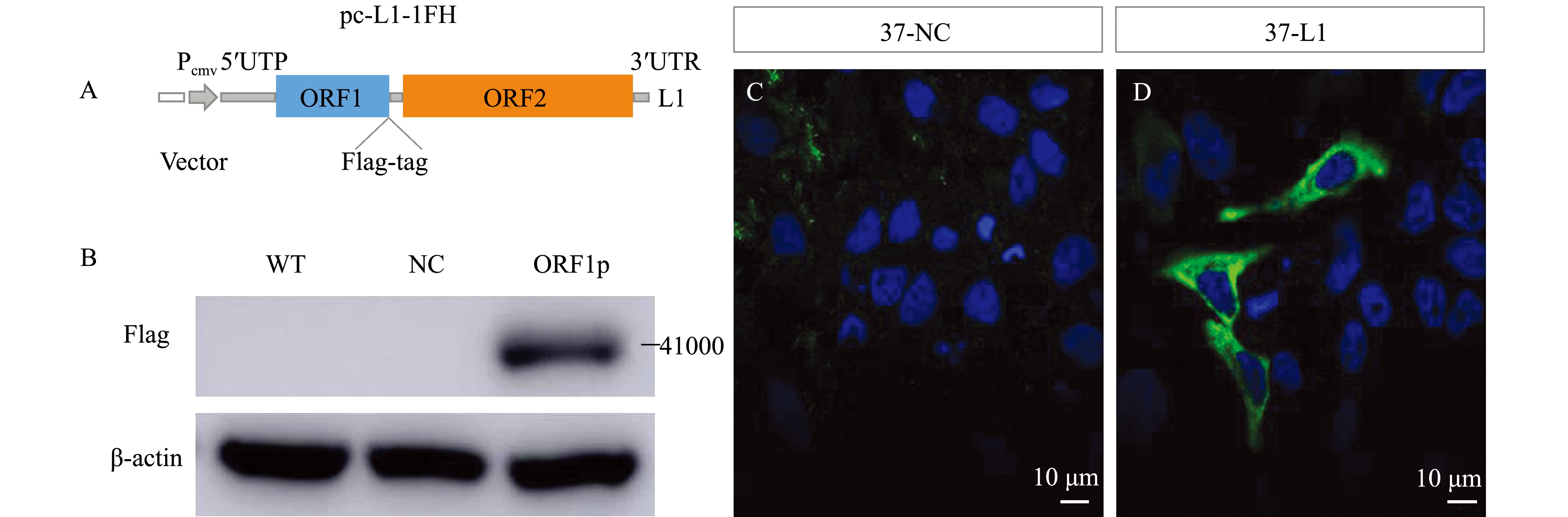
注:A为L1表达载体简图,含有Flag标签;B为Hela细胞转染人类L1的ORF1p表达,利用Flag抗体;C、D为免疫荧光反应,C为对照组细胞,D为转染人类L1细胞;蓝色为DAPI染的核,绿色为anti-FLAG
Note:A,Schematic diagrams depicting the L1 expression plasmid with Flag-tag;B,Western blot showing ORF1p production in Hela cells transfected with human L1; C and D, immunofluorescence reaction; C,Control Hela cells; D,Overexpression of L1 in Hela cells. Nuclei stained with DAPI shown in blue and anti-FLAG is shown in green
图1 L1在Hela细胞中的表达及定位
Fig.1 Expression and location of L1 in Hela cells
采用 Illumina HiSeqTM 2000 测序共获得36 497 978条原始读序,经过质量预处理后最终获得平均长度为106 bp的有效读序33 589 851条(表1)。各测序样品的比对效率均在 93.69%以上,比对效果较好,所选的参考基因组组装能满足信息分析要求,比对数据可用于后续的功能注释及分析。
2.3 差异表达基因筛选
利用 DESeq进行样品组间的差异表达分析,根据基因的表达量,取|log2(fold change)|≥1,差异倍数大于2,P<0.05,筛选出了391个差异表达基因。以差异基因表达变化倍数的对数[log2(fold change)]为横坐标,以差异基因表达显著性值的校正值P的对数[lg(P value)]为纵坐标,绘制基因火山图,结果显示,有205个基因下调,186个基因上调(图2-A)。其中,上调基因中含倍数大于8[即|log2(fold change)|≥8]的差异表达基因有13个,上调倍数最大的基因是DANT1。其中,下调基因中倍数大于8的有11个,下调倍数最大的基因是TRNL1(表2),并对这些基因进行聚类分析(图2-B)。
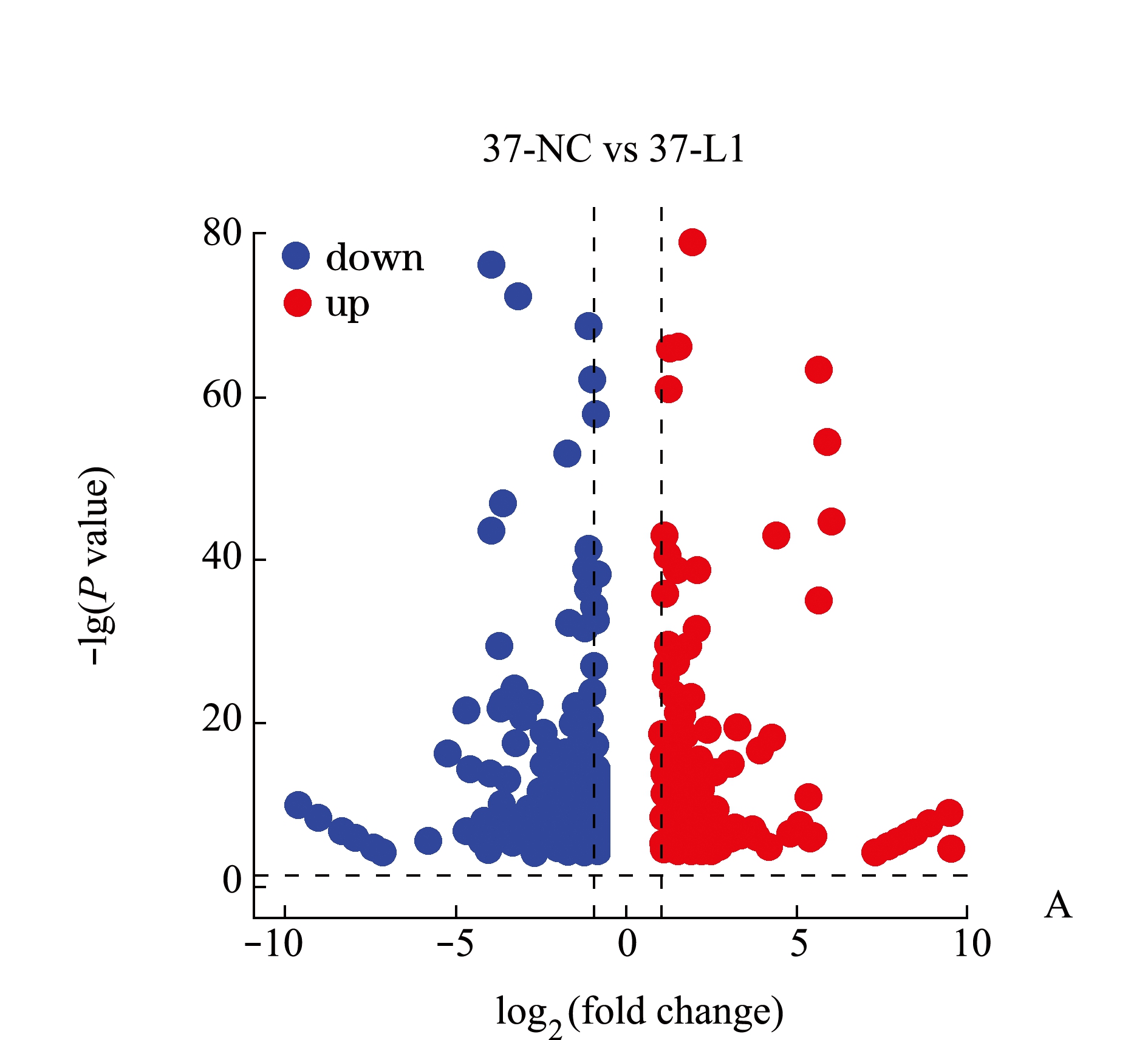
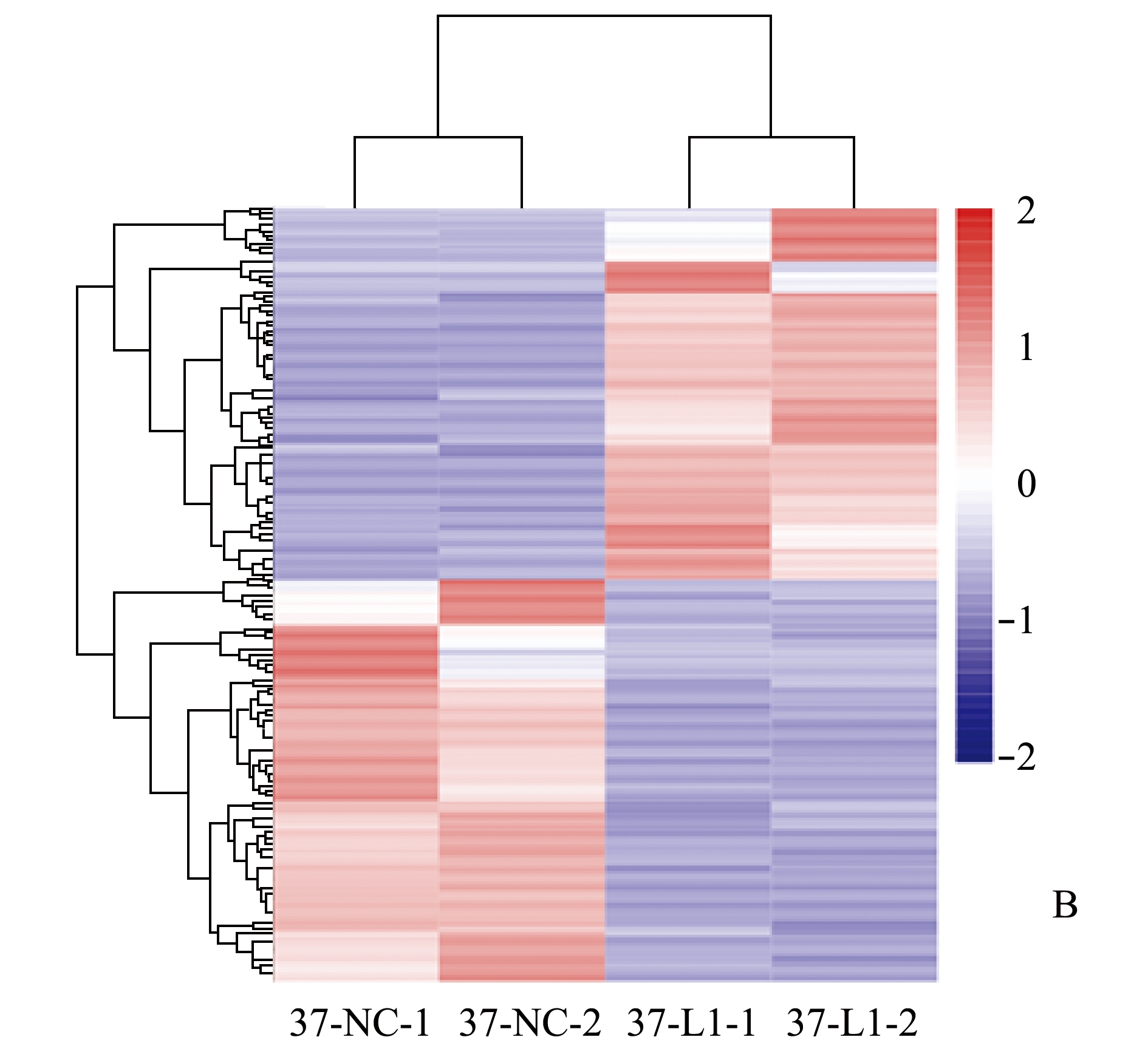
注:A为转染L1细胞和对照组的差异表达基因火山图;B为差异表达基因热图
Note:A,Valcano chart of differential expression of genes between Hela cells transfected with L1 and in the control;B,Heat map of differentially expressed genes
图2 差异表达基因火山图及热图分析
Fig.2 Valcano chart and heat map of differentially expressed genes
表1 RNA-seq样本质量评估
Tab.1 An overview of RNA-seq samples

组别group原始段数raw read修正后段数trimmed read比对上的段数mapped read37-NC-1865565480045557499571(93.69%)37-NC-2875573880909277582110(93.71%)37-L1-1948223186950258146598(93.69%)37-L1-2960435587993448246053(93.71%)
在391个差异表达基因中,有5个基因在数据库中未有相应的注释,且这5个差异表达基因均有显著高表达。初步推测这些基因可能与L1的逆转座有关,具体功能有待进一步研究。
2.4 差异表达基因GO功能分析和KEGG Pathway富集分析
因为差异表达基因较多,为了更清楚地认识L1对Hela细胞的作用机制,可通过生物信息学方法对差异表达基因进行功能注释分析。对差异表达基因进行GO功能富集分析,涉及生物学过程、分子功能、细胞组分3大类14个小类。其中,生物学过程(14.29%,biological processes,BP)的有溶酶体蛋白定位、羧酸跨膜转运;分子功能(7.14%,molecular functions,MF)的有肝素结合;细胞组分(78.57%,cellular component,CC)的有肌动蛋白纤维、膜的褶皱、细胞突出膜、高尔基体膜囊、黏着斑、细胞基质黏着连接、细胞基质连接、AP型膜被接合复合体、外显子复合物、反面高尔基体网络、网格蛋白外被。参与基因较多的是细胞组分中的黏着斑、细胞基质黏着连接、细胞基质连接(图3)。
表2 试验组和对照组表达倍数大于10和小于-10的差异表达基因
Tab.2 Differentially expressed genes in experimental and control groups at more than 10-folds and less than -10
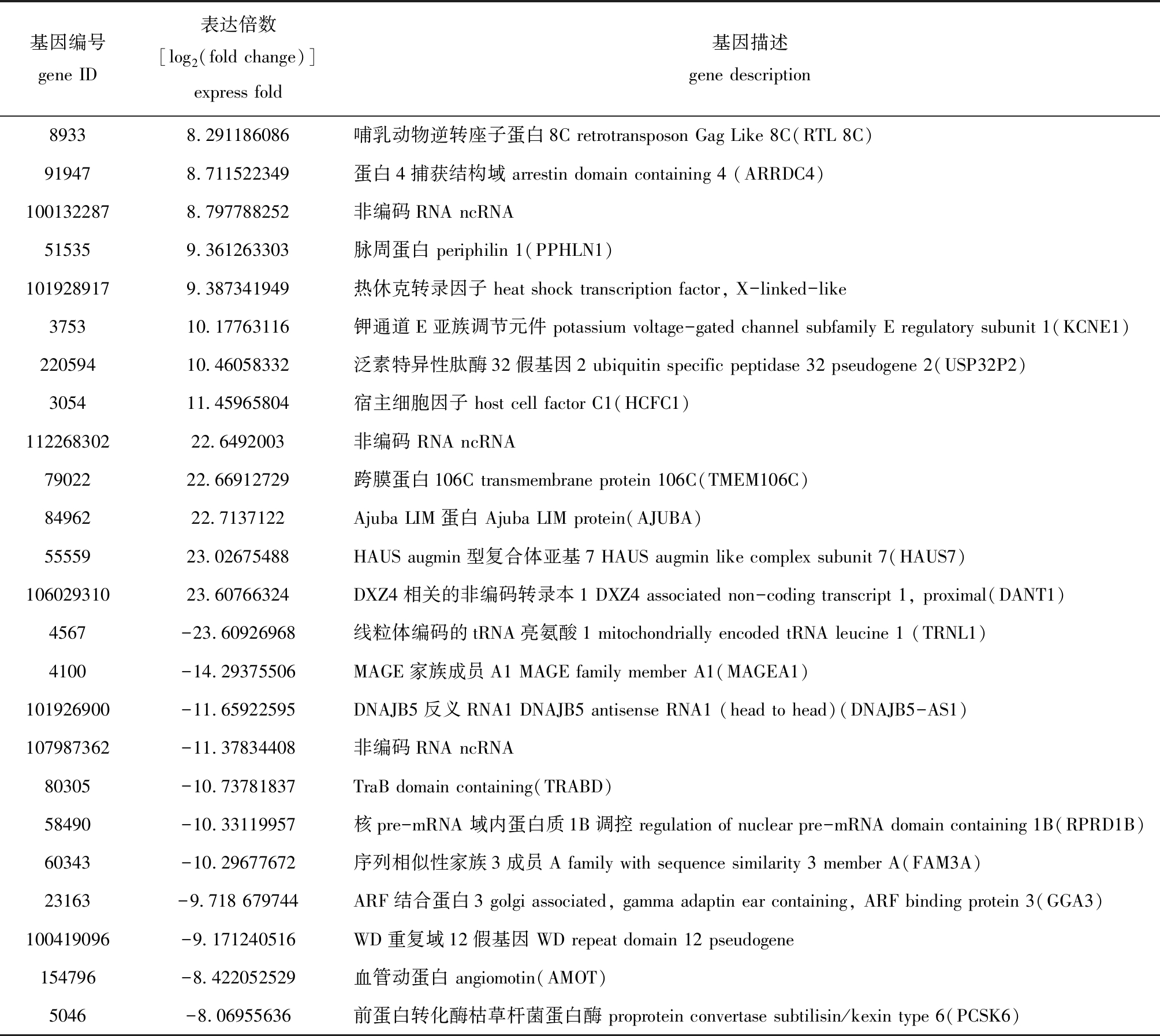
基因编号gene ID表达倍数[log2(fold change)]express fold基因描述gene description89338.291186086哺乳动物逆转座子蛋白8C retrotransposon Gag Like 8C(RTL 8C)919478.711522349蛋白4捕获结构域 arrestin domain containing 4 (ARRDC4)1001322878.797788252非编码RNA ncRNA515359.361263303脉周蛋白 periphilin 1(PPHLN1)1019289179.387341949热休克转录因子 heat shock transcription factor, X-linked-like375310.17763116钾通道E亚族调节元件 potassium voltage-gated channel subfamily E regulatory subunit 1(KCNE1)22059410.46058332泛素特异性肽酶32假基因2 ubiquitin specific peptidase 32 pseudogene 2(USP32P2)305411.45965804宿主细胞因子 host cell factor C1(HCFC1)11226830222.6492003非编码 RNA ncRNA7902222.66912729跨膜蛋白106C transmembrane protein 106C(TMEM106C)8496222.7137122Ajuba LIM蛋白 Ajuba LIM protein(AJUBA)5555923.02675488HAUS augmin型复合体亚基7 HAUS augmin like complex subunit 7(HAUS7)10602931023.60766324DXZ4相关的非编码转录本1 DXZ4 associated non-coding transcript 1, proximal(DANT1)4567-23.60926968线粒体编码的tRNA亮氨酸1 mitochondrially encoded tRNA leucine 1 (TRNL1)4100-14.29375506MAGE家族成员A1 MAGE family member A1(MAGEA1)101926900-11.65922595DNAJB5反义RNA1 DNAJB5 antisense RNA1 (head to head)(DNAJB5-AS1)107987362-11.37834408非编码RNA ncRNA80305-10.73781837TraB domain containing(TRABD)58490-10.33119957核pre-mRNA 域内蛋白质1B调控 regulation of nuclear pre-mRNA domain containing 1B(RPRD1B)60343-10.29677672序列相似性家族3成员A family with sequence similarity 3 member A(FAM3A)23163-9.718 679744ARF结合蛋白3 golgi associated, gamma adaptin ear containing, ARF binding protein 3(GGA3)100419096-9.171240516WD重复域12假基因 WD repeat domain 12 pseudogene154796-8.422052529血管动蛋白 angiomotin(AMOT)5046-8.06955636前蛋白转化酶枯草杆菌蛋白酶 proprotein convertase subtilisin/kexin type 6(PCSK6)
注:基因ID为Entrez-gene ID
Note: The gene ID is Entrez-gene ID
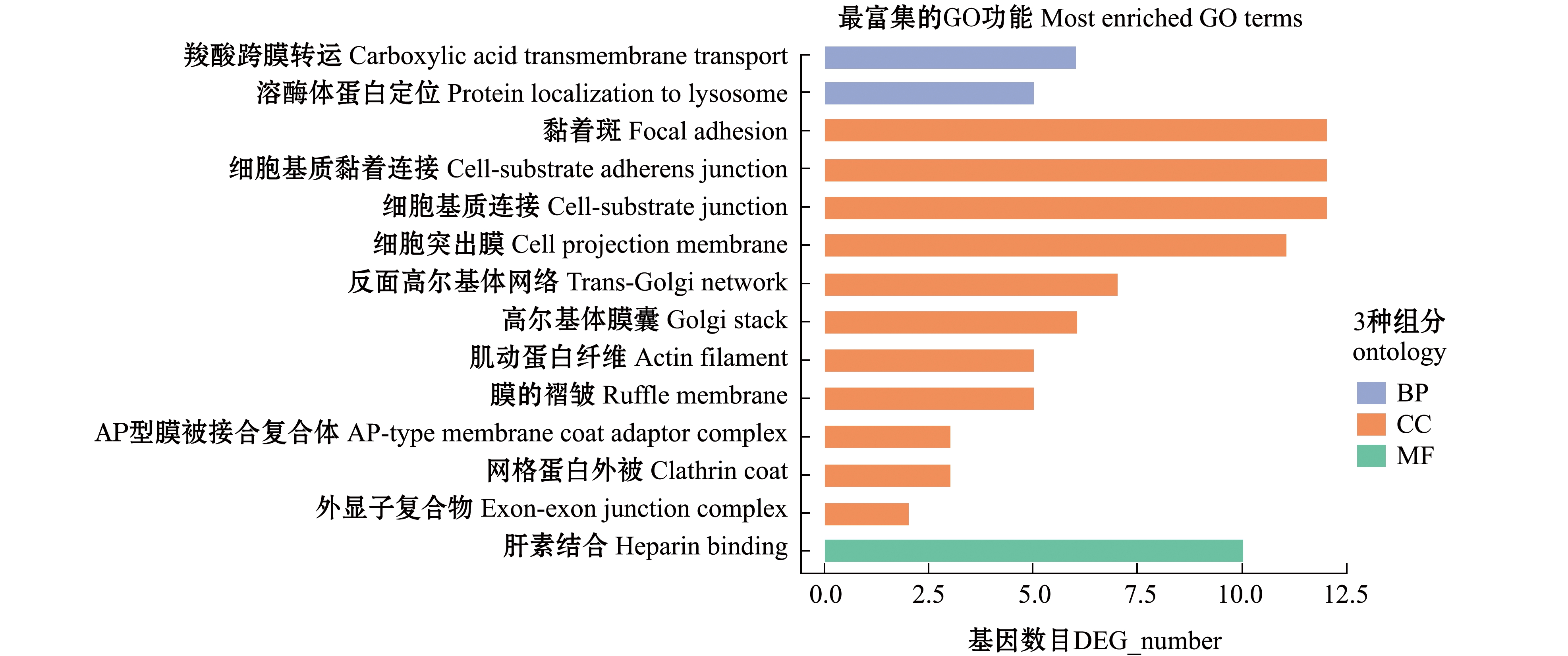
图3 GO功能注释及分类统计
Fig.3 Function annotation and classification of GO
在大多数生物学过程中,基因常通过相互作用参与其中,为了进一步探索这些差异表达基因的生物学功能,对差异表达基因进行KEGG Pathway 代谢通路注释。共得到5条显著富集的通路,包括破骨细胞分化、色氨酸代谢、乙型肝炎、病毒致癌作用、可卡因成瘾。其中,骨细胞分化、乙型肝炎、病毒致癌作用富集基因数均超过6个(图4)。

图4 差异表达基因KEGG富集分析
Fig.4 KEGG pathway analysis of differentially expressed genes
3 讨论
3.1 L1逆转座子在细胞生长发育过程中的作用
长散在重复元件(L1)是哺乳动物基因组中主要的逆转座子,占人类基因组的17%[12]。L1能够将non-L1的转录本复制到基因组DNA中,产生基因多样性和各种基因改变[6,13-14]。首先,L1在Hela细胞中的表达蛋白ORF1p符合预测的分子质量40 000,表达位置在细胞质中。
为了进一步探究L1在Hela细胞中的作用,本研究中通过RNA-seq做了进一步的分析。通过对转录组数据的分析,共筛选获得391个差异表达基因,表达量显著性上调的基因与细胞代谢(ARRDC4)、表皮完整性(PPHLN1)、细胞反应(HSFX3)、钾通道、转录因子假基因(USP32P2)、转膜蛋白(TMEM106c)、细胞连接复合体(AJUBA)、DNA修复(HAUS7)和控制细胞周期(HCFC1)有关。值得关注的是表达量上调最大的基因DANT1,通常在人类胚胎干细胞(ESCs)中有高表达,在体细胞和其他细胞系未有表达,是一种可以作为染色质之间的绝缘体或促进染色体间相互作用的因子[15]。在Hela细胞过表达L1后,DANT1表达量显著上升,初步推测是因为L1发生逆转座影响了DANT1高表达,并进一步促进了染色体之间相互作用。
3.2 L1逆转座子与致癌作用的联系
本研究中,通过对差异表达基因进行GO功能富集分析发现,99个差异表达基因中有78.57%参与了细胞组分的形成。有文献报道,在非恶性的乳腺癌细胞中,因为上皮细胞钙黏蛋白(E-cadherin)的缺失会引起细胞微妙的形态变化,细胞基质附着力减弱,细胞骨架中的肌动蛋白和顶端微管网络发生了变化[16]。分析GO的注释结果,发现多个基因与肌动蛋白纤维、黏着斑、细胞基质连接等相关。在差异表达基因中,与其相关的因子,如脉周蛋白(PPHLN1)、Ajuba LIM蛋白(AJUBA)的基因发生了250倍以上的显著上调。因此,猜测L1对细胞结构的影响是通过激活癌细胞导致的。同时,通过对差异表达基因进行KEGG Pathway 代谢通路注释共得到5条显著富集的通路,注释结果显示,L1主要参与了乙型肝炎和病毒致癌作用。有文献报道,L1的去甲基化可导致如前列腺癌症、胃癌等疾病[7-10]。本试验结果也证实了L1逆转座子的确与致癌作用具有一定的联系,也进一步证明L1对细胞结构的影响很有可能是其致癌作用引起的,其具体机制还需要进一步试验验证。
3.3 L1逆转座子功能研究进展
由于L1在人类基因组中所占比例较大,所以L1在机体中的作用受到高度关注,其功能研究显得尤为重要。L1作为一类转座子其作用非常广泛。已有研究报道,L1通过参与假基因的生成、缺失、外显子化和选择性剪接[16],可导致DNA 双链断裂、重排等基因组改变,进而影响DNA的多态性[17-18]。另外,L1启动子区域的去甲基化在肺、结直肠、前列腺、卵巢、骨髓瘤和肝肿瘤中均有发现[19-23],这说明L1的去甲基化对组织器官发育也具有一定的影响。更多的功能,如关于L1活性的调控、L1复制的生物化学过程和L1与宿主间的相互作用都是未知的,这对于理解哺乳动物的基因功能有较大阻碍。逆转座子与宿主间怎样共同进化达到逆转座、特定目的地的插入及插入突变间的平衡且实现双赢,仍需深入研究。
4 结论
本研究中,在Hela细胞中对逆转座子L1进行了表达和定位,并利用转录组分析L1在Hela细胞中的作用。通过对差异表达基因进行分析,一方面对L1的作用进行了初步探究,部分结果与已有报道相符,说明L1的确在细胞组分形成过程及致癌过程中发挥重要作用。另一方面,本试验中同时分析发现了L1可能参与的一些新的生物过程,如破骨细胞分化、色氨酸代谢、肝素结合等,这也为进一步研究L1的作用提供了思路和理论依据。
[1] De Koning A P J,Gu W J,Castoe T A,et al.Repetitive elements may comprise over two-thirds of the human genome[J].PLoS Genetics,2011,7(12):e1002384.
[2] Yang Fang,Wang P J.Multiple LINEs of retrotransposon silencing mechanisms in the mammalian germline[J].Seminars in Cell & Developmental Biology,2016,59:118-125.
[3] Denli A M,Narvaiza I,Kerman B E,et al.Primate-specific ORF0 contributes to retrotransposon-mediated diversity[J].Cell,2015,163(3):583-593.
[4] Baba Y,Yagi T,Sawayama H,et al.Long interspersed element-1 methylation level as a prognostic biomarker in gastrointestinal cancers[J].Digestion,2018,97(1):26-30.
[5] Zhao K,Du J,Peng Y F,et al.LINE1 contributes to autoimmunity through both RIG-I- and MDA5-mediated RNA sensing pathways[J].Journal of Autoimmunity,2018,90:105-115.
[6] Beck C R,Garcia-Perez J L,Badge R M,et al.LINE-1 elements in structural variation and disease[J].Annual Review of Genomics and Human Genetics,2011,12:187-215.
[7] Fiano V,Zugna D,Grasso C,et al.LINE-1 methylation status in prostate cancer and non-neoplastic tissue adjacent to tumor in association with mortality[J].Epigenetics,2017,12(1):11-18.
[8] Tahara T,Tahara S,Horiguchi N,et al.Methylation status of IGF2 DMR and LINE1 in leukocyte DNA provides distinct clinicopathological features of gastric cancer patients[J].Clinical and Experimental Medicine,2018,18(2):215-220.
[9] Roman-Gomez J,Jimenez-Velasco A,Agirre X,et al.Promoter hypomethylation of the LINE-1 retrotransposable elements activates sense/antisense transcription and marks the progression of chronic myeloid leukemia[J].Oncogene,2005,24(48):7213-7223.
[10] Saito K,Kawakami K,Matsumoto I,et al.Long interspersed nuclear element 1 hypomethylation is a marker of poor prognosis in stage IA non-small cell lung cancer[J].Clinical Cancer Research,2010,16(8):2418-2426.
[11] Svensson V,Vento-Tormo R,Teichmann S A.Exponential scaling of single-cell RNA-seq in the past decade[J].Nature Protocols,2018,13(4):599-604.
[12] Cordaux R,Batzer M A.The impact of retrotransposons on human genome evolution[J].Nature Reviews Genetics,2009,10(10):691-703.
[13] Huang C R L,Schneider A M,Lu Yunqi,et al.Mobile interspersed repeats are major structural variants in the human genome[J].Cell,2010,141(7):1171-1182.
[14] Hancks D C,Kazazian Jr H H.Active human retrotransposons:variation and disease[J].Current Opinion in Genetics & Development,2012,22(3):191-203.
[15] Yang Fan,Deng Xinxian,Ma Wenxiu,et al.The lncRNA Firre anchors the inactive X chromosome to the nucleolus by binding CTCF and maintains H3K27me3 methylation[J].Genome Biology,2015,16:52.
[16] Chen A,Beetham H,Black M A,et al.E-cadherin loss alters cytoskeletal organization and adhesion in non-malignant breast cells but is insufficient to induce an epithelial-mesenchymal transition[J].BMC Cancer,2014,14:552.
[17] Kazazian Jr H H,Moran J V.Mobile DNA in health and disease[J].New England Journal of Medicine,2017,377(4):361-370.
[18] Chen Shue,Yu Mengchao,Chu Xu,et al.Cold-induced retrotransposition of fish LINEs[J].Journal of Genetics and Genomics,2017,44(8):385-394.
[19] Iskow R C,Mccabe M T,Mills R E,et al.Natural mutagenesis of human genomes by endogenous retrotransposons[J].Cell,2010,141(7):1253-1261.
[20] Shukla R,Upton K R,Mu oz-Lopez M,et al.Endogenous retrotransposition activates oncogenic pathways in hepatocellular carcinoma[J].Cell,2013,153(1):101-111.
oz-Lopez M,et al.Endogenous retrotransposition activates oncogenic pathways in hepatocellular carcinoma[J].Cell,2013,153(1):101-111.
[21] Solyom S,Ewing A D,Rahrmann E P,et al.Extensive somatic L1 retrotransposition in colorectal tumors[J].Genome Research,2012,22(12):2328-2338.
[22] Liu Xinneng,Jin Yongtang,Xu Peiwei,et al.Relationship between Line 1 methylation and clinical data of non-small cell lung cancer[J].Chinese Journal of Medical Genetics,2014,31(3):307-311.
[23] Zelic R,Fiano V,Zugna D,et al.Global hypomethylation (LINE-1) and gene-specific hypermethylation (GSTP1) on initial negative prostate biopsy as markers of prostate cancer on a rebiopsy[J].Clinical Cancer Research,2016,22(4):984-992.