 (pir)中,与鮰爱德华菌野生菌株ATCC33202共培养。经同源重组和蔗糖负筛获得鮰爱德华菌eivpP基因缺失突变阳性克隆,经PCR鉴定结果显示,突变株未检测出eivpP基因,即eivpP基因彻底敲除成功。本试验结果可为研究eivpP基因在鮰爱德华菌致病过程中的作用机制提供数据资料,为鮰爱德华菌弱毒疫苗的研发提供理论依据。
(pir)中,与鮰爱德华菌野生菌株ATCC33202共培养。经同源重组和蔗糖负筛获得鮰爱德华菌eivpP基因缺失突变阳性克隆,经PCR鉴定结果显示,突变株未检测出eivpP基因,即eivpP基因彻底敲除成功。本试验结果可为研究eivpP基因在鮰爱德华菌致病过程中的作用机制提供数据资料,为鮰爱德华菌弱毒疫苗的研发提供理论依据。鮰爱德华菌Edwardsiella ictaluri是革兰氏阴性短杆菌,隶属于肠杆菌科Enterobacteriaceae、爱德华氏菌属Edwardsiella,于1979年在美国斑点叉尾鮰肠型败血症(Enteric Septicaemia of Catfish,ESC)上首次报道[1]。鮰爱德华菌是鲶形目鱼类的主要致病菌[2],随着该菌的大面积暴发,其宿主范围也逐渐扩大,包括鮰科鱼类及黄颡鱼Pelteobagrus fulvidraco、欧洲鲇Silurus glanis、虹鳟Oncorhynchus mykiss、大鳞大麻哈鱼Oncorhynchus tshawytscha、泰国蟾胡鲇Clarias batrachus、中华鳖Trionyx sinensis等[3-5],鮰爱德华菌主要通过感染鱼类神经系统引发炎症(头盖穿孔型)和由消化道侵入血液循环引发急性败血症(肠道败血型)两种方式感染鱼体[6],对鱼类养殖业会造成重大的经济损失[7]。鮰爱德华菌病具有季节性[8],多发于每年的5—6月和9—10月,可通过降低水温抑制该菌的生长,也可用超声粉碎菌体疫苗和福尔马林灭活疫苗刺激鱼体产生抗体形成保护免疫。
分泌系统是细菌在长期进化过程中形成的一种与外界环境进行相互作用的工具,细菌通过分泌系统将毒力蛋白或效应因子释放到外界环境中,或者直接作用于原核或真核生物细胞。研究表明,许多革兰氏阴性细菌通过分泌系统分泌毒力因子使机体患病。对鮰爱德华菌全基因组测序发现,其存在一种Ⅵ型分泌系统(T6SS)[9],该分泌系统在鮰爱德华菌致病过程中具有重要作用[10-11]。对与鮰爱德华菌同属的迟缓爱德华菌E.tarda的研究表明,该菌的T6SS包含3种分泌蛋白:EvpC(Hcp同系物)、EvpI(VgrG同系物)和EvpP[12],迟缓爱德华菌的evpP位于T6SS基因簇最前端;Hcp的分泌依赖于EvpP[12],evpP基因的缺失可导致Hcp和VgrG的表达量显著降低。有报道表明,鮰爱德华菌的EivpP可以阻碍宿主胞浆中Ca2+的增加,使Ca2+依赖性的c-Jun N-末端激酶(Jnk)无法被激活,进而影响炎症小体接合器ASC寡聚化和效应亚基caspase-1的裂解,致使炎症小体NLRP3激活失败,且宿主无法分泌白细胞介素-1β(IL-1β)和白细胞介素-18(IL-18),最终,EivpP通过抑制炎症小体的激活促进鮰爱德华菌在宿主体内的增殖[13]。
基因敲除又称基因打靶,是指外源打靶基因与基因组目标基因通过基因工程手段,在转染细胞中发生DNA同源重组,使外源基因定点整合到基因组目标基因中[14]。基于该技术,可以从分子水平上将一个基因去除或替代,并从整体观察试验对象,进而推测相应基因的功能[15]。本研究中,通过同源重组法和融合PCR技术成功构建鮰爱德华菌eivpP基因缺失突变菌株,旨在为研究EivpP及T6SS在鮰爱德华菌致病过程中的作用机制、寻找有效防控措施奠定基础。
菌株和质粒:试验用菌株和质粒见表1。
表1 试验菌株和质粒
Tab.1 Strains and plasmids used in this experiment
试验试剂:氨苄青霉素Amp(100 μg/mL)、硫酸黏菌素(50 μg/mL)、氯霉素Cmr(34 μg/mL)、IPTG(1.0 mmol/L)和蔗糖(10 %);质粒提取试剂盒和PCR产物胶回收试剂盒购于天根生化科技有限公司,限制性内切酶XhoⅠ、XbaⅠ、BamHⅠ购于Thermo公司,Prime STAR GXL DNA Ploynerase体系购于TaKaRa公司。
1.2.1 eivpP上、下同源臂及融合PCR扩增 根据鮰爱德华菌全基因组序列获得eivpP及其上、下臂序列,设计引物eivpP-a-F/R和eivpP-b-F/R,引物序列见表2。
以鮰爱德华菌基因组DNA为模板,以eivpP-a-F/R和eivpP-b-F/R为引物,分别对eivpP上臂(545 bp)和下臂(500 bp)进行PCR扩增,用胶回收试剂盒对扩增产物进行纯化。PCR反应体系(共25 μL):Prime STAR GXL DNA Ploynerase 0.5 μL,5×Prime STAR GXL Buffer(Mg2+ plus)5 μL,dNTP Mixture 2 μL,模板1 μL,上、下游引物(10 μmol/L)各1 μL,ddH2O 14.5 μL。PCR反应程序:98 ℃下预变性5 min;98 ℃下变性10 s,68 ℃下退火复性40 s,共进行30个循环;最后在68 ℃下再延伸5 min。
将纯化后的上、下同源臂片段按1∶1混合。反应程序为:98 ℃下预变性5 min;98 ℃下循环变性10 s,68 ℃下退火复性1 min 20 s,共进行10个循环。以混合片段作为模板,以eivpP-a-F和eivpP-b-R为引物进行融合PCR,反应程序同上。
表2 试验用引物
Tab.2 Primers used in this experiment

引物primer酶切位点restriction enzyme cutting site序列(5′-3′)sequence(5′-3′)eivpP-a-FXho I TTCTCGAGCAGTGCGTTCCACTGGCeivpP-a-RATTATTTTTGAAGTAATTAGTGAATGCGCTATCCTTTTTCAGTA eivpP-b-FTACTGAAAAAGGATAGCGCATTCACTAATTACTTCAAAAATAAT eivpP-b-RXba I TTTCTAGAGTGCGGAATGACAGATGT eivpP-FBamH I ATGGATCCATGTCTATTTTGAACTCTG eivpP-RXho I ATCTCGAGCTATTTCAATGCTGAAAAG M13-F CGCCAGGGTTTTCCCAGTCACGAC M13-R AGCGGATAACAATTTCACACAGGA RV-M-R GAGCGGATAACAATTTCACACAGG
注:下划线标记的碱基为酶切位点
Note:The restriction enzyme cutting sites are marked by underlined letters
1.2.2 重组自杀质粒pDM4-ab的构建 上述扩增产物经切胶回收后进行A碱基反应:融合片段20.25 μL,10×A-Tailing Buffer 2.5 μL,dNTP Mixture 2 μL,A-Tailing Enzyme 0.25 μL,72 ℃下反应20 min,于冰上静置2 min。
将上述产物进行切胶回收并与pMD19-T连接,并通过热激法将连接产物转化到DH5α感受态细胞中,于LB液体培养基中37 ℃下以180 r/min活化1 h,涂布于含有Amp抗性的LB固体培养基上,37 ℃下过夜培养。挑取阳性菌落,以M13-F/R为引物进行PCR验证。选取PCR验证为阳性克隆送北京华大基因有限公司进行测序,获得含有融合片段的pMD19-T-ab质粒。
提取质粒pMD19-T-ab和pDM4,经XhoⅠ和XbaⅠ双酶切后分别回收目的片段,将酶切后的pDM4质粒与目的片段混合后,经T4连接酶于16 ℃下连接过夜。将连接产物热激转化到大肠杆菌S17-1 (pir)感受态细胞中,于LB液体培养基中在37 ℃下以180 r/min活化1 h,然后涂布于含有氯霉素抗性的LB固体培养基上,37 ℃下过夜培养。挑取单克隆菌落,以eivpP-a-F和eivpP-b-R为引物进行PCR验证,产物送北京华大基因有限公司进行测序。
(pir)感受态细胞中,于LB液体培养基中在37 ℃下以180 r/min活化1 h,然后涂布于含有氯霉素抗性的LB固体培养基上,37 ℃下过夜培养。挑取单克隆菌落,以eivpP-a-F和eivpP-b-R为引物进行PCR验证,产物送北京华大基因有限公司进行测序。
1.2.3 基因缺失菌株的构建
(1) 结合转移。自杀质粒借助Mob基因从S17-1 (pir)菌转移到野生鮰爱德华菌中。将含有pDM4-ab质粒的S17-1
(pir)菌转移到野生鮰爱德华菌中。将含有pDM4-ab质粒的S17-1 (pir)(氯霉素)与野生鮰爱德华菌分别接种于LB和NB液体培养基中培养至对数期,以4000 r/min低速离心去除培养基,用新鲜培养基重悬菌体并按1∶1混合后,在NA培养基上滴4滴,每滴50 μL,于28 ℃下共同培养,至长出菌斑。
(pir)(氯霉素)与野生鮰爱德华菌分别接种于LB和NB液体培养基中培养至对数期,以4000 r/min低速离心去除培养基,用新鲜培养基重悬菌体并按1∶1混合后,在NA培养基上滴4滴,每滴50 μL,于28 ℃下共同培养,至长出菌斑。
(2) 初筛。刮取菌斑于NB液体培养基中培养至一定浓度,涂布于含有氯霉素和硫酸黏菌素的双抗NA培养基上,28 ℃下培养至长出单菌落。将单菌落接种于含有硫酸黏菌素的NB液体培养基中培养,并用引物eivpP-a-F和eivpP-b-R进行PCR验证。
(3) 负筛。借助sacB基因反向筛选出基因缺失菌株。初筛完成后,挑取单菌落在含有硫酸黏菌素的NB液体培养基中培养,将菌液涂布到含有硫酸黏菌素和10%蔗糖的固体培养基上,28 ℃下培养至长出单菌落,挑取单菌落并接种于含有硫酸黏菌素的NB液体培养基中培养,用引物eivpP-a-F和eivpP-b-R进行PCR验证。若未扩增出eivpP基因,则表明eivpP基因敲除成功。
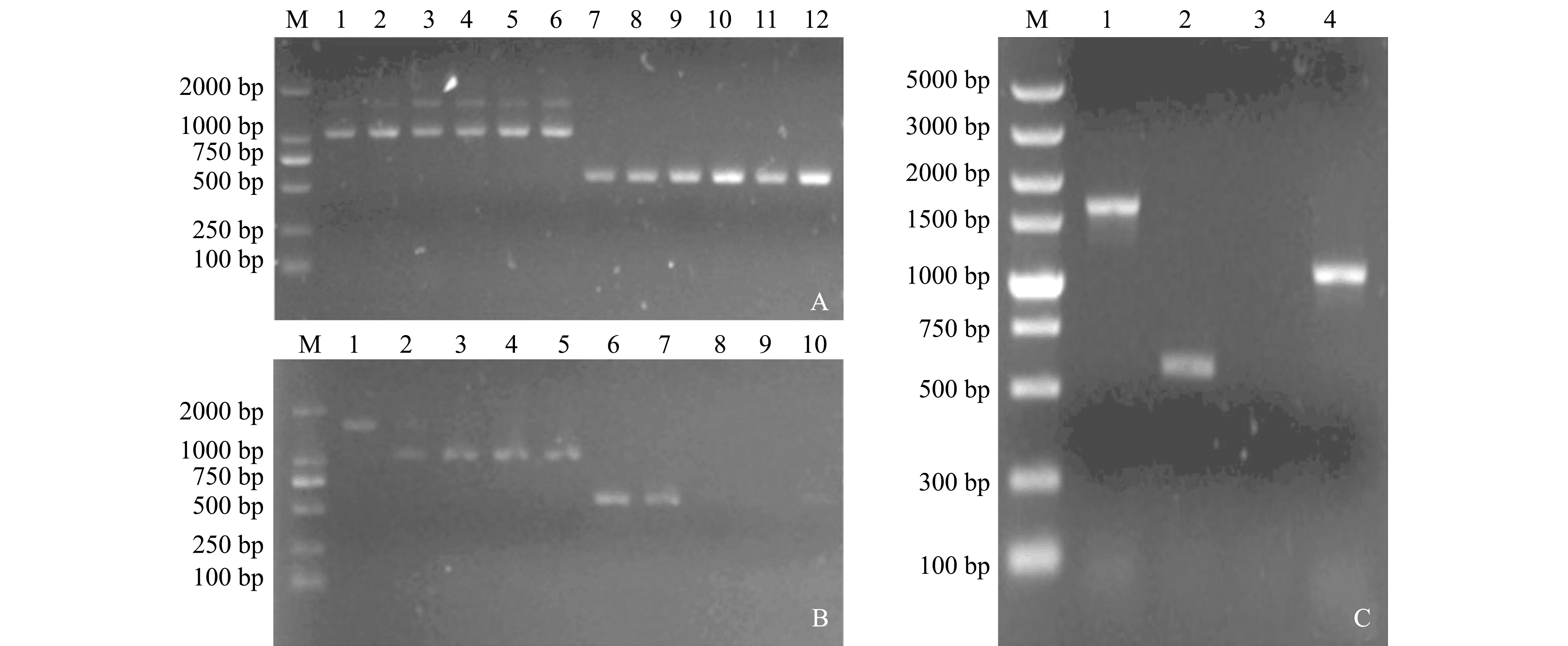
本试验中以eivpP-a-F/R和eivpP-b-F/R为引物,分别扩增出eivpP上游同源臂(约为545 bp)和下游同源臂(约为500 bp)。以eivpP-a-F和eivpP-b-R为引物,PCR扩增出融合片段ab(约为1026 bp),用10 g/L琼脂糖凝胶电泳,结果如图1-A所示。
融合片段ab与pMD19-T连接后命名为pMD19-T-ab,以M13-F和RV-M-R为引物,pMD19-T-ab(DH5 )为模板进行扩增,产物大小约为1026 bp,如图1-B所示。
)为模板进行扩增,产物大小约为1026 bp,如图1-B所示。
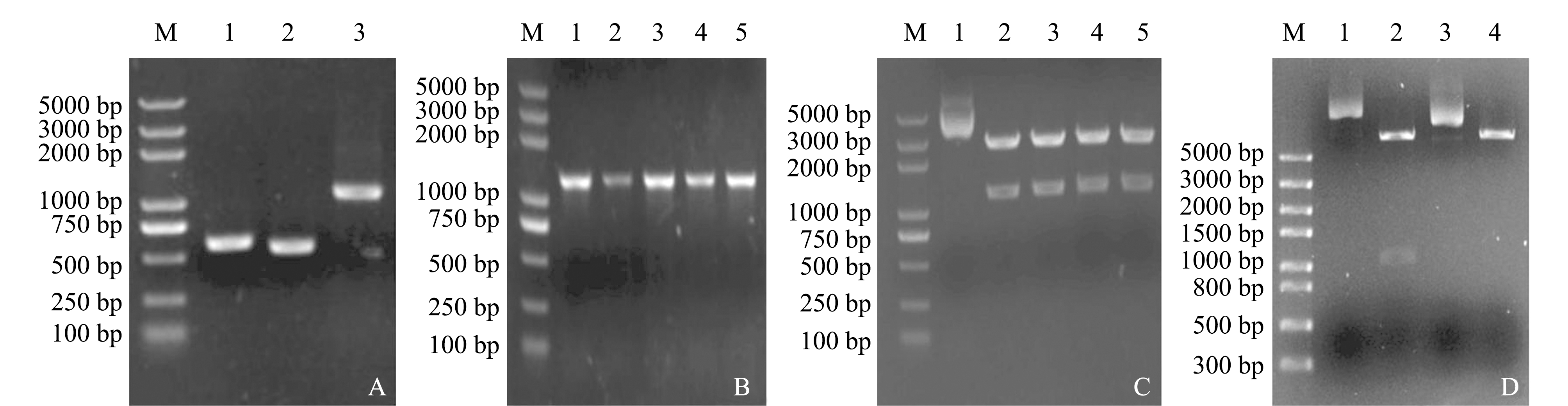
注:A为上下同源臂及融合片段扩增结果,A中1为上游同源臂a,2为下游同源臂b,3为融合片段ab;B为载体pMD19-T-ab的鉴定结果,B中1~5均以M13-F/R为引物;C为载体pMD19-T-ab的双酶切结果,C中1为载体pMD19-T-ab,2~5均为载体pMD19-T-ab的双酶切结果;D为载体pDM4-ab及pDM4的双酶切结果,D中1~4分别为载体pDM4-ab、pDM4-ab双酶切、pDM4和pDM4双酶切结果;M为DNA Marker
Note:A, The amplification of upper and lower homologous arms and fusion fragments, 1, upstream homologous arm a, 2, downstream homologous arm b, 3, fusion fragment ab; B, The identification of the vector pMD19-T-ab, 1-5, M13-F/R as primer; C, The double enzymes digestion of vector pMD19-T-ab, 1, vector pMD19-T-ab, 2-5, double enzymes digestion of vector pMD19-T-ab; D, The double enzymes digestion of vector pDM4-ab and pDM4, 1-4, the results of vector pDM4-ab, double enzymes digestion of pDM4-ab, pDM4 and double enzymes digestion of pDM4,respectively; M, DNA Marker
图1 eivpP同源臂融合及重组自杀质粒的构建
Fig.1 eivpP homologous arm fusion and the construction of recombinant suicide plasmids
分别提取质粒pMD19-T-ab和pDM4,使用Xho Ⅰ和Xba Ⅰ双酶切并回收,结果如图1-C和图1-D所示。将回收后的目的片段(约为1026 bp)与pDM4载体连接,连接产物命名为pDM4-ab。使用Xho Ⅰ和Xba Ⅰ对质粒pDM4-ab进行双酶切,并得到约为1026 bp的片段,进一步验证了重组自杀质粒构建成功,如图1-D所示。
经结合转移,自杀质粒借助Mob基因从S17-1 (pir)菌转移到野生鮰爱德华菌中。通过氯霉素和硫酸黏菌素进行初筛,以野生ATCC33202为模板,以eivpP-a-F/eivpP-b-R和eivpP-F/eivpP-R为引物进行验证,经10 g/L琼脂糖凝胶电泳显示,扩增出两条大小分别为1603、1026 bp的条带,且该菌中仍存在eivpP基因。说明重组质粒与鮰爱德华菌全基因组完成一个同源臂交换,结果如图2-A所示。
(pir)菌转移到野生鮰爱德华菌中。通过氯霉素和硫酸黏菌素进行初筛,以野生ATCC33202为模板,以eivpP-a-F/eivpP-b-R和eivpP-F/eivpP-R为引物进行验证,经10 g/L琼脂糖凝胶电泳显示,扩增出两条大小分别为1603、1026 bp的条带,且该菌中仍存在eivpP基因。说明重组质粒与鮰爱德华菌全基因组完成一个同源臂交换,结果如图2-A所示。
初筛完成后,通过10%蔗糖和硫酸黏菌素进行负筛,以野生ATCC33202全基因组为模板,以eivpP-a-F/eivpP-b-R和eivpP-F/eivpP-R为引物进行PCR验证,经琼脂糖凝胶电泳显示,只扩增出一条约为1026 bp的DNA条带,且未扩增出eivpP基因,结果初步表明,eivpP基因敲除成功,如图2-B所示,其中8、9、10泳道为阳性结果。
以野生ATCC33202全基因组为模板,以eivpP-a-F/eivpP-b-R为引物,可扩增出1603 bp片段;以eivpP-F/eivpP-R为引物,可扩增出558 bp片段,即eivpP基因。以ATCC33202- eivpP基因组为模板,以eivpP-a-F/eivpP-b-R为引物,可扩增出1026 bp片段;以eivpP-F/eivpP-R为引物,未能扩增出eivpP基因,如图2-C所示。结合测序结果,确定eivpP基因缺失突变菌株已构建成功。
eivpP基因组为模板,以eivpP-a-F/eivpP-b-R为引物,可扩增出1026 bp片段;以eivpP-F/eivpP-R为引物,未能扩增出eivpP基因,如图2-C所示。结合测序结果,确定eivpP基因缺失突变菌株已构建成功。
基因敲除技术是利用微生物体内的同源重组系统,在一定选择压力下使体外改造的某功能基因与受体细胞染色体上的功能基因发生同源重组[16]。
注:A为初筛菌株的鉴定结果,A中1~6均为以eivpP-a-F/eivpP-b-R为引物的扩增,7~12均为eivpP基因的扩增; B为负筛菌株的鉴定结果,B中1为以野生菌株为模板的鉴定,2~5均为以敲除菌株为模板的鉴定,6~7均为以野生菌株为模板的eivpP基因扩增,8~10均为以敲除菌株为模板的eivpP基因扩增(无扩增条带,即为敲除成功);C为基因敲除菌株ATCC33202-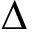 eivpP的鉴定结果,C中1为以野生菌株为模板的鉴定,2为以野生菌株为模板的eivpP基因扩增,3为以敲除菌株为模板的eivpP基因扩增(无扩增条带,即为敲除成功),4为以敲除菌株为模板的鉴定;M为DNA Marker
eivpP的鉴定结果,C中1为以野生菌株为模板的鉴定,2为以野生菌株为模板的eivpP基因扩增,3为以敲除菌株为模板的eivpP基因扩增(无扩增条带,即为敲除成功),4为以敲除菌株为模板的鉴定;M为DNA Marker
Note:A, The identification of the primary screening strain, 1-6, the amplification with eivpP-a-F/eivpP-b-R as the primer, 7-12, the amplification of eivpP gene; B, The identification of the reverse screening strain, 1, the identification results with wild strain as template, 2-5, the identification with knockout strain as template, 6-7, the eivpP gene amplification with wild strain as template, 8-10, the eivpP gene amplification with knockout strain as template (no amplification band, that is, the knockout is successfull); C, The identification of the knochout strain ATCC33202- eivpP, 1, the identification with wild strain as template; 2, the eivpP gene amplification with wild strain as template; 3, the eivpP gene amplification with knockout strain as template (no amplification band, that is, the knockout is successfull); 4, the identification with knockout strain as template; M, DNA Marker
eivpP, 1, the identification with wild strain as template; 2, the eivpP gene amplification with wild strain as template; 3, the eivpP gene amplification with knockout strain as template (no amplification band, that is, the knockout is successfull); 4, the identification with knockout strain as template; M, DNA Marker
图2 eivpP基因缺失菌株的构建与鉴定
Fig.2 Construction and identification of eivpP in-frame deletion strain
基因敲除技术起源于20世纪80年代,为研究基因功能创造了新的途径,大大推动了生物学和医学研究的发展。传统的基因敲除方法包括同源重组法和随机插入突变法:同源重组法精准度高,对大多数细菌来说较为简单;随机插入突变法具有效率高、基因完全失活等优点,但插入位点的随机性较高,且常引起染色体重排。随着功能基因组学研究的深入发展,也产生了一些新兴的基因敲除技术,包括RNA干扰技术、锌指核酸酶基因打靶技术、TALEN切割特定核苷酸靶序列技术和Cas9基因敲除技术[17]。本研究中,采用同源重组法和融合PCR技术,将eivpP基因上下游同源臂融合并克隆到自杀载体pDM4上,将重组质粒转化到大肠杆菌S17-1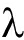 (pir)中,再结合转移到鮰爱德华菌菌株内,经pDM4质粒上的sacB基因反向筛选得到突变株。
(pir)中,再结合转移到鮰爱德华菌菌株内,经pDM4质粒上的sacB基因反向筛选得到突变株。
同源重组法构建敲除株的关键步骤之一是目的基因上、下臂的融合。常见的融合方法有双酶切法和融合PCR法:双酶切法是分别以目的基因上、下臂为模板,设计含有酶切位点的引物,双酶切后连接;融合PCR法是采用含有互补末端的引物扩增出具有重叠链的PCR产物,通过PCR产物重叠链的延伸,从而将两段DNA片段连接起来。融合PCR技术不需要内切酶消化和连接酶处理,相比较双酶切法更加快速简洁[18]。同源重组法敲除的另一个关键步骤是蔗糖负筛。自杀质粒pDM4上存在sacB基因,该基因编码的产物可分解蔗糖产生有毒物质,使没有完成同源重组且含有自杀质粒的受体菌无法在蔗糖平板上生长,从而筛选出敲除成功的菌株[19]。在试验过程中,需严格控制蔗糖浓度,以降低假阳性的概率,并通过多次筛选确保基因被彻底敲除。
另外,抗性筛选是筛选阳性克隆的常规操作方法。本试验中共涉及4次抗性筛选:在克隆载体pMD19-T-ab的构建过程中,根据T载体所携带的抗性基因,将转化后的菌液涂布于含有Amp抗性的LB固体培养基上以筛选阳性克隆;在重组自杀载体pDM4-ab的构建过程中,采用同样的方法使用氯霉素进行阳性筛选;在基因缺失菌株的构建过程中,根据鮰爱德华菌对硫酸黏菌素不敏感、大肠杆菌对其敏感这一特性,在初筛阶段利用氯霉素和硫酸黏菌素筛选得到含有重组自杀质粒的野生鮰爱德华菌,并在负筛阶段利用硫酸黏菌素和蔗糖筛选得到eivpP基因缺失突变菌株。
Ⅵ型分泌系统与细菌致病性密切相关,其在细菌侵染宿主细胞及细菌在宿主细胞内繁殖存活的过程中均发挥了重要作用[20]。EvpP和Hcp、VgrG是Ⅵ型分泌系统的主要效应蛋白[3],直接影响Ⅵ型分泌系统功能的行使。研究表明,EvpP的表达影响细菌内化及侵入宿主细胞的过程,从而影响细菌对宿主的致病作用[21]。鮰爱德华菌是鲇形目鱼类如斑点叉尾鮰和其他部分非鲇形目鱼类的重要病原。本研究为首次报道鮰爱德华菌EivpP,并采用同源重组法将eivpP基因敲除,成功构建鮰爱德华菌eivpP基因缺失菌株,为研究eivpP基因在鮰爱德华菌致病过程中的作用机制提供了数据资料,同时也为鮰爱德华菌弱毒疫苗的研发提供理论依据[22]。
[1] Hawke J P.A bacterium associated with disease of pond cultured channel catfish,Ictalurus punctatus[J].Journal of the Fisheries Research Board of Canada,1979,36(12):1508-1512.
[2] Dong H T,Nguyen V V,Phiwsaiya K,et al.Concurrent infections of Flavobacterium columnare and Edwardsiella ictaluri in striped catfish,Pangasianodon hypophthalmus in Thailand[J].Aquaculture,2015,448:142-150.
[3] Iwanowicz L R,Griffin A R,Cartwright D D,et al.Mortality and pathology in brown bullheads Amieurus nebulosus associated with a spontaneous Edwardsiella ictaluri outbreak under tank culture conditions[J].Diseases of Aquatic Organisms,2006,70(3):219-225.
[4] Keskin O,Seçer S,Izgür M,et al.Edwardsiella ictaluri infection in rainbow trout (Oncorhynchus mykiss)[J].Turkish Journal of Veterinary and Animal Sciences,2004,28(4):649-653.
[5] 肖克宇,江为民,舒新华,等.鮰爱德华氏菌变异株C9605及对鳖的致病性研究[J].微生物学通报,1998,25(5):262-265.
[6] 战文斌.水产动物病害学[M].北京:中国农业出版社,2004.
[7] 殷战,徐伯亥.鱼类细菌性疾病的研究[J].水生生物学报,1995,19(1):76-83.
[8] WooP T K,Bruno D W.Fish diseases and disorders:Vol.3 Vieal,Bacterial and Fungal Infections [M].2nd ed.Wallingford:CABI,2011.
[9] Williams M L,Gillaspy A F,Dyer D W,et al.Genome sequence of Edwardsiella ictaluri 93-146,a strain associated with a natural channel catfish outbreak of enteric septicemia of catfish[J].Journal of Bacteriology,2012,194(3):740-741.
[10] 魏畅,李华,叶仕根,等.鮰爱德华菌hcp基因的克隆及重组表达[J].大连海洋大学学报,2013,28(5):424-430.
[11] 张利娟,叶仕根,杨晓宇,等.革兰氏阴性细菌Ⅵ型分泌系统的研究进展[J].大连海洋大学学报,2015,30(6):692-698.
[12] Zheng Jun,Leung K Y.Dissection of a type VI secretion system in Edwardsiella tarda[J].Molecular Microbiology,2007,66(5):1192-1206.
[13] Chen Hao,Yang Dahai,Han Fajun,et al.The bacterial T6SS effector EvpP prevents NLRP3 inflammasome activation by inhibiting the Ca2+-dependent MAPK-jnk pathway[J].Cell Host & Microbe,2017,21(1):47-58.
[14] 熊烈,钱淑岚,朱成云,等.假单胞菌基因敲除方法研究进展[J].化学与生物工程,2017,34(8):5-9.
[15] 万海英,汤华.基因敲除技术现状及应用[J].医学分子生物学杂志,2007,4(1):86-90.
[16] 谢承佳,何冰芳,李霜.基因敲除技术及其在微生物代谢工程方面的应用[J].生物加工过程,2007,5(3):10-14.
[17] 陶果,信吉阁,肖晶,等.基因敲除技术最新研究进展及其应用[J].安徽农业科学,2013,41(29):11605-11608,11644.
[18] 李敏,杨谦.一种高效构建同源重组DNA片段的方法——融合PCR[J].中国生物工程杂志,2007,27(8):53-58.
[19] 刘霞,高鹤,杨琳,等.副溶血性弧菌基因敲除方法的建立及应用[J].中国实验动物学报,2011,19(3):188-192.
[20] Sana T G,Lugo K A,Monack D M.T6SS:the bacterial ‘fight club’ in the host gut[J].PLoS Pathogens,2017,13(6):e1006325.
[21] Wang Xin,Wang Qiyao,Xiao Jingfan,et al.Edwardsiella tarda T6SS component evpP is regulated by esrB and iron,and plays essential roles in the invasion of fish[J].Fish & Shellfish Immunology,2009,27(3):469-477.
[22] Peterson B C,Flora C,Wood M,et al.Vaccination of full-sib channel catfish families against enteric septicemia of catfish with an oral live attenuated Edwardsiella ictaluri vaccine[J].Journal of the World Aquaculture Society,2016,47(2):207-211.