日本鳗鲡Anguilla japonica隶属于鳗鲡目Anguilliformes 、鳗鲡科Anguillidae 、鳗鲡属Anguilla,为典型的降海洄游性鱼类,广泛分布于东亚水域[1],一直是中国主要的养殖和出口水产品[2]。由于其人工繁育遇到技术瓶颈,规模化繁育尚未取得突破,因此,养殖日本鳗鲡苗种仍完全依靠天然捕捞[3]。从20世纪后期至今,鳗苗捕捞一直是中国沿海和河口,特别是长江口的重要渔业生产活动。随着中国社会经济快速发展,水域生态环境总体上不断恶化,捕捞强度不断增强,加之极端气候频发,多种因素叠加影响,导致中国主要河流的日本鳗鲡资源急剧衰退,各大河口的鳗苗资源量也大幅下滑[2,4]。鉴于日本鳗鲡具有重要的经济价值和生态价值,国内外学者针对其种群结构、遗传结构和资源量变化等进行了较为全面的研究[3,5-7]。长江靖江段地处长江中下游近河口区,是长江干流水域的重要组成部分,常年水流平稳,在长江径流和潮汐的双重影响下形成自然流态复杂的水域环境[8],不仅是鱼类栖息和繁衍的良好场所,也是鳗苗溯河洄游的必经之路。目前,针对日本鳗鲡遗传结构的研究大多数偏重于地理种群间的差异[3,7,9-10],而对同一水域不同时段、不同规格的日本鳗鲡遗传结构特征的相关研究报道则较少。
AFLP是一种新型、高效、稳定的分子标记技术,现已应用于鲢Hypophthalmichthys molitrix[11]、虾夷扇贝Patinopecten yessoensis[12]、罗非鱼Oreochromis niloticus[13]等遗传结构的分析。本研究中,通过AFLP标记技术研究长江靖江段不同时间、不同规格日本鳗鲡的遗传多样性特征,旨在了解同一水域、不同规格的日本鳗鲡种群分化程度,丰富日本鳗鲡洄游种群遗传信息,为长江日本鳗鲡的洄游及遗传结构研究提供基础资料。
1 材料与方法
1.1 材料
试验用日本鳗鲡样本共计71尾,标记为C1~C71,均于2015年3月—11月采自长江靖江段
(31°57′23.6952″N、120°7′22.926″E),捕鱼的鱼簖长为50 m,宽为3 m,网目为1 cm(图1),捕获的鱼生物学体长为189~517 mm,平均为302.92 mm,体质量为10~252 g,平均为44.46 g。根据谢刚等[14]对鳗鲡生物学特征的总结,将体长<400 mm的日本鳗鲡样本定义为小规格组(A组),而体长>400 mm的则定义为大规格组(B组)。现场采集新鲜样本的肌肉组织用体积分数为95%的乙醇固定,于冰箱(4 ℃)中密封保存备用。
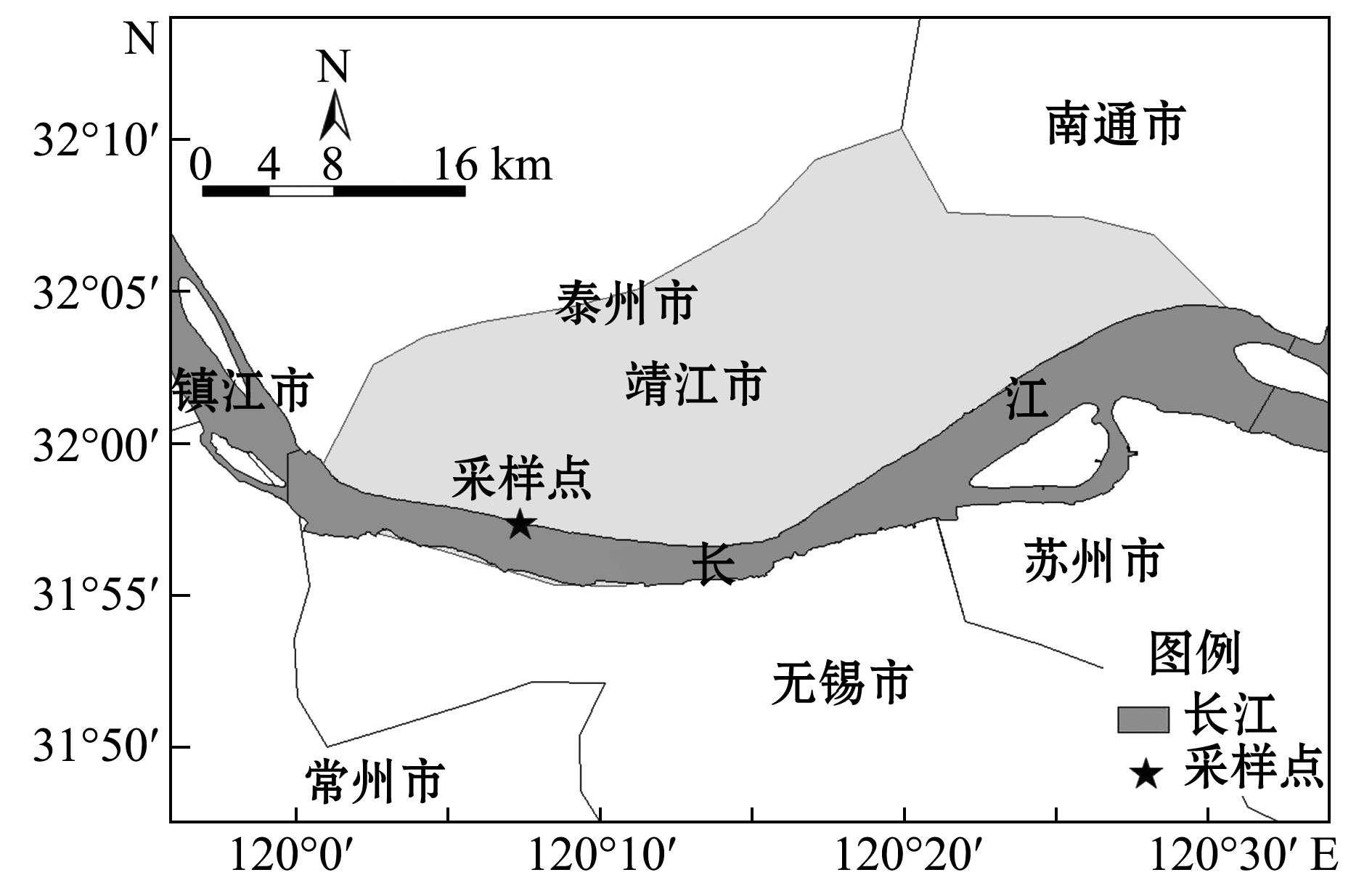
图1 日本鳗鲡采样点
Fig.1 Sampling sites of Japanese eel Anguilla japonica
1.2 方法
1.2.1 基因组DNA的提取 样本DNA均采用CTAB提取法,并依据Vos等[15]和Wang等[16]的方法构建AFLP指纹图库。表1为接头引物、预扩增引物和选择性扩增引物。限制性内切酶为EcoRⅠ和MseⅠ,预试验从8种EcoRⅠ引物(E-AA、E-AC、E-AG、E-AT、E-TA、E-TC、E-TG 和E-TT)和8种MseⅠ引物(M-CAA、M-CAC、M-CAG、M-CAT、M-CTA、M-CTC、M-CTG和M-CTT)共64对引物中筛选特异性较强的9对(E-AAC/M-CAC、E-AAC/M-CAG、E-AAC/M-CTG、E-AAG/M-CTT、E-ACA/M-CAG、E-ACA/M-CAT、E-ACT/M-CTG、E-ACC/M-CAA、E-ACC/M-CTG)进行AFLP分子标记。AFLP试剂盒由北京鼎国昌盛生物技术有限责任公司提供。
1.2.2 限制性酶切及连接反应 酶切体系(20 μL)主要包括:DNA模板4 μL,接头引物1 μL,EcoRⅠ/MseⅠ引物2 μL,10×Reaction buffer 2.5 μL,10 mmol/L ATP 2.5 μL,T4 Ligase 1 μL,ddH2O 7 μL。混合均匀后离心数秒,37 ℃下保温5 h,8 ℃下保温4 h,4 ℃下过夜,最后用15 g/L琼脂糖凝胶电泳检测酶切连接产物。
1.2.3 预扩增反应 预扩增反应体系(25 μL)主要包括:连接DNA 2 μL,预扩增引物1 μL,dNTPs (10 mmol/L)1 μL,10×PCR buffer 2.5 μL,Taq酶0.5 μL, ddH2O 18 μL。预扩增PCR反应条件:94 ℃下预变性2 min; 94 ℃下循环变性30 s,56 ℃下退火复性30 s, 72 ℃下延伸30 s,共进行30个循环;最后在72 ℃下再延伸5 min,于4 ℃下保存。预扩增反应结束后,通过15 g/L琼脂糖凝胶检测预扩增产物。
表1 AFLP接头引物、预扩增引物和选择性扩增引物
Tab.1 Adaptor,pre-,and selective amplification primers in AFLP analysis
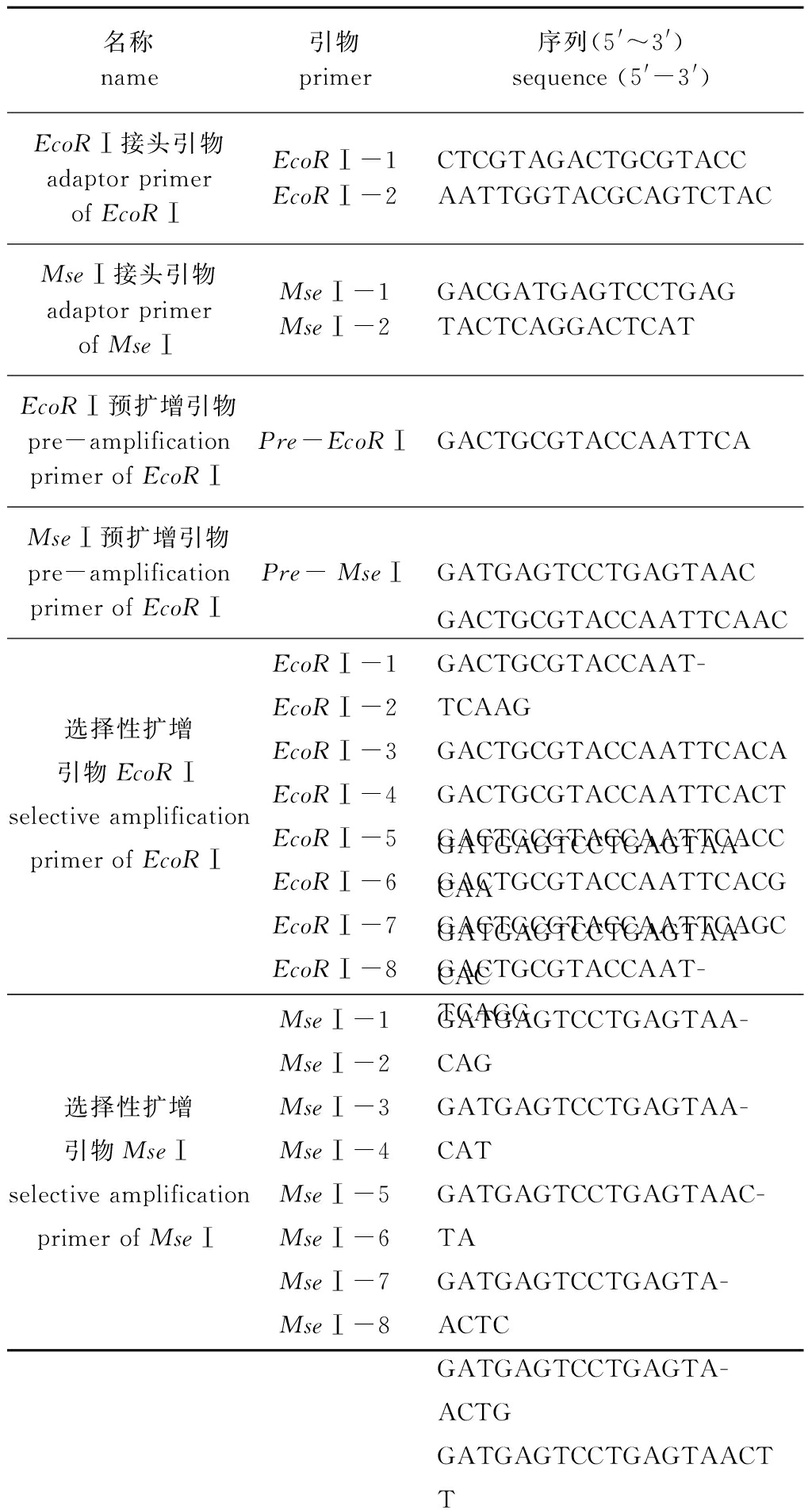
名称name引物 primer序列(5'~3') sequence (5'-3')EcoRⅠ接头引物adaptor primerof EcoRⅠEcoRⅠ-1EcoRⅠ-2CTCGTAGACTGCGTACCAATTGGTACGCAGTCTACMseⅠ接头引物adaptor primerof MseⅠMseⅠ-1MseⅠ-2GACGATGAGTCCTGAGTACTCAGGACTCATEcoRⅠ预扩增引物pre-amplification primer of EcoRⅠPre-EcoRⅠGACTGCGTACCAATTCAMseⅠ预扩增引物pre-amplification primer of EcoRⅠPre- MseⅠGATGAGTCCTGAGTAAC选择性扩增引物EcoRⅠselective amplification primer of EcoRⅠEcoRⅠ-1EcoRⅠ-2EcoRⅠ-3EcoRⅠ-4EcoRⅠ-5EcoRⅠ-6EcoRⅠ-7EcoRⅠ-8GACTGCGTACCAATTCAACGACTGCGTACCAAT-TCAAGGACTGCGTACCAATTCACAGACTGCGTACCAATTCACTGACTGCGTACCAATTCACCGACTGCGTACCAATTCACGGACTGCGTACCAATTCAGCGACTGCGTACCAAT-TCAGG选择性扩增引物MseⅠselective amplification primer of MseⅠMseⅠ-1MseⅠ-2MseⅠ-3MseⅠ-4MseⅠ-5MseⅠ-6MseⅠ-7MseⅠ-8GATGAGTCCTGAGTAA-CAAGATGAGTCCTGAGTAA-CACGATGAGTCCTGAGTAA-CAGGATGAGTCCTGAGTAA-CATGATGAGTCCTGAGTAAC-TAGATGAGTCCTGAGTA-ACTCGATGAGTCCTGAGTA-ACTGGATGAGTCCTGAGTAACT T
1.2.4 选择性扩增反应 将预扩增产物按1∶20的比例用双蒸水稀释后作为选择性扩增模板DNA。选择性扩增体系(25 μL)包括:模板DNA 2 μL,10×PCR buffer 2.5 μL,dNTPs (10 mmol/L)0.5 μL, EcoRⅠ 引物 (10 μmoL/L)1 μL,MseⅠ 引物 (10 μmoL/L)1 μL,Taq酶0.5 μL,ddH2O 17.5 μL 。PCR反应条件: 94 ℃下预变性30 s;65 ℃下循环变性30 s,72 ℃下延伸80 s,共进行12个循环(每一循环退火温度减少0.7 ℃);然后在94 ℃下预变性30 s,55 ℃下退火复性30 s, 72 ℃下延伸80 s,共进行23个循环;最后于4 ℃下保存备用。
1.3 数据处理
根据AFLP的显性标记特征,对所有扩增清晰的条带进行01矩阵统计,有条带记为“1”,无条带记为“0”,从而将构建的指纹DNA图库转换成数据01数字矩阵,利用Popgene 1.32软件[17]统计遗传学参数[18-19],包括位点总数、多态位点数、多态位点频率、有效等位基因比例、Nei’s 基因多样性指数、Shannon多样性指数、种群基因多样性、基因分化系数 (Gst)、基因流系数 (Nm)、遗传相似度和遗传距离等。使用AMOVA软件[20]统计组内和组间差异来源,使用NTSYSpc 2.02软件[21]统计样本组群间的相似系数,并构建样本聚类图。
2 结果与分析
2.1 日本鳗鲡DNA凝胶电泳
用8 g/L琼脂糖凝胶电泳检测样品基因组DNA完整性(附件),电泳结果显示,样品DNA条带清晰,纯度较高,无明显的拖尾迹象,满足AFLP对基因组DNA的标记要求。
2.2 AFLP扩增结果
经预试验筛选,获得条带清晰、多态性好的引物9对,对71尾日本鳗鲡进行AFLP遗传多样性
标记,标记片段大小为70~500 bp。结果显示:9对引物共获得26 259个位点,平均有效等位基因为1.378 2;其中引物组合E-ACA/M-CTC和E-ACC/M-CAA扩增的多态位点比率最高(均为98.15%),引物组合E-ACC/M-CTG扩增的多态位点比率最低(91.2%)(表2)。
表2 AFLP引物检测结果
Tab.2 Primers combination sets of AFLP
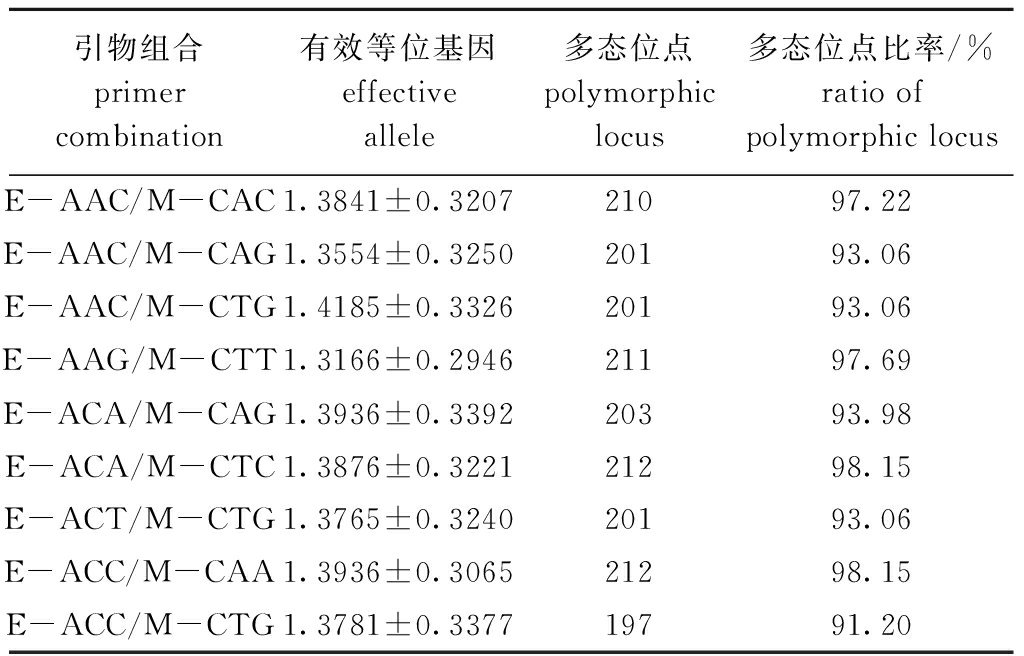
引物组合primer combination有效等位基因effective allele多态位点polymorphic locus多态位点比率/%ratio of polymorphic locusE-AAC/M-CAC1.3841±0.320721097.22E-AAC/M-CAG1.3554±0.325020193.06E-AAC/M-CTG1.4185±0.332620193.06E-AAG/M-CTT1.3166±0.294621197.69E-ACA/M-CAG1.3936±0.339220393.98E-ACA/M-CTC1.3876±0.322121298.15E-ACT/M-CTG1.3765±0.324020193.06E-ACC/M-CAA1.3936±0.306521298.15E-ACC/M-CTG1.3781±0.337719791.20
2.3 日本鳗鲡的遗传多样性
根据不同渔获月份将样本分成9组,有效等位基因、Nei’s基因多样性指数、Shannon多样性指数、多态位点及其比率见表3。其中平均多态位点比率为95.06%,但7、8、9月的多态位点比率为100%;Nei’s基因多样性指数最高为7月(0.297 3±0.123 1),最低为10月(0.259 6±0.127 3),平均为0.277 2;Shannon多样性指数最高为7月(0.463 9±0.149 0),平均为0.433 2。另外,小规格的A组有效等位基因、Nei’s基因多样性指数和Shannon多样性指数均高于大规格的B组。
表3 AFLP增结果及遗传学参数
Tab.3 Amplification and parameters of genetic diversity by AFLP
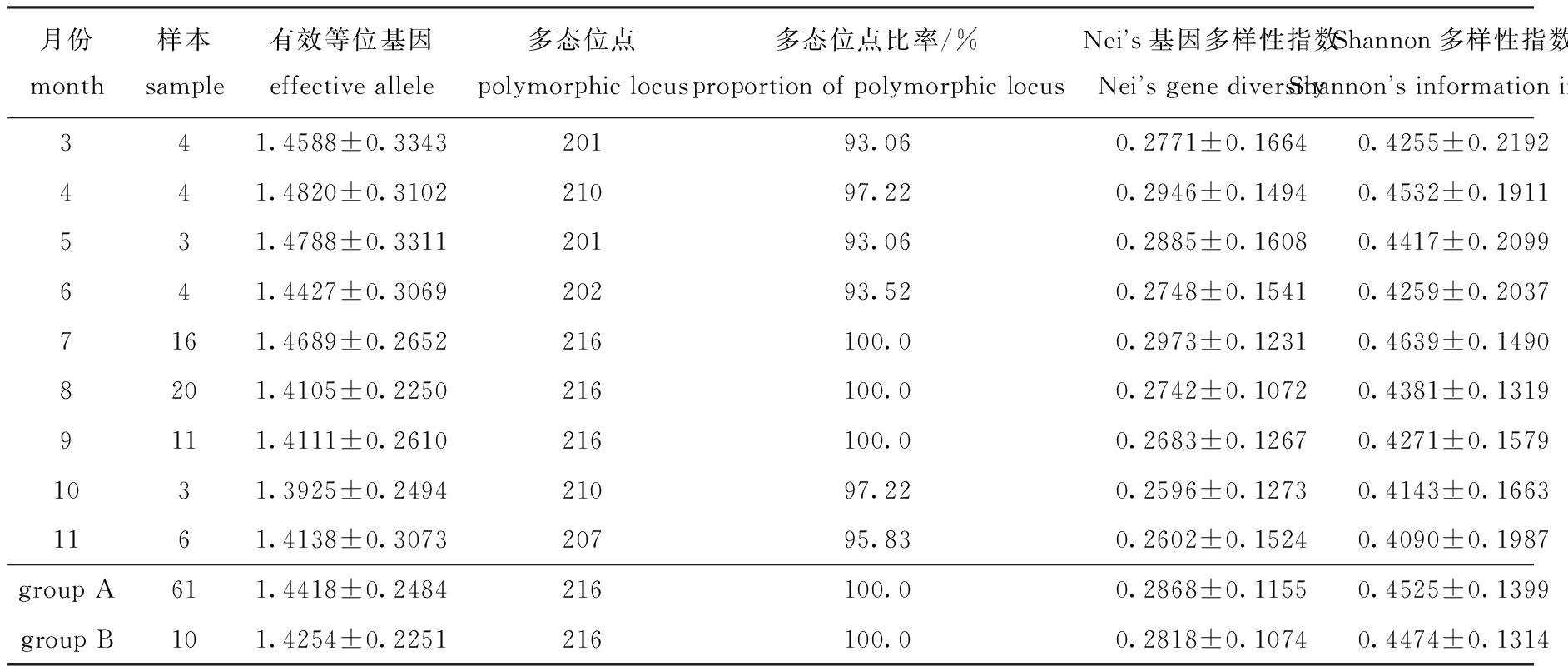
月份month样本sample有效等位基因effective allele多态位点polymorphic locus多态位点比率/%proportion of polymorphic locusNeis基因多样性指数Neis gene diversityShannon多样性指数Shannons information index341.4588±0.334320193.060.2771±0.16640.4255±0.2192441.4820±0.310221097.220.2946±0.14940.4532±0.1911531.4788±0.331120193.060.2885±0.16080.4417±0.2099641.4427±0.306920293.520.2748±0.15410.4259±0.20377161.4689±0.2652216100.00.2973±0.12310.4639±0.14908201.4105±0.2250216100.00.2742±0.10720.4381±0.13199111.4111±0.2610216100.00.2683±0.12670.4271±0.15791031.3925±0.249421097.220.2596±0.12730.4143±0.16631161.4138±0.307320795.830.2602±0.15240.4090±0.1987group A611.4418±0.2484216100.00.2868±0.11550.4525±0.1399group B101.4254±0.2251216100.00.2818±0.10740.4474±0.1314
研究表明:Gst值为0~0.05时,表明种群间遗传分化较弱; Gst为0.05~0.15时,表明种群间遗传分化中等; Gst为0.15~0.25时,表明种群间遗传分化较大;Gst>0.25时, 表明分化极大[9,22]。通过Popgene 32软件分析组间基因分化系数(Gst)和基因流系数(Nm)(表4),结果显示,不同月份之间的基因分化系数为0~0.05,其中8月与9月的基因分化系数最小(0.006 7)。曲若竹等[23]研究表明:种群基因流系数Nm<1时,表明种群间基因交流受限;而Nm> 1时,说明种群间存在基因交流。本研究结果表明,日本鳗鲡不同月份间的Nm均大于1,其中8月与9月的基因流系数最大(74.441 7)。另外,遗传距离结果显示,8月与9月的遗传距离最小(0.005 0);根据体长规格差异将日本鳗鲡分为A、B两组,其个体组成在种群时间变异上并无显著的相关关系,但两组间的遗传距离仅为0.002 6(表5)。用AMOVA软件对变异来源进行分析,结果显示,靖江段日本鳗鲡的遗传变异主要来源于个体间(表 6)。
表4 日本鳗鲡基因分化系数(对角线上方)和基因流(对角线下方)
Tab.4 Parameters of gene differentiation (above diagonal) and Nm (below diagonal) for Japanese eel Anguilla japonica
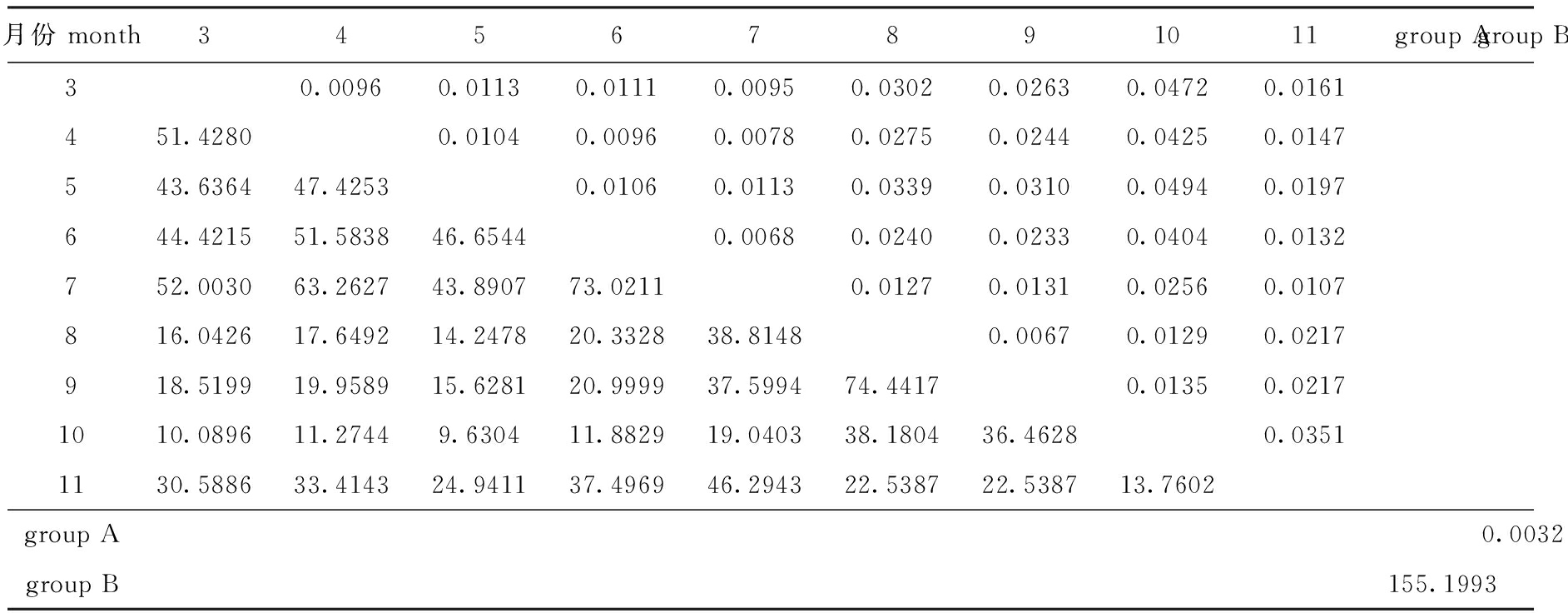
月份 month34567891011group Agroup B30.00960.01130.01110.00950.03020.02630.04720.0161451.42800.01040.00960.00780.02750.02440.04250.0147543.636447.42530.01060.01130.03390.03100.04940.0197644.421551.583846.65440.00680.02400.02330.04040.0132752.003063.262743.890773.02110.01270.01310.02560.0107816.042617.649214.247820.332838.81480.00670.01290.0217918.519919.958915.628120.999937.599474.44170.01350.02171010.089611.27449.630411.882919.040338.180436.46280.03511130.588633.414324.941137.496946.294322.538722.538713.7602group A0.0032group B155.1993
表5 日本鳗鲡基因相似度(对角线上方)及遗传距离(对角线下方)
Tab.5 Genetic identity (above diagonal) and genetic distance (below diagonal) for Japanese eel Anguilla japonica
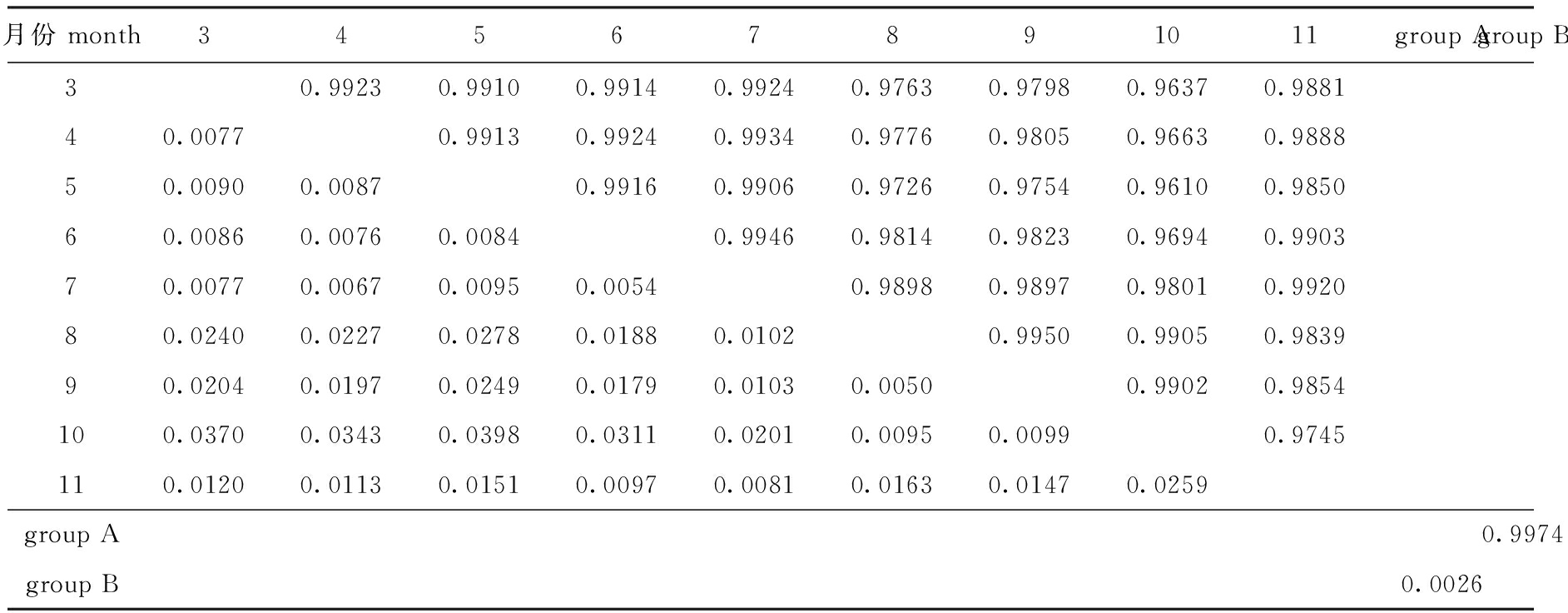
月份 month34567891011group Agroup B30.99230.99100.99140.99240.97630.97980.96370.988140.00770.99130.99240.99340.97760.98050.96630.988850.00900.00870.99160.99060.97260.97540.96100.985060.00860.00760.00840.99460.98140.98230.96940.990370.00770.00670.00950.00540.98980.98970.98010.992080.02400.02270.02780.01880.01020.99500.99050.983990.02040.01970.02490.01790.01030.00500.99020.9854100.03700.03430.03980.03110.02010.00950.00990.9745110.01200.01130.01510.00970.00810.01630.01470.0259group A0.9974group B0.0026
表6 日本鳗鲡的AMOVA分析
Tab.6 Analysis of molecular variance (AMOVA) in Japanese eel Anguilla japonica

分组group变异来源source of variation自由度degree of freedom方差总和sum of sguare deviation变异组成variance component所占比例%percent3—11月群体间 among populations81678.380948.201318.983-11 month群体内 within populations623593.3387205.726781.02A/B组群体间 among populations1361.0967.53043.15group A/B 群体内 within populations69361.096231.699996.85
2.4 组间和个体之间的进化树
利用NTSYSpc 2.02软件构建系统进化树,结果显示,9个组群的日本鳗鲡明显地聚为两支,其中8、9、10月份的组群聚为一支,其他月份的组群聚为另一支(图2)。针对日本鳗鲡个体构建的系统进化树也显示,日本鳗鲡个体同样明显地聚为两支,一支有34个个体,另一支有37个个体(图3)。
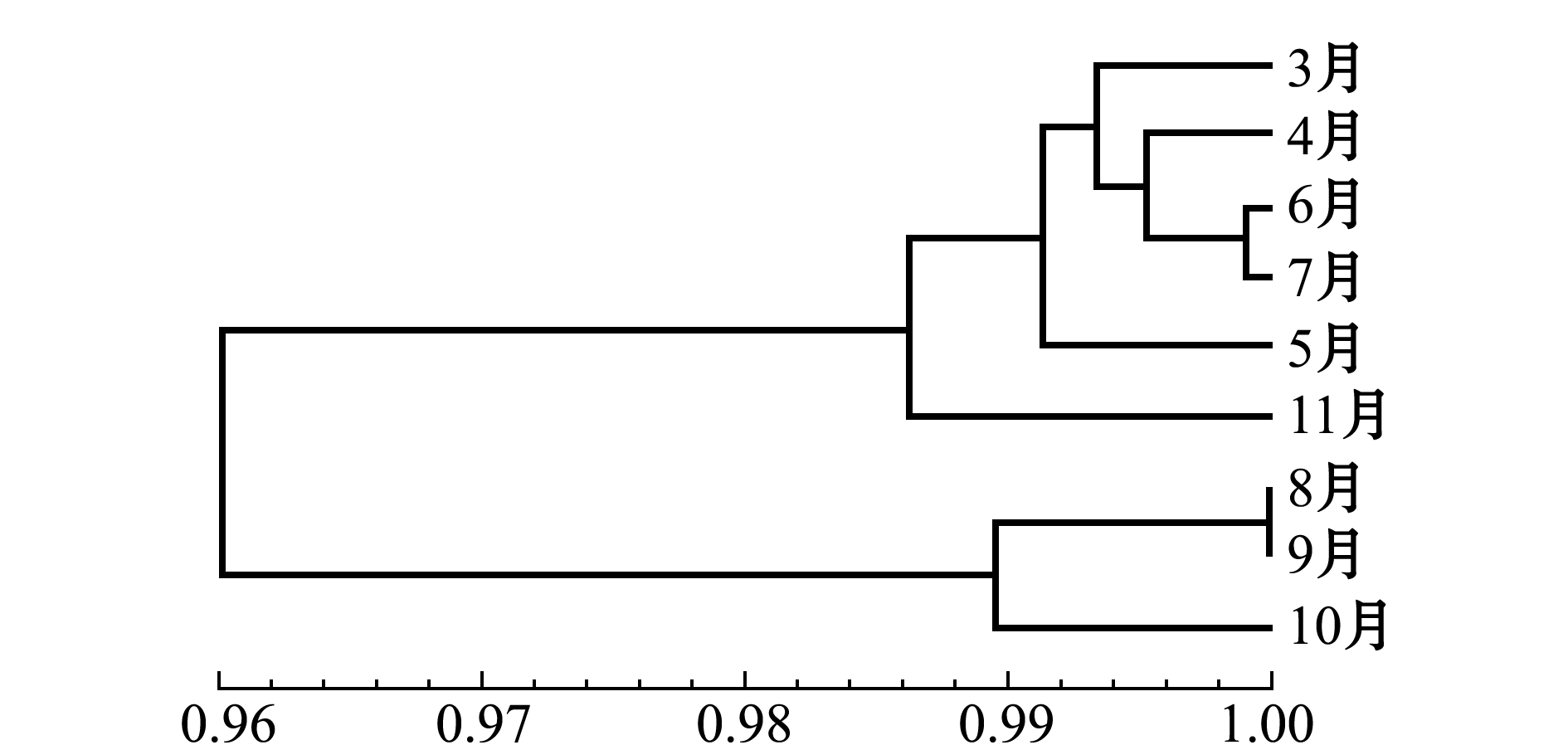
图2 不同月份日本鳗鲡系统进化树
Fig.2 NTSYS dendrogram of Japanese eel Anguilla japonica in different months
3 讨论
3.1 日本鳗鲡遗传多样性水平分析
本研究中采用的AFLP标记技术是一种新型分子标记方法,与RFLP和RAPD技术相比,其检测范围更广、灵敏度更好、多态位点更加丰富,因而已广泛地应用于多种水生生物遗传多样性的研究中[7-8,24-27]。
诸多学者采用AFLP对日本鳗鲡遗传多样性的研究,主要集中于不同地理种群,而针对同一水域不同时段、不同规格的日本鳗鲡遗传多样性的研究极少。龚小玲等[3]研究发现,长江口日本鳗鲡不同规格间存在相当程度的遗传距离和遗传分化,说明该水域日本鳗鲡遗传多样性水平较为丰富;张辛等[7]研究发现,长江口玻璃鳗与闽江流域成鳗(由玻璃鳗养殖而成)两个群体具有明显的群体分化现象,且东海日本鳗鲡的遗传多样性水平相对较高。而本研究中日本鳗鲡AFLP的多态位点比率平均值>95%,高于花鳗鲡Anguilla marmorata[9]和长江口日本鳗鲡(86.36%)[3];Shannon多样性指数平均值>0.4,说明长江靖江段日本鳗鲡的遗传信息同样丰富,种群遗传多样性水平较高,与龚晓玲等[3]、张辛等[7]对长江口日本鳗鲡的研究结果基本一致。长江靖江段位于长江下游距河口区约160 km的长江干流,是长江及东海日本鳗鲡洄游的必经之路。优越的地理位置为日本鳗鲡的资源补充提供了便利,推测是靖江段日本鳗鲡种群遗传多样性维持较高水平的一个重要原因。程兴华[28]在研究靖江段沿岸群居型鱼类多样性时指出,靖江上下游均有沙洲分布,形成了营养盐丰富的独特自然环境,复杂的流态生境为水生生物提供多样化的栖息地、洄游通道、产卵场和索饵场等。这一研究结果客观地阐述了靖江段独特的生态环境(水位、岸线潮水和饵料生物等),为日本鳗鲡提供了良好的洄游通道和充足饵料,也是靖江段日本鳗鲡种群遗传多样性维持在较高水平的一个重要因素。

图3 日本鳗鲡个体系统进化树
Fig.3 NTSYS dendrogram in individuals of Japanese eel Anguilla japonica
3.2 日本鳗鲡遗传结构及聚类分析
靖江段日本鳗鲡的基因分化系数为0<Gst<0.05,表明种群自身可抵制遗传漂变的发生;基因流系数Nm>1且遗传距离平均值为0.02,表明日本鳗鲡种群内的基因交流不会随捕获时间而停止,彼此间的遗传联系依然紧密,种群分化程度维持在较低水平。这也预示着个体间存在着随机交配的可能性,不利于种群种质资源的保护。有学者早在2006年的研究中提出,应将中国东海水域附近的日本鳗鲡分成3个不同组群,说明同一水域的同一种群间存在遗传结构差异,这与本研究中的聚类结果相一致[7]。
本研究中9个分组和71个个体的系统进化树均明显地聚为两支,说明靖江段日本鳗鲡群体内存在不同亲缘关系的两个群体。综合基因分化程度及基因流系数可以推测,日本鳗鲡群体存在亲鱼产卵后死亡的可能性[7,29],表现为群体分化程度较低,基因间存在信息交流,但群体间亲缘关系逐渐分离。本研究中进化树聚类结果显示,8、9、10月日本鳗鲡3个组聚为一支,其他组聚为一支,可能是因为同一繁殖种群最佳苗汛期内的幼鳗个体由于受到洋流等自然因素影响[1],不能同时逆流而上到达靖江水域而表现为时间上的延续。虽然长江流域的潮水规模不及海洋暖流,但丰、枯水季节的潮水变化差异较大,且靖江地处长江下游,受上游径流及下游潮汐的双重影响较为显著,河相关系复杂[30],这也是干扰幼鳗溯河洄游的一个关键因素,这与本研究结果相符合。有数据显示,8月和9月是长江三峡大坝主要的泄洪期[31-32],使得长江中下游水位升高,而该时段渔获的样本数量显著地高于其他月份且聚为一支,这种现象一定程度上预示着靖江段不同时段日本鳗鲡种群遗传结构受不同时间长江水流河相关系的综合影响。
此外,AMOVA分析显示,该群体的变异主要来源于个体之间,组群间无显著差异。由此可见,靖江段日本鳗鲡个体在不同时段均存在信息交流,而样本规格对改变种群遗传结构并无贡献,不会造成种群遗传分化,这与龚晓玲等[3]的结果不一致,不排除靖江段不同规格的日本鳗鲡来自同一繁殖群体的可能性。谢刚等[14]对鳗鲡繁殖生物学特性的研究表明,体长<400 mm的全为雌性, 介于400~700 mm则雌、雄均有,本研究结果则在一定程度上验证了该结论,表明同一繁殖群体分批洄游的生态习性。同时,不同规格的群体在种群遗传结构上并无差异,说明日本鳗鲡同一繁殖群体在某一水域存在“定点洄游”的可能性,这更有助于日本鳗鲡种群遗传结构的研究。
综上所述,靖江段日本鳗鲡的遗传分化程度较低,但种群遗传多样性水平相对较高,这与该江段地理位置及生态环境密切相关。本研究成果有助于掌握靖江段,甚至整个长江流域日本鳗鲡的种群分化程度、遗传结构和种群多样性水平,丰富日本鳗鲡种群的遗传结构,为其科学研究提供理论补充,同时也为日本鳗鲡种质资源的保护提供科学依据。