秀丽白虾Exopalaemon modestus隶属于甲壳纲Crustacea、十足目Decapoda、长臂虾科Palaemonidae、长臂虾亚科Palaemoninae、白虾属Exopalaemon,在中国分布广泛,是大中型淡水湖泊的虾类优势种[1]。秀丽白虾具有分布广、产量高、壳薄味美、营养丰富、经济价值高的特点,在中国淡水湖泊渔业中占有非常重要的地位。作为“太湖三宝”之一,秀丽白虾在全太湖均有分布,年产量占太湖虾类总产量的50%以上[2]。市场供应的秀丽白虾基本上来自野生资源,人工育苗和池塘养殖还处于试验探索阶段[3-4]。关于秀丽白虾的研究主要集中在食性[5]、生长[6]、耗氧率[7]、种群形态差异[8]、繁殖生物学[9]和资源变动[10]等方面。而关于其分子生物学、代谢功能基因研究[11],以及基于RAPD[12]、AFLP标记[13]的遗传多样性分析较少,但有关不同地理种群秀丽白虾线粒体DNA 16S rRNA基因序列的研究目前尚未见报道。
与核基因相比,动物线粒体DNA为母系遗传,且具有进化速度快、不同基因片段核苷酸替代速率不同等特点,被广泛应用于物种鉴定、种群遗传和系统发育等研究[14]。线粒体16S rRNA基因由于进化速度适中,可用通用引物扩增,当前被广泛应用于鱼、虾、贝的种群差异及分化研究[15-17]。本研究中,以太湖、鄱阳湖和兴凯湖3个地理种群秀丽白虾作为研究对象,对其线粒体16S rRNA基因序列及种群遗传分化进行初步研究,旨在为秀丽白虾种质资源保护提供基础数据。
1 材料与方法
1.1 材料
试验用秀丽白虾于2016年5—8月采集自太湖、鄱阳湖和兴凯湖,每个种群采集约200尾个体。采样网具为地龙网。样品采集后立即用体积分数为95%的乙醇固定,带回实验室备用。
试验所用试剂购自大连宝生物科技有限公司。
1.2 方法
1.2.1 基因组DNA的提取 DNA抽提采用MiniBEST Universal Genomic DNA Extraction Kit(TaKaRa),具体方法参照说明书。抽提所得DNA于-20 ℃下保存备用。
1.2.2 PCR 扩增、电泳与测序 用于扩增的引物[16]分别为16SA:5′CGC CTG TTTAAC AAA AACAT 3′,16SB:5′CCG GTT GAA CTC AGA TCA3′。
引物由铂尚生物技术(上海)有限公司合成。
PCR扩增在ABI Veriti 96 Well Thermal Cycler多重控温PCR仪上进行。扩增反应体系(共20 μL):Premix (TaKaRa TaqTM Version 2.0 plus dye) 10 μL,16SA、16SB引物(20 mmol)各1 μL,模板DNA(20 ng/μL) 4 μL,ddH2O 4 μL。反应条件为:94 ℃下预变性3 min;94 ℃下变性40 s,53 ℃下退火40 s,72 ℃下延伸50 s,共进行35个循环;最后在72 ℃下再延伸8 min。
PCR扩增产物经12 g/L琼脂糖凝胶检测,电泳缓冲液为1×TBE(pH 8.0),电压为5 V/cm,常温下电泳,用E.B染色,相对分子质量标记为DL 2000 DNA Ladder Marker。PCR产物由铂尚生物技术(上海)有限公司纯化测序,测序引物与扩增引物相同。共成功测序秀丽白虾样品129尾,其中,太湖种群(TH)54尾,鄱阳湖种群(PYH)42尾,兴凯湖种群(XKH)33尾。
1.3 数据处理
采用Clustal X1.81软件对测得的序列进行DNA Star、序列拼接、比对。采用MEGA 3.1软件完成碱基组成、变异位点、转换(si)与颠换(sv)比值(r=si/sv)、遗传距离的分析。采用DNASP 5.10软件分析单倍型。采用Arlequin 3.1软件中的分子变异分析(Analysis of molecular variance,AMOVA)估算种群间和种群内遗传变异、遗传分化系数(Gst)和基因流(Nm)。
2 结果与分析
2.1 秀丽白虾mtDNA 16S rRNA基因
秀丽白虾样本经PCR扩增后得到单一清晰的16S rRNA基因片段,长度约580 bp(图1)。
测序得到的序列经Clustal X比对和人工校对后,经NCBI中Blast比对,获得486 bp的同源片断,该片断与秀丽白虾线粒体16S rRNA部分序列(DQ 194971.1)一致性高达99%,确认所测序列为秀丽白虾线粒体16S rRNA的基因片断。

注:M为DL2000 DNA Marker;1~10分别为10个秀丽白虾样本
Note:M,DL2000 DNA Marker;1-10, 10 samples of Siberian prawn Exopalaemon modestus
图1 秀丽白虾mtDNA16S rRNA基因片断PCR扩增电泳图
Fig.1 PCR amplification electrophoresis of mtDNA 16S rRNA gene fragment of Siberian prawn Exopalaemon modestus
2.2 序列变异及单倍型分析
从129尾秀丽白虾样本中共检出8种单倍型,单倍型Ⅰ序列见表1。
从所有样本的486 bp序列中,共检测到10个变异位点(占所测序列的2.06%),分别位于第95、114、158、159、163、295、335、444、483和484位点上(表2)。其中,第484位为颠换位点(transversional pairs),其余9个为转换位点(transitional paris)。转换位点个数和颠换位点个数的比值(r=si/sv)为9,与不同群体鲇Silurus asotus的16S rRNA基因序列的转换、颠换位点数比值(r=9.14∶1)[15]接近。
表1 秀丽白虾mtDNA 16S rRNA基因序列(单倍型Ι)
Tab.1 HapⅠsequence of mtDNA 16S rRNA gene of Siberian prawn Exopalaemon modestus
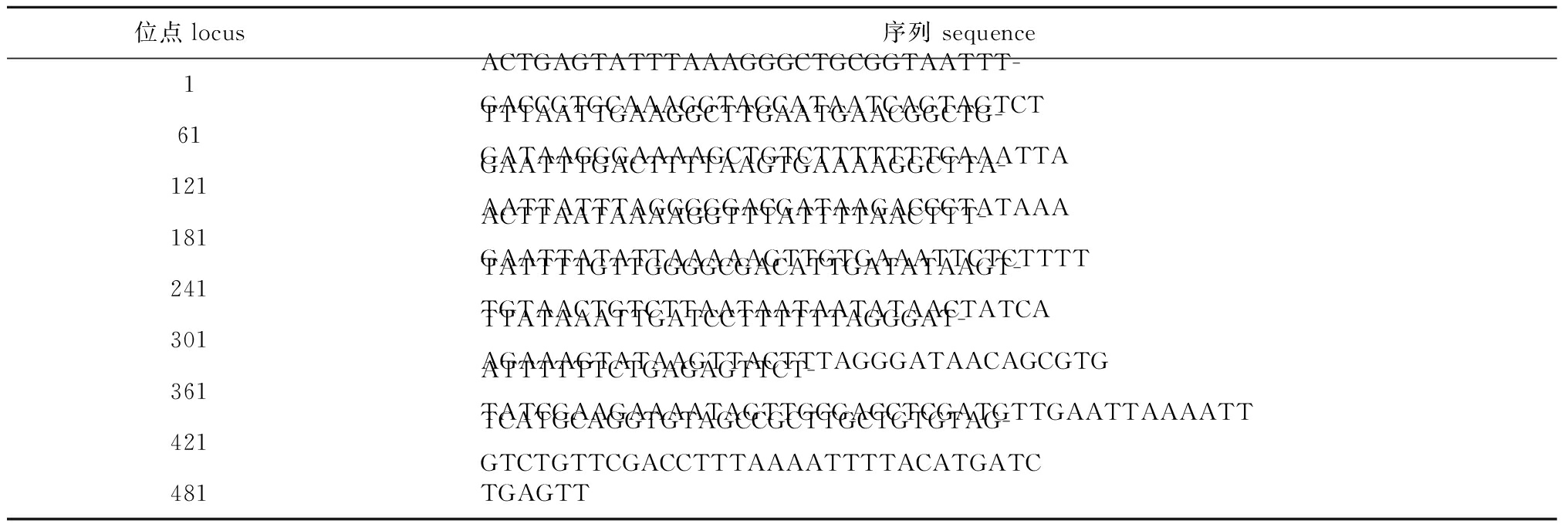
位点locus序列 sequence 1ACTGAGTATTTAAAGGGCTGCGGTAATTT-GACCGTGCAAAGGTAGCATAATCAGTAGTCT 61TTTAATTGAAGGCTTGAATGAACGGCTG-GATAAGGGAAAAGCTGTCTTTTTTTCAAATTA121GAATTTGACTTTTAAGTGAAAAGGCTTA-AATTATTTAGGGGGACGATAAGACCCTATAAA181ACTTAATAAAAGGTTTATTTTAACTTT-GAATTATATTAAAAAGTTGTGAAATTCTCTTTT241TATTTTGTTGGGGCGACATTGATATAAGT-TGTAACTGTCTTAATAATAATATAACTATCA301TTATAAATTGATCCTTTTTTAGGGAT-AGAAAGTATAAGTTACTTTAGGGATAACAGCGTG361ATTTTTTCTGAGAGTTCT-TATCGAAGAAAATAGTTGCGACCTCGATGTTGAATTAAAATT421TCATGCAGGTGTAGCCGCTTGCTGTGTAG-GTCTGTTCGACCTTTAAAATTTTACATGATC481TGAGTT
表2 秀丽白虾mtDNA 16S rRNA基因多态位点及单倍型
Tab.2 Polymorphic loci and haplotypes in mtDNA 16S rRNA gene of Siberian prawn Exopalaemon modestus
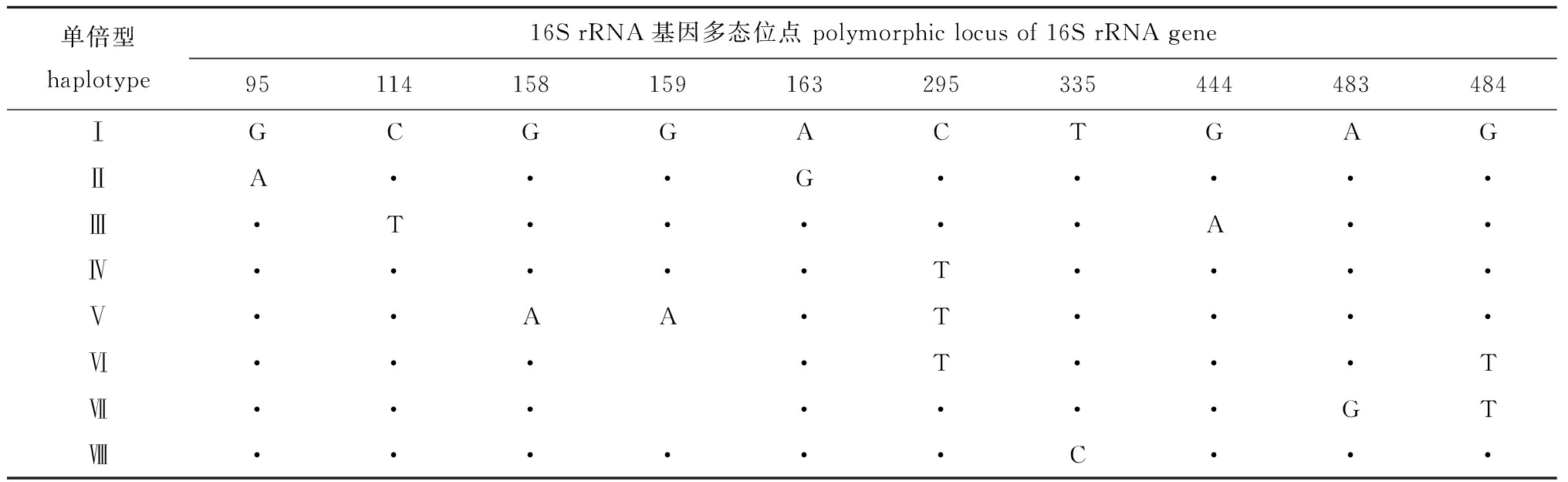
单倍型haplotype16S rRNA基因多态位点 polymorphic locus of 16S rRNA gene95114158159163295335444483484ⅠGCGGACTGAGⅡA···G·····Ⅲ·T·····A··Ⅳ·····T····Ⅴ··AA·T····Ⅵ····T···TⅦ·······GTⅧ······C···
注:·表示碱基相同
Note: · indicate the same base
从单倍型分布(表3)可见:太湖种群出现5种单倍型,鄱阳湖种群出现4种单倍型,兴凯湖种群出现1种单倍型(Ⅳ);其中,单倍型Ⅳ为第一优势单倍型,占样本总数的41.86%,其次为单倍型Ⅰ,占样本总数的37.20%;3个种群间未有共享单倍型,但2个种群间有共享单倍型,太湖和鄱阳湖种群共享单倍型Ⅰ,鄱阳湖和兴凯湖种群共享单倍型Ⅳ。
表3 秀丽白虾3个种群线粒体16S rRNA基因单倍型分布
Tab.3 Distribution of haplotypes in mtDNA 16S rRNA gene of three populations of Siberian prawn Exopalaemon modestus
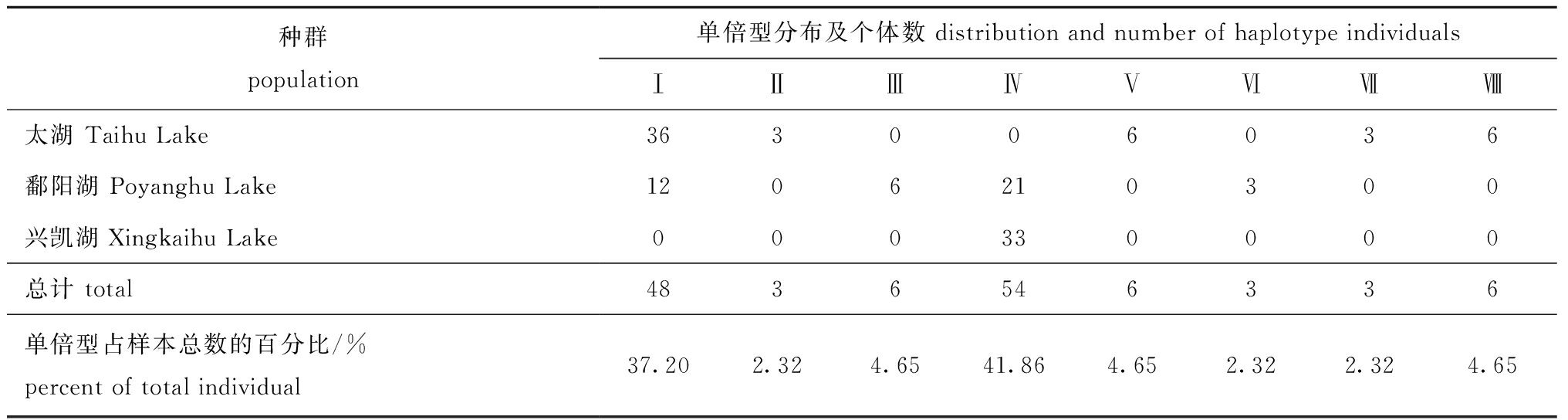
种群population单倍型分布及个体数 distribution and number of haplotype individualsⅠⅡⅢⅣⅤⅥⅦⅧ太湖 Taihu Lake363006036鄱阳湖 Poyanghu Lake1206210300兴凯湖Xingkaihu Lake000330000总计 total4836546336单倍型占样本总数的百分比/% percent of total individual37.202.324.6541.864.652.322.324.65
2.3 种群遗传多样性及中性检验
狭义的遗传多样性是指生物种内(包括同一种群内及不同种群间)基因的变化。多态位点数(S)、单倍型多样性(Hd)、核苷酸多样性(Pi)和平均核苷酸差异数(K)常被用来度量种群遗传多样性,S、Hd、Pi和K值越大,表明种群的遗传多样性越丰富。3个种群秀丽白虾的遗传多样性及中性检验(Tajima’s D)结果表明(表4):兴凯湖种群只有一种单倍型,多态性参数(S、Hd、Pi和K)均为0;太湖种群的多态位点数S大于鄱阳湖种群,单倍型多样性Hd略低于鄱阳湖种群,3个种群的核苷酸多样性Pi均较低;太湖种群的平均核苷酸差异数K为1.366 01,鄱阳湖种群的K值为1.197 80;虽然太湖种群、鄱阳湖种群的Tajima’s D值均为负值,但在统计学上均未达到显著偏离中性进化的假说(P>0.1)。
表4 秀丽白虾3个种群的遗传多样性及Tajima’s D检验
Tab.4 Genetic diversity index and Tajima’s D test of three populations of Siberian prawn Exopalaemon modestus
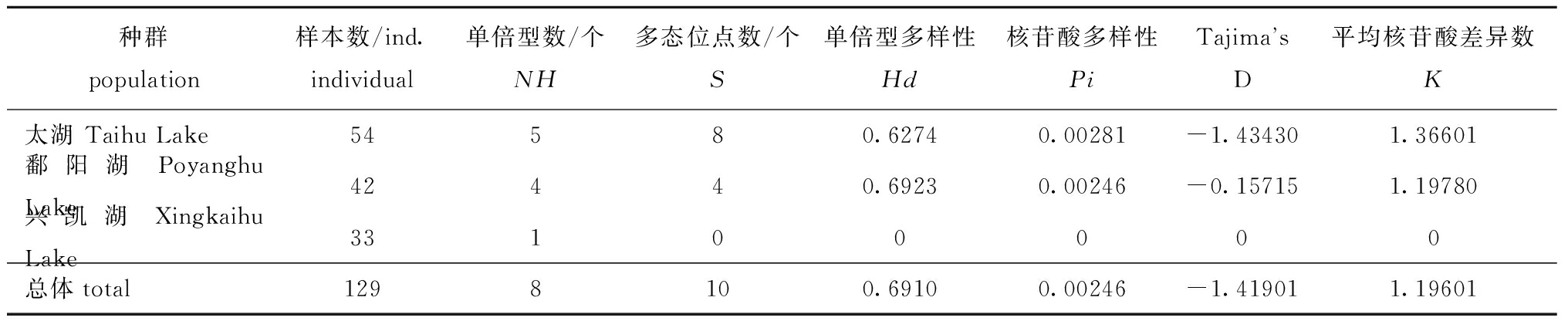
种群population样本数/ind.individual单倍型数/个NH多态位点数/个S单倍型多样性Hd核苷酸多样性PiTajimas D平均核苷酸差异数K太湖 Taihu Lake54580.62740.00281-1.434301.36601鄱阳湖 Poyanghu Lake42440.69230.00246-0.157151.19780兴凯湖Xingkaihu Lake33100000总体total1298100.69100.00246-1.419011.19601
2.4 种群遗传距离及AMOVA分析
基于Kimura 2-parameter的种群间和种群内遗传距离显示,太湖种群和兴凯湖种群间遗传距离最小(0.001 62),鄱阳湖种群内的遗传距离最大(0.002 83)(表5)。
将3个秀丽白虾种群归为一组进行AMOVA分析,结果显示,种群间的遗传变异为29.58%,种群内的遗传变异为70.42%,遗传分化系数Gst为0.295 8,基因流Nm为0.595 2。F0.01(2,126)=4.780,F=6.897> F0.01(2,126),3个种群间遗传存在极显著性差异(P<0.01)(表6)。
表5 秀丽白虾3个种群16S rRNA基因遗传距离
Tab.5 Genetic distance among three populations based on 16S rRNA gene of Siberian prawn Exopalaemon modestus
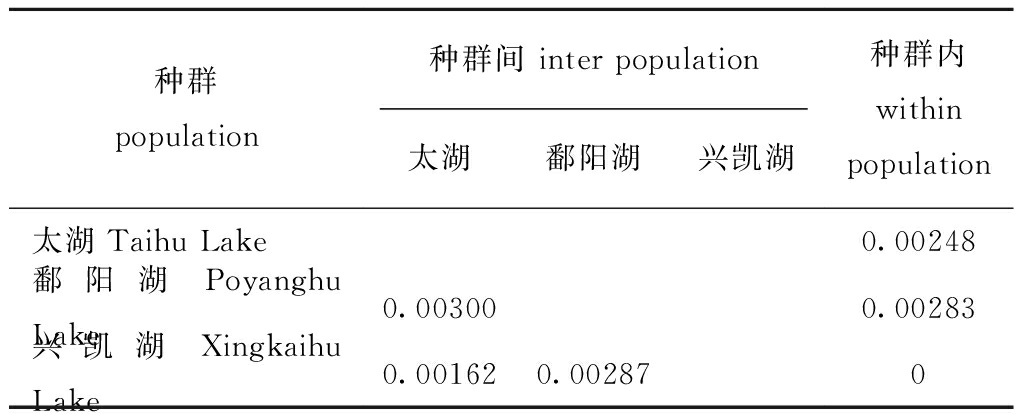
种群population种群间 inter population太湖鄱阳湖兴凯湖种群内within population太湖Taihu Lake0.00248鄱阳湖Poyanghu Lake0.003000.00283兴凯湖Xingkaihu Lake0.001620.002870
表6 秀丽白虾3个种群线粒体16S rRNA基因序列AMOVA分析
Tab.6 Analysis of molecular variance (AMOVA) of mtDNA 16S rRNA gene of three populations of Siberian prawn Exopalaemon modestus
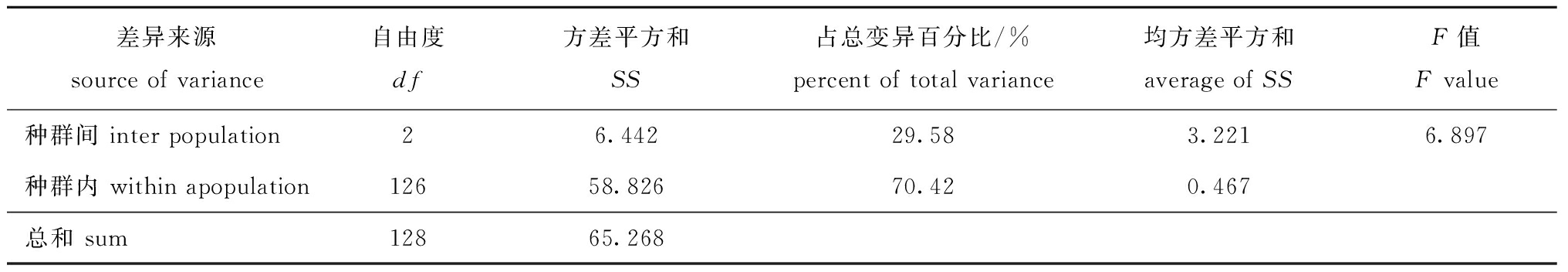
差异来源source of variance自由度df方差平方和SS占总变异百分比/%percent of total variance均方差平方和average of SSF值F value 种群间 inter population26.44229.583.2216.897种群内 within apopulation12658.82670.420.467总和 sum12865.268
3 讨论
3.1 基于16S rRNA基因的序列变异
动物mtDNA仅编码少量蛋白质,结构相对简单。一方面,mtDNA呈母系遗传,且进化速度快,较快的进化速率使种群间(及近缘种间)的遗传变异更易被检测;另一方面,mtDNA不同区域的变异率存在差异,即使是同一段基因片断,不同的物种其遗传变异解析能力也不同。因此,mtDNA序列分析已成为研究种群遗传结构及变异的一种有效方法[18]。
虽然在一些物种中,16S rRNA 序列被证明在属水平上有相对较高的变异水平和较好的分辨能力,但绝大部分的研究均表明,16S rRNA序列保守性较COⅠ基因序列高,COⅠ基因比16S rRNA基因更适合于分析种群遗传变异及物种鉴别[19-20]。本研究中,通过对3个群体129尾秀丽白虾个体16S rRNA基因的序列测定,发现秀丽白虾不同种群间基于16S rRNA基因的序列变异较小,486 bp 序列中共检测到8种单倍型和10个变异位点(占所测序列的2.06%),变异位点比例低于鲇的5个野生群体16S rRNA基因序列(变异位点占所测序列的4.40%)[15]。
目前,关于秀丽白虾基于线粒体的种群遗传多样性研究报道较少,仅见陈杰[21]基于COⅠ基因对来自5个种群的25个个体的秀丽白虾进行了初步研究,与其研究结果相比,本研究中的变异位点数和单倍型数均低于基于COⅠ基因的秀丽白虾的遗传变异,印证了16S rRNA基因比COⅠ基因更保守的结论。
3.2 秀丽白虾单倍型多样性和核苷酸多样性
单倍型多样性(Hd)与核苷酸多样性(Pi)常被用来评估种群遗传多样性及推断种群的进化历史,当单倍型多样性较高而核苷酸多样性较低(Hd>0.5,Pi<5%)时,可能是由于受瓶颈效应后种群数量的迅速扩张导致[22]。高单倍型多样性而低核苷酸多样性的情况在鱼类及虾蟹类中较为常见,如基于线粒体Cyt b基因序列的7个群体南海短尾大眼鲷Priacanthus macracanthus的单倍型多样性为0.813 0~0.901 2,核苷酸多样性为0.004 0~0.005 3[23];长吻鮠Leiocassis longirostris野生群体平均单倍型多样性为0.973 6±0.007 0,平均核苷酸多样性为0.008 7±0.001 5,而养殖群体的单倍型多样性为0.886 7±0.001 3, 核苷酸多样性为0.005 6±0.001 3[24];中国南海野生斑节对虾Penaeus monodon 5个野生群体的单倍型多样性为0.521 1~0.721 1,核苷酸多样性为0.0043 5~0.0116 8[25];戴艳菊等[26]对三疣梭子蟹Portunus trituberculatus 4个野生群体的研究表明,基于COⅠ基因片段的4个野生群体的平均单倍型多样性为0.608,平均核苷酸多样性为0.001 09。
本研究中,秀丽白虾种群的单倍型多样性为0.691 0,而其核苷酸多样性仅为0.002 46,这说明本研究中的秀丽白虾种群遗传多样性较高,该种群在进化中可能经历了瓶颈效应,是从一个有效种群数量较小的群体快速扩张而来,种群单倍型多样性较丰富, 但还未能积累足够的核苷酸变异。
3.3 秀丽白虾种群中性检验及3种群间的遗传分化
中性理论(neutral theory),又称中性突变与随机漂移理论(neutral mutation and random genetic drift theory),是分子进化的重要理论之一。该理论认为:大部分对种群的遗传结构与进化有贡献的分子突变在自然选择的意义上都是中性或近中性的,因而自然选择对这些突变并不起作用;中性突变的进化是随机漂移的过程,或被固定在种群中,或消失。Tajima[27]设计了D值检验(Tajima’s D),通过统计学模型来验证中性突变假说。
应用Tajima’s D值中性检验推测种群曾经历的历史时,如果Tajima’s D值为负,且在统计学上达到显著标准,则说明群体中存在许多低频率的等位基因(稀有等位基因),可能预示着被研究种群曾经经历过一个定向选择和规模扩张的历史[27]。本研究中,太湖、鄱阳湖种群Tajima’s D值为负(表4),若将包括兴凯湖在内的3个种群看作一个整体,则Tajima’s D值仍为负,但各种群在单独进行中性检验时,并没有达到显著水平(P>0.1),说明秀丽白虾可能在历史上发生过扩张,但基于16S rRNA基因的秀丽白虾种群并未有显著偏离中性进化模型。
遗传分化系数是反映各亚群间遗传分化的重要指标。由种群AMOVA分析可知,3个种群间的遗传分化系数为0.2958,基因流为0.595 2,检验表明,3个种群间存在极显著分化,这和基于AFLP分子标记的秀丽白虾3个野生群体间呈现中高度遗传分化(遗传分化系数为0.252)的结果基本一致[13]。另外,太湖种群和鄱阳湖种群分别发现5种和4种单倍型,兴凯湖种群仅有一种单倍型Ⅳ,推测兴凯湖种群在进化过程中由于其独特的生态环境(冷水性湖泊)导致某些稀有等位基因丢失,单倍型多样性缺乏。
综上,本研究结果初步揭示兴凯湖秀丽白虾种群与太湖、鄱阳湖种群间的基因交流较少,种群间已经出现了极显著分化。一个物种抵御不良环境的能力和进化潜力有赖于种群的遗传结构,也取决于种群内遗传变异的大小[28]。总体来说,秀丽白虾虽然为自由游泳生活,但个体较小,迁移活动范围有限,地理隔离较明显,不同地理种群间分化程度较高。
[1] 谈奇坤,董秀英,吴家瑾.巢湖秀丽白虾渔业生物学初步调查[J].动物学杂志,1982,17(6):4-7.
[2] 王克文,余方德,嵇发根.湖州市志[M].北京:昆仑出版社,1999:702-704.
[3] 胡廷尖,周志明,黄鲜明,等.秀丽白虾繁殖特性与人工育苗技术的研究[J].中国水产,2001(1):46-47.
[4] 胡廷尖,叶金云,周志明,等.秀丽白虾池塘养殖技术[J].科学养鱼,2002(2):31.
[5] 施炜纲,严小梅,邴旭文.太湖秀丽白虾生物学与食性的研究[J].湖泊科学,1995,7(1):69-76.
[6] 温周瑞,谢平.太湖秀丽白虾生长研究[J].水产科技情报,2013,40(3):139-142.
[7] 姜叶琴,韩志萍,杨万喜.日本沼虾和秀丽白虾耗氧率的比较[J].水产科学,2003,22(2):15-17.
[8] 张敏莹,徐东坡,刘凯,等.中国五大湖秀丽白虾地理种群的形态差异与判别分析[J].长江流域资源与环境,2010,19(6):678-684.
[9] 陈思宝,廖传松,赵修江,等.三峡水库秀丽白虾生长与繁殖生物学特征研究[J].水生生物学报,2015,39(5):989-996.
[10] 温周瑞,张大文,谢平,等.太湖日本沼虾、秀丽白虾现存量的周年动态研究——以梅梁湾、贡湖湾为例[J].长江流域资源与环境,2011,20(9):1035-1039.
[11] 王敏,吴萍,叶元土,等.秀丽白虾4种C型凝集素基因的克隆、组织表达与进化分析[J].水产学报,2013,37(11):1632-1644.
[12] 张敏莹,徐东坡,段金荣,等.太湖秀丽白虾遗传多样性的RAPD分析[J].中国农学通报,2008,24(3):459-462.
[13] 许志强,葛家春,曹廷,等.江苏3个秀丽白虾野生群体遗传多样性的AFLP分析[J].江苏农业学报,2009,25(1):160-164.
[14] 陈双雅,王嘉鹤,陈伟玲,等.16S rRNA基因和COI基因序列分析在石斑鱼物种鉴定中的应用[J].生物技术通报,2012,28(10):124-130.
[15] 王庆容,宋培勇,唐立俊,等.不同地理群体的鲇线粒体DNA 16S rRNA基因序列比较分析[J].广东农业科学,2013,40(1):150-153.
[16] 孙悦娜,冯建彬,李家乐,等.日本沼虾三群体线粒体16S rRNA基因片段序列的差异与系统进化[J].动物学杂志,2007,42(1):59-66.
[17] 刘亚军,喻子牛,姜艳艳,等.栉孔扇贝16S rRNA基因片段序列的多态性研究[J].海洋与湖沼,2002,33(5):477-483.
[18] 胡文革,王金富.动物线粒体DNA多态研究及其在鱼类群体遗传结构上的应用(综述)[J].石河子大学学报:自然科学版,2001,5(3):253-258.
[19] 石林波,张小燕,汪雁,等.福建华溪蟹线粒体DNA COI和16S rRNA基因序列的遗传多样性[J].中国人兽共患病学报,2013,29(7):671-675.
[20] 产久林,姜华鹏,刘一萌,等.CO I和16S rRNA基因在高原裂腹鱼物种鉴定中的应用[J].水生态学杂志,2015,36(4):98-104.
[21] 陈杰.秀丽白虾五群体形态和遗传特征初步研究[D].上海:华东师范大学,2006.
[22] Bowen B W,Grant W S.Phylogeography of the sardines (Sardinops spp.):assessing biogeographic models and population histories in temperate upwelling zones[J].Evolution,1997,51(5):1601-1610.
[23] 熊丹,李敏,李永振,等.南海短尾大眼鲷线粒体Cyt b基因序列及种群判别分析[J].中国水产科学,2016,23(1):188-197.
[24] 肖明松,崔峰,康健,等.长吻鮠养殖群体与野生群体遗传多样性分析[J].水生生物学报,2013,37(1):90-99.
[25] 周发林,江世贵,姜永杰,等.中国南海野生斑节对虾5个地理群体线粒体16S rRNA基因序列比较分析[J].水产学报,2009,33(2):208-214.
[26] 戴艳菊,刘萍,高保全,等.三疣梭子蟹4个野生群体线粒体16S rRNA和COⅠ基因片段的比较分析[J].中国海洋大学学报,2010,40(3):54-60.
[27] Tajima F.Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J].Genetics,1989,123(3):585-595.
[28] Grant P R,Grant B R.Hybridization of bird species[J].Science,1992,256(5054):193-197.