扇贝是中国海水贝类养殖的主要品种之一,因其具有味道鲜美、营养丰富等特点,深受人们的喜爱。目前,国内广泛养殖的扇贝品种主要有中国土著的栉孔扇贝Chlamys farreri、从美国引进的海湾扇贝Argopecten irradians[1]和从日本引进的虾夷扇贝Patinopecten yessoensis[2]。然而,伴随着扇贝养殖产业的迅猛发展,纵观整个产业链条从苗种生产到成贝养殖均存在诸多问题,如种质衰退严重、疾病灾害频发、死亡率升高、品质下降等[3]。因此,新品种的引进将成为解决上述问题的有效途径之一。自2000年以来,先后由大连碧龙海产有限公司于2005年将海扇贝Placopecten magellanicus引入中国并繁育成功[4]和青岛农业大学王春德教授于2008年从秘鲁引进紫扇贝Argopecten purpuratus并繁育成功[5]。但目前这两种扇贝在国内均未被广泛推广养殖,具体原因尚不清楚。岩扇贝Crassadoma gigantea隶属于软体动物门Mollusca、双壳纲Bivalvia、扇贝目 Pectinida、扇贝科 Pectinidae,原产于北美太平洋沿岸,因其具有个体大、生长速度快、抗逆性强、肉质鲜美等优点,在国际市场备受青睐。大连海洋大学曹善茂等于2012年从加拿大温哥华引进美洲岩扇贝,于2015年完成了全人工室内育苗试验并获得成功[6]。目前,国内外有关岩扇贝的研究主要集中在生理生态、生产养殖等方面[7-13],对其遗传学背景的研究在国内外却鲜见报道。作为外来引进物种,查明其遗传学及种质资源资料将有益于其养殖生产和育种推广。
线粒体DNA(mtDNA)作为核外遗传物质,具有结构简单、分子量小、易于分离、进化速度快等特点,适用于种群遗传分析[14-15]。在mtDNA中,相比rRNA基因和非编码区,蛋白质的编码基因更易发生重排,其进化速度适中,且在试验过程中容易扩增[16]。已有的研究表明,细胞色素b(Cytochrome b,Cyt b)在mtDNA众多蛋白质的编码基因中是最为可靠的分子标记之一,目前,对其结构和功能的研究较为深入[17-18],普遍被认为是研究群体遗传多样性和系统进化的理想工具[16,19-20]。为此,本研究中拟测定岩扇贝及3种国内主要养殖扇贝 (栉孔扇贝、海湾扇贝和虾夷扇贝)的mtDNA Cyt b基因序列,分析其遗传变异,计算4种扇贝的基因差异和遗传距离,结合GenBank中扇贝科其他物种的相应基因序列进行系统进化分析,从分子水平上了解岩扇贝的遗传背景及其与3种主要经济扇贝的亲缘关系,以期为岩扇贝的引种与推广养殖,以及今后的遗传育种研究工作提供参考信息。
1 材料与方法
1.1 材料
试验用岩扇贝为从加拿大温哥华引进种贝的F1代个体,为1龄贝,养殖于大连龙王塘海域 (简记为Y);虾夷扇贝 (简记为X)和栉孔扇贝 (简记为Z)均购自于大连新长兴海产品市场,均为2龄贝;海湾扇贝 (简记为H)采自山东莱州某养殖场,为1龄贝。采样时间均为2017年5月,每种扇贝随机取10个样本,经形态学鉴定后取闭壳肌保存于无水乙醇中备用。
1.2 方法
1.2.1 基因组DNA提取 取约50 mg扇贝闭壳肌组织,参考Sambrook等[21]的酚/氯仿抽提法并略做修改进行基因组DNA的提取。将沉淀后的基因组DNA溶解于200 μL的TE溶液中保存,取溶解后的2 μL基因组DNA进行琼脂糖凝胶电泳,检测基因组DNA的完整性,并取1 μL,在 NanoDrop 2000微量分光光度计上测定浓度,最后稀释成约50 ng/μL的工作液于4℃下保存备用。
1.2.2 引物设计与合成 试验用引物使用Primer 5软件[22]进行设计,最后由生工生物工程 (上海)股份有限公司合成。其中虾夷扇贝、栉孔扇贝和海湾扇贝的Cyt b基因序列扩增引物,分别根据Gen-Bank数据库公布的该物种线粒体基因组全序列进行设计,而岩扇贝的Cyt b基因序列则根据本实验室测得的岩扇贝线粒体基因组序列设计 (表1)。
1.2.3 PCR扩增及序列测定 PCR反应总体系为25 μL, 包括模板 DNA(50 ng/μL)1.0 μL, 10×reaction buffer 2.5 μL, dNTP MIX(2.5 mmol/L)1.0 μL,上下游混合引物 (各 10 μmol/L)1.0 μL,1 U Taq DNA聚合酶0.2 μL,用ddH2O补足至25 μL。PCR反应条件为:94℃下预变性5 min;94℃下变性35 s,50℃下退火40 s,72℃下延伸40 s,共进行38个循环;最后在72℃下充分延伸10 min。PCR产物用10 g/L琼脂糖凝胶电泳检测,经BioSpin Gel Extraction Kit纯化回收后,连接到pMD18-T载体上,再转化至大肠杆菌感受态细胞DH5α中。挑选阳性克隆送天一辉远 (武汉)生物科技有限公司进行双向测序。
表1 试验用引物序列及其退火温度
Tab.1 Sequences and annealing temperatures of the primers uesd in the expriment
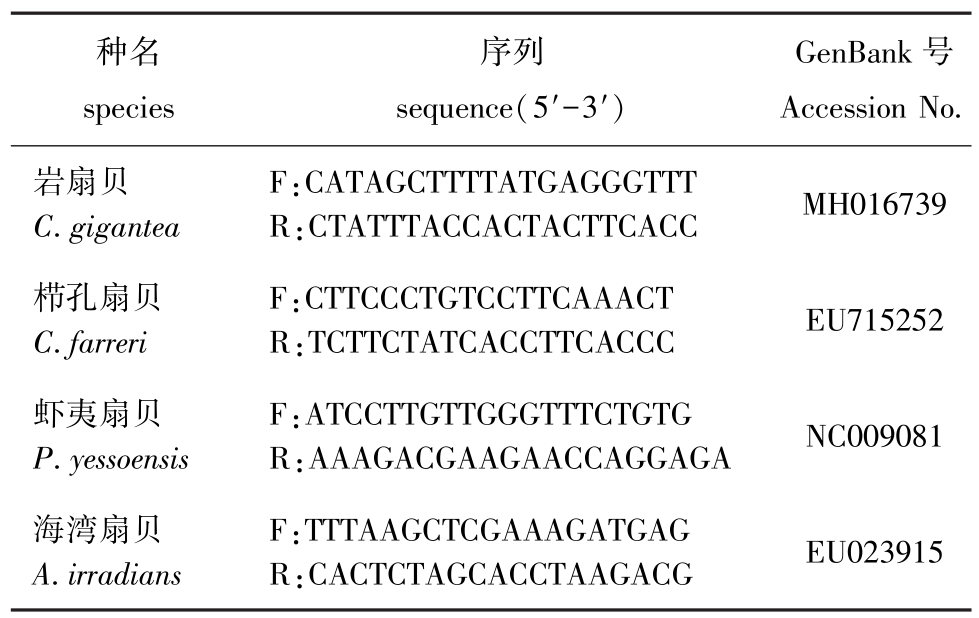
种名species GenBank号Accession No.岩扇贝C.gigantea序列sequence(5′-3′)F:CATAGCTTTTATGAGGGTTT R:CTATTTACCACTACTTCACC MH016739栉孔扇贝C.farreri F:CTTCCCTGTCCTTCAAACT R:TCTTCTATCACCTTCACCC EU715252虾夷扇贝P.yessoensis F:ATCCTTGTTGGGTTTCTGTG R:AAAGACGAAGAACCAGGAGA NC009081海湾扇贝A.irradians F:TTTAAGCTCGAAAGATGAG R:CACTCTAGCACCTAAGACG EU023915
1.3 数据分析
首先,根据测序峰结果进行人工校对;使用NCBI的BLAST工具搜索其他贝类的同源序列,下载扇贝科8种扇贝和贻贝科两种贻贝的Cyt b基因的同源序列 (图1);使用ClustalX 1.83程序[23]对不同物种DNA序列进行多重比对和人工校准;使用GeneMarkS 4.6b[24]对岩扇贝的测序结果进行Cyt b基因的预测;使用DNAsp 5[25]分析核酸基本参数;使用MEGA 5.1[26]计算碱基组成和Kimura双参数遗传距离,并利用获得的4种扇贝Cyt b基因序列 (所有单倍型序列),结合GenBank数据库中扇贝科其他扇贝的相应序列,基于邻接法 (Neighbor-Joining,NJ) 和最大简约法 (Maximum-Parsimory,MP)分别构建系统发育树,采用重抽样法(Bootstrap)1000检验系统树分支的置信度,选取贻贝科的两个种作为外源群。
2 结果与分析
2.1 Cyt b基因的序列组成
经PCR扩增及测序,获得了4种扇贝的Cyt b基因全序列 (已提交至GenBank数据库),其中栉孔扇贝、海湾扇贝和虾夷扇贝的Cyt b基因序列与GenBank数据库中相应序列的一致性达98%以上。从表2可见:4种扇贝的Cyt b基因序列长度为1146~1173 bp,岩扇贝序列长度最长,其次为栉孔扇贝,海湾扇贝序列长度最短;4种扇贝的A、T、G、C含量分别为19.65%~22.39%、35.23%~37.80%、25.94%~29.41% 和12.71%~17.29%,A+T含量为57.12%~59.64%,栉孔扇贝A+T含量最高,岩扇贝最低,但均高于G+C含量。此外,栉孔扇贝、海湾扇贝、岩扇贝的启动密码子均为ATG,虾夷扇贝为ATT;虾夷扇贝、海湾扇贝、岩扇贝的终止密码子均为TAG,栉孔扇贝为TAA。
表2 4种扇贝Cyt b基因的序列特征及碱基组成Tab.2 Sequence characteristics and base composition of Cyt b gene in four scallop species
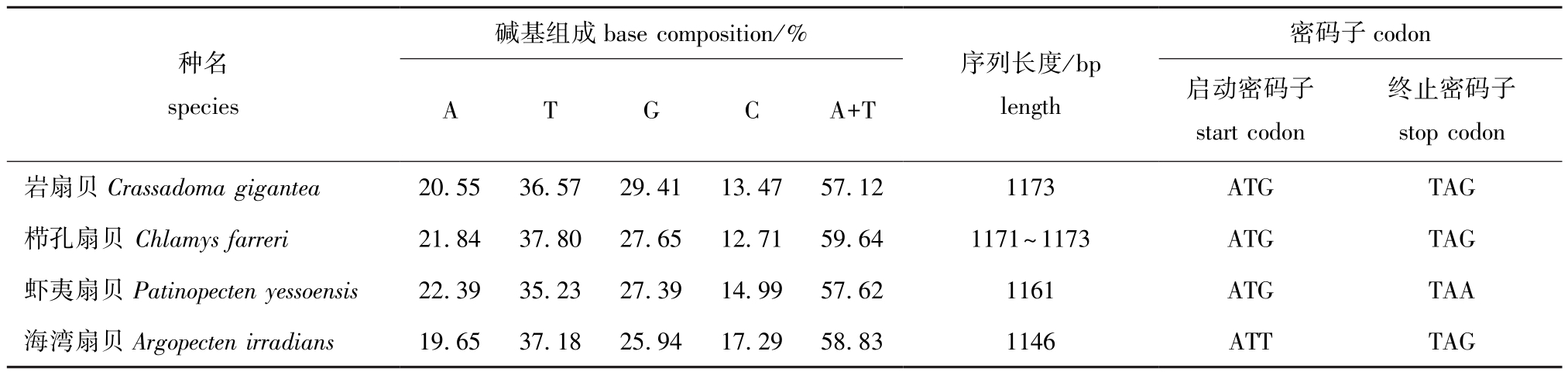
碱基组成base composition/%终止密码子stop codon岩扇贝Crassadoma gigantea 20.55 36.57 29.41 13.47 57.12 1173 ATG TAG栉孔扇贝 Chlamys farreri 21.84 37.80 27.65 12.71 59.64 1171~1173 ATG TAG虾夷扇贝Patinopecten yessoensis 22.39 35.23 27.39 14.99 57.62 1161 ATG TAA海湾扇贝Argopecten irradians 19.65 37.18 25.94 17.29 58.83 1146 ATT TAG种名species A T G C A+T序列长度/bp length密码子codon启动密码子start codon
2.2 Cyt b基因序列的遗传变异
从表3可见:从岩扇贝、栉孔扇贝、虾夷扇贝和海湾扇贝中分别检测到3、3、4、4种单倍型,多态性位点数依次为2、4、5、14,4种扇贝的单倍型多样性指数 (Hd)、核苷酸多样性指数 (π)和平均核苷酸差异 (k)分别为 0.545~0.800、0.001 00~0.005 72和0.788~6.545; Hd值为虾夷扇贝最高,栉孔扇贝最低,而π值和k值均为海湾扇贝最高,岩扇贝最低。
表4为4种扇贝Cyt b基因序列多态性核苷酸位点及单倍型分布情况。从表4可见:海湾扇贝的单倍型H4仅为某一个体所独有;除栉孔扇贝单倍型Z3含有两2个插入/缺变异位点外,且同样为某一个体所独有,较为特殊;4种扇贝的其余变异位点均由转换和颠换引起。
表3 4种扇贝Cyt b基因序列的遗传变异参数
Tab.3 Parameters of genetic variation in the sequences of Cyt b gene in four scallop species
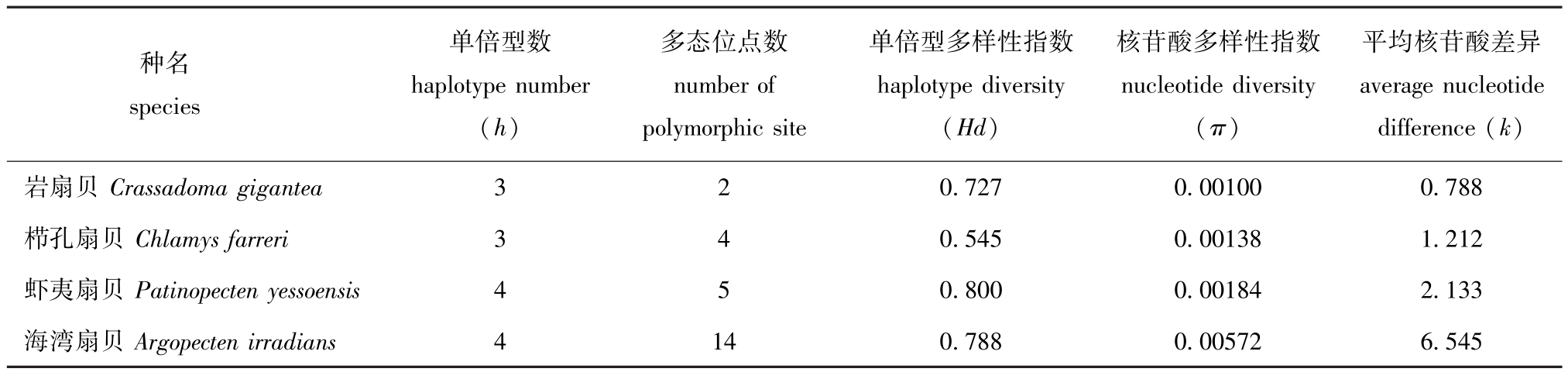
平均核苷酸差异average nucleotide difference(k)岩扇贝Crassadoma gigantea 3 2 0.727 0.00100 0.788栉孔扇贝Chlamys farreri 3 4 0.545 0.00138 1.212虾夷扇贝Patinopecten yessoensis 4 5 0.800 0.00184 2.133海湾扇贝Argopecten irradians 4 14 0.788 0.00572 6.545种名species单倍型数haplotype number(h)多态位点数number of polymorphic site单倍型多样性指数haplotype diversity(Hd)核苷酸多样性指数nucleotide diversity(π)
2.3 种间遗传距离及系统发育树
4种扇贝基于Cyt b基因序列的种间遗传距离如表5所示。4种扇贝间的遗传距离为0.255~0.429,岩扇贝与虾夷扇贝的遗传距离最近,与海湾扇贝的遗传距离最远。
基于Cyt b基因序列的NJ树与MP树如图1、图2所示。用两种方法获得的系统发育树拓扑结构几近一致,且大分支节点的支持率均较高。扇贝科的12种扇贝明显分为两个单系群。其中,Mimachlamys属的两个种单独聚为一个单系群;而另外一个单系群则包括Argopecten、Pecten、Placopecten、 Chlamys、 Patinopecten和Crassadoma 6个属的10种扇贝,该单系群又分为两个主要的分支,具体为P.yessoensis的4种单倍型与C.gigantea的3种单倍型先聚为一支,再与C.farreri的3种单倍型聚类为一大支,而A.irradians的4种单倍型先与Argopecten系的3个种聚为一支,再与Pecten属的2个种进行聚类,最后与Patinopecten属聚为另一个大支。
3 讨论
3.1 4种扇贝Cyt b基因的序列组成分析
4种扇贝Cyt b基因序列的种间同源性较低,变异较大,但未表现出明显的种间长度多态,该现象同样存在于扇贝科的其他物种,因此,笔者认为,序列的长度多态无法作为物种鉴定的依据。此外,4种扇贝Cyt b基因序列的碱基组成均表现为T含量最高,其次为G含量,C含量最低,并且A+T含量均明显高于G+C含量,其符合动物细胞色素b的碱基组成特征[27],与其他贝类及鱼类的碱基组成偏向性相似[28-29]。胡丽萍等[15]推测认为,mtDNA具有较高的A+T含量在无脊椎动物中是一种普遍现象。
表4 4种扇贝Cyt b基因序列多态性核苷酸位点及各单倍型分布情况
Tab.4 Distribution of variable nucleotides and haplotypes in Cyt b gene fragments of four scallop species
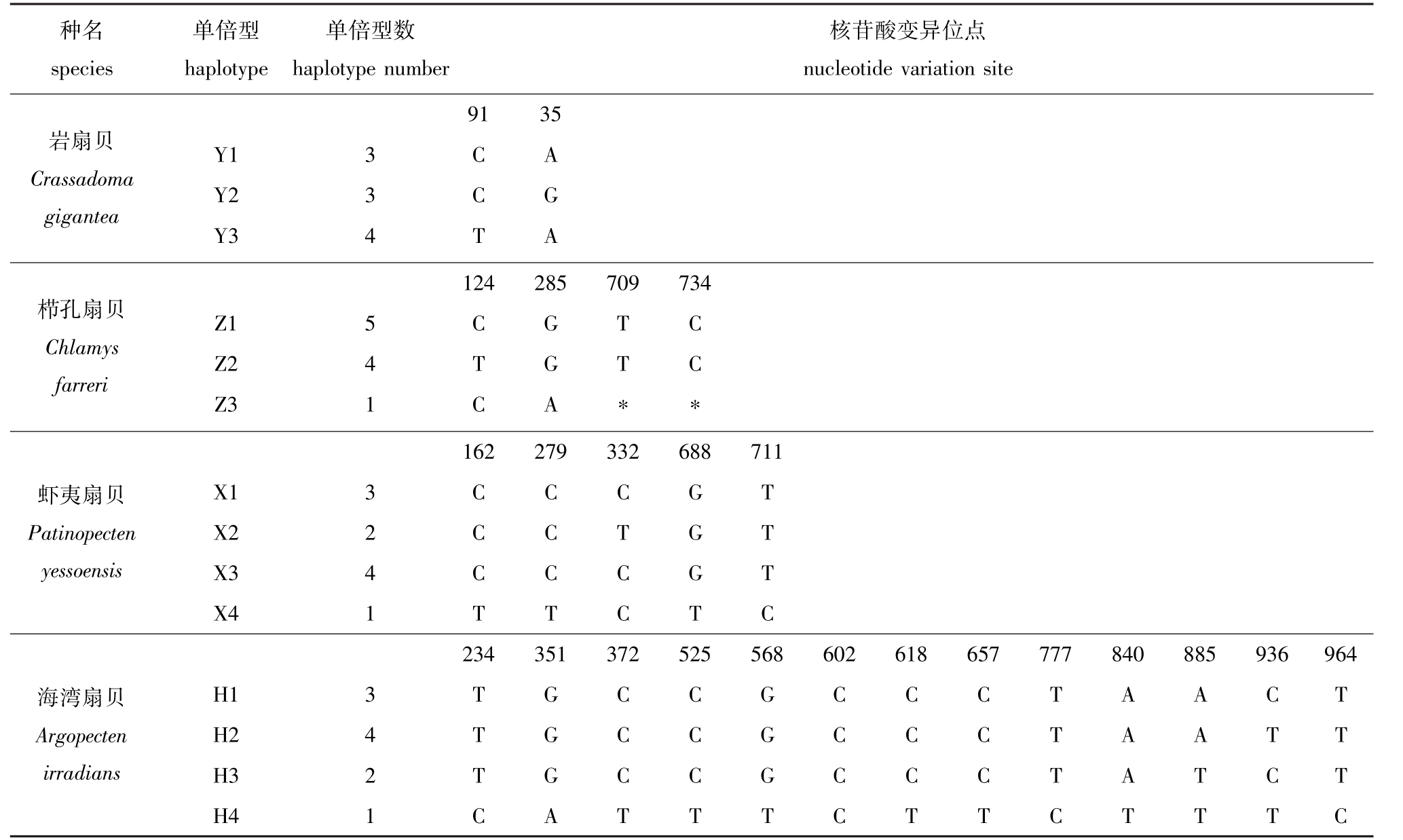
种名species单倍型haplotype单倍型数haplotype number核苷酸变异位点nucleotide variation site岩扇贝Crassadoma gigantea 91 35 Y1 3 C A Y2 3 C G Y3 4 T A栉孔扇贝Chlamys farreri 124 285 709 734 Z1 5 C G T C Z2 4 T G T C Z3 1 CA∗∗虾夷扇贝Patinopecten yessoensis 162 279 332 688 711 X1 3 C C C G T X2 2 C C T G T X3 4 C C C G T X4 1 T T C T C海湾扇贝Argopecten irradians 234 351 372 525 568 602 618 657 777 840 885 936 964 H1 3 T G C C G C C C T A A C T H2 4 T G C C G C C C T A A T T H3 2 T G C C G C C C T A T C T H4 1 C A T T T C T T C T T T C
表5 4种扇贝种间Kimura双参数遗传距离
Tab.5 Kimura 2-parameter genetic distance among four scallop species

种名species 岩扇贝Crassadoma gigantea 栉孔扇贝Chlamys farreri 虾夷扇贝Patinopecten yessoensis栉孔扇贝Chlamys farreri 0.260虾夷扇贝Patinopecten yessoensis 0.255 0.289海湾扇贝Argopecten irradians 0.429 0.413 0.407
3.2 4种扇贝的种内遗传变异分析
遗传多样性是物种适应环境变化、维持生存与进化的必要条件,遗传多样性越高,遗传变异越丰富,对环境适应能力就越强,可适宜生存的环境就越广泛[16,29-30]。 通常来说, 核苷酸多样性指数(π)和单倍型多样性指数 (Hd)是评估种群遗传多样性的两个重要指标,但由于单个碱基的变异就能生成一种新的单倍型,Hd值可在短时间内积累变异而快速提高,而π值的提高需要长时间的积累,因此,在评估种群遗传多样性和遗传变异时,π 值比 Hd值更具代表性[17,31-32]。 本研究中, 4种扇贝的π值在0.001 00~0.005 72之间,均表现出较低的遗传多样性水平。虾夷扇贝、栉孔扇贝和海湾扇贝表现出较低的多样性水平,可能与试验样本均为养殖群体有关;而引进种岩扇贝可能是由于试验样本为F1代个体,仅由少数几个引进亲本繁育而来,其遗传背景相对单一,导致较低的遗传变异水平。胡丽萍等[15]曾在其他引进种扇贝中也报道了类似现象。
3.3 亲缘关系及系统发育树分析
本研究中的种间遗传距离结果表明,引进的岩扇贝与虾夷扇贝的亲缘关系较近,而与海湾扇贝间的亲缘关系较远。其余3种扇贝间则是虾夷扇贝与栉孔扇贝之间的亲缘关系较近,该研究结果与秦艳杰等[4]应用AFLP技术对3种扇贝亲缘关系的研究结果一致。
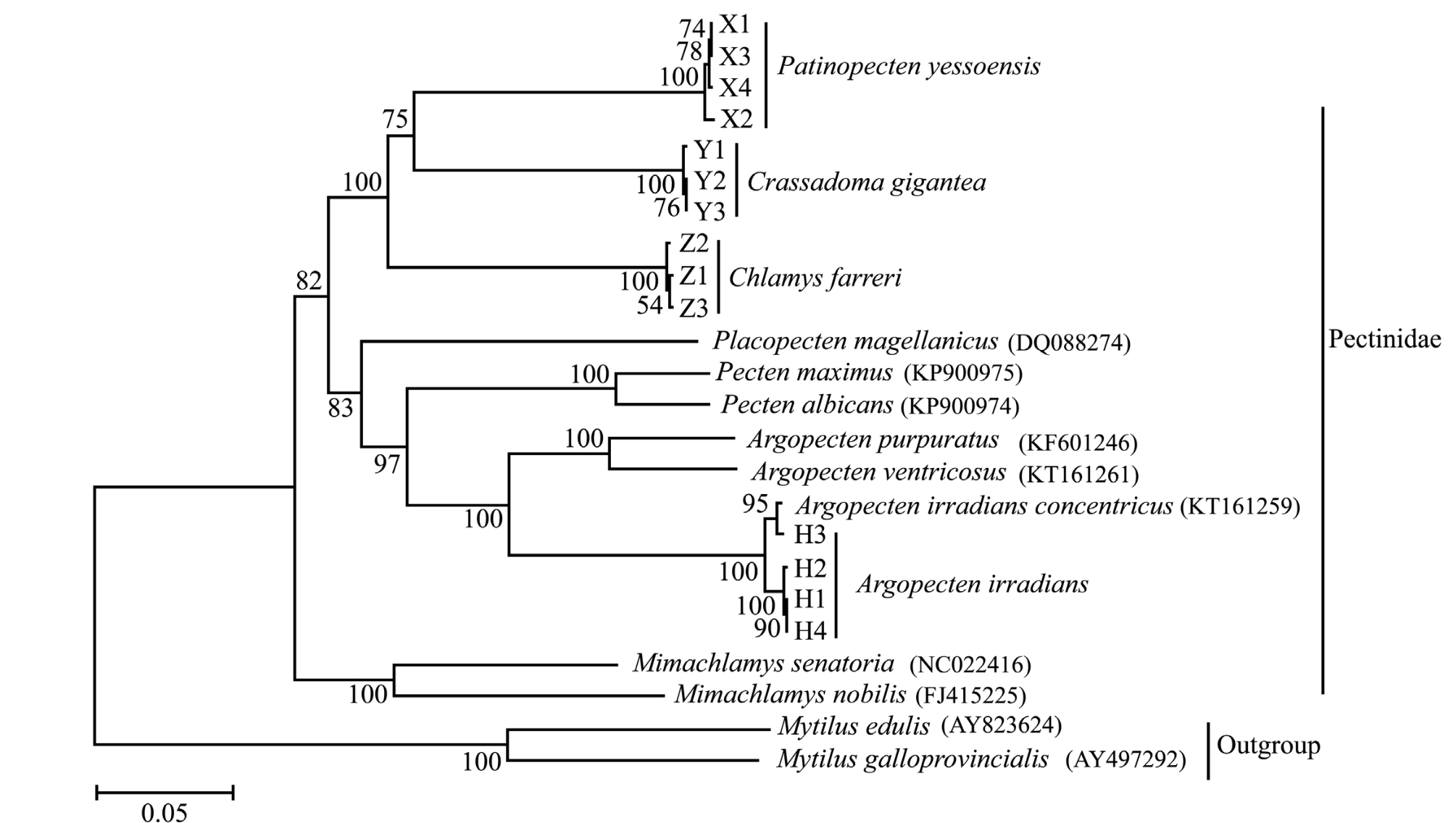
图1 应用邻接法 (NJ)构建基于Cyt b基因序列的扇贝科系统发育树
Fig.1 Neighbor-joining(NJ)phylogenetic tree constructed based on the sequences of Cyt b gene in Pectinidae
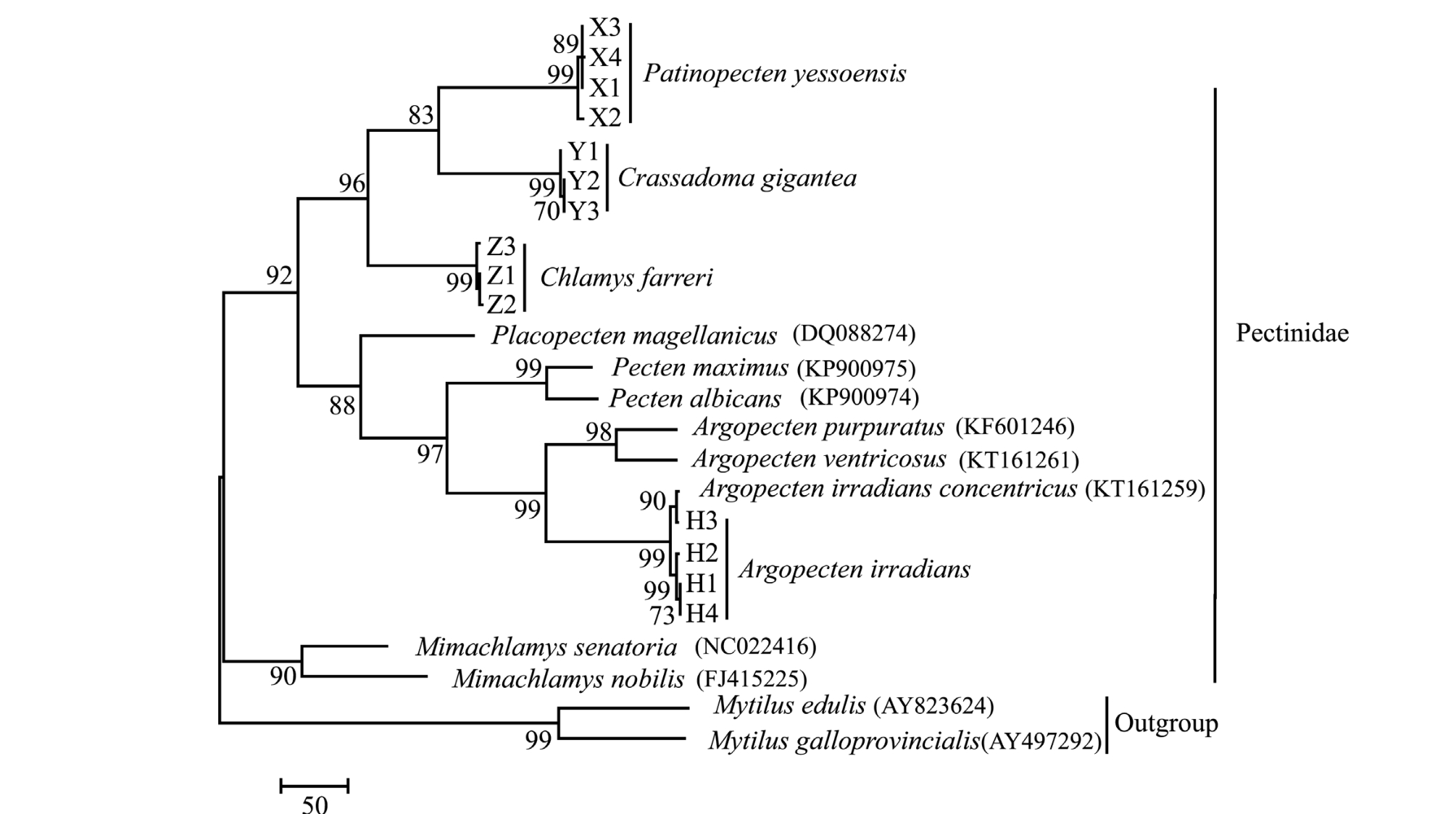
图2 应用最大简约法 (MP)构建基于Cyt b基因序列的扇贝科系统发育树
Fig.2 Maximum-parsimory(MP)phylogenetic tree constructed based on the sequences of Cyt b gene in Pectinidae
本研究中,基于两种方法 (NJ和MP)构建的系统进化树的拓扑结构几近一致,表明了结果的准确性和可靠性。系统进化树显示,扇贝科中同属的物种先单独聚为一个分支,再与其他属物种进行聚类,表明了mtDNA Cyt b基因作为一种分子标记在种属间系统发生关系研究中的适用性[33]。基于mtDNA基因片段序列探讨扇贝科物种系统进化关系有较多的研究和报道,如Barucca等[34]通过对16S rDNA和12S rDNA部分序列的比较分析,分别构建了23种扇贝的系统进化树;Saavedra等[35]利用mtDNA 16S rDNA和12S rDNA部分序列对扇贝科27种扇贝进行了系统进化研究;而Puslednik等[36]同时利用两个线粒体基因 (16S和12S)及一个核基因 (H3组蛋白)的部分序列构建了46种扇贝的系统进化树,其结果表明,C.gigantea与P.yessoensis的亲缘关系较近,而 C.gigantea与A.irradians的亲缘关系较远,该部分结果与本研究结果一致。但上述研究中均未对 C.gigantea、A.irradians、P.yessoensis和C.farreri 4种扇贝的系统进化及分子遗传学分析进行阐述,并且构建系统进化树所用序列均为相应基因的部分序列,在分析上存在一定局限性。本研究中则通过Cyt b基因全序列,阐明了引进种岩扇贝与国内3种主要经济扇贝间的亲缘关系,将为岩扇贝今后的遗传育种研究工作提供基础信息。
参考文献:
[1] 张福绥,何义朝,刘祥生,等.海湾扇贝引种、育苗及试养[J].海洋与湖沼,1986,17(5):367-374.
[2] 张福绥,何义朝,马江虎,等.虾夷扇贝的引种、育苗及试养[J].海洋科学,1984,8(5):38-45.
[3] 寇凌霄.我国四个主要扇贝品种介绍及养殖发展对策[J].河北渔业,2012(6):56-59.
[4] 秦艳杰,李霞,周伯文,等.利用AFLP技术分析海扇贝与三种扇贝的亲缘关系[J].东北农业大学学报,2009,40(10):69-74.
[5] 王春德,刘宝忠,李继强,等.紫扇贝与海湾扇贝种间杂交的研究[J].海洋科学,2009,33(10):84-87,91.
[6] 曹善茂,汪健,王谦,等.岩扇贝人工育苗的初步研究[J].大连海洋大学学报,2017,32(1):1-6.
[7] MacDonald B A,Bourne N F.Growth of the purple-hinge rock scallop,Crassadoma gigantea Gray,1825 under natural conditions and those associated with suspended culture[J].Journal of Shellfish Research,1989,8(1):179-186.
[8] 曹善茂,王昊,陈炜,等.岩扇贝闭壳肌营养成分的分析及与中国3种扇贝的比较[J].大连海洋大学学报,2016,31(5):544-550.
[9] 曹善茂,梁伟锋,刘钢,等.不同生态因子对岩扇贝幼贝耗氧率和排氨率的影响[J].大连海洋大学学报,2017,32(3):280-286.
[10] Beitler M K.Toxicity of adductor muscles from the purple hinge rock scallop(Crassadoma gigantea)along the Pacific coast of North America[J].Toxicon,1991,29(7):889-894.
[11] Culver C S,Richards J B,Page H M.Manipulation of the cementing process of the purple-hinge rock scallop,Crassadoma gigantea[J].Journal of Shellfish Research,2000,19(6):651-652.
[12] Culver C S,Richards J B,Page H M.Plasticity of attachment in the purple-hinge rock scallop,Crassadoma gigantea:implications for commercial culture[J].Aquaculture,2006,254(1-4):361-369.
[13] 曹善茂,梁伟锋,汪健,等.岩扇贝幼贝滤食率的基础研究[J].大连海洋大学学报,2016,31(6):612-617.
[14] 李霞,秦艳杰,初冠囡,等.采用16S rRNA基因序列分析海扇贝与3种扇贝的亲缘关系[J].大连海洋大学学报,2009,24(3):269-273.
[15] 胡丽萍,姜黎明,黄晓婷,等.基于16S rDNA基因序列探讨引进物种紫扇贝(Argopecten purpuratus)在海湾扇贝属(Argopecten)中的分类地位[J].海洋与湖沼,2016,47(6):1149-1157.
[16] 李青,王波,郑风荣,等.星斑川鲽、黄盖鲽和石鲽线粒体基因Cytb和COⅠ片段序列的比较研究[J].水产科学,2016,35(6):625-632.
[17] 胡静,侯新远,尹绍武,等.基于mtDNA COⅠ和Cytb基因序列对南中国海不同海域波纹唇鱼群体遗传多样性的研究[J].水生生物学报,2014,38(6):1008-1016.
[18] Irwim D M,Kocher T D,Wilson A C.Evolution of the cytochrome b gene of mammals[J].Journal of Molecular Evolution,1991,32(2):128-144
[19] 高天翔,毕潇潇,赵林林,等.基于线粒体Cytb基因全序列的松江鲈群体遗传结构分析[J].水生生物学报,2013,37(2):199-207.
[20] 郝桂英,杨应东,古小彬,等.基于线粒体cox1和cytb基因对四川地区多头带绦虫的种群遗传多样性研究[C]//中国畜牧兽医学会家畜寄生虫学分会第七次代表大会暨第十二次学术研讨会论文集.郑州:中国畜牧兽医学会,2013.
[21] Sambrook J,Fritsch E F,Maniatis T,et al.Molecular Cloning:A Laboratory Manual[M].New York:Cold Spring Harbor Laboratory Press,1989.
[22] Lincoln A L,Daly M,Lander E.PRIMER:a computer program for automatically selecting PCR primers[R].Cambridge:Whitehead Institute Technical Report,1991.
[23] Larkin M A,Blackshields G,Brown N P,et al.Clustal W and Clustal X version 2.0[J].Bioinformatics,2007,23(21):2947-2948
[24] Besemer J,Lomsadze A,Borodovsky M.GeneMarkS:a self-training method for prediction of gene starts in microbial genomes.Implications for finding sequence motifs in regulatory regions[J].Nucleic Acids Research,2001,29(12):2607-2618.
[25] Librado P,Rozas J.DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J].Bioinfomatics,2009,25(11):1451-1452.
[26] Tamura K,Peterson D,Peterson N,et al.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Molecular Biology and Evolution,2011,28(10):2731-2739.
[27] 祁得林,晁燕,杨成,等.川陕哲罗鲑Cyt b基因克隆及其在鲑亚科中的系统发育关系[J].四川动物,2009,28(6):805-809.
[28] Ren J F,Shen X,Jiang F,et al.The mitochondrial genomes of two scallops,Argopecten irradians and Chlamys farreri(Mollusca:Bivalvia):the most highly rearranged gene order in the family Pectinidae[J].Journal of Molecular Evolution,2010,70(1):57-68.
[29] 沈朕,关洪斌,郑风荣,等.基于cytb和D-loop的4个大泷六线鱼群体遗传多样性分析[J].海洋科学进展,2017,35(4):524-534.
[30] 古小彬,朱俊扬,王保健,等.四川地区鸡异刺线虫核糖体转录间隔区(ITS1/2)序列的遗传变异分析[J].畜牧兽医学报,2016,47(4):796-804.
[31] Wu S G,Wang G T,Xi B W,et al.Population genetic structure of the parasitic nematode Camallanus cotti inferred from DNA sequences of ITS1 rDNA and the mitochondrial COI gene[J].Veterinary Parasitology,2009,164(2-4):248-256.
[32] 郝桂英,杨应东,古小彬,等.基于线粒体cox1和Cyt b基因对四川地区多头带绦虫的种群遗传多样性研究[J].畜牧兽医学报,2014,45(4):631-638.
[33] Saccone C,De Giorgi C,Gissi C,et al.Evolutionary genomics in Metazoa:the mitochondrial DNA as a model system[J].Gene,1999,238(1):195-209.
[34] Barucca M,Olmo E,Schiaparelli S,et al.Molecular phylogeny of the family Pectinidae(Mollusca:Bivalvia)based on mitochondrial 16S and 12S rRNA genes[J].Molecular Phylogenetics and E-volution,2004,31(1):89-95.
[35] Saavedra C,Peña J B.Phylogenetics of American scallops(Bivalvia:Pectinidae)based on partial 16S and 12S ribosomal RNA gene sequences[J].Marine Biology,2006,150(1):111-119.
[36] Puslednik L,Serb J M.Molecular phylogenetics of the Pectinidae(Mollusca:Bivalvia)and effect of increased taxon sampling and outgroup selection on tree topology[J].Molecular Phylogenetics and Evolution,2008,48(3):1178-1188.