在一定的环境条件下,由于外界环境因素的负面影响,使动物无法维持正常的生理状态,称为环境胁迫 (environmental stress),也叫环境应激。生物总会受到生存环境中各种环境因子的刺激,且刺激与生物有机体总是处于相互作用的动态过程中。近年来,随着工厂化水产养殖模式迅速发展,其集约化程度越来越高。在集约化养殖中,养殖密度的提高会产生一系列不利于鱼类生长的环境胁迫,引起水质恶化,影响鱼类的成活率、生长、健康、福利和产量[1]。
经典遗传学认为,遗传的分子基础是核酸,核酸的碱基序列上包含生物体的所有遗传信息,几乎所有的生命活动均由基因调控。然而,生物体是一个复杂的系统,不是一种因素就能决定整个生命活动。随着遗传学的发展,人们发现基因表达模式的改变是由一些DNA或染色质水平的修饰引起的。表观遗传 (epigenetics)是指DNA序列不发生变化,但基因表达却发生了可遗传的改变。这种改变是细胞内除遗传信息外其他可遗传物质发生的改变,且这种改变在发育和细胞增殖过程中能稳定传递[2]。它是一种全新的遗传机制,因为它不涉及DNA序列的改变,不符合孟德尔的遗传方式。在遗传学研究中,人们发现了表观遗传有多种修饰方式,包括DNA甲基化作用、组蛋白修饰作用、非编码RNA作用等,而且探究了其错综复杂的生物学作用。研究发现,表观遗传与外界环境刺激有密切的关系[3],由于外界环境对机体刺激的影响,任何一种调节机制发生紊乱都能导致细胞活动发生异常,从而引起各种疾病[4]。已有研究表明,环境胁迫能调控水生生物基因的表达,影响水生生物的表型性状,并且在一定程度上改变遗传信息并传递到下一代,进而影响下一代的表型性状。本研究中,就水产养殖中环境胁迫与表观遗传调控的研究进展做一综述,以期为进一步研究环境因素与基因互作关系提供理论参考。
1 表观遗传学的基本要素
表观遗传是指基因的表达发生可遗传的变异现象,而基因序列不发生改变[5],是生物体内遗传物质和环境因素共同作用的结果,主要包括4个方面:DNA甲基化、组蛋白修饰、染色质重塑和非编码RNA调控等[6]。
1.1 DNA甲基化修饰
1.1.1 DNA甲基化 DNA甲基化是指在DNA甲基转移酶 (DNA methyltransferase,DNMT)的催化作用下,通过S-腺苷-L-甲硫氨酸 (S-adenymethionine,SAH)提供甲基供体,将甲基转移到脱氧胞嘧啶环第5位碳原子上形成甲基化脱氧胞嘧啶共价修饰[7],DNA甲基化过程由DNA转移酶家族(DNMTS)中的维持甲基化转移酶 (DNMT1)和重新甲基化转移酶 (DNMT3A和DNMT3B)参与完成,其中DNMT1主要负责DNA复制后的甲基化,以保证子链和亲链具有相同的甲基化模式;DNMT3A和DNMT3B则主要负责对未甲基化两条链的甲基化过程,以及发育过程中DNA的重新甲基化,也参与了异常甲基化过程[8-10]。
DNA甲基化主要发生在CPG岛上,CPG岛通常存在许多重要的基因启动子区。DNA的高度甲基化会影响基因的结构,进而影响RNA转录,引起基因沉默。正常的DNA甲基化是调控生物体基因表达的重要方式之一,参与较多生理过程,如维持染色体结构、胚胎发育、细胞分化、X染色体失活、肌生成等。而异常的DNA甲基化则会引发疾病,近年来研究表明,遗传过程中存在类似DNA甲基化作用的其他表观遗传修饰 (如羟基化),这些作用可能与鱼类的生长发育存在密切的关系[11]。1.1.2 DNA去甲基化 DNA的去甲基化同DNA的甲基化一样,在基因的表达过程中扮演着非常重要的作用。DNA去甲基化的两个主要过程是主动去甲基化 (Active DNA demethylation)和被动去甲基化 (Passive DNA demethylation)。在 DNA去甲基酶参与过程下形成的去甲基化为主动去甲基化,而在DNA的复制过程中,由于DNA的甲基化维持机制受损导致的去甲基化为被动去甲基化。
DNA的去甲基化主要是由10-11转置蛋白(Ten-Eleven Translocation,TET)介导的去甲基化过程[12]。 TET1可催化 5-MEC形成 5-HMC(5-hydroxymethylcytosine)。 5-HMC 中间体在 5-MEC变成非甲基化C中起着重要作用[13]。沉默抑制因子 (Repressor of Silencing 1,ROSl)是拟南芥Arabidopsis thaliana体内的一种DNA去甲基化酶,具有裂解酶和糖基化酶的活性,同时含有核酸内切酶结合域,从而介导DNA去甲基化。ROS1突变会导致一些内源位点和转基因位点的高度甲基化,同时也会对DNA损伤试剂敏感[14]。ROS4具有乙酰转移酶的活性,是催化组蛋白H3乙酰化的主要活性物质,ROS4倾向于结合不含有修饰的核小体的特异甲基化DNA,组蛋白的乙酰化为染色体创造一个相对松散的环境,利于ROS1发挥作用,同时DNA甲基化减少蛋白 (Decrease in DNA methylation1,DDM1) 的功能也需要 ROS1的参与[15-16]。ROS5具有Α晶体状结构规域,编码一个热激蛋白,可以同ROS4和ROS1互化,同时也影响DNA去甲基化和组蛋白的修饰[17-18]。近些年相关研究发现,AP核酸内切酶和碱基切除修复途径X线修复交叉互补基因 (X-ray repair cross-complementing1 XRCC1)也参与 DNA去甲基化途径[19-20]。同时也有研究表明,甲基化胞嘧啶结合蛋白(Methylcytosine-Binding Protein, MBD4) 可结合含有DNA甲基化的位点,指导DNA的去甲基化[21-22]。
1.2 组蛋白修饰
1.2.1 组蛋白修饰 组蛋白修饰是另一个主要的表观遗传过程 (图1)。组蛋白修饰包括一系列的转录后修饰,如甲基化、乙酰化、生物酰化、磷酸化、泛素化等[23-24]。乙酰化组蛋白与活化基因相关,而去乙酰化通常与转录沉默相关[25]。组蛋白甲基化对基因活性有着不同的影响,组蛋白H3第4位赖氨酸 (K4)甲基化通常和增加基因活性相关,而组蛋白H3第9位赖氨酸 (K9)甲基化可能导致转录抑制。更重要的可能是DNA甲基化与组蛋白修饰通常共同起作用[26],比如,胞嘧啶的甲基化可能有助于H3K9的甲基化。同时,H3K9甲基化可能促进胞嘧啶的甲基化进而导致基因下调。通常DNA甲基化和组蛋白甲基化修饰相互依赖的起作用进而调控整体染色体的状态。早期研究发现,高乙酰化组蛋白和转录活化基因相关,组蛋白乙酰化有助于基因的转录[27-28]。实际上,组蛋白乙酰化的化学性质表明了其可能促进基因表达的一个机制。组蛋白甲基化被当作调控元件,在许多真核生物启动子区域富集H3K4me3,但H3K4甲基转移酶较少[29]。在体外,H3K4me3通过果蝇Drosophila melanogaster、人类多梳抑制性复合物 2(Polycomb RepressiveComplex2, PRC2) 抑制H3K27 三甲基化[30]。此外,H3K9me3 和H3K27me3也与异染色质的形成以及多梳沉默相关。组蛋白磷酸化影响其修饰位点残基的正电荷,表明组蛋白磷酸化与乙酰化有相似的调节核小体动力学机制[31]。
1.2.2 组蛋白去甲基化去乙酰化调节机制 组蛋白甲基化也是动态的,赖氨酸特异性去甲基化酶1(Lysine Specific Demethylase1,LSD1)是一种组蛋白去甲基化酶,也是一种胺氧化酶,可在辅基存在的条件下进行胺氧化反应催化去除H3K4结构位点的二甲基化和单甲基化,进一步研究发现,LSD1在雄激素受体引导下也可去除H3K4的二甲基化[32]。另一类组蛋白去甲基化酶是含有JmjC结构域的蛋白 (JmjC Domain Containing Protein,JMJ)类蛋白,也是最大的一类去甲基化酶,可去除多甲基化修饰包括无法去除的三甲基化蛋白,并通过活性中心结构域对甲基基团进行氧化反应,再脱去水释放甲酸而将甲基去除[33]。类蛋白通过去组蛋白甲基化以及与其他染色质修饰相互协作等方式参与了众多的基因表达和染色质活动的调控,涉及到了代谢、发育和环境适应等生物学过程。
组蛋白的乙酰化和去乙酰化发生在不同组蛋白的不同位点。DNA与组蛋白间、核小体与核小体间相互作用的强度可通过组蛋白赖氨酸残基加上乙酰化修饰后中和自身的正电荷来改变[34]。每种生物均含有多种组蛋白乙酰转移酶和去乙酰化酶,组蛋白乙酰转移酶和去乙酰化酶识别的位点一般不止一个,这与组蛋白甲基转移酶有较大不同。基因的转录激活同样离不开组蛋白的去乙酰化修饰。HOS2是Ⅰ型组蛋白去乙酰化酶,关于HOS2对基因的转录调控机制的观点认为:一般表达量高的基因均需要组蛋白的多种修饰以及多个转录激活因子的激活作用,但这些修饰可能成为后期转录的障碍[35]。因此,对于需要多轮转录的基因来说,组蛋白去乙酰化酶HOS2负责恢复转录后染色质的结构,以方便下一轮转录。
1.3 染色质重塑
染色质重塑是指在整个细胞分裂周期中染色质的位置和结构发生改变[36]。染色质重塑发生时,在核小体连接处染色质状态变得相对疏松,这使得启动子区中的顺式作用元件暴露,为顺式作用元件与转录因子结合提供尽可能多的接触空间。染色质重塑与部分依赖能量供给的组蛋白修饰密切联系,会诱导许多蛋白-DNA及蛋白-蛋白间的关联受到破坏。2种蛋白复合体 (ATP依赖型核小体重构复合体和组蛋白修饰复合体)会通过水解作用诱导核小体的构型发生变化[37],后者可以催化核心组蛋白N端尾部的共价修饰 (赖氨酸的乙酰化及泛素化、精氨酸和赖氨酸的甲基化、谷氨酸的多聚ADP核糖基化等)[38]。通过组蛋白修饰酶的作用破坏基因组DNA与组蛋白尾部及核小体之间的相互作用也可以引起染色质重塑[39-40];另外,染色质重塑也可为基因表达相关的蛋白及高级染色质结构的组织者提供识别位点,经过修饰的组蛋白也可作为染色质特异位点的标志[23,41]。ATPase亚基存在于所有ATP依赖型核小体重构复合体中,根据亚基的同源性可分为 ISWI、SWI/SNF、CHD、INO80、RAD54亚家族[42]。酵母交换型转换/蔗糖不发酵复合物 (SWI/SNF)是第一个在酿酒酵母Saccharomyces cerevisiae中发现并被确认的ATP依赖的重塑复合体[43]。
1.4 非编码RNA调控
非编码RNA是各种不翻译成蛋白质的RNA的统称,非编码RNA在基因的表达过程中同样扮演重要的角色[44],包括参与基因转录、翻译等调控过程,催化RNA前体中内含子的剪切、DNA复制、调控细胞发育和分化等[45-48](图2)。
1.4.1 小分子RNA对基因转录的调控 小分子RNA主要有microRNA、siRNA、piRNA和启动子附近产生的一类RNA,micRNA和siRNA通过在细胞质中组建RISC复合物诱导Argonaute蛋白与特定区域的靶标RNA结合实现对基因的沉默,然而Argonaute蛋白在 RNA干扰 (RNA interference,RNAi)通路及在细胞核内的存在形式在表观遗传调控上的作用为这套系统赋予了多层面、更复杂的功能。miR-320由基因POLR3D启动子区域反义方向转录产生,miR-320可以介导 Argonaute-1(AGO1)蛋白与RRC2复合物组分EZH2之间的相互作用,也可以将启动子区域组蛋白H3上第27位赖氨酸的三甲基化 (H3K27me3),从而抑制POLR3D基因的转录[49]。
1.4.2 长链非编码RNA对基因转录的调控 长链非编码RNA是一类不具有编译蛋白质能力的RNA,研究发现,影响DNA甲基化的RNA许多为编码蛋白质的mRNA的反义转录产物,如LUC7L和P53的反义RNA,WRAP53、HBA1(Haemoglobin α1)的反义RNA。WRAP53是通过与CTCF结合调节P53基因的表达,而LUC7L则是通过甲基化启动子区CPG岛抑制基因转录。作为支架招募组蛋白修饰相关的复合物是长链非编码RNA调控基因转录的一种重要方式,通过对染色质组蛋白进行甲基化或乙酰化修饰调节基因的表达[50]。
2 环境胁迫对养殖鱼类表观遗传的影响
水产养殖环境胁迫按性质可分为:物理、化学、生物和人为因子等4种[51]。物理胁迫因子是由物理因素对机体造成的应激,包括高温、低温及其温度的剧变,以及光照、噪音等;化学胁迫因子源包括有机物和重金属污染,亚硝酸盐及氨氮浓度的过高,以及盐度、pH变化等;生物胁迫因子包括养殖密度过高导致的拥挤应激、营养缺乏或过剩、鱼类雌雄分化和生活习性等;人为胁迫因子包括养殖过程中人为的捕捉、分池、运输和病害处理等造成的应激。
2.1 主要物理胁迫对水产养殖动物的表观遗传效应
2.1.1 温度 鱼类属于变温动物,有适宜的生长与繁育温度,存在生命活动的低温与高温极限。Liu等[52]研究发现,高温应激可显著提高团头鲂Megalobrama amblycephala血清促肾上腺激素和皮质醇水平,并诱导氧化应激,促进 HSP60、HSP70、HSP90基因的表达。在对欧洲鲈欧洲鲈Dicentrarchus labrax的研究中发现,在性腺成熟前温度可以影响性别比例,在水温较高情况下,雄鱼比例大于雌鱼,雄性鲈鱼幼鱼性腺内的CYP19A启动子甲基化水平显著高于雌性幼鱼,在高温刺激下,雌鱼CYP19A基因的启动子甲基化水平明显上升,说明DNA甲基化参与基因调控诱导性别分化,并且在该过程中CYP19A基因的表达与DNA甲基化水平呈现反比的关系[53],个体CYP19A基因的启动子甲基化水平的变化过程发生在生殖腺而非其他组织中,说明这不是一个普通的温度效应,而且这些影响在性别未分化和未经雌激素处理的鱼中同样可以检测到,因此,在温度诱导雄鱼产生的过程中,CYP19A基因启动子甲基化扮演了重要的角色。在体外条件下,温度会诱导欧洲鲈鱼个体CYP19A基因启动子发生甲基化,SF-1和FoxI2基因的表达会受到抑制,推测CYP19A基因启动子甲基化可能是生物体在漫长的进化过程中,形成的一种对温度适应的机制[53]。Jabbari等[54]发现,变温的鱼类和两栖类动物DNA甲基化水平是恒温的鸟类和哺乳类动物的2倍,并且他还发现,南极鱼类DNA甲基化水平高于热带鱼类。有研究表明,鱼类的肌纤维生成过程及鱼类肌肉分化相关的基因表达与孵化温度有密切的关系,斑马鱼Danio rerio胚胎在26℃孵化条件下,其成年个体肌纤维数量比低温 (22℃)和高温 (31℃)条件孵化的个体增加了14%~19%[55]。在对刺参Apostichopus japonicus的研究中发现,高温应激会促进 DNMT1、HDAC3和MLL5基因的表达[56]。河豚鱼胚胎孵化后期,温度升高会促进FoxK1基因转录水平[57]。斑马鱼胚胎在23、27、31℃条件下孵化,选择发育过程中的6个时间点检测DNMTS基因的表达情况,结果显示,DNMT3基因表达在发育前期有显著的动态变化趋势,尤其是DNMT3A的mRNA的转录水平在孵化时期显著增加,同时在相同发育阶段,发现不同孵化温度组中DNMT3A和DNMT3B基因表达有显著差异,由此推测,鱼类发育前期阶段,DNMT3A和DNMT3B基因在温度的表观遗传调控中发挥着不同的作用[58]。罗非鱼 Oreochromis mossambicus耐寒品系DNA甲基化总体水平相比于对照组基因组明显下降,经过连续多代的低温胁迫可引起尼罗罗非鱼DNA甲基化水平及状态发生改变,这说明罗非鱼抗寒性能与DNA甲基化密切相关[59]。
总的来说,温度是水产养殖过程中的主要胁迫因子,长期的进化过程中鱼类已衍化出抵御温度应激的生理调节机制。综上所述,机体可能会通过表观遗传修饰方式的变化以适应温度应激,通过进一步研究温度胁迫对鱼类表观遗传的影响,有助于寻找与环境适应力相关的重要基因标志物,从而解释表观遗传的作用机理,为表观遗传学在鱼类抗逆育种及其抗胁迫中的应用提供新的思路和理论参考。2.1.2 光照 在自然界,所有生物的能量均来源于光照,光的变化具有稳定性和规律性,光的周期性变化可影响鱼类的繁殖、代谢等生理机能。已有研究表明,光的周期性变化可通过表观遗传影响鱼类的表型。干旱期间,低耗能状态和极端温度下穴居的有条纹青蛙骨骼肌基因的转录沉默,DNA甲基转移酶和DNA甲基化显著扩增。夏眠成年蛙DNA甲基转移酶DNMT1的转录水平和转录协同抑制因子SIN3A显著高于非成年蛙。与此类似,光周期性变化通过DNA甲基化调节肌肉生长速率的机制在大西洋鳕鱼Gadus morhus中也有报道,连续暴露在光照下的大西洋鳕肌肉中DNMT1和DNMT3A基因的表达量显著高于自然光周期下饲养的鱼。这种现象会一直维持下去,直到自然光周期等于连续照明的光周期,这表明,饲养期间连续的光照对于DNMT基因的表达影响可能是持久的[60]。目前,光照对鱼类表观遗传具体作用机制还有待进一步研究。
2.2 主要化学胁迫对水产动物的表观遗传效应
2.2.1 有机污染物 在集约化的高密度水产养殖环境中,环境污染物的存在是不可避免的。已有研究表明,多数环境污染物能通过基因组水平调控表观遗传机制,如调控基因启动子甲基化水平和组蛋白修饰等,这在生物生长、发育过程中具有重要的作用。这种表观遗传状态的变化可以通过生殖系细胞中的一系列调控从亲本传递到生殖细胞,最后引起后代表型的变化。已有大量研究表明,生物生长后期相关基因的疾病是由基因组甲基化状态的变化引起。苯菌灵 (Benomy1)处理后的斑马鱼Danio rerio胚胎将会有一半死亡,存活的胚胎也会出现畸形、心脏伸长、脊柱弯曲、心包水肿、心率降低等症状[61]。用雌二醇 (E2)处理后的雄性三刺鱼性腺基因组会处于高甲基化状态[7]。水蚤暴露于烯菌酮中DNA甲基化水平会发生持续稳定的降低,且这种变化会遗传给后代[62]。用烯菌酮处理F0代小鼠,可在F3代小鼠的生殖细胞中检测到DNA甲基化状态的改变[63]。暴露17α-炔雌醇一段时间后的成年斑马鱼,其肝脏卵黄蛋白原Ⅰ基因的5′侧翼甲基化水平明显下降,这表明雌激素可通过诱导DNA甲基化水平调控软黄蛋白原的表达[64]。暴露于E2或BPA环境下的小鼠,其前列腺4型磷酸二酯酶变体4酶基因会处于低甲基化状态,这表明发育时期小鼠的前列腺表观基因组会受到低浓度雌激素的影响[65]。
野生鱼在污染水域中性腺增长相关基因的甲基化水平会异常升高,且其性腺增长速度显著低于在正常水环境中的野生鱼[66]。在阿特拉津和毒死蜱农药胁迫下,鲤Cyprinus carpio的性腺组织和脑甲基化水平也会随之发生改变[67]。先前有研究表明,当环境中的苯并 [a]芘浓度达到1 μg/L时能够传代影响F3代青鳉Oryzias latipes幼鱼的骨骼发育,苯并 [a]芘处理后的子三代雄性成鱼通过Ct测量和组织病理学分析发现,骨骼的厚度减少,微裂纹增加,通过比对青鳉鱼对照组和处理组miRNA的表达,检测到5种方式mRNA/miRNA(Osx/Mir-214, Col2a1b/Mir-29b, Runx2/miR-204, Sox9b/miR-199a-3p, Apc/Mir-27b)[68], 这表明苯并[a]芘能通过miRNA调节表观遗传机制。有研究表明,Cr(VI)可与组蛋白去乙酰化酶1-DNA甲基化转移酶1的复合物 (HDAC-DNMTL)相结合,通过交联到Cypla1启动子区域的染色质的方式,抑制由AhR调节的基因转录,引起组蛋白H3甲基化、磷酸化等,从而促进了B[a]P和DNA形成加合物,导致DNA损伤[69](图3)。有机污染物对鱼类表观遗传影响的数据越来越多,并引起广大学者的重视,更多的作用机制还有待进一步研究。2.2.2 重金属 环境中的重金属,如镉、镍、铬和甲基汞等可以引起机体发生氧化还原反应,产生大量氧自由基,可氧化损伤DNA,干扰DNMTs与DNA的相互作用,从而影响DNA甲基化状态[70]。有研究表明,毛蚶Scapharca subcrenata在经过Cd2+和Cu2+处理之后,鳃组织甲基化水平异常升高[71]。也有研究显示,在经砷暴露数周后的大鼠Rattus norvegicus肝脏全基因组DNA呈低甲基化状态[72〛。同样,当金鱼Carassius auratus经砷暴露数周后,肝脏全基因组呈高甲基化状态[73]。周新文等[74]研究发现,鲫鱼Carassius auratus在经0.02 mg/L的Pb2+染毒48 h后,肾脏和肝脏中DNA总甲基化显著提高。王丙莲等[75]研究发现,泥鳅 Misgurnus anguillicaudatus在经 Pb2+染毒 14 d后,肝胰脏DNA甲基化水平明显升高,但在染毒28 d后,肾脏DNA甲基化水平异常降低。将围产期大鼠经饮水暴露乙酸铅2个月后发现,新生小鼠海马体全基因组呈高甲基化状态[76]。在用镉对欧洲鳗鱼Anguilla anguilla的慢性胁迫试验中发现,相关基因通过甲基化水平的变化对基因表达产生负调控[77]。通过研究重金属对水生动物表观遗传效应的影响,阐明相关的作用机制,可为重金属污染防治提供一些参考依据。
2.2.3 盐度 盐度对于水生动物在淡水湖泊、河流和海洋间的迁移是十分重要的。洄游鱼类必须通过调节基因的表达途径,以适应从出生地到进入海洋 (或从海洋返回产卵场)间不断变化的盐度。从孵化场迁往海洋的鱼存活率往往较低,这可能与适应孵化环境有关。在对鲑鱼Oncorhynchus的研究中发现,不同生长条件的体质下降现象与基因组的甲基化有关[78],但是这种变化在淡水孵化场养殖的鱼中未被发现[79]。在对三疣梭子蟹Portunus trituberculatus的低盐胁迫试验中发现,其鳃中有12个差异表达的mRNA,这些mRNA的靶基因主要参与甲壳素的代谢及离子转运等渗透压的调节过程[80]。渗透胁迫转录因子1(Osmotic Stress Transcription Factor 1,OSTF1)在水生动物的渗透压调节过程中起着重要作用。Yan等[81]研究发现,尼罗罗非鱼miR-429可通过调控OSTF1的表达来调控血浆中的离子浓度。Flynt等[82]研究发现,在斑马鱼Danio rerio的胚胎发育过程中,miR-8家族可通过调节Nherf1基因的表达来调控离子的转运,并且在离子细胞中高度表达。
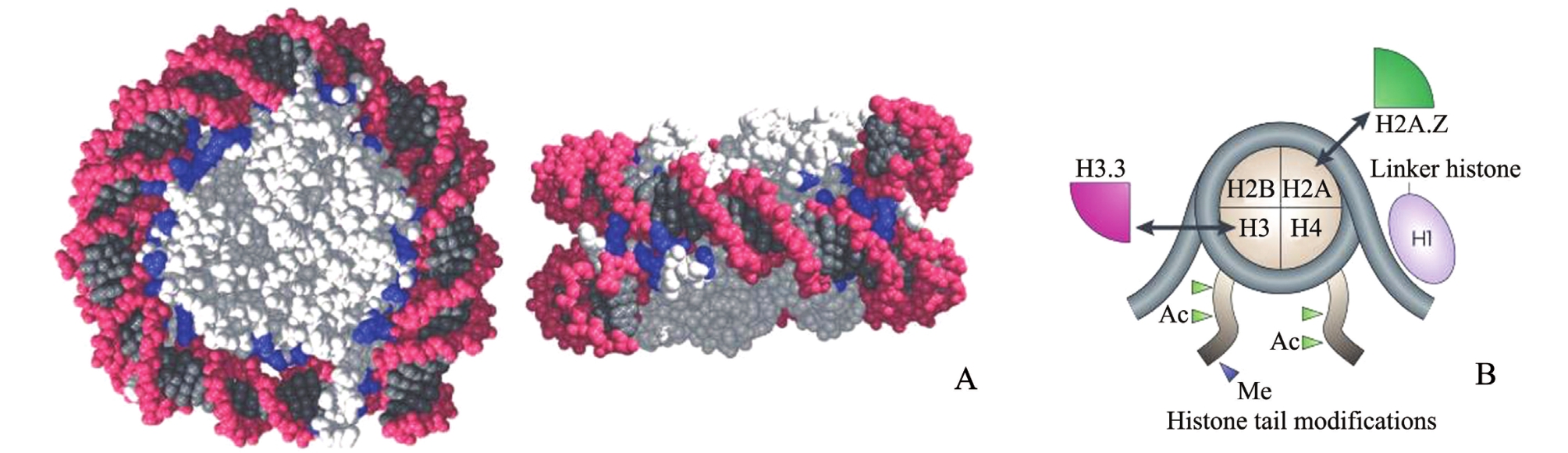
图1 组蛋白修饰[24]
Fig.1 Histone tail modifications[24]
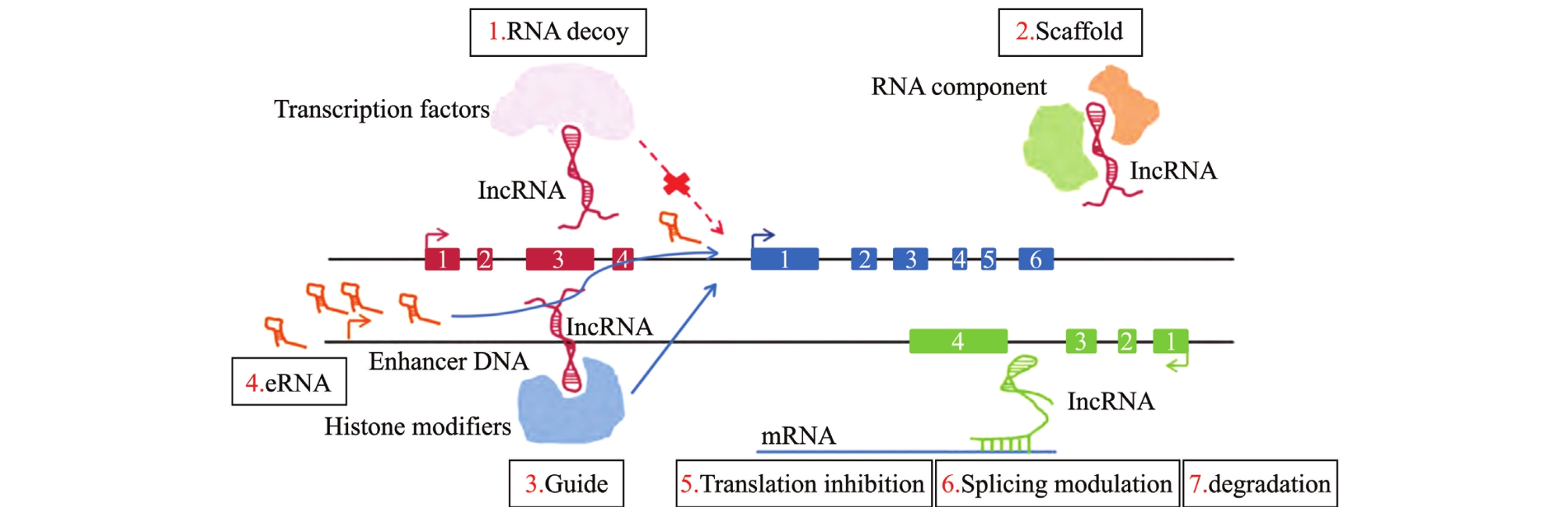
图2 非编码RNA的调控机制[49]
Fig.2 Regulation of gene transcription by non-coding RNAs[49]
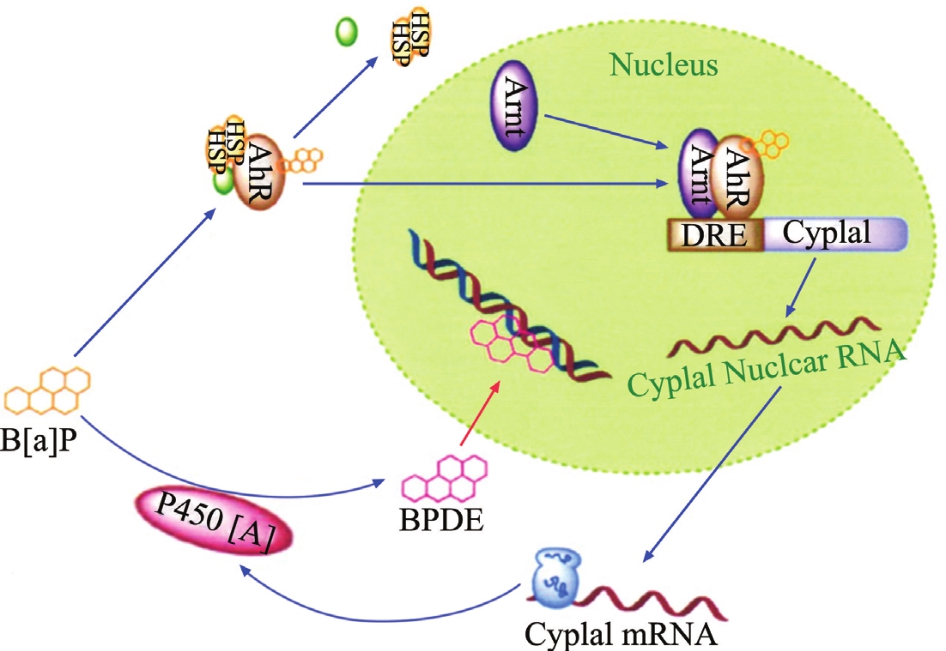
图3 B[a]P通过与DNA共价形成DNA加合物导致基因损伤[71]
Fig.3 B[a]P can cause gene damage by covalent binding of DNA adducts with DNA[71]
一些广盐性的水生动物如凡纳滨对虾Litopenaeus vannamei、罗非鱼、梭鱼Mugil soiuy等在淡水和海水中均可进行人工养殖,目前对其渗透压的调节机制尚不明确,从上述研究结果来看,盐度的变化能影响水生动物的一些miRNA及其相关基因的表达。针对具体物种的渗透压调节机制,分析养殖水体环境的水质、离子等成分可在必要时补充相关离子,对水生动物的生长具有重要的意义。因此,研究盐度对水产动物的表观遗传调控机制显得尤为重要,相关内容还待进一步地解析。
2.2.4 亚硝酸盐、氨氮 亚硝酸盐和氨氮是普遍存在于水环境中的污染物,亚硝酸盐由细菌消化过程和反硝化过程产生,氨氮是水产动物蛋白质代谢的最终产物。当水体中的亚硝酸盐和氨氮浓度过高时,会降低水产动物的生长率、免疫力,并造成其生理功能紊乱,甚至导致死亡。Guo等[83]对凡纳滨对虾的研究发现,亚硝酸盐暴露显著上调caspase-3 mRNA水平,同时诱导血细胞凋亡。Sun等[84]对团头鲂的研究发现,亚硝酸盐暴露诱导鳃细胞凋亡,并伴随着caspase-8和caspase-9 mRNA的上调。肿瘤抑制因子P53参与多种生物学过程,包括DNA损伤修复,在对暗纹东方鲀Takifugu obscurus的研究中发现,在氨氮应激下,机体会通过诱导NO和ROS的产生并上调P53的表达量来对抗应激带来的损伤[85]。目前,关于亚硝酸盐、氨氮对水产动物的表观遗传机制的影响研究较少,但根据表观遗传的作用机制可以分析发现,表观遗传修饰可影响多种生物学过程,包括DNA的损伤修复,诱导相关抗氧化基因的上调,从而抵抗外界环境胁迫对水产动物机体造成的损伤。开展此方面的研究有助于了解亚硝酸盐、氨氮对水生动物表观遗传修饰的作用规律,为揭示水产动物氧化应激调节机制具有重要意义。
2.3 主要生物应激对水生动物的表观遗传效应
2.3.1 营养素 许多营养刺激可以通过改变特定细胞的表观遗传标记,影响鱼类发育过程中细胞的分化,目前,营养与表观遗传学的研究也受到广大学者的关注。虹鳟Oncorhynchus mykiss的幼体发育需要经历内源性营养、混合营养和外源性营养3个阶段,有研究发现,虹鳟肌肉中miR-1/133在这3个阶段中特异表达,且表达量逐渐下降,这表明随着鱼类的食性变化,miR-1/133会通过调控肌细胞的形成参与鱼类的生长过程[86]。对禁食和饱食后的鳜Siniperca chuatsi研究发现,miR-10c等7个miRNA在肌肉中差异表达,这表明食物摄取能通过miRNA调控骨骼肌的发育[87]。有研究发现,饲喂高碳水化合物饲料能使虹鳟患有糖尿病,通过检测mRNA转录水平发现,H3K9处于低乙酰化状态、G6pcb2处于高甲基化状态[88]。缺乏维生素(A、D、C)的鲈鱼其生长因子相关基因表达的时序和协调性会受到破坏,影响成骨细胞的分化,从而使某些细胞转化成脂肪细胞,导致鱼体畸形[88]。成骨细胞转化成脂肪细胞也可以发生在海洋鱼类早期生命阶段膳食中不饱和脂肪酸较高的情况下[89]。鲑鱼脂肪前体细胞具有根据环境条件分化免疫谱系细胞 (最有可能是类巨噬细胞)的能力[90]。目前,日粮蛋白质缺乏对机体表观遗传影响的报道相对较多,Rees等[91]研究发现,给妊娠期小鼠饲喂低蛋白质的日粮会导致子代肝脏组织DNA高度甲基化,这也是动物胚胎时期营养缺乏与子代表观遗传修饰变化关系的较早报道。有诸多试验表明,在动物妊娠期间,日粮蛋白质缺乏会导致子代DNA某些核心区域甲基化模式发生改变,且这种变化在动物成年后会变得非常稳定。总的来说,现有研究表明,母体蛋白质缺乏会通过表观遗传修饰的方式影响子代基因的表达。Burdge等[92]研究表明,脂肪酸,尤其是多不饱和脂肪酸 (n-3 HUFA's)可以改变表观基因组。n-3 HUFA是类花生酸类的重要前体物质,对鱼类的生长和发育至关重要,其以ATP的形式为鱼类代谢提供能量,且参与细胞膜的形成[93]。雌性鲑鱼性腺发育过程中,日粮中的脂肪源发生改变,会影响鱼卵的脂肪酸成分,结果导致胚胎发育异常[94]。增加真鲷Pagrosomus major亲鱼日粮中的鱼油添加比例,会影响孵化3 d后仔鱼的存活率、鱼苗的长度,卵的直径,以及蛋白质和脂肪含量,并影响脂肪细胞的代谢和形态[95-96]。对人类的研究表明,妊娠期α-亚麻酸的摄取会诱发后代肝脏出现表观遗传学变异[97]。Benatti等[98]研究发现,一些脂肪酸会作为激素控制转录因子的活性。Seierstad等[99]研究发现,饲喂不同脂肪源的大西洋鲑Salmo salar长至3~5 kg时,就已经出现与典型生活方式相关的症状,如动脉硬化、肝脏和心脏等器官中的脂肪异常沉积,导致鱼类器官功能受损和各种循环问题。
日粮营养与有机体的关系一直是学者重点研究的方向,有关日粮营养素对动物DNA甲基化的影响已有较多的研究,甲基供体对DNA甲基化的调控也受到了较多关注,如甜菜碱对肉鸡脂蛋白酯酶(LPL)[100]和脂肪合成酶 (FAS)[101]启动子甲基化的影响。研究营养素对水产动物表观遗传影响的分子机制,有助于从源头上阐述营养素对鱼类的影响,但目前营养素对鱼类表观遗传调控的影响研究刚刚起步,随着研究的逐步深入,其中的具体调控机制将会逐步明确。
2.3.2 雌、雄个体 有研究表明,雌鱼哺育后代能通过表观遗传影响后代,通过对三刺鱼Gasterosteus aculeatus的研究,雄鱼哺育后代同样能增加后代对早期环境压力的抗性,提高后代成活率,而这种变化与脑组织中DNA甲基化密切相关[102]。欧洲鲈鱼的雄鱼相比于雌鱼芳香化酶启动子表现出较高水平的DNA甲基化[103]。类似的研究在两栖类动物红耳龟Trachemys scripta和密西西比短吻鳄Alligator mississippiensis中也有报道[104]。一些种类的海龟 Chelonia mydas、蜥蜴和鳄鱼 Crocodylus siamensis的性别会受到温度的影响,在对鳄鱼的研究中发现,雄性性腺的SOX9基因的启动子序列基因相比于雌性表现出较高的DNA甲基化水平[105]。在遗传育种方面,红鲫与鲤的异源四倍体杂交过程同样被证实受到DNA甲基化调控[106]。研究表明,CYP11a1基因在卵巢和精巢发育过程中的表达规律不同,在精巢成熟期表达量最高,CYP11a1基因在一定程度上与斑马鱼的性腺发育相关,其可能在促进斑马鱼精子成熟中发挥一定作用[107]。不同种鱼类有不同的生活习性及生长特性,通过研究其与表观遗传的作用机制,对于理解表观遗传机制的多功能性和解析水产动物生长特性均具有重要意义。
2.3.3 养殖密度 在集约化水产养殖中,养殖密度的提高可有效增加养殖效益,但随着养殖密度的提高,鱼类也受到了更多的生存压力。Sadhu等[108]研究发现,海鲈Lateolabrax japonicus血糖含量在高密度组显著高于低密度组,Xie等[109]研究发现,鲤的血液皮质醇、葡萄糖、溶菌酶和肝丙二醛含量在拥挤胁迫后显著升高,肝脏中过氧化氢酶和超氧化物歧化酶活性在拥挤胁迫后减少。Costas等[110]研究发现,塞内加尔舌鳎Solea senegalensis养殖63 d后,高密度组中鱼血浆渗透压显著提高,这表明过高的养殖密度会导致机体渗透压功能和离子平衡的改变。Caipang等[111]研究表明,大西洋鳕在拥挤胁迫24 h和72 h后,HSP70基因表达显著上调。在对养殖密度的研究中发现,高密度组的海鲈和虹鳟HSP70基因表达显著高于低密度组[112-113]。而对塞内加尔鳎的研究中,高密度组鱼HSP70和HSP90基因的表达显著下调[114]。Gornati等[112]研究发现,海鲈 MT mRNA水平在高密度养殖条件下显著上调,表明MT可作为一种应激指标来评估机体的生理状态。CYP1A是混合功能氧化酶,可作生物标志物,用来评估机体生理状态。研究发现,海鲈肝、脾和肾CYP1A基因在高密度养殖条件下均显著上调[112]。促性腺激素释放激素GnRH1是一种十肽结构的神经激素,具有刺激腺垂体释放促性腺激素 (FSH)和黄体生成素 (LH)释放的功能,控制着脊椎动物的繁殖[115]。在对非洲慈鲷的研究中发现,妊娠期雌鱼正常生长情况下和拥挤胁迫条件下脑组织中GnRH1基因甲基化程度在一些CG位点明显不同,且其后代中CpG岛-1146、-1128和-1110位点呈低甲基化状态[116]。在对虹鳟的研究发现,高密度能显著提高其鳃和肾脏中PYGMA基因的表达,使肝脏中HIF1A信号通路上调,且与拥挤胁迫下血糖浓度的变化相一致[117]。从以上结果可以发现,拥挤胁迫可以影响鱼的生长,以及代谢酶和基因的表达。虽然某些基因变化的具体调节机制尚不明确,但可以明确的是,基因表达水平的变化是通过外界环境刺激诱导表观遗传修饰,进而影响基因的表达。目前,密度应激与酶及应激相关信号通路 (如Nrf2-Keap1)的关系已有报道,但与表观遗传变化的具体调节机制还有待研究。
2.4 人为应激的表观遗传效应
管理胁迫是指在水产养殖过程中由于人为管理操作而对水产动物所造成的胁迫。高强度的管理胁迫会对养殖动物的生理状态、繁殖、免疫力和生长造成不利影响。如在拉网、分池、捕捉、苗种运输、麻醉、消毒、病害用药等。生物前期生活经历的环境效应和前期母体效应将会影响各种精神机能的发育。对大鼠的研究表明,母体效应的变化信息能够稳定的影响后代DNA甲基化[118]。研究发现,亲代长期受到电离辐射会通过生殖细胞将辐射危害信息传递给下一代,增加癌症发病率和突变的可能性。研究人员推测,这可能是环境中的电离辐射会影响机体基因组DNA甲基化水平,且该影响具有相对稳定的遗传特性[118]。对表观遗传机制与人为应激的研究较少,许多具体机制有待进一步研究。
综上所述,表观遗传修饰主要包括DNA甲基化、组蛋白修饰、染色质重塑和非编码RNA调控等4个方面。这4个方面既相互作用又相互独立,共同调节着胚胎期及出生后整个生命周期中的基因表达。DNA甲基化是表观遗传修饰最主要的方式,近些年,在对哺乳动物的研究中已获得了较多数据支持,对水生生物的研究结果表明,有较多的应激方式均可引起DNA甲基化的变化,如高温可诱导鲈鱼CYP19A启动子和刺参相关促甲基化酶的表达,光照、营养成分、水环境变化等均会影响相关基因的甲基化程度。污染物B[a]P可引起组蛋白H3甲基化、磷酸化等,从而促进了B[a]P和DNA形成加合物,影响基因的表达。目前,关于非编码RNA对水生动物表观遗传效应的影响研究已有较多的报道,亚硝酸盐、氨氮、盐度的变化等均会引起相关非编码RNA的变化。总之,水生动物在生长过程中会受到物理、化学、生物、人为等应激因素影响,并通过DNA甲基化、组蛋白修饰、染色质重塑和非编码RNA调控等的表观遗传调控,最终影响水生动物的生长,以及发育障碍、疾病等表型性状 (图4)。
4 展望
外界环境刺激会影响动物生长发育,引起表现型发生改变,其未涉及遗传信息的改变,这属于表观遗传学的范畴。在宏观上,生物体通过表现型的变化适应生活环境的改变,在分子水平上,环境胁迫和表观遗传间的关系还有待深入研究。近年来,随着蛋白质组学技术和基因组测序技术的快速发展,研究基因调控和基因功能与环境应激的关系已经成为可能,如运用二代测序技术可系统检测水生动物的基因组及转录组,但研究表观基因组需要考虑的因素也许多,如研究处于什么发展阶段的表观基因组,判断这些表观遗传变化是否遗传就必须研究亲本和子代早期生活阶段的表观基因组。在某些情况下,在鱼的发育过程中进行表观遗传标记是必要的。通过对表观遗传机理的深入研究,能让养殖工作者更多地了解环境因素对鱼类健康的影响,从而针对不同生长阶段的水产动物,设计更精细、更合理的养殖环境,使鱼类的生产性能得到更大程度发挥的同时减少鱼类患病几率,提高经济效益,同时为育种学家提供新的思路,如寻找鉴定新的miRNA,并对靶基因进行标识,实现对水产动物的组织定向分化。或将DNA甲基化用作分子遗传标记手段对目标性状进行辅助选择标记,调控特异性基因实现对性别的控制,从而改善水产动物品质,加快推动集约化高效养殖的现代化渔业进程。
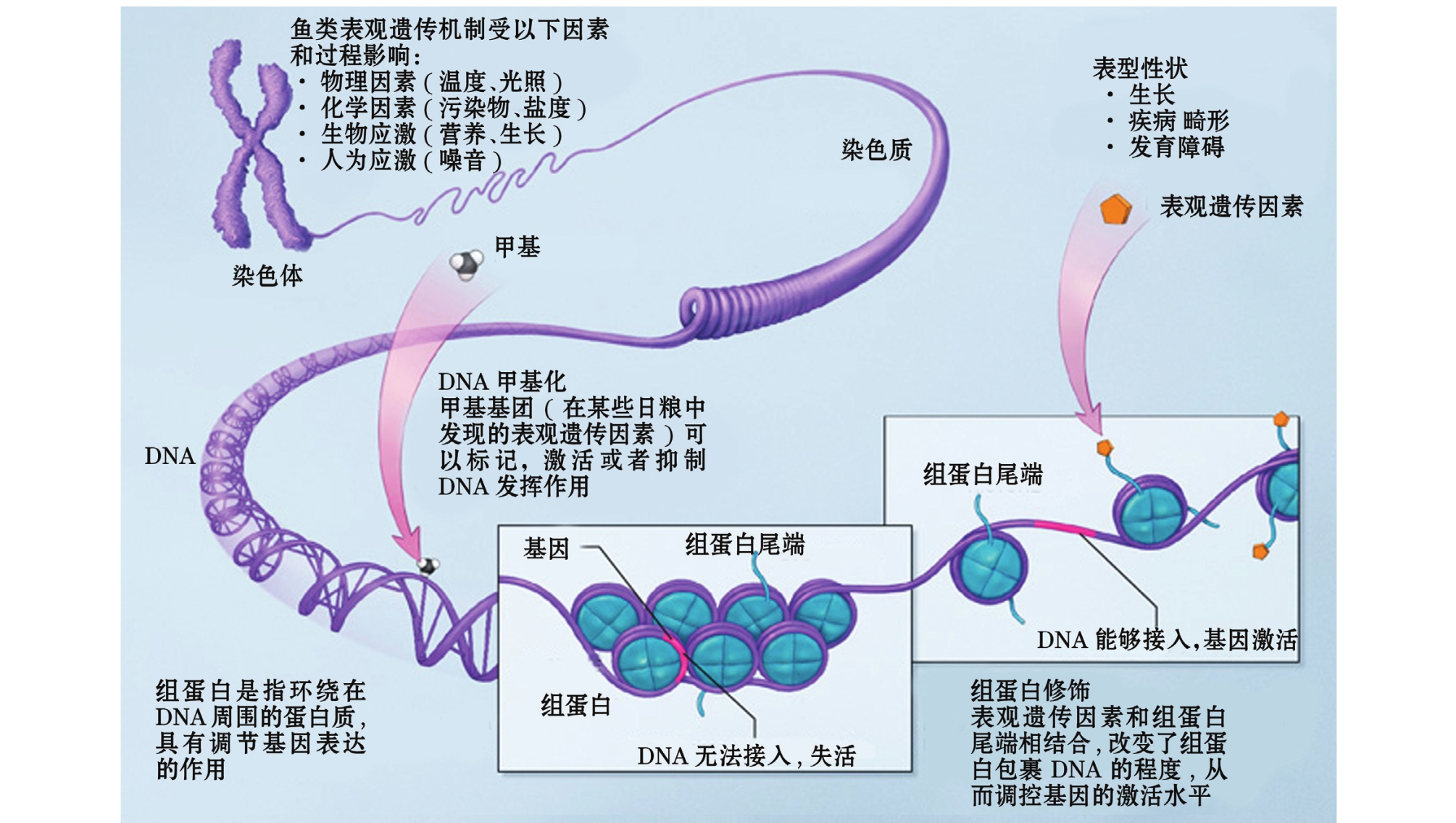
图4 表观遗传调节机制
Fig.4 Epigenetic regulation mechanisms
参考文献:
[1] 麦康森,艾庆辉,徐玮,等.水产养殖中的环境胁迫及其预防——营养学途径[J].中国海洋大学学报,2004,34(5):767-774.
[2] Deans C,Maggert K A.What do you mean,“epigenetic”?[J].Genetics,2015,199(4):887-896.
[3] Seo M K,Ly N N,Lee C H,et al.Early life stress increases stress vulnerability through BDNF gene epigenetic changes in the rat hippocampus[J].Neuropharmacology,2016,105:388-397.
[4] Weidman J R,Dolinoy D C,Murphy S K,et al.Cancer susceptibility:epigenetic manifestation of environmental exposures[J].Cancer Journal,2007,13(1):9-16.
[5] Goldberg A D,Allis C D,Bernstein E.Epigenetics:a landscape takes shape[J].Cell,2007,128(4):635-638.
[6] Cox B D.On the difficulty in getting out of historical ruts:waddington and an argument for behavioral epigenetics[J].New Ideas in Psychology,2013,31(3):374-389.
[7] Aniagu S O,Williams T D,Allen Y,et al.Global genomic methylation levels in the liver and gonads of the three-spine stickleback(Gasterosteus aculeatus)after exposure to hexabromocyclododecane and 17-β oestradiol[J].Environment International,2008,34(3):310-317.
[8] Nelissen E C M,van Montfoort A P A,Dumoulin J C M.Epigenetics and the placenta[J].Human Reproduction Update,2010,17(3):397-417.
[9] 王杰,徐友信,刁其玉,等.非孟德尔遗传模式:表观遗传学及其应用研究进展[J].中国农学通报,2016,32(14):37-43.
[10] Kurdyukov S,Bullock M.DNA methylation analysis:choosing the right method[J].Biology,2016,5(1):3.
[11] Si Yufeng,Ding Yuxia,He Feng,et al.DNA methylation level of cyp19a1a and Foxl2 gene related to their expression patterns and reproduction traits during ovary development stages of Japanese flounder(Paralichthys olivaceus)[J].Gene,2016,575(2):321-330.
[12] Rasmussen K D,Helin K.Role of TET enzymes in DNA methylation,development,and cancer[J].Genes & Development,2016,30(7):733-750.
[13] Kohli R M,Zhang Yi.TET enzymes,TDG and the dynamics of DNA demethylation[J].Nature,2013,502(7472):472-479.
[14] Gong Zhizhong,Morales-Ruiz T,Ariza R R,et al.ROS1,a re-pressor of transcriptional gene silencing in Arabidopsis,encodes a DNA glycosylase/lyase[J].Cell,2002,111(6):803-814.
[15] Li Xiaojie,Qian Weiqiang,Zhao Yusheng,et al.Antisilencing role of the RNA-directed DNA methylation pathway and a histone acetyltransferase in Arabidopsis[J].Proceedings of the National Academy of Sciences of the United States of America,2012,109(28):11425-11430.
[16] Qian Weiqiang,Miki D,Zhang Heng,et al.A histone acetyltransferase regulates active DNA demethylation in Arabidopsis[J].Science,2012,336(6087):1445-1448.
[17] Zhao Yusheng,Xie Shaojun,Li Xiaojie,et al.Repressor of silencing5 encodes a member of the small heat shock protein family and is required for DNA demethylation in arabidopsis[J].Plant Cell,2014,26(6):2660-2675.
[18] Qian Weiqiang,Miki D,Lei Mingguang,et al.Regulation of active DNA demethylation by an α-crystallin domain protein in Arabidopsis[J].Molecular Cell,2014,55(3):361-371.
[19] Li Yan,Córdoba-Cañero D,Qian Weiqiang,et al.An AP endonuclease functions in active DNA demethylation and gene imprinting in Arabidopsis[J].PLoS Genetics,2015,11(1):e1004905.
[20] Lee J,Jang H,Shin H,et al.AP endonucleases process 5-methylcytosine excision intermediates during active DNA demethylation in Arabidopsis[J].Nucleic Acids Research,2014,42(18):11408-11418.
[21] Lang Zhaobo,Lei Mingguang,Wang Xingang,et al.The Methyl-CpG-binding protein MBD7 facilitates active DNA demethylation to limit DNA hyper-methylation and transcriptional gene silencing[J].Molecular Cell,2015,57(6):971-983.
[22] Li Qi,Wang Xiaokang,Sun Han,et al.Regulation of active DNA demethylation by a methyl-CpG-binding domain protein in Arabidopsis thaliana[J].PLoS Genetics,2015,11(5):e1005210.
[23] Kouzarides T.Chromatin modifications and their function[J].Cell,2007,128(4):693-705.
[24] Jiang C,Pugh B F.Nucleosome positioning and gene regulation:advances through genomics[J].Nature Reviews Genetics,2009,10(3):161-172.
[25] Sun Weijian,Zhou Xiang,Zheng Jihang,et al.Histone acetyltransferases and deacetylases:molecular and clinical implications to gastrointestinal carcinogenesis[J].Acta Biochimica et Biophysica Sinica,2012,44(1):80-91.
[26] Clements E G,Mohammad H P,Leadem B R,et al.DNMT1 modulates gene expression without its catalytic activity partially through its interactions with histone-modifying enzymes[J].Nucleic Acids Research,2012,40(10):4334-4346.
[27] Allfrey V G,Faulkner R,Mirsky A E.Acetylation and methylation of histones and their possible role in the regulation of RNA synthesis[J].Proceedings of the National Academy of Sciences of the United States of America,1964,51(5):786-794.
[28] Pogo B G T,Allfrey V G,Mirsky A E.RNA synthesis and histone acetylation during the course of gene activation in lymphocytes[J].Proceedings of the National Academy of Sciences of the United States of America,1966,55(4):805-812.
[29] Hon G C,Hawkins R D,Ren B.Predictive chromatin signatures in the mammalian genome[J].Human Molecular Genetics,2009,18(R2):R195-R201.
[30] Schmitges F W,Prusty A B,Faty M,et al.Histone methylation by PRC2 is inhibited by active chromatin marks[J].Molecular Cell,2011,42(3):330-341.
[31] Banerjee T,Chakravarti D.A peek into the complex realm of histone phosphorylation[J].Molecular and Cellular Biology,2011,31(24):4858-4873.
[32] He Yingzi,Tang Dongmei,Cai Chengfu,et al.LSD1 is required for hair cell regeneration in zebrafish[J].Molecular Neurobiology,2016,53(4):2421-2434.
[33] Wang Yang,Li Yuejiao,Guo Chen,et al.ISL1 and JMJD3 synergistically control cardiac differentiation of embryonic stem cells[J].Nucleic Acids Research,2016,44(14):6741-6755.
[34] Tse C,Sera T,Wolffe A P,et al.Disruption of higher-order folding by core histone acetylation dramatically enhances transcription of nucleosomal arrays by RNA polymerase III[J].Molecular and Cellular Biology,1998,18(8):4629-4638.
[35] Wang A,Kurdistani S K,Grunstein M.Requirement of Hos2 histone deacetylase for gene activity in yeast[J].Science,2002,298(5597):1412-1414.
[36] Riddihough G,Pennisi E.The evolution of epigenetics[J].Science,2001,293(5532):1063.
[37] Racki L R,Narlikar G J.ATP-dependent chromatin remodeling enzymes:two heads are not better,just different[J].Current Opinion in Genetics & Development,2008,18(2):137-144.
[38] Wang G G,Allis C D,Chi Ping.Chromatin remodeling and cancer,part I:covalent histone modifications[J].Trends in Molecular Medicine,2007,13(9):363-372.
[39] Barrett R M,Wood M A.Beyond transcription factors:The role of chromatin modifying enzymes in regulating transcription required for memory[J].Learning & Memory,2008,15(7):460-467.
[40] Taverna S D,Li Haitao,Ruthenburg A J,et al.How chromatinbinding modules interpret histone modifications:Lessons from professional pocket pickers[J].Nature Structural&Molecular Biology,2007,14(11):1025-1040.
[41] Li Bing,Carey M,Workman J L.The role of chromatin during transcription[J].Cell,2007,128(4):707-719.
[42] Gangaraju V K,Bartholomew B.Mechanisms of ATP dependent chromatin remodeling[J].Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis,2007,618(1-2):3-17.
[43] Peterson C L,Herskowitz I.Characterization of the yeast SWI1,SWI2,and SWI3 genes,which encode a global activator of transcription[J].Cell,1992,68(3):573-583.
[44] Mattick J S,Makunin I V.Non-coding RNA.Human Molecular Genetics,2006,15(S1):R17-R29.
[45] Ma Lina,Bajic V B,Zhang Zhang.On the classification of long non-coding RNAs[J].RNA Biology,2013,10(6):924-933.
[46] Quinn J J,Chang H Y.Unique features of long non-coding RNA biogenesis and function[J].Nature Reviews Genetics,2016,17(1):47.
[47] Cech T R,Steitz1 J A.The noncoding RNA revolution—trashing old rules to forge new ones[J].Cell,2014,157(1):77-94.
[48] 刘旭,单革.非编码RNA对基因转录的调控[J].生命科学,2016,28(5):569-575.
[49] Kim D H,Saetrom P,Snøve Jr O,et al.MicroRNA-directed transcriptional gene silencing in mammalian cells[J].Proceedings of the National Academy of Sciences of the United States of America,2008,105(42):16230-16235.
[50] Saldaña-Meyer R,González-Buendía E,Guerrero G,et al.CTCF regulates the human p53 gene through direct interaction with its natural antisense transcript,Wrap53[J].Genes & Development,2014,28(7):723-734.
[51] Pickering A D.Environmental stress and the survival of brown trout,salmo trutta[J].Freshwater Biology,1989,21(1):47-55.
[52] Liu B,Xu P,Brown P B,et al.The effect of hyperthermia on liver histology,oxidative stress and disease resistance of the Wuchang bream,Megalobrama amblycephala[J].Fish& Shellfish Immunology,2016,52:317-324.
[53] Navarro-Martín L,Viñas J,Ribas L,et al.DNA methylation of the gonadal aromatase(cyp19a)promoter is involved in temper ature-dependent sex ratio shifts in the european sea bass[J].PLoS Genetics,2011,7(12):e1002447.
[54] Jabbari K,Cacciò S,de Barros J P P,et al.Evolutionary changes in CpG and methylation levels in the genome of vertebrates[J].Gene,1998,205(1-2):109-118.
[55] Johnston I A,Lee H T,Macqueen D J,et al.Embryonic temperature affects muscle fibre recruitment in adult zebrafish:genomewide changes in gene and microRNA expression associated with the transition from hyperplastic to hypertrophic growth phenotypes[J].Journal of Experimental Biology,2009,212(Pt 12):1781-1793.
[56] 李尚俊,孙国华,李雪燕,等.高温胁迫下仿刺参表观遗传调控相关基因的表达特征[J].中国水产科学,2017,24(3):470-476.
[57] Fernandes J M,MacKenzie M G,Kinghorn J R,et al.FoxK1 splice variants show developmental stage-specific plasticity of expression with temperature in the tiger pufferfish[J].Journal of Experimental Biology,2007,210(Pt 19):3461-3472.
[58] Campos C,Valente L M P,Fernandes J M O.Molecular evolution of zebrafish dnmt3 genes and thermal plasticity of their expression during embryonic development[J].Gene,2012,500(1):93-100.
[59] 朱华平,卢迈新,黄樟翰,等.低温对罗非鱼基因组DNA甲基化的影响[J].水产学报,2013,37(10):1460-1467.
[60] Giannetto A,Nagasawa K,Fasulo S,et al.Influence of photoperiod on expression of DNA(cytosine-5)methyltransferases in Atlantic cod[J].Gene,2013,519(2):222-230.
[61] Kim D J,Seok S H,Baek M W,et al.Benomyl induction of brain aromatase and toxic effects in the zebrafish embryo[J].Journal of Applied Toxicology,2009,29(4):289-294.
[62] Vandegehuchte M B,Lemière F,Vanhaecke L,et al.Direct and transgenerational impact on Daphnia magna of chemicals with a known effect on DNA methylation[J].Comparative Biochemistry and Physiology Part C:Toxicology & Pharmacology,2009,151(3):278-285.
[63] Guerrero-Bosagna C,Settles M,Lucker B,et al.Epigenetic transgenerational actions of vinclozolin on promoter regions of the sperm epigenome[J].PLoS One,2010,5(9):e13100.
[64] Strömqvist M,Tooke N,Brunström B.DNA methylation levels in the 5′flanking region of the vitellogenin I gene in liver and brain of adult zebrafish(Danio rerio)—Sex and tissue differences and effects of 17α-ethinylestradiol exposure[J].Aquatic Toxicology,2010,98(3):275-281.
[65] Prins G S,Tang W Y,Belmonte J,et al.Perinatal exposure to oestradiol and bisphenol a alters the prostate epigenome and increases susceptibility to carcinogenesis[J].Basic&Clinical Pharmacology & Toxicology,2008,102(2):134-138.
[66] Pierron F,Colombier S B D,Moffett A,et al.Abnormal ovarian DNA methylation programming during gonad maturation in wild contaminated fish[J].Environmental Science & Technology,2014,48(19):11688-11695.
[67] Xing Houjuan,Wang Chao,Wu Hongda,et al.Effects of atrazine and chlorpyrifos on DNA methylation in the brain and gonad of the common carp[J].Comparative Biochemistry and Physiology Part C:Toxicology & Pharmacology,2015,168:11-19.
[68] Seemann F,Jeong C B,Zhang Ge,et al.Ancestral benzo[a]pyrene exposure affects bone integrity in F3 adult fish(Oryzias latipes)[J].Aquatic Toxicology,2016,183:127-134.
[69] 夏菠.Cr(Ⅵ)和B[a]P对16HBE细胞的联合毒性效应及表观遗传改变的研究[D].长沙:中南大学,2012.
[70] Baccarelli A,Bollati V.Epigenetics and environmental chemicals[J].Current Opinion in Pediatrics,2009,21(2):243-251.
[71] Lee Y W,Broday L,Costa M.Effects of nickel on DNA methyltransferase activity and genomic DNA methylation levels[J].Mutation Research/Genetic Toxicology and Environmental Mutagenesis,1998,415(3):213-218.
[72] Uthus E O,Davis C.Dietary arsenic affects dimethylhydrazine-induced aberrant crypt formation and hepatic global DNA methylation and DNA methyltransferase activity in rats[J].Biological Trace Element Research,2005,103(2):133-145.
[73] Bagnyukova T V,Luzhna L I,Pogribny I P,et al.Oxidative stress and antioxidant defenses in goldfish liver in response to shortterm exposure to arsenite[J].Environmental and Molecular Mutagenesis,2007,48(8):658-665.
[74] 周新文,朱国念,Mwalilino J,等.Cu、Zn、Pb、Cd 及其混合重金属离子对鲫鱼(Carassius auratus)DNA甲基化水平的影响[J].中国环境科学,2001,21(6):546-552.
[75] 王丙莲,张迎梅,谭玉凤,等.镉铅对泥鳅DNA甲基化水平的影响[J].毒理学杂志,2006,20(2):78-80.
[76] Sánchez-Martín F J,Lindquist D M,Landero-Figueroa J,et al.Sex-and tissue-specific methylome changes in brains of mice perinatally exposed to lead[J].NeuroToxicology,2015,46:92-100.
[77] Pierron F,Baillon L,Sow M,et al.Effect of low-dose cadmium exposure on DNA methylation in the endangered European eel[J].Environmental Science & Technology,2014,48(1):797-803.
[78] Chilcote M W C,Goodson K W G,Falcy M R.Reduced recruitment performance in natural populations of anadromous salmonids associated with hatchery-reared fish[J].Canadian Journal of Fisheries and Aquatic Sciences,2011,68(3):511-522.
[79] Blouin M S,Thuillier V,Cooper B,et al.No evidence for large differences in genomic methylation between wild and hatchery steelhead(Oncorhynchus mykiss)[J].Canadian Journal of Fisheries and Aquatic Sciences,2010,67(2):217-224.
[80] Lv Jianjian,Liu Ping,Gao Baoquan,et al.The identification and characteristics of salinity-related microRNAs in gills of Portunus trituberculatus[J].Cell Stress and Chaperones,2016,21(1):63-74.
[81] Yan Biao,Zhao Lihui,Guo Jinliang,et al.miR-429 regulation of osmotic stress transcription factor 1(OSTF1)in tilapia during osmotic stress[J].Biochemical and Biophysical Research Communications,2012,426(3):294-298.
[82] Flynt A S,Thatcher E J,Burkewitz K,et al.miR-8 microRNAs regulate the response to osmotic stress in zebrafish embryos[J].The Journal of Cell Biology,2009,185(1):115-127.
[83] Guo Hui,Xian Jian'an,Li Bin,et al.Gene expression of apo ptosis-related genes, stress protein and antioxidant enzymes in hemocytes of white shrimp Litopenaeus vannamei under nitrite stress[J].Comparative Biochemistry and Physiology Part C:Toxicology & Pharmacology,2013,157(4):366-371.
[84] Sun Shengming,Ge Xiangping,Zhu Jian,et al.Identification and mRNA expression of antioxidant enzyme genes associated with the oxidative stress response in the Wuchang bream(Megalobrama amblycephala Yih)in response to acute nitrite exposure[J].Comparative Biochemistry and Physiology Part C:Toxicology&Pharmacology,2014,159:69-77.
[85] Cheng Changhong,Yang Fangfang,Ling Renzhi,et al.Effects of ammonia exposure on apoptosis,oxidative stress and immune response in pufferfish(Takifugu obscurus)[J].Aquatic Toxicology,2015,164:61-71.
[86] Mennigen J A,Skiba-Cassy S,Panserat S.Ontogenetic expression of metabolic genes and microRNAs in rainbow trout alevins during the transition from the endogenous to the exogenous feeding period[J].The Journal of Experimental Biology,2013,216(Pt 9):1597-1608.
[87] Zhu Xin,Chen Dunxun,Hu Yi,et al.The microRNA signature in response to nutrient restriction and refeeding in skeletal muscle of Chinese perch(Siniperca chuatsi)[J].Marine Biotechnology,2015,17(2):180-189.
[88] Mazurais D,Darias M J,Gouillou-Coustans M F,et al.Dietary vitamin mix levels influence the ossification process in European sea bass(Dicentrarchus labrax)larvae[J].American Journal of Physiology.Regulatory, Integrative and Comparative Physiology,2007,294(2):R520-R527.
[89] Škugor S,Škugor A,Todorˇcevic'M,et al.Exposure to lipopolysaccharide induces immune genes in cultured preadipocytes of Atlantic salmon[J].Fish & shellfish immunology,2010,29(5):817-824.
[90] Marandel L,Lepais O,Arbenoits E,et al.Remodelling of the hepatic epigenetic landscape of glucose-intolerant rainbow trout(Oncorhynchus mykiss)by nutritional status and dietary carbohydrates[J].Scientific Reports,2016,6:32187.
[91] Rees W D,Hay S M,Brown D S,et al.Maternal protein deficiency causes hypermethylation of DNA in the livers of rat fetuses[J].Journal of Nutrition,2000,130(7):1821-1826.
[92] Burdge G C,Lillycrop K A.Fatty acids and epigenetics[J].Current Opinion in Clinical Nutrition and Metabolic Care,2014,17(2):156-161.
[93] Sargent J R,Bell M V,Bell J G,et al.Origins and functions of n-3 polyunsaturated fatty acids in marine organisms[M].Champaign:Ceve G and Paltauf F,1995:248-259.
[94] Pickova J,Kiessling A,Pettersson A,et al.Fatty acid and carotenoid composition of eggs from two nonanadromous Atlantic salmon stocks of cultured and wild origin[J].Fish Physiology and Biochemistry,1999,21(2):147-156.
[95] Cruz-Garcia L,Sánchez-Gurmaches J,Bouraoui L,et al.Changes in adipocyte cell size,gene expression of lipid metabolism markers,and lipolytic responses induced by dietary fish oil replacement in gilthead sea bream(Sparus aurata L.)[J].Comparative Biochemistry and Physiology Part A:Molecular&Integrative Physiology,2011,158(4):391-399.
[96] Zakeri M,Kochanian P,Marammazi J G,et al.Effects of dietary n-3 HUFA concentrations on spawning performance and fatty acids composition of broodstock,eggs and larvae in yellowfin sea bream,Acanthopagrus latus[J].Aquaculture,2011,310(3-4):388-394.
[97] Niculescu M D,Lupu D S,Craciunescu C N.Perinatal manipulation of α-linolenic acid intake induces epigenetic changes in maternal and offspring livers[J].The FASEB Journal,2013,27(1):350-358.
[98] Benatti P,Peluso G,Nicolai R,et al.Polyunsaturated fatty acids:biochemical,nutritional and epigenetic properties[J].Journal of the American College of Nutrition,2004,23(4):281-302.
[99] Seierstad S L,Svindland A,Larsen S,et al.Development of intimal thickening of coronary arteries over the lifetime of Atlantic salmon,Salmo salar L.,fed different lipid sources[J].Journal of Fish Diseases,2008,31(6):401-413.
[100] Xing Jinyi,Kang Li,Jiang Yunliang.Effect of dietary betaine supplementation on lipogenesis gene expression and CpG methylation of lipoprotein lipase gene in broilers[J].Molecular Biology Reports,2011,38(3):1975-1981.
[101] Xing Jinyi,Jiang Yunliang.Effect of dietary betaine supplementation on mRNA level of lipogenesis genes and on promoter CpG methylation of fatty acid synthase(FAS)gene in laying hens[J].African Journal of Biotechnology,2012,11(24):6633-6640.
[102] McGhee K E,Bell A M.Paternal care in a fish:epigenetics and fitness enhancing effects on offspring anxiety[J].Proceedings of the Royal Society B:Biological Sciences,2014,281(1794):20141146.
[103] Mizukami T,Kanai Y,Fujisawa M,et al.Five azacytidine,a DNA methyltransferase inhibitor,specifically inhibits testicular cord formation and Sertoli cell differentiation in vitro[J].Molecular Reproduction & Development,2008,75(6):1002-1010.
[104] Matsumoto Y,Buemio A,Chu R,et al.Epigenetic control of gonadal aromatase(cyp19a1)in temperature-dependent sex determination of red-eared slider turtles[J].PLoS One,2013,8(6):e63599.
[105] Merchant-Larios H,Díaz-Hernández V.Environmental sex determination mechanisms in reptiles[J].Sexual Development,2013,7(1-3):95-103.
[106] Xiao Jun,Song Can,Liu Shaojun,et al.DNA methylation analysis of allotetraploid hybrids of red crucian carp(Carassius auratus red var.)and common carp(Cyprinus carpio L.)[J].PLoS One,2013,8(2):e56409.
[107] 王智诚,周晓旭,王昊泽,等.长链非编码RNA及其在斑马鱼中的研究进展[J].大连海洋大学学报,2017,32(2):248-254.
[108] Sadhu N,Sharma S R K,Joseph S,et al.Chronic stress due to high stocking density in open sea cage farming induces variation in biochemical and immunological functions in Asian seabass(Lates calcarifer,Bloch)[J].Fish Physiology and Biochemistry,2014,40(4):1105-1113.
[109] Xie Jun,Liu Bo,Zhou Qunlan,et al.Effects of anthraquinone extract from rhubarb Rheum officinale Bail on the crowding stress response and growth of common carp Cyprinus carpio var.Jian[J].Aquaculture,2008,281(1-4):5-11.
[110] Costas B,Aragão C,Mancera J M,et al.High stocking density induces crowding stress and affects amino acid metabolism in Senegalese sole Solea senegalensis(Kaup 1858)juveniles[J].Aquaculture Research,2008,39(1):1-9.
[111] Caipang C M A,Brinchmann M F,Berg I,et al.Changes in selected stress and immune-related genes in Atlantic cod,Gadus morhua, following overcrowding[J].Aquaculture Research,2008,39(14):1533-1540.
[112] Gornati R,Papis E,Rimoldi S,et al.Rearing density influences the expression of stress-related genes in sea bass(Dicentrarchus labrax,L.)[J].Gene,2004,341:111-118.
[113] Aksakal E,Ekinci D,Erdoˇgan O,et al.Increasing stocking density causes inhibition of metabolic-antioxidant enzymes and elevates mRNA levels of heat shock protein 70 in rainbow trout[J].Livestock Science,2011,141(1):69-75.
[114] Salas-Leiton E,Anguis V,Martín-Antonio B,et al.Effects of stocking density and feed ration on growth and gene expression in the Senegalese sole(Solea senegalensis):potential effects on the immune response[J].Fish & Shellfish Immunology,2010,28(2):296-302.
[115] Bouligand J,Ghervan C,Tello J A,et al.Isolated familial hypogonadotropic hypogonadism and a GNRH1 mutation[J].New England Journal of Medicine,2009,360(26):2742-2748.
[116] Alvarado S G,Lenkov K,Williams B,et al.Social crowding during development causes changes in GnRH1 DNA methylation[J].PLoS One,2015,10(10):e0142043.
[117] Rebl A,Zebunke M,Borchel A,et al.Microarray-predicted marker genes and molecular pathways indicating crowding stress in rainbow trout(Oncorhynchus mykiss)[J].Aquaculture,2017,473:355-365.
[118] Zhang Tieyuan,Hellstrom I C,Bagot R C,et al.Maternal care and DNA methylation of a glutamic acid decarboxylase 1 promoter in rat hippocampus[J].Journal of Neuroscience,2010,30(39):13130-13137.