体外受精是绝大多数海洋动物的繁殖方式,在开放水体中,同种配子间的精准识别是体外受精成功的关键,也是保障海洋生物物种独立性和稳定性的重要环节。大量研究显示,配子识别蛋白是海洋生物体外受精过程中保证同种配子准确识别并快速融合的决定性因子之一,也是同种海洋生物不同群体间形成生殖隔离的必需条件[1]。大量研究显示,精子细胞溶素 (lysin)和精子结合素 (bindin)是海洋生物配子识别蛋白的两个重要种类。有研究显示,在贝类配子识别蛋白研究方面,在进化研究尺度内,鲍Abalone的lysin蛋白的氨基酸在快速替换过程中存在较大的适应值,符合达尔文的正向选择规律,也正是这种配子识别蛋白的快速分化促进了鲍不同种间生殖隔离分子屏障的形成,从而确保了鲍的种类多样性的稳定[2]。通过对巨牡蛎属Crassostrea种类的长牡蛎Crassostrea gigas、熊本牡蛎Crassostrea sikamea和葡萄牙牡蛎Crassostrea angulata bindin蛋白基因的研究发现,巨牡蛎属种类bindin蛋白无论在分子大小还是在序列结构上,均存在比较明显的种内多样性,此外,在正向选择压力下,牡蛎bindin蛋白可通过快速替换、可变剪切等以适应其相对应的卵子表面受体的变化,从而达到二者的协同进化,保障牡蛎配子的种间生殖隔离和种内配子兼容性[3]。
棘皮动物Echinodermata属于后口动物Deuterostomia,在进化上属于无脊椎动物的高等类别,其中,海胆纲Echinoidea的部分种类作为发育生物学和繁殖生物学的模式生物已经有近百年的历史。与贝类等其他海洋生物相比,棘皮动物配子识别蛋白的研究起步较早。在20世纪初期,Lillie[4]就发现,海胆纲动物的精子能够凝集在同种卵子的表面,继而诱发顶体反应;Vacquier等[5]发现,海胆精子中含有一种特殊的凝集素样不溶性颗粒蛋白,因其可以特异性地识别相对应卵子表面的糖蛋白受体,促进精-卵的识别和融合,故被称为结合素(bindin)。目前,关于棘皮动物配子识别蛋白的研究主要集中于bindin蛋白基因的分子结构、蛋白质功能、适应性进化特点等方面。
1 棘皮动物bindin的序列特征
关于棘皮动物bindin分子结构和序列特征的研究最早始于20世纪80年代。1986年,Gao等[6]利用cDNA文库构建、基因组印记杂交 (Genome Blot Hybridization)、体内翻译 (in vitro translation)和免疫沉淀反应 (immunoprecipitation)、RNA印记杂交 (RNA Blot Hybridizations)等技术,从紫球海胆Strongylocentrotus purpuratus中首次获得了紫球海胆bindin(Spbindin,NCBI登录号:M14487.1)基因的全长cDNA序列,结果显示,紫球海胆bindin基因的cDNA序列全长为1873 bp,其中,包含一个1446 bp的开放阅读框,一个141 bp的5′端非编码区和一个286 bp的3′端非编码区,经生物信息学分析,该序列编码成熟的bindin蛋白 (含有236个氨基酸),其N端起始于Spbindin基因mRNA编码多肽的第245个氨基酸。通过这一研究成果,第一次对海胆纲动物bindin基因的序列信息和特征有了初步的了解。随后,大量关于棘皮动物bindin的分子克隆和生物信息学分析等研究工作相继开展。迄今为止,据NCBI数据库 (https://www.ncbi.nlm.nih.gov/nucleotide/)显示,已经登录的棘皮动物bindin基因序列共有2266条,其中,完整序列5条,部分序列2261条,所涉及种类主要来自于海胆纲和海星纲。
通过比较NCBI数据库中已经登录的5条完整的棘皮动物bindin基因序列可以发现,不同棘皮动物的bindin基因序列全长以及编码的相应蛋白长度存在较大差异 (表1)。在海胆纲Echinoidea动物中,海胆 Lytechinus variegatus的 bindin基因序列(Lvbindin,NCBI登录号:M59489.1)最长,为2584 bp,该基因包含一个162 bp的5′端非编码区和一个1033 bp的3′端非编码区,其开放阅读框为1389 bp,编码一个含有462个氨基酸的多肽,其中,信号肽序列由19个氨基酸组成。海胆Strongylocentrotus franciscanus和Echinometra lucunter的bindin基因序列 (Sfbindin, NCBI登录号:M59490.1; Elbindin,NCBI登录号: AY452683.1)全长分别为2307 bp和1944 bp,开放阅读框为1458 bp和1620 bp,二者均含有一个20个氨基酸的信号肽序列,分别编码485和539个氨基酸的多肽,这两种海胆bindin基因的5′端非编码区分别是4种海胆中最长和最短的,分别为193 bp和75 bp;3′端非编码区分别为656 bp和249 bp。通过比较发现,Elbindin基因的开放阅读框最长编码氨基酸数目最多。值得注意的是,虽然海胆S.purpuratus的bindin基因序列全长只有1873 bp,是现有海胆bindin基因cDNA全长序列中最短的,但是,其开放阅读框所含的核苷酸数目却大于海胆L.variegatus和E.lucunter, 为1446 bp, 编码481个氨基酸,具有一个包含20个氨基酸的信号肽序列。以上结果提示,与牡蛎种类相似,海胆纲动物bindin基因的DNA和蛋白序列存在较大的种内差异。
在海星纲Asteroidea动物中,已经获得全长bindin基因序列的仅海星 Patiria miniata一种(Pmbindin,NCBI登录号:FJ439659.1)。海星P.miniata的bindin基因cDNA序列全长为3819 bp,包含一个3075 bp的开放阅读框,编码含有1024个氨基酸的多肽,其信号肽序列由23个氨基酸组成,Pmbindin的3′端非编码区为744 bp,不具有5′端非编码区。与海胆纲的4个种类相比,海星P.miniata具有较长的序列全长和开放阅读框,编码的氨基酸序列远大于海胆纲的4种动物,通过比较可以发现,只有海星P.miniata不具有5′端非编码区,即开放阅读框起始于bindin mRNA的第一位核苷酸,其信号肽序列也与海胆纲物种具有明显差异,这一结果可从一定程度上证实了棘皮动物bindin基因序列的种间变异性特征。
表1 部分棘皮动物bindin基因及蛋白序列
Tab.1 Sequence characterization of bindin gene in some echinoderm species
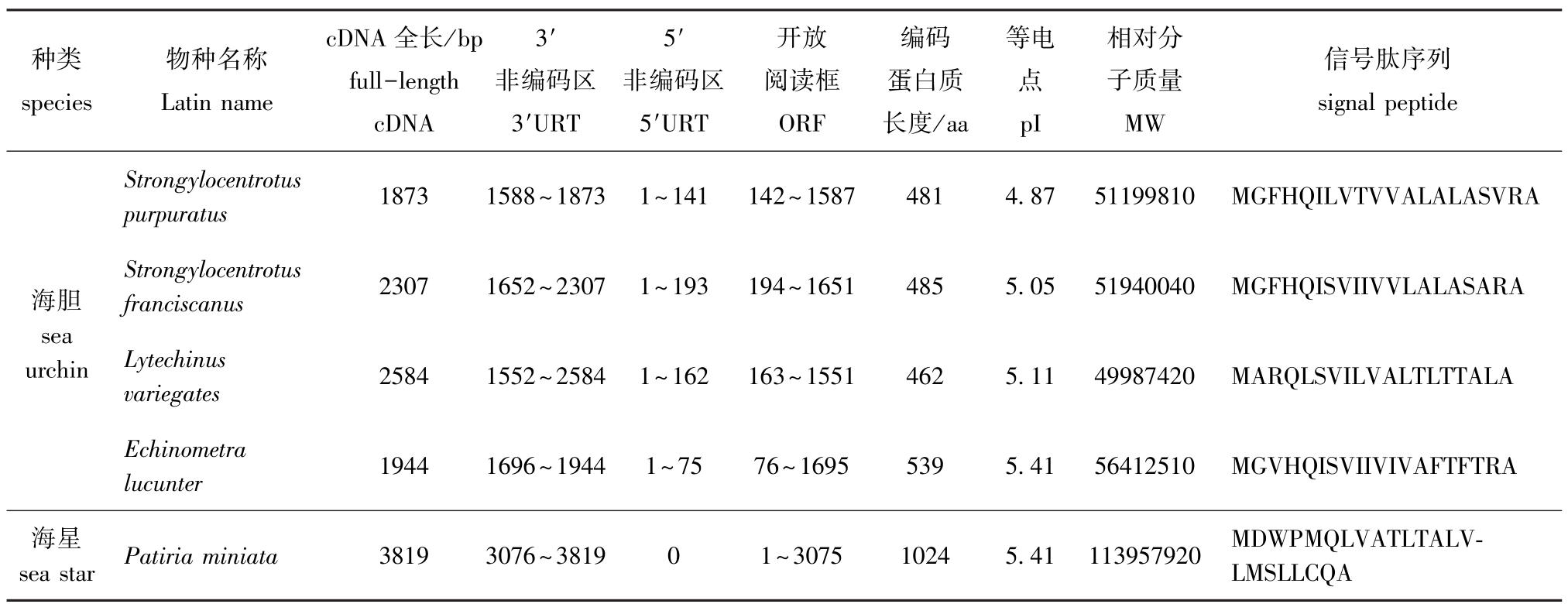
种类species物种名称Latin name cDNA全长/bp full-length cDNA 3′非编码区3′URT 5′非编码区5′URT开放阅读框ORF编码蛋白质长度/aa等电点pI相对分子质量MW信号肽序列signal peptide Strongylocentrotus purpuratus 1873 1588~1873 1~141 142~1587 481 4.87 51199810 MGFHQILVTVVALALASVRA海胆sea urchin Strongylocentrotus franciscanus 2307 1652~2307 1~193 194~1651 485 5.05 51940040 MGFHQISVIIVVLALASARA Lytechinus variegates 2584 1552~2584 1~162 163~1551 462 5.11 49987420 MARQLSVILVALTLTTALA Echinometra lucunter 1944 1696~1944 1~75 76~1695 539 5.41 56412510 MGVHQISVIIVIVAFTFTRA海星sea star Patiria miniata 3819 3076~3819 0 1~3075 1024 5.41 113957920 MDWPMQLVATLTALVLMSLLCQA
关于bindin蛋白的早期研究证实,棘皮动物bindin蛋白最初是以一种保守的前体蛋白原 (prepropeptide)形式存在,该前体蛋白原包含有一个成对碱性氨基酸蛋白酶 (furin)型的剪切位点、一个含有55~60个氨基酸的高度保守核心区以及两个位于核心区两侧的高度可变重复区[7-8]。通过对不同种类棘皮动物bindin蛋白序列比较发现,无论是海星纲动物还是海胆纲动物的bindin蛋白序列中,均存在大小不同的保守bindin结构域,且该结构域大都位于蛋白序列的后段 (图1),与海胆纲动物不同的是,海星P.miniata的蛋白序列中还包含两个核糖核酸酶E(ribonuclease E)结构域。研究显示,不同种类棘皮动物的bindin前体蛋白原经翻译后加工,可形成长度不一的成熟bindin蛋白,成熟的海胆bindin蛋白长度范围一般在193~418个氨基酸之间,是一个相对分子质量约为30 500的糖蛋白[9]。bindin蛋白多序列比对结果显示,bindin蛋白氨基酸序列在海胆纲动物中相似性较高,海星纲动物则与之相差较大 (图2)。通过对bindin蛋白二级结构的比较发现,其大部分卷曲、螺旋等结构位点较为一致,但不同种间仍存在差异(图3)。海胆S.purpuratus的bindin蛋白二级结构中含有19个卷曲、5个折叠和13个螺旋。同为Strongylocentrotus属的海胆 S.franciscanus的 bindin蛋白二级结构与之相比,则多1个折叠结构和2个螺旋结构。海胆L.variegatus的bindin蛋白二级结构由18个卷曲、3个折叠和14个螺旋组成。海胆E.lucunter的bindin蛋白二级结构则由21个卷曲、4个折叠和17个螺旋构成。而海星P.miniata的bindin蛋白二级结构包含的卷曲、折叠和螺旋数目分别为17个、4个和14个。
2 棘皮动物bindin蛋白的生物学功能
早在1977年,Glabe等[10]就已发现,纯化后的bindin蛋白可以与同种的卵细胞特异性识别并结合,具有帮助精子黏附于卵子上的作用。研究显示,bindin蛋白是一种不溶性的凝集素样颗粒物质,特异性地分布于棘皮动物精子的顶体中。利用过氧化物酶偶联抗体的方法对海胆bindin蛋白在顶体反应前后的转运途径进行定位分析发现,当海胆卵子胶膜上的海藻硫酸盐激发精子的顶体反应后,成熟的bindin蛋白颗粒便从顶体囊泡中释放至精子顶端的膜表面,与卵子表面相对应的糖蛋白受体发生相互作用,即配子的相互识别,从而使精子最终能够凝集在同种类卵子的卵黄膜上[11]。大量实验已经证明,bindin蛋白在配子识别过程中具有物种特异性,即bindin蛋白与卵子的黏附具有物种特异性,一种bindin蛋白只有被同种卵子有效识别,才能进一步完成受精作用[10,12-13]。1998年,Ulrich等[13-14]在研究中发现,在海胆成熟bindin蛋白的核心保守区存在一个由18个氨基酸组成的多肽。2004年,Afonin等[15]研究发现,该多肽可形成一个 “螺旋-断裂-螺旋 (helix-break-helix)”的超二级结构,该结构两端的α螺旋具有不同的疏水性,其中,疏水性较强一端的α螺旋可插入脂质双分子层,进而破坏磷脂细胞膜,而另一端的α螺旋则仍留在磷脂膜表面替代磷脂头部的水分子。综上,“B18区”(氨基酸序列为:LGLLLRHLRHHSNLLANI)多肽的作用主要是便于磷脂质膜的聚集和融合。
上述研究结果表明,不仅提示bindin蛋白可能在棘皮动物受精过程中兼具配子识别和配子融合两种作用[4],同时,也在一定程度上解释了,为什么在自然环境下,虽然有些异源的配子可以 “侥幸”识别,甚至能够诱导顶体反应的发生,但是,随后的配子融合却不能正常进行,即bindin的种属特异性与配子识别和配子融合密切相关,也是种群多样性产生和维持种群生殖隔离稳定性的重要原因[3,11]。最新的研究显示,位于夏威夷卡内奥赫湾沿岸的两种近缘夏威夷海胆Echinometra mathaei和E.oblonga之间存在比较严重的生殖隔离现象,通过研究发现,这两种海胆虽然在线粒体基因和核基因上几乎没有差异,即呈现近缘效应,但是,这两种亲缘关系很近的海胆在bindin基因序列上却存在很大差异,导致其编码的bindin蛋白在蛋白序列和空间结构上存在较大差异,最终导致近缘精卵之间不能有效识别,进而产生合子前生殖隔离,但其具体的分子机制仍有待深入研究[16]。
3 棘皮动物bindin的进化特点
一般说来,某一基因的进化速率与其生物功能的重要性密切相关,即越具有重要功能的基因其进化速率相对越慢[17],然而,大量研究表明,在海洋生物配子识别以及精卵融合过程中发挥重要作用的配子识别蛋白却经历着相对快速的适应性进化,因此,研究配子识别蛋白的进化规律以及进化机制,已成为海洋生物繁殖生物学的热点领域之一。
通过序列挖掘技术,收集NCBI数据库中现有的部分棘皮动物已知的bindin基因序列信息 (表2)
并利用邻接法 (neighbor-joining,NJ)构建基于bindin基因核苷酸序列的分子系统进化树 (图4),经聚类分析发现,Paracentrotus属两种海胆聚为一支,Strongylocentrotus属两种海胆聚为一支,并与其他种海胆最终聚为一支。所有海星最终也聚为一支,且分别同属 Pisaster、Cryptasterina、Parvulastra、Patiria属的物种均分别聚为一支。这一结果表明,bindin基因在海胆纲和海星纲动物的种内和
种间均存在明显的分化,其进化规律以及具体的进化机制可能具有种属特异性,这一结果也在一定程度上支持了Wu等[18]提出的 “bindin的分化可代表生殖隔离和物种形成的历史过程”这一假设。


图1 部分棘皮动物bindin核苷酸及其推导氨基酸序列的比较
Fig.1 Nucleotide and deduced amino acid sequences of bindin in some echinoderm species
注:左边的数字标明了核苷酸序列及推导的氨基酸序列。∗表示终止密码子,灰色部分为bindin保守结构域,加粗部分为EB病毒核抗原3(EBNA-3)结构域,下划线部分为核糖核酸酶E结构域。A为海胆Strongylocentrotus purpuratus;B为海胆Strongylocentrotus franciscanus;C为海胆Lytechinus variegatus;D为海胆Echinometra lucunter;E为海星Patiria miniata
Note: The numbers on the left indicate the nucleotide and deduced amino acid sequences.∗ indicates the stop codon, conserved bindin domain is shaded in gray,bolding shows Epstein-Barr virus nuclear antigen 3(EBNA-3)and the ribonuclease E domain is underlined.A,sea urchin Strongylocentrotus purpuratus; B, sea urchin Strongylocentrotus franciscanus; C, sea urchin Lytechinus variegatus; D, sea urchin Echinometra lucunter; E,sea star Patiria miniata

图2 部分棘皮动物bindin蛋白多序列比对
Fig.2 Multiple sequence alignment of deduced amino acid sequences from some echinoderm species

图3 部分棘皮动物bindin蛋白二级结构比较
Fig.3 Secondary structure prediction of bindin proteins from some echinoderm species
注:黑线 (C)、黄色箭头 (E)和粉红色的圆柱体 (H)分别代表卷曲、折叠和螺旋。A为海胆Strongylocentrotus purpuratus bindin蛋白;B为海胆Strongylocentrotus franciscanus bindin蛋白;C为海胆Lytechinus variegatus bindin蛋白;D为海胆Echinometra lucunter bindin蛋白;E为海星Patiria miniata bindin蛋白
Note: Black lines(C),yellow arrows(E)and pink cylinders(H)represent coils,strands and helices,respectively.A,sea urchin Strongylocentrotus purpuratus; B, sea urchin Strongylocentrotus franciscanus; C, sea urchin Lytechinus variegatus; D, sea urchin Echinometra lucunter; E, sea star Patiria miniata
有研究表明,棘皮动物bindin基因的起源至少可追溯到二亿五千万年前[19],石笔海胆Heterocentrotus mamillatus与海胆纲其他种类出现分离的时期。研究显示,在二亿五千万年的进化历程中,棘皮动物bindin蛋白保守的核心区一段氨基酸序列为LGLLLRHLRHHSNLLANI的区域呈现出了高度的进化保守性,被称为 “B18区”,该区域的生物功能现已被证实与配子识别后的膜融合密切相关[15]。通过比较现存的海胆纲动物bindin蛋白 “B18区”氨基酸组成发现,不同种类海胆纲生物 “B18区”氨基酸组成完全一致。与保守的bindin蛋白 “B18区”不同,位于bindin核心区两侧的重复区,在进化上呈现比较快速的替换趋势。目前,已有研究证实,驱动棘皮动物bindin核酸序列多态性的形成以及bindin蛋白进化的动力是达尔文的正向选择[20-21]。基于海胆bindin蛋白编码区 (不包含内含子)的研究显示,驱动不同种类海胆bindin蛋白快速进化的机制可涉及4个主要方面:增强效应(reinforcement),即增强前合子隔离的选择效应,用以防止异种受精和避免劣质杂交种产生;细胞层次的性别选择 (sexual selection),即繁殖性状(reproductive traits)的进化,包括 “隐秘的雌性选择 (cryptic female choice)”;两性冲突 (sexual conflict),即精子竞争 (sperm competition)的平衡和防止多精入卵 (egg polyspermy)的发生;免疫防御 (immunological defense),即拮抗协同进化(antagonistic coevolution with pathogens)[21]。最新的研究显示,除了遗传因素外,海胆bindin基因的进化还可能受到地理环境和空间密度高低对特定配偶的影响。值得注意的是,Hart等[22]的研究显示,与其他种类的海胆相比,海胆Heliocidaris的bindin基因在进化上呈现一种相对较低的速率,这一结果与鱼类的性别分化机制有一定相似之处[23]。但是,关于棘皮动物不同种类之间以及种间bindin分子特异性进化的具体机制至今仍不是十分明确。
Patino等[7]运用来自13个物种的DNA和RNA序列数据研究海星的性选择是否会影响bindin的进化。这一研究不仅证实了bindin在海星中也有分布,而且表明了bindin蛋白是海星性选择中的关键蛋白。交配系统的差异会导致bindin在分子进化过程中部分变异。bindin在不同物种之间具有多样性,其多样性程度几乎令人难以置信,但是仍存在一个明显的规律:在雌雄同体中的多样性少于雌雄异体的物种。这一差异表明,bindin的进化是由性选择,特别是繁殖成功的精子竞争或者其他差异所驱使的。对于bindin多样性的性选择来源,可能包括不同bindin基因型雄性之间的精子竞争,精卵接触最佳比率下 (雄性高雌性低)雌雄之间的性冲突以及致死性多精入卵的配子消耗 (雄性低雌性高)。实验室中的受精研究以及在雌雄中分别表达基因的比较,揭示了雄性基因型、雌雄基因型以及受精率之间功能上和遗传上的联系。这些结果表明,性选择有利于特定的等位基因或基因型组合,并且能够导致bindin与其卵子受体的共同进化[24-28]。有研究分析了13种不同海星的精子顶体bindin基因的编码序列和重复域的变化,结果表明,单个氨基酸差异与重复的差异可能影响精卵结合以及生殖兼容性[12]。
表2 系统进化树中bindin基因序列信息
Tab.2 Bindin nucleotide sequences used for phylogenetic analysis

海胆sea urchin Lytechinus williamsi AY183328.1 Tripneustes gratilla KU355783.1 Paracentrotus gaimardi FJ713315.1 Paracentrotus lividus FJ713312.1 Lytechinus variegatus M59489.1 Echinometra lucunter AY452683.1 Strongylocentrotus franciscanus M59490.1 Strongylocentrotus purpuratus M14487.1 A.punctulata X54155.1 Moira clotho AY126484.1 Diadema antillarum AY126485.1海星sea star Pisaster giganteus KJ481934.1 Pisaster ochraceus KJ481933.1 Pisaster brevispinus KJ481935.1 Dermasterias imbricata KT318447.1 Evasterias troschelii KT318446.1 Cryptasterina hystera KT318450.1 Cryptasterina pentagona KT318451.1 Parvulastra parvivipara KT318452.1 Parvulastra vivipara KT318453.1 Patiria pectinifera KT318449.1 Patiria miniata FJ439659.1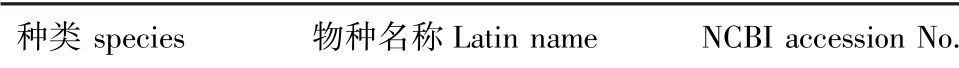
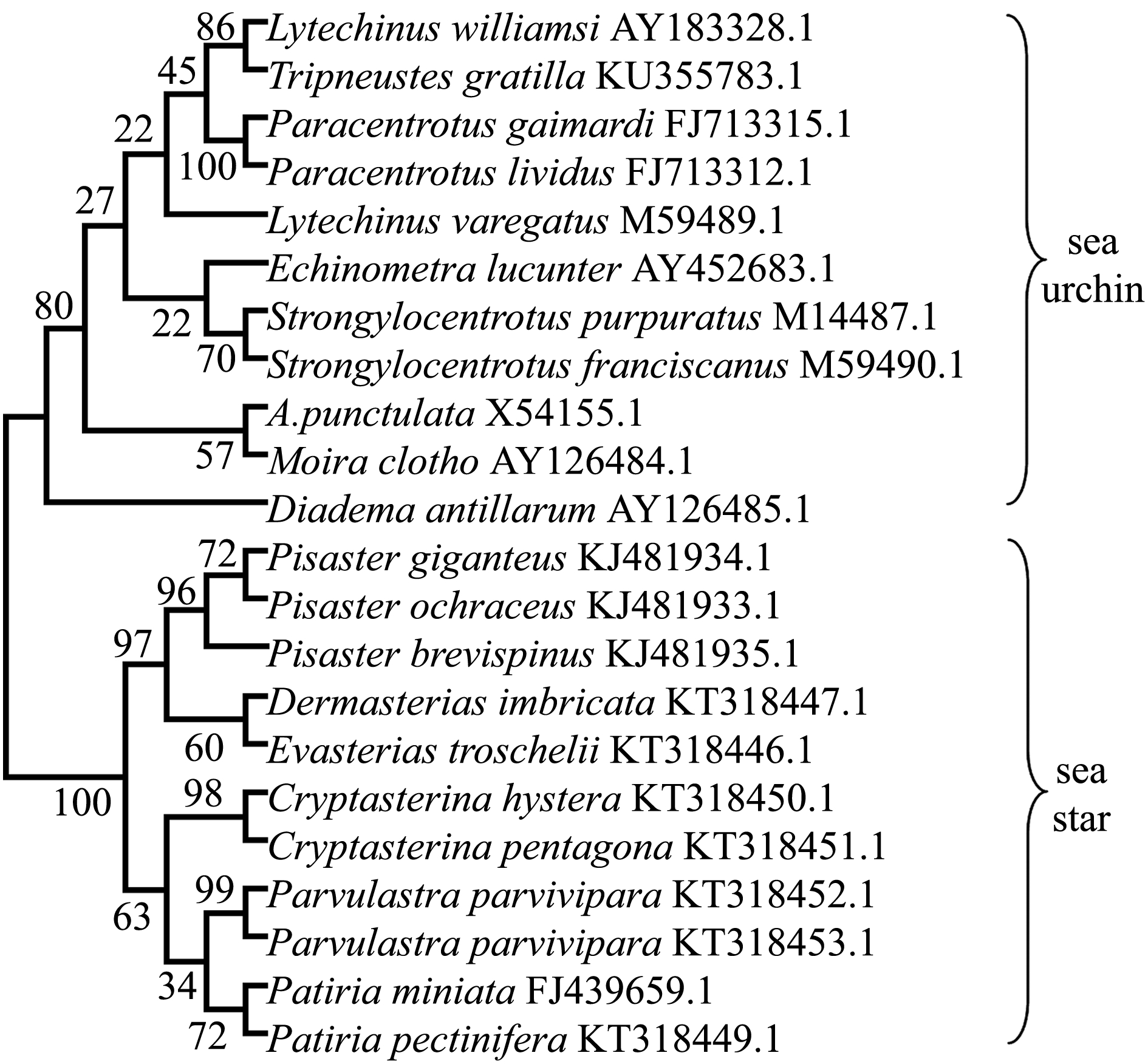
图4 部分棘皮动物bindin基因核苷酸序列的聚类分析
Fig.4 Phylogenetic analysis of bindin nucleotide sequences of some echinoderm species
2014年,Popovic等[29]比较分析了海星纲Pisaster属3种不同海星 (Pisaster ochraceus、Pisaster brevispinus和Pisaster giganteus)的bindin基因种内和种间的分子进化规律及趋势,通过对3种海星的bindin基因中3个外显子、3个蛋白结构域 (包括一个富含半胱氨酸的50个氨基酸组成的bindin前体蛋白区域、一个中心重复区域以及一个由30个氨基酸组成的保守功能核心区)的比较分析发现,Pisaster属3种不同海星的bindin基因的结构具有高度相似性。多序列比对结果显示,Pisaster属3种海星的bindin基因与目前已经获得序列信息的其他种属海星的bindin基因在结构上具有一定的相似性,但是,在位于bindin前体蛋白区域和保守功能核心区之间的中心重复区域Pisaster属的3种海星却存在较大差异。
上述结果与牡蛎种类 bindin基因的进化特征具有一定相似之处,Wu等[18]在研究牡蛎 Crassostrea angulata bindin基因 (Cabindin)的进化特征时发现,位于Cabindin蛋白F-lectin重复区发现了11个正向选择位点,这些正向选择位点都位于F-lectin的表面,并且围绕在组氨酸37(H37)、精氨酸64(R64)和精氨酸70(R70)3个重要的识别氨基酸周围。
4 展望
棘皮动物不仅是胚胎发育生物学和繁殖生物学的重要模式种类,其中海胆纲、海参纲的部分品种还具有较高的经济价值,是重要的海洋渔业资源和养殖对象。近年来,随着海洋环境的不断恶化,海洋棘皮类生物资源量逐年减少,经济棘皮动物的种质衰退现象日益加剧,如何有效保护棘皮动物的种群多样性以及提高经济棘皮动物的种质质量、改良养殖品种,已经成为海洋生物学和水产养殖学研究的热点。bindin基因作为与棘皮动物种群生殖分离(配子识别)以及性别选择高度相关的重要基因,不仅可以作为种群特异性标志物来深入研究棘皮动物生物多样性的产生和变化规律,还可以有助于认识和了解棘皮动物种间杂交屏障产生的分子机制。因此,通过不断丰富棘皮动物bindin基因的结构信息,深入研究棘皮动物bindin蛋白的进化规律及机制,掌握bindin蛋白及其相应卵膜受体的协同进化规律,可制定有效的棘皮动物多样性保护措施,还可打破生殖隔离的分子屏障,利用种间杂交优势繁育出性状更优的经济棘皮动物新品种。
参考文献:
[1] 黄晓红.长牡蛎(Crassostrea gigas)配子及受精前后的蛋白质组学研究[D].青岛:中国科学院研究生院(海洋研究所),2014.
[2] 刘慧慧,李太武,苏秀榕.鲍配子识别蛋白的进化[J].浙江海洋学院学报:自然科学版,2004,23(2):140-143.
[3] 吴琪.熊本牡蛎(Crassostrea sikamea)、葡萄牙牡蛎(Crassostrea angulata)精子bindin蛋白基因研究[D].青岛:中国科学院研究生院(海洋研究所),2010.
[4] Lillie F R.Studies of fertilization.VI.The mechanism of fertilization in arbacia[J].Journal of Experimental Zoology,1914,16(4):523-590.
[5] Vacquier V D,Moy G W.Isolation of bindin:the protein responsible for adhesion of sperm to sea urchin eggs[J].Proceedings of the National Academy of Sciences of the United States of America,1977,74(6):2456-2460.
[6] Gao Boning,Klein L E,Britten R J,et al.Sequence of mRNA coding for bindin,a species-specific sea urchin sperm protein required for fertilization[J].Proceedings of the National Academy of Sciences of the United States of America,1986,83(22):8634-8638.
[7] Patiño S,Keever C C,Sunday J M,et al.Sperm bindin divergence under sexual selection and concerted evolution in sea stars[J].Molecular Biology and Evolution,2016,33(8):1988-2001.
[8] Vacquier V D,Swanson W J,Hellberg M E.What have we learned about sea urchin sperm bindin?[J].Development,Growth& Differentiation,1995,37(1):1-10.
[9] Zigler K S.The evolution of sea urchin sperm bindin[J].The International Journal of Developmental Biology,2008,52(5-6):791-796.
[10] Glabe C G,Vacquier V D.Species specific agglutination of eggs by bindin isolated from sea urchin sperm[J].Nature,1977,267(5614):836-838.
[11] Glabe C G,Lennarz W J.Isolation of a high molecular weight glycoconjugate derived from the surface of S.purpuratus eggs that is implicated in sperm adhesion[J].Journal of Cellular Biochemistry,1981,15(4):387-394.
[12] Glabe C G,Lennarz W J.Species-specific sperm adhesion in sea urchins.A quantitative investigation of bindin-mediated egg agglutination[J].The Journal of Cell Biology,1979,83(3):595-604.
[13] Ulrich A S,Otter M,Glabe C G,et al.Membrane fusion is induced by a distinct peptide sequence of the sea urchin fertilization protein bindin[J].J Biol Chem,1998,273(27):16748-16755.
[14] Ulrich A S,Tichelaar W,Förster G,et al.Ultrastructural characterization of peptide-induced membrane fusion and peptide self-assembly in the lipid bilayer[J].Biophysical Journal, 1999, 77(2):829-841.
[15] Afonin S,Dürr U H N,Glaser R W,et al.‘Boomerang’-like insertion of a fusogenic peptide in a lipid membrane revealed bysolid-state19F NMR[J].Magnetic Resonance in Chemistry,2004,42(2):195-203.
[16] Metz E C,Palumbi S R.Positive selection and sequence rearrangements generate extensive polymorphism in the gamete recognition protein bindin[J].Molecular Biology and Evolution,1996,13(2):397-406.
[17] 叶子弘.生物信息学[M].杭州:浙江大学出版社,2011:136.
[18] Wu Q,Li L,Zhang G F.Crassostrea angulata bindin gene and the divergence of fucose-binding lectin repeats among three species of Crassostrea[J].Marine Biotechnology,2011,13(2):327-335.
[19] Zigler K S,Lessios H A.250 million years of bindin evolution[J].The Biological Bulletin,2003,205(1):8-15.
[20] Vacquier V D,Swanson W J.Selection in the rapid evolution of gamete recognition proteins in marine invertebrates[J].Cold Spring Harb Perspect Biol,2011,3(11):a002931.
[21] Balakirev E S,Anisimova M,Pavlyuchkov V A,et al.DNA polymorphism and selection at the bindin locus in three Strongylocentrotus sp.(Echinoidea)[J].BMC Genetics,2016,17:66.
[22] Hart M W,Popovic I,Emlet R B.Low rates of bindin codon evolution in lecithotrophic Heliocidaris sea urchins[J].Evolution,2012,66(6):1709-1721.
[23] 闫浩,梁利群,常玉梅,等.遗传和温度因素对鱼类性别分化相关基因表达及性别比例的影响[J].大连海洋大学学报,2017,32(1):111-118.
[24] Palumbi S R.All males are not created equal:fertility differences depend on gamete recognition polymorphisms in sea urchins[J].Proceedings of the National Academy of Sciences of the United States of America,1999,96(22):12632-12637.
[25] Levitan D R,Ferrell D L.Selection on gamete recognition proteins depends on sex, density, and genotype frequency[J].Science,2006,312(5771):267-269.
[26] Palumbi S R.Speciation and the evolution of gamete recognition genes:pattern and process[J].Heredity,2009,102(1):66-76.
[27] Levitan D R.Contemporary evolution of sea urchin gamete-recognition proteins:experimental evidence of density-dependent gamete performance predicts shifts in allele frequencies over time[J].Evolution,2012,66(6):1722-1736.
[28] Stapper A P,Beerli P,Levitan D R.Assortative mating drives linkage disequilibrium between sperm and egg recognition protein loci in the sea urchin Strongylocentrotus purpuratus[J].Molecular Biology and Evolution,2015,32(4):859-870.
[29] Popovic I,Marko P B,Wares J P,et al.Selection and demographic history shape the molecular evolution of the gamete compatibility protein bindin in Pisaster sea stars[J].Ecology and Evolution,2014,4(9):1567-1588.