斑马鱼Danio rerio个体较小,性成熟期短,繁殖力强,胚胎透明且体外发育,易于活体观察,便于研究其遗传发育、生理病理和疾病免疫功能[1]。目前,斑马鱼已成为研究人类、动物和鱼类疾病的一种水生模式动物[1-2]。
鱼类肠道微生物是影响其生长发育等生命活动的重要因素之一。研究表明,鱼类肠道正常菌群在营养物质消化吸收、免疫反应和器官发育等方面具有重要作用,且影响鱼类的生长、发育、生理和病理[3-4]。目前,国内外对肠道微生物的研究主要集中在畜禽动物,而对鱼类肠道菌群组成的研究报道尚少[3-5]。对于鱼类肠道微生物群落结构的分析鉴定需要建立快速、准确的研究技术,其中单链构象多态性分析 (SSCP)是一种基于DNA构象差别来检测点突变的方法[6]。而PCR-SSCP方法被广泛用于未知基因突变的检测,近年来在肠道微生物多样性的分析上也有应用[7-11]。为此,本试验中通过对斑马鱼肠道细菌进行分离纯化、16S rDNA基因克隆和V3区的PCR-SSCP等系统研究,揭示斑马鱼肠道细菌种类,旨在为探讨鱼类肠道菌群结构对其生命活动的影响提供科学参考。
1 材料与方法
1.1 材料
试验用斑马鱼购自江苏省徐州市某花鸟市场。
LB固体培养基、LB液体培养基、4%NaCl嗜盐菌培养基由天津市水产生态及养殖重点实验室配制。
TCBS琼脂、SS琼脂、伊红美蓝EMB选择培养基购自杭州微生物试剂公司。UNIQ-10柱式细菌基因组DNA抽提试剂盒购自上海生工生物工程公司,dNTP、Taq DNA聚合酶、10×PCR反应试剂、PCR产物胶回收试剂盒购自上海捷瑞生物工程公司,pMD 18-T载体购自大连宝生物工程公司。
1.2 方法
1.2.1 斑马鱼肠道细菌的分离与培养特性 参照胡秀彩等[5]的方法,在无菌条件下分别将菌落接种到 LB平板、4%NaCl营养琼脂、TCBS琼脂、SS琼脂、EMB选择培养基上,于29℃下倒置培养24 h。分别挑取菌落形态特征不同的单个菌落接种到LB平板上,无菌条件下挑取单菌落多次划线进行分离纯化。获得纯化细菌菌株于4℃下保存(试管斜面)。观察细菌在普通琼脂培养基与选择培养基上的生长情况,并记录菌体培养特性。
1.2.2 细菌基因组DNA提取及16S rDNA基因PCR扩增 根据斑马鱼肠道菌株在不同平板上的菌落形态、颜色、大小等,选取12株细菌进行16S rDNA序列测定,采用UNIQ-10柱式细菌基因组DNA抽提试剂盒提取基因组DNA,经琼脂糖电泳后,将12株细菌的总DNA于-20℃下保存备用。采用大肠杆菌通用引物对16S rDNA进行扩增,正向引物序列为 5′AGAGTTTGATCATGGCTCAG 3′, 反向引物序列为 5′TACGGTTACCTTGTTACGACTT 3′。 PCR反应体系 (共 15 μL): 模板1.0 μL, 正、 反向引物各 0.5 μL, Taq PCR Master Mix 7.5 μL, 加 ddH2O 5.5 μL。 反应条件: 94 ℃下预变性4 min;94℃下变性30 s,55℃下退火30 s,72℃下延伸1 min,共进行30个循环;最后在72℃下再延伸10 min,于4℃下保存备用。
将PCR产物连接pMD-18T载体,并转化至大肠埃希菌DH5α感受态细胞中,筛选阳性克隆,经pMD-18T载体 M13的正向引物序列 5′ACTCCTACGGGAGGCAGCAG 3′、 反向引物序列 5′ATTACCGCGGCTGCTGG 3′进行PCR扩增,验证阳性克隆由上海生工生物工程有限公司测序。采用10 g/L琼脂糖凝胶电泳,EB染色检测,用紫外线分析仪拍照观察。
1.2.3 16S rDNA序列及系统发育学分析 将测序获得的16S rDNA序列,利用NCBI进行BLAST序列比对 (http://www.ncbi.nlm.nih.gov/BLAST/),基于16S rDNA序列分析,所获得序列与数据库中的参考序列应用CLUSTALX 1.8软件进行匹配比对,然后采用MEGA 4.1软件构建系统发育树。
1.2.4 16S rDNA基因V3序列的PCR-SSCP分析
以细菌总DNA为模板,用正向引物序列5′CAGTCACGACGTTGTAA 3′、 反 向 引 物 序 列5′CAGGAAACAGCTATGAC 3′扩增16S rDNA 序列。PCR反应体系 (共15 μL):正、反向引物各0.5 μL, Buffer 1.5 μL, Mg2+0.6 μL, dNTP 0.2 μL,Taq 酶 0.1 μL, 模板 1.5 μL, 加 ddH2O 至 15 μL。反应条件:94℃下预变性4 min;94℃下变性30 s,51℃下退火30 s,72℃下延伸1 min,共进行30个循环;最后在72℃下再延伸10 min,于4℃下保存备用。PCR扩增产物采用10 g/L琼脂糖凝胶电泳检测。
SSCP分析依照文献[5-6]进行,简述如下:取6 μL的PCR产物和4 μL变性缓冲液混匀,98℃下变性10 min后立即冰浴10 min,于10%PAGE中电泳,4℃、180 V条件下电泳2.5 h(Bio-Rad);电泳后先用蒸馏水洗1~2次,再用0.1%AgNO3染色15 min,最后利用2.0%NaOH(0.2%甲醛)显色,直至条带清晰为止,拍照进行SSCP带型分析。
2 结果与分析
2.1 斑马鱼肠道细菌形态及培养特征
从斑马鱼肠道中分离纯化出12株细菌,分别命名为 Zf1、 Zf2、 Zf3、 Zf4、 Zf5、 Zf6、 Zf7、 Zf8、Zf9、Zf10、Zf11和 Zf12,在 LB培养基培养24 h后12株分离细菌均为中小型菌落,多为透明或乳白色菌落。其中,菌株 Zf5、Zf6、Zf7、Zf8、Zf9在4%NaCl平板上生长缓慢,菌落形态小而圆、多为乳白色,其余7个菌株生长不良;菌株Zf1、Zf2、 Zf3、 Zf4分别在 TCBS、 TCBS、 EMB、 SS选择培养基上生长较快,并呈现出不同的颜色;菌株Zf10、Zf11和Zf12在LB培养基上生长较快,呈现为乳白色的圆形菌落 (表1)。
2.2 肠道菌株l6S rDNA基因克隆及分子鉴定
斑马鱼肠道细菌基因组总DNA电泳检测显示,12株细菌基因组DNA均大于2000 bp,可用作PCR扩增16S rDNA的模板 (图1)。
以所提取的细菌总DNA为模板进行16S rDNA PCR扩增,PCR产物用琼脂糖凝胶电泳检测,表明扩增出Zf1~Zf12菌株的16S rDNA基因片段,大小约为1500 bp(图2),用PCR检测pMD18-T/16S rDNA阳性克隆并进行测序。对斑马鱼肠道细菌测序获得的7个菌株的16S rDNA序列,利用NCBI进行BLAST序列相似性检索,结果见表2。同时结合肠道细菌形态学和培养特征,确定菌株Zf1与Zf11属于气单胞菌属Aeromonas,与菌株Aeromonas veronii为同一种细菌;Zf4与Zf8属于鞘氨醇单胞菌属 Sphingomonas,与菌株 Sphingomonas sp.为同一种菌;Zf5属于芽孢杆菌属Bacillus,与菌株Bacillus subtilis为同一种菌;Zf7属于气单胞菌属Aeromonas,与菌株Aeromonas sp.M10为同一种菌;而Zf10与非培养菌株uncultured bacterium clone GI3-M-5-G01为同一种菌,可能是一种新的菌种。
表1 细菌的形态和培养特征
Tab.1 Morphological and culture characteristics of all strains

菌种编号strain No.培养特征characteristics Zf1 TCBS选择培养基:菌落为黄色,形态中等,呈圆形,湿润,隆起,不透明,边缘整齐Zf2 TCBS选择培养基:菌落为绿色,形态较小,呈圆形,湿润,隆起,半透明,边缘整齐Zf3 EMB选择培养基:菌落为粉红色,形态较小,呈圆形,湿润,隆起,不透明,边缘整齐Zf4 SS选择培养基:菌落微黄,形态中等,呈圆形,湿润,扁平,中间呈黑色,透明,边缘整齐Zf5 4%NaCl平板:菌落为浅黄色,形态中等,呈圆形,湿润,隆起,半透明,边缘整齐Zf6 4%NaCl平板:菌落形态较小,呈圆形,湿润,隆起,半透明,乳白色,混浊,边缘整齐Zf7 4%NaCl平板:菌落形态较小,呈圆形,干燥,扁平,不透明,乳白色,边缘不整齐Zf8 4%NaCl平板:菌落形态较小,呈圆形,湿润,隆起,半透明,乳白色,边缘整齐Zf9 4%NaCl平板:菌落形态中等,呈圆形,湿润,隆起,不透明,乳白色,边缘整齐Zf10 LB培养基:菌落形态较小,呈圆形,湿润,隆起,半透明,乳白色,边缘整齐Zf11 LB培养基:菌落形态中等,呈圆形,干燥,扁平,不透明,乳白色,边缘不整齐Zf12 LB培养基:菌落形态中等,呈圆形,湿润,扁平,半透明,乳白色,边缘整齐
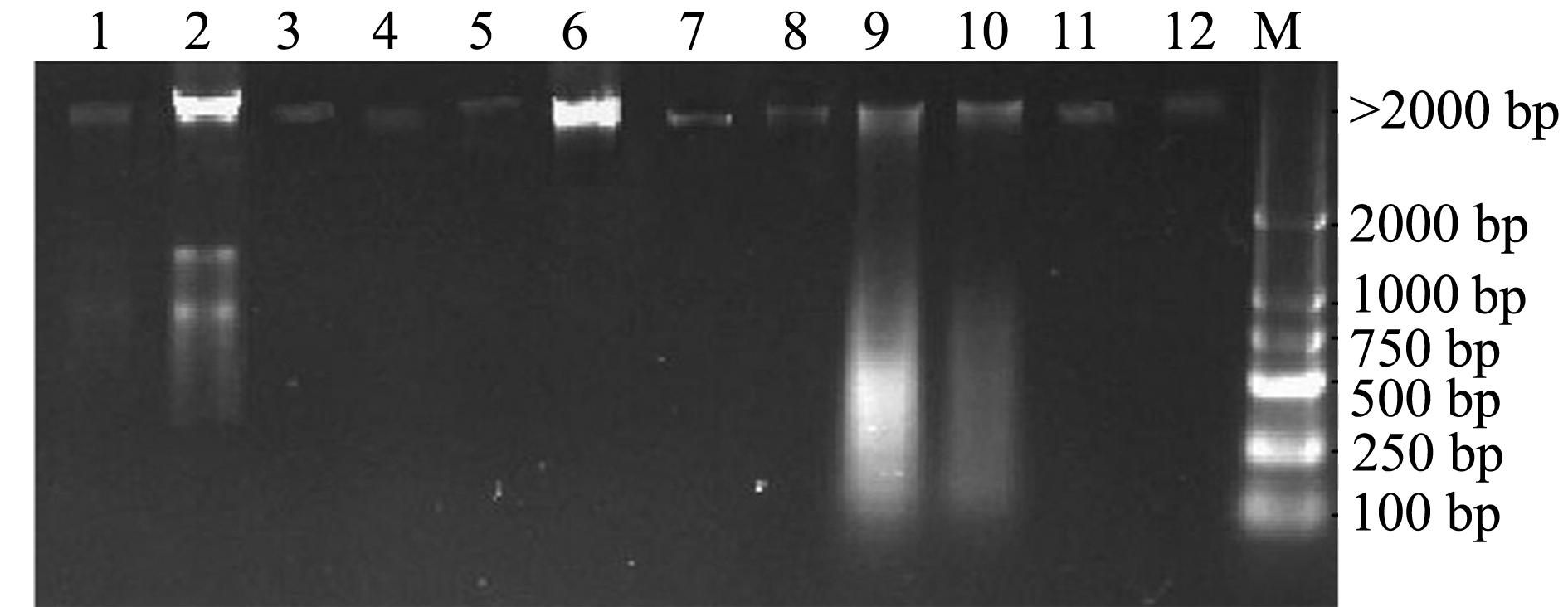
图1 菌株Zf1-Zf12基因组总DNA
Fig.1 Total DNA of genome from strains Zf1 to strain Zf12 in the intestine of zebrafish
注:M为 DL2000 DNA Marker;1~12分别为Zf1~Zf12菌株样品
Note: M, DL2000 DNA Marker;1-12,bacteria of Zf1-Zf12
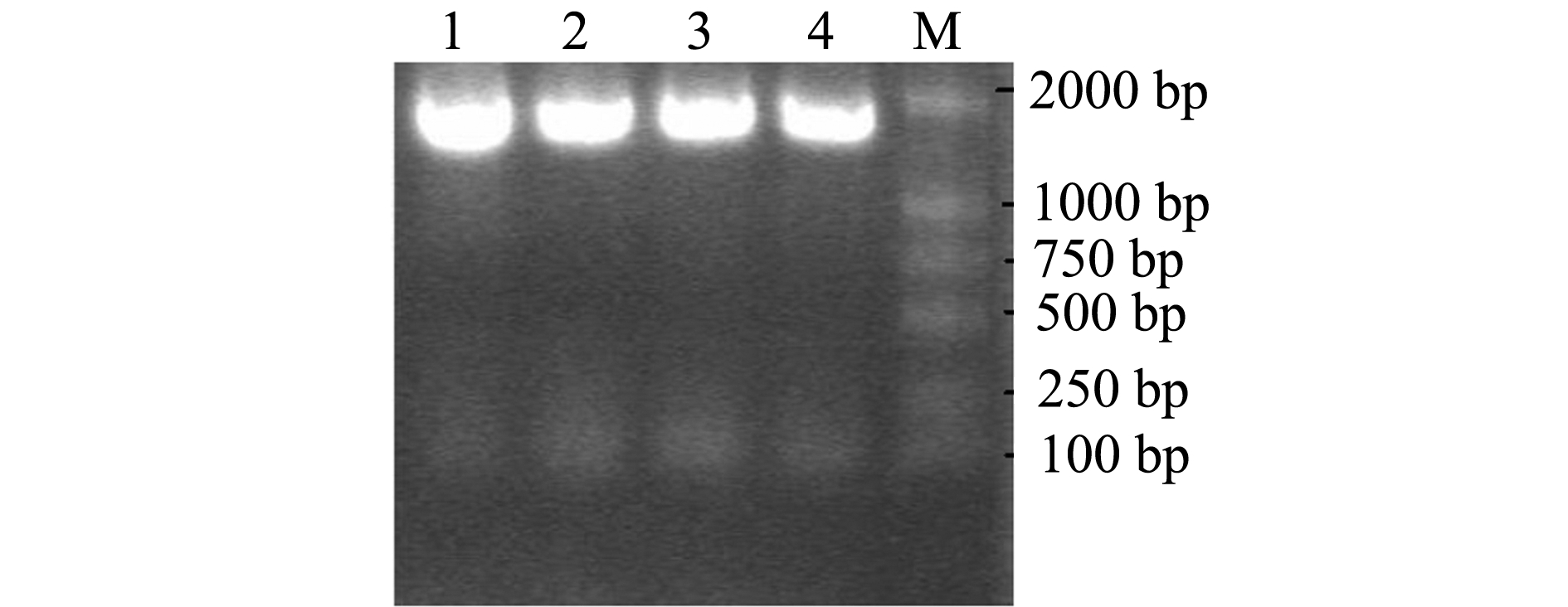
图2 菌落PCR检测pMD18-T/16S rDNA阳性克隆Fig.2 Positive clones of pMD18-T/16S rDNA in the strains by PCR
注:M为DL2000 DNA Marker;1~4,PCR阳性克隆产物
Note: M, DL2000 DNA Marker l-4, PCR products of positive clones
表2 16S rDNA序列测序结果分析
Tab.2 Sequencing of the 16S rDNA from different isolates

注:1)为BLAST搜索中最接近相对序列的登录号
Note: 1), accession number of the closest relative sequence found by BLAST search
菌种号strain No.登录号1)accession No.最似细菌closest relative bacterium一致性/%indentity Zf1 FJ808727.1 Aeromonas veronii strain QXL0711B 99 Zf4 AF385529.1 Sphingomonas sp.oral clone AV069 99 Zf5 FJ876834.1 Bacillus subtilis strain EBS05 99 Zf7 DQ200865.1 Aeromonas sp.M10 99 Zf8 AF385529.1 Sphingomonas sp.oral clone AV069 99 Zf10 FJ191530.1 uncultured bacterium clone GI3-M-5-G01 92 Zf11 FJ808727.1 Aeromonas veronii strain QXL0711B 99
通过构建16S rDNA基因序列系统发育树分析显示,以Zf1、Zf11为代表的菌株与已知种的细菌Aeromonas veronii聚为一支,亲缘关系最近,归在同一类群,且一致性达99%;而菌株Zf5则与Bacillus subtilis聚为一支,一致性达99%;而以Zf4、Zf8为代表的菌株则与Sphingomonas sp.未知细菌聚为一支,归在同一类群,一致性达99%;Zf7与Aeromonas sp.未知细菌聚为一支,一致性达99%;另外一个代表菌株Zf10则与尚未获得纯培养的菌株聚为一支,一致性达92%。以肠道大肠埃希氏菌Escherichia cdi作为参考菌株,与各菌株分支较远,可知分离的菌种与大肠埃希氏菌亲缘关系较远(图3)。
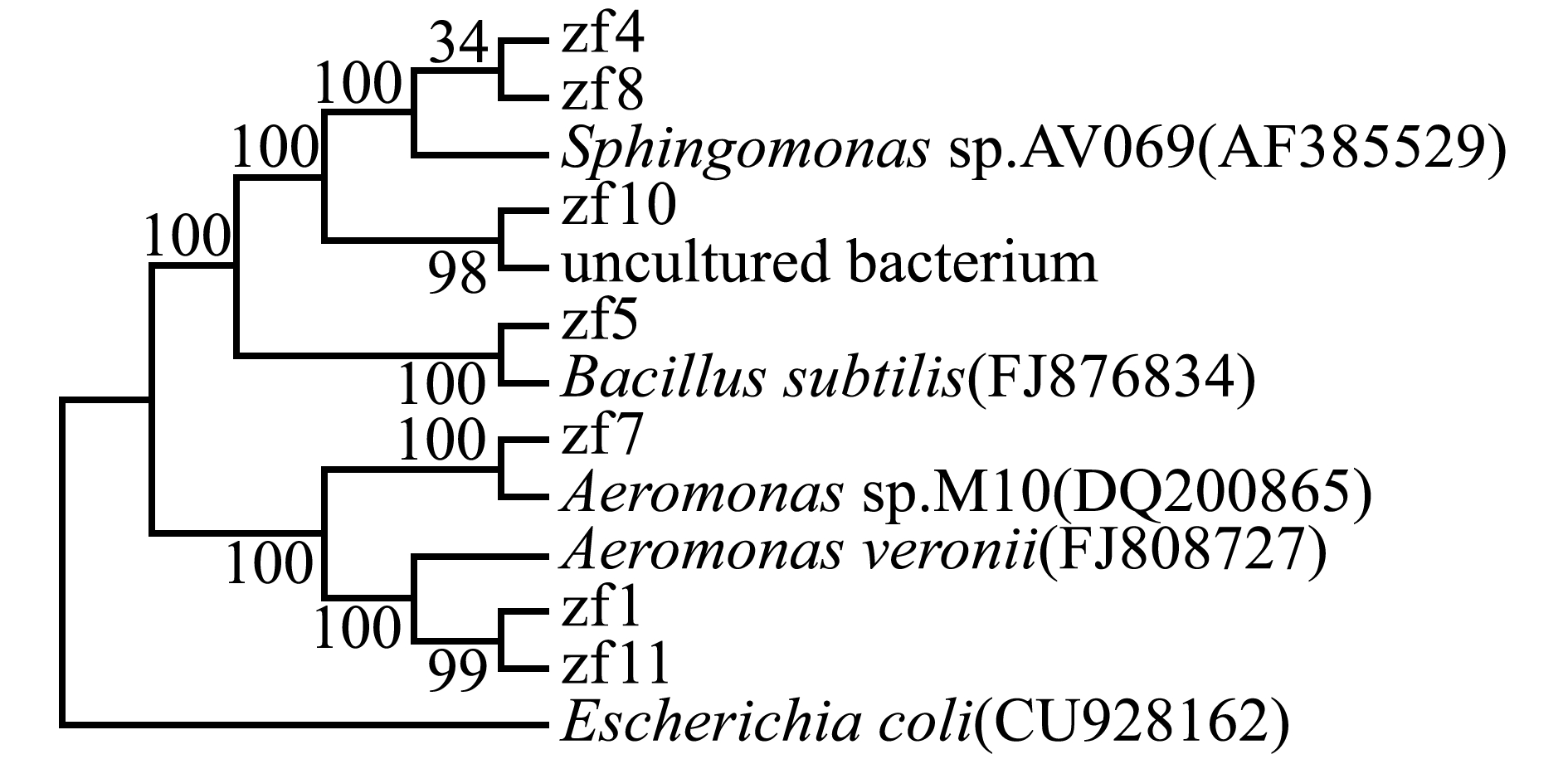
图3 以斑马鱼肠道细菌16S rDNA基因序列构建的系统发育树
Fig.3 Phylogenetic tree analysis of different bacterial isolates from the intestine of zebrafish based on 16S rDNA gene sequence
2.3 16S rDNA基因V3区的PCR-SSCP分析
对12株分离菌16S rDNA V3区基因的PCRSSCP图谱显示,斑马鱼肠道细菌Zf1~Zf12的16S rDNA V3区基因存在多态性:Zf1与Zf11带型一致;Zf4与Zf8带型一致;而Zf5、Zf7、Zf10带型各不相同,与前两种带型也有所不同 (图4)。进一步对其中7株肠道细菌16S rDNA V3区基因序列进行比对 (图5),结果显示,Zf1、Zf11序列完全相同,表明为同一种菌;Zf4、Zf8序列基本相同,为同一种菌。Zf5、Zf7、Zf10序列互不相同,且与Zf1、Zf11以及Zf4、Zf8均有较大差别,初步鉴定为不同菌种,此结果与16S rDNA-SSCP图谱一致。

图4 16S rDNA V3区基因的PCR-SSCP图谱
Fig.4 PCR-SSCP results of the V3 region gene of l6S rDNA of the bacteria from the intestine of zebrafish
注:1~12分别为Zf1~Zf12菌株样品
Note:Lanes 1-12 represent samples of bacterial strains from Zf1 to Zf12

图5 16S rDNA基因V3区基因序列比对
Fig.5 Homological analysis of 16S rDNA V3 region sequences
注:Con为大肠杆菌的6S rDNA V3区基因序列
Note: Con, sequences of 16S rDNA V3 region in Escherichia coli
3 讨论
3.1 鱼类肠道细菌的多样性研究
近年来,对动物肠道微生物的研究主要集中于哺乳类动物[12-14],而对模式生物斑马鱼肠道菌群进行分离鉴定的研究则鲜有文献报道[1,3]。国内对鱼类肠道菌群组成的研究起步相对较晚。迄今,相关学者对大弹涂鱼[15]、青石斑鱼[16]肠道菌群进行鉴定分析,并发现了肠道优势菌群与细菌种属,认为这些优势菌群可能是参与消化生理活动的主要菌群。樊海平等[17]研究发现,日本鳗鲡肠道中细菌数量与栖息水体、生理状态、饵料状况等有关。赵庆新[18]以草鱼、鲤等鲤科Cyprinidate鱼类为研究对象,分析了其肠道细菌的菌群结构,结果表明,鱼类肠道中主要有致病杆菌属Xenorhabdus、气单胞菌属Aeromonas、柠檬酸杆菌属Citrobacter、假单胞杆菌属Pseudomonas等。本研究中,采用PCRSSCP的分子技术鉴定方法研究了斑马鱼肠道细菌多样性,16S rDNA系统发育树分析显示,以Zf1、Zf11为代表的菌株与已知种细菌Aeromonas veronii,Zf5与 Bacillus subtilis, Zf4、 Zf8与 Sphingomonas sp.,Zf7与Aeromonas sp.分别聚为一支,且16S rDNA序列一致性高达99%;同时从斑马鱼肠道中分离获得Zf10菌株与uncultured bacterium clone一致性为92%,表明有可能为一种新分离菌株。斑马鱼肠道细菌16S rDNA分子鉴定表明,Zf1、Zf7和Zf11属于气单胞菌属Aeromonas,Zf4、Zf8属于鞘氨醇单胞菌属Sphingomonas,Zf5属于芽孢杆菌属Bacillus。此外,斑马鱼肠道细菌Zf2、Zf3、Zf6、Zf9和Zf12等分离株构建的阳性克隆,测序尚未成功,这可能与鱼类肠道细菌多样性、菌株生物学特性和试验技术条件因素有关。已有研究报道,利用宏基因组技术可进行微生物多样性结构和功能基因组研究[7]。本试验中通过对斑马鱼肠道细菌16S rDNA的基因克隆测序分析,发现Zf10与尚未获得纯培养的菌株uncultured bacterium GI3-M-5-G01聚为一支,一致性为92%,推测菌株Zf10可能是一种新分离菌种,有待进一步研究鉴定,以丰富斑马鱼肠道微生物物种资源。
3.2 鱼类肠道细菌PCR-SSCP分析
有研究表明,16S rDNA序列分析在细菌鉴定方面具有不可替代的作用,结合SSCP技术可以提高检测的特异性[9-10]。细菌16S rDNA既有保守区,也有相对变异区,因此,可以采用PCR-SSCP技术进行鉴定分析[8]。王永等[9]对于4种食源性致病菌16S rDNA的可变区进行SSCP分析,结果表明,4种致病菌16S rDNA单链构象多态性具有种属特异性。本试验结果表明,斑马鱼肠道分离菌种的SSCP图谱无论从条带的数目、相对迁移率及条带之间的间距看,相互间均存在明显差异,其中Zf1与Zf11带型一致,Zf4与 Zf8带型一致,而Zf5、Zf7、Zf10带型各不相同,与前两种带型也有所不同。通过构建16S rDNA系统进化树分析发现,其SSCP图谱差异似与亲缘关系成正比,即亲缘关系越远,差异越明显。此外,16S rDNA基因V3区片段大小约为200 bp,利用SSCP分析V3区基因片段分辨率较高,得到的图谱也较清晰;尽管如此,利用SSCP技术进行分析时也有其缺陷,比如,当分离片段较大 (>400 bp)时,会出现分辨率低、共迁移等现象[9,19]。此外,有学者利用PCR-DGGE、PCR-RFLP技术进行肠道微生物、病原菌分析鉴定研究[19-20]。本研究中,斑马鱼肠道细菌16S rDNA基因V3区的PCR-SSCP结果与16S rDNA基因序列比对结果一致,值得进一步研究。
综上所述,本研究中从斑马鱼肠道分离纯化出的12株细菌,其中气单胞菌属Aeromonas等优势菌株与其他学者研究报道结果一致[15,18],这为今后进行斑马鱼肠道菌群及其微生态功能研究提供了科学依据,也为PCR-SSCP技术应用于斑马鱼肠道细菌多样性分析提供了参考。
参考文献:
[1] 全珊珊,吴新荣.斑马鱼,人类疾病研究的理想模式动物[J].生命的化学,2008,28(3):260-263.
[2] 王智诚,周晓旭,王昊泽,等.长链非编码RNA及其在斑马鱼中的研究进展[J].大连海洋大学学报,2017,32(2):248-254.
[3] 宋增福,吴天星.鱼类肠道正常菌群研究进展[J].水产科学,2007,26(8):471-474.
[4] Liu Yukun,Yao Yayun,Li Huan,et al.Influence of endogenous and exogenous estrogenic endocrine on intestinal microbiota in zebrafish[J].PLoS One,2016,11(10):e0163895.
[5] 胡秀彩,王艺,吕爱军.弗氏柠檬酸杆菌的分离鉴定与PCR-SSCP 分析[J].微生物学杂志,2011,31(4):12-18.
[6] Orita M,Iwahana H,Kanazawa H,et al.Detection of polymorphisms of human DNA by gel electrophoresis as single-strand conformation polymorphisms[J].Proc Natl Acad Sci USA,1989,86(8):2766-2770.
[7] 李丽娟,张殿昌,龚世园.宏基因组技术在开发未培养微生物资源中的应用[J].水利渔业,2007,27(3):7-9,31.
[8] 刘文强,贾玉萍,赵宏坤.16S rRNA在细菌分类鉴定研究中的应用[J].动物医学进展,2006,27(11):15-18.
[9] 王永,赵新,兰青阔,等.4种食源性致病菌的PCR-SSCP检测技术研究[J].天津农业科学,2009,15(1):13-15.
[10] Callon C, Delbès C, Duthoit F,et al.Application of SSCP-PCR fingerprinting to profile the yeast community in raw milk Salers cheeses[J].Systematic and Applied Microbiology,2006,29(2):172-180.
[11] 刘莉,王中康,俞和韦,等.贡嘎蝠蛾幼虫肠道细菌多样性分析[J].微生物学报,2008,48(5):616-622.
[12] 邱索平,凌华枢,陈文.食蟹猴肠道志贺氏菌的分离和鉴定[J].安徽农业科学,2007,35(33):10730-10736.
[13] 邱立,朱占烨,杨增岐,等.关中奶山羊肠道正常菌群的分离鉴定[J].黑龙江畜牧兽医,2007(10):77-78.
[14] 张振瑞,张力,陈桂银,等.健康家兔肠道益生菌的分离鉴定[J].中国养兔杂志,2007(2):21-24.
[15] 王伟霞,李福后,李信书.大弹涂鱼肠道细菌学分析[J].安徽农业科学,2007,35(12):3545-3549.
[16] 覃志坚,卢冬,常正义,等.PCR-SSCP法快速检测鉴定临床病原菌的探讨[J].广西医科大学学报,2003,20(3):341-343.
[17] 樊海平,曾占壮,林煜,等.养殖的日本鳗鲡肠道中细菌的数量和组成[J].台湾海峡,2005,24(4):515-519.
[18] 赵庆新.鲤科(Cyprinidate)鱼肠道菌群分析[J].微生物学杂志,2001,21(2):18-20.
[19] 罗雯,万雅各,彭宣宪,等.采用通用引物PCR配合SSCP及RFLP技术快速检测常见病原菌[J].中华微生物学和免疫学杂志,2001,21(6):687-689.
[20] 李可,郑天凌,田蕴,等.南美白对虾肠道微生物群落的分子分析[J].微生物学报,2007,47(4):649-653.