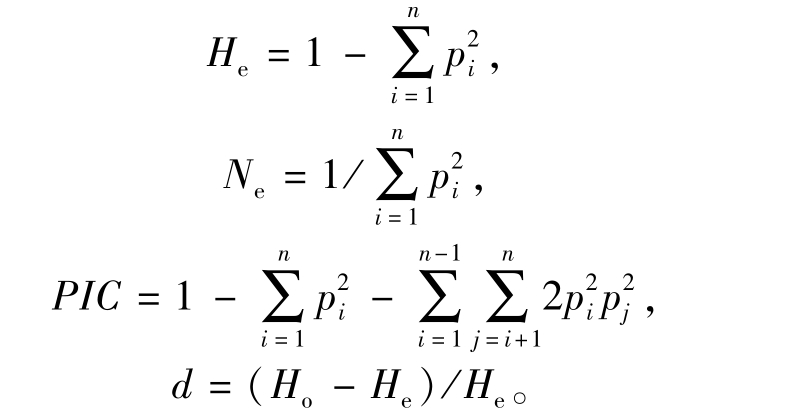
摘要:为获得可用于小体鲟Acipenser ruthenus种群遗传多样性分析的微卫星分子标记,采用跨种PCR扩增的方法从已发表的近缘种微卫星DNA标记引物中筛选得到13个具有多态性的位点,同时为了探讨更适合多倍体样品的微卫星数据读取方法,分别采用比例法和补齐法对微卫星数据进行分析。结果表明:两种数据读取方法的分析结果无显著性差异(P>0.05);本研究中选用补齐法对小体鲟种群遗传多样性进行分析,结果显示,小体鲟种群等位基因数(Na)从5个到30个不等,表观杂合度(HO)范围为0.089 6~0.940 3,平均为0.648 0,期望杂合度(He)范围为0.085 2~0.912 6,平均为0.719 3,种群Hardy-Weinberg平衡遗传偏离指数(d)平均为-0.098 0,种群多态信息含量(PIC)平均为0.702 8;筛选得到的13个微卫星位点是适用于小体鲟种群遗传研究的具有多态性的良好分子标记。研究表明,被检测的小体鲟养殖种群存在一定程度的近交现象,建议通过引进不同来源亲本加以改善,以增加种群遗传多样性的丰富度。
关键词:小体鲟;微卫星;跨种扩增;遗传多样性
鲟形目Acipenseriformes隶属于硬骨鱼纲、辐鳍亚纲、硬鳞总目,共包括27种[1]。所有鲟鱼都是四倍体以上的多倍体鱼类,种属间易于杂交。鲟鱼肉质鲜美、营养全面,且富含多种人体必需氨基酸和不饱和脂肪酸[2-3]。然而,由于过度开采、环境污染和生态破坏,影响了鲟鱼的野外栖息环境,导致其野外资源急剧下降,已处于濒危状态[4]。目前,人工养殖鲟鱼已经成为鲟鱼相关制品的重要来源。对养殖鲟鱼种群进行遗传多样性评估是避免其近亲繁殖、保障苗种质量的重要手段,对鲟鱼产业发展具有重要意义。
微卫星DNA标记(microsatellite),也称为简单重复序列(simple sequence repeats,SSRs)或短串联重复序列(simple tandem repeats,STRs),是均匀分布于真核生物基因组中的简单重复序列,由2~6个核苷酸的串联重复片段构成,符合孟德尔遗传模式,具有共显性表达、多态性丰富等特点。由于重复单位的重复次数在个体间呈高度变异性且数量丰富,因此,微卫星标记的应用非常广泛[5]。目前,微卫星DNA标记被广泛应用于鲟鱼种质鉴定、亲子关系鉴定和遗传多样性分析等研究中[6-7]。
小体鲟Acipenser ruthenus是鲟形目鱼类中倍性最少的种类之一,为四倍体鱼类[8],具有个体较小、性成熟时间短等特点,在养殖中常作为鲟鱼杂交育种的亲本。以小体鲟为父本、欧洲鳇Huso huso为母本的杂交后代Bester,属于广受欢迎的具备双亲优点的杂交鲟鱼[9]。因此,对作为亲本的人工养殖小体鲟种群进行遗传多样性分析,对保障苗种质量尤为重要。本研究中,利用在近缘种中已发表的微卫星引物,在小体鲟人工养殖群体中进行跨种PCR扩增,并利用获得的具有多态性的微卫星位点对该小体鲟人工养殖群体进行种群遗传多样性分析,旨在为进一步进行种群遗传学研究和苗种繁育奠定基础。
1.1 材料
试验用67尾小体鲟采自北京市水产科学研究所房山十渡国家级鲟鱼良种场,为同一批次人工繁殖的F2代群体。
1.2 方法
1.2.1 样品的采集及基因组DNA的提取 剪取鲟鱼尾鳍鳍条放入2 mL无菌离心管中,加入体积分数为95%的乙醇带回实验室,于冰箱(4℃)中保存待用。利用TaKaRa Mini BEST Universal Genomic DNA Extraction Kit 5.0试剂盒从鳍条中提取基因组DNA,用Nanodrop2000紫外分光光度计测定DNA含量。
1.2.2 跨种扩增 从近缘种湖鲟A.fulvescens[10]、高首鲟A.transmontanus[11]和短吻鲟A.brevirostrum(GenBank序列号:EU379637~EU379641)已被发表的微卫星序列中选取25个微卫星标记位点进行跨种扩增,从中筛选出适用于小体鲟的微卫星位点。
PCR反应体系(共10μL):ddH2O 7μL,10×Buffer(Mg2+)1.0μL,Primers(5μmol/L)0.3μL×2,dNTPs(2.5 mmol/L)0.8μL,Ex Taq DNA polymerase(5 U/μL)0.1μL,DNA(30 ng/μL)0.5μL。PCR反应程序:95℃下预变性5 min;95℃下变性30 s,50~65℃下退火30 s,72℃下延伸30 s,共35个循环;72℃下再延伸10 min,4℃下保存。反应产物经15 g/L琼脂糖凝胶电泳,用紫外凝胶成像系统观察并记录。
1.2.3 微卫星位点的确认及多态性分析 选择扩增条带清晰、与原物种中产物大小接近(150~450 bp)的PCR产物进行进一步分析,以确认产物中是否存在微卫星重复序列。使用胶回收试剂盒(Axygen)从琼脂糖凝胶中分别回收纯化扩增产物,然后连接pMD19-T载体(大连宝生物工程有限公司),再转入大肠杆菌Escherichia coli DH10B感受态细胞中。挑出含有转化质粒的菌液送北京擎科新业生物技术有限公司进行DNA序列测定。
对确认扩增产物中含有微卫星重复序列的正向PCR引物进行荧光标记,并与原反向引物形成引物对,再次对基因组DNA进行PCR扩增,反应条件同上(表1)。将3种引物标有不同荧光染料(FAM、HEX和TMARA),并且扩增片段大小相差50~100 bp的PCR产物分为一组,使用ABI PRISM 3100型遗传分析仪(Applied Biosystems)对扩增片断进行测定。
1.3 数据处理
利用GeneMapper 3.7软件对基因型数据进行处理。利用SPAGeDi1.4软件计算种群的等位基因数(observed number of alleles,Na)、期望杂合度(expected heterozygosity,He)和表观杂合度(observed heterozygosity,Ho)。通过计算有效等位基因数(effective number of alleles,Ne)、多态信息含量(polymorphism information content,PIC)、哈迪-温伯格遗传偏离指数(Hardy-Weinberg departure value,d)等数据,对小体鲟种群遗传多样性进行分析。计算公式分别为
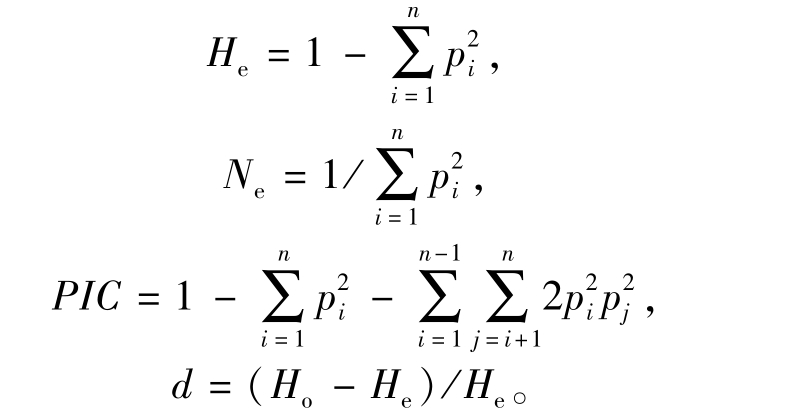
其中:pi、pj分别为群体中某基因座第i、j个等位基因的频率;n为等位基因数。
2.1 微卫星标记的筛选
在25对来源于已发表的其他鲟鱼微卫星标记中,有13对在小体鲟中可以扩增出清晰且符合预期大小的目的条带。测序结果表明,这13对引物的扩增产物均含有微卫星DNA序列(表1)。因此,可利用这13对引物对采自北京市水产科学研究所房山十渡国家级鲟鱼良种场的67尾小体鲟进行扩增和遗传多样性分析。
2.2 微卫星数据读取方法的比较
利用GeneMapper 3.7软件,依据等位基因大小读取小体鲟种群各微卫星位点的基因型数据,其中一个样品在位点Afu19的结果见图1,以便演示用比例法和补齐法的读图结果。
从图1可以看出,该样品在该位点具有两个等位基因,分别为121 bp和137 bp。由于小体鲟为四倍体,因此,使用补齐法进行数据处理的读取结果为121 137 000 000。由于等位基因121 bp和137bp荧光信号的比例约为1∶3,考虑到小体鲟为四倍体,所以使用比例法进行数据处理的读取结果为121 137 137 137。利用SPAGeDi1.4软件对两种数据读取方法得到的数据进行分析,并通过Excel软件对数据进行统计计算,结果见表2。
表1 13对微卫星引物信息及退火温度筛选结果
Tab.1 Characterizations and annealine tem perature choice of the 13 m icrosatellite primers
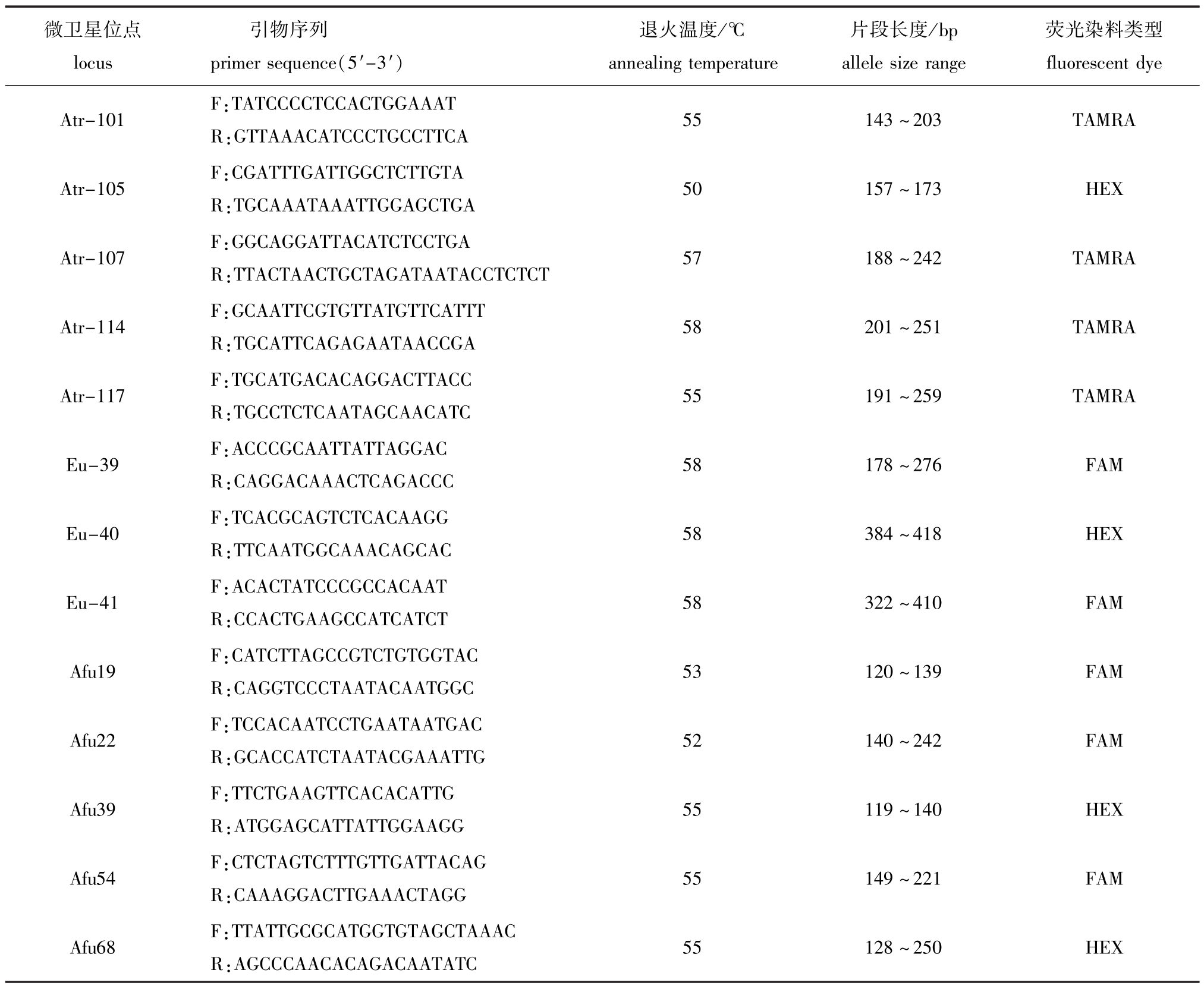
微卫星位点locus荧光染料类型fluorescent dye Atr-101 F:TATCCCCTCCACTGGAAAT R:GTTAAACATCCCTGCCTTCA引物序列primer sequence(5′-3′)退火温度/℃annealing temperature片段长度/bp allele size range 55 143~203 TAMRA Atr-105 F:CGATTTGATTGGCTCTTGTA R:TGCAAATAAATTGGAGCTGA 50 157~173 HEX Atr-107 F:GGCAGGATTACATCTCCTGA R:TTACTAACTGCTAGATAATACCTCTCT 57 188~242 TAMRA Atr-114 F:GCAATTCGTGTTATGTTCATTT R:TGCATTCAGAGAATAACCGA 58 201~251 TAMRA Atr-117 F:TGCATGACACAGGACTTACC R:TGCCTCTCAATAGCAACATC 55 191~259 TAMRA Eu-39 F:ACCCGCAATTATTAGGAC R:CAGGACAAACTCAGACCC 58 178~276 FAM Eu-40 F:TCACGCAGTCTCACAAGG R:TTCAATGGCAAACAGCAC 58 384~418 HEX Eu-41 F:ACACTATCCCGCCACAAT R:CCACTGAAGCCATCATCT 58 322~410 FAM Afu19 F:CATCTTAGCCGTCTGTGGTAC R:CAGGTCCCTAATACAATGGC 53 120~139 FAM Afu22 F:TCCACAATCCTGAATAATGAC R:GCACCATCTAATACGAAATTG 52 140~242 FAM Afu39 F:TTCTGAAGTTCACACATTG R:ATGGAGCATTATTGGAAGG 55 119~140 HEX Afu54 F:CTCTAGTCTTTGTTGATTACAG R:CAAAGGACTTGAAACTAGG 55 149~221 FAM Afu68 F:TTATTGCGCATGGTGTAGCTAAAC R:AGCCCAACACAGACAATATC 55 128~250 HEX

图1 GeneM apper 3.7软件处理微卫星数据图
Fig.1 M icrosatellite data readed by GeneM apper 3.7
经T-test分析表明,利用比例法和补齐法计算得到的养殖小体鲟种群的有效等位基因数(Ne)、期望杂合度(He)、多态信息含量(PIC)和Hardy-Weinberg平衡遗传偏离指数(d)等遗传多样性指标均无显著性差异(P>0.05),因此,在对小体鲟种群进行遗传多样性分析时仅使用补齐法进行数据处理即可。
表2 用比例法和补齐法得到数据的比较分析
Tab.2 Comparison analysis of the data between proportion method and fillingmethod

微卫星位点NeHe PIC d locus 比例法补齐法比例法补齐法比例法补齐法比例法补齐法Atr-101 2.0597 1.9849 0.5145 0.4962 0.4992 0.4822-0.1297-0.0976 Atr-105 3.4507 3.4855 0.7102 0.7131 0.6842 0.6881 0.0508 0.0465 Atr-107 10.309 10.559 0.9030 0.9053 0.8951 0.8975 0.0248 0.0222 Atr-114 6.1614 6.7935 0.8377 0.8528 0.8205 0.8371-0.1091-0.1249 Atr-117 10.427 11.441 0.9041 0.9126 0.8963 0.9058 0.0400 0.0304 Eu-39 8.3893 8.7796 0.8808 0.8861 0.8684 0.8744 0.0506 0.0443 Eu-40 2.5700 2.5813 0.6109 0.6126 0.5815 0.5825-0.8045-0.8051 Eu-41 6.0496 6.0790 0.8347 0.8355 0.8168 0.8180-0.3026-0.3033 Afu19 1.9372 1.9395 0.4838 0.4844 0.4580 0.4586 0.0798 0.0784 Afu22 9.5969 15.873 0.8958 0.9026 0.8883 0.9329 0.0164 0.0087 Afu39 2.1815 2.2212 0.5416 0.5498 0.4481 0.4581-0.0630-0.0770 Afu54 1.0846 1.1104 0.0780 0.0852 0.0775 0.0987 0.1481 0.0511 Afu68 9.0253 9.2678 0.8892 0.8921 0.8791 0.8822-0.1440-0.1467平均值5.9319 6.6777 0.7142 0.7193 0.6928 0.7028-0.0844-0.0980 P值0.1754 0.1375 0.0686 0.2294
2.3 遗传多样性分析
利用SPAGeDi1.4软件对通过补齐法读取的小体鲟种群数据进行遗传多样性分析,结果见表3。从表3可以看出,13个微卫星位点在小体鲟种群内共检测到224个等位基因,平均等位基因数为17.58,每个位点的等位基因数从5个到30个不等,大小在122~418 bp之间,位点Afu39在该种群的等位基因数最少,为5个,位点Atr-107在该种群的等位基因数最多,为30个。小体鲟种群有效等位基因数平均为6.677 7,表观杂合度平均为0.648 0,期望杂合度平均为0.719 3,Hardy-Weinberg平衡遗传偏离指数平均为-0.098 0,种群多态信息含量平均为0.702 8。结果表明,筛选出的13个微卫星位点是适用于小体鲟种群遗传研究的具有多态性的良好分子标记。
表3 小体鲟种群的13个微卫星位点遗传多样性分析
Tab.3 Genetic diversity of 13 m icrosatellite loci in sterlet population
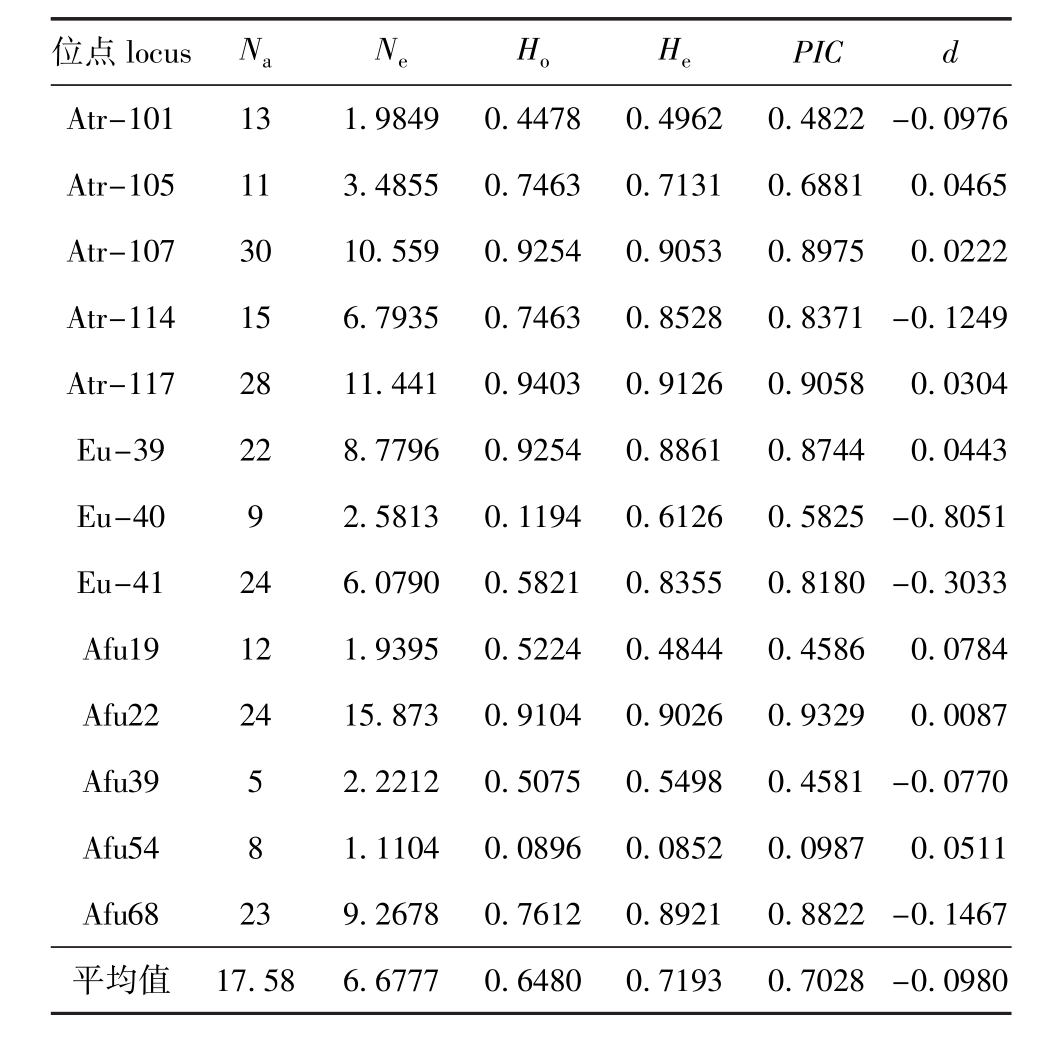
位点locus NaNeHoHePIC d Atr-101 13 1.9849 0.4478 0.4962 0.4822-0.0976 Atr-105 11 3.4855 0.7463 0.7131 0.6881 0.0465 Atr-107 30 10.559 0.9254 0.9053 0.8975 0.0222 Atr-114 15 6.7935 0.7463 0.8528 0.8371-0.1249 Atr-117 28 11.441 0.9403 0.9126 0.9058 0.0304 Eu-39 22 8.7796 0.9254 0.8861 0.8744 0.0443 Eu-40 9 2.5813 0.1194 0.6126 0.5825-0.8051 Eu-41 24 6.0790 0.5821 0.8355 0.8180-0.3033 Afu19 12 1.9395 0.5224 0.4844 0.4586 0.0784 Afu22 24 15.873 0.9104 0.9026 0.9329 0.0087 Afu39 5 2.2212 0.5075 0.5498 0.4581-0.0770 Afu54 8 1.1104 0.0896 0.0852 0.0987 0.0511 Afu68 23 9.2678 0.7612 0.8921 0.8822-0.1467平均值17.58 6.6777 0.6480 0.7193 0.7028-0.0980
3.1 微卫星数据读取方法在多倍体基因组生物中
的比较分析
微卫星DNA标记是种群遗传学研究中较常用的分子标记,可以直接方便地区分杂合子和纯合子[12]。由于多倍体基因组进化的复杂性,导致其数据分析处理也比较复杂。与二倍体微卫星数据单一的处理方法不同,在对鲟鱼等多倍体样品进行基因型读取的时候,由于试验方法和数据处理习惯的不同,存在比例法、补齐法和1、0法等多种微卫星数据读取方法[10,13]。
用聚丙烯酰胺凝胶电泳得到的微卫星结果只能用1、0法读取,由于聚丙烯酰胺凝胶电泳具有操作不便、误差较大等缺点[14],越来越多的实验室特别是国外实验室都使用灵敏度更高的荧光毛细管电泳法进行微卫星标记的研究[15],而比例法、补齐法常见于荧光毛细管电泳数据读取,且在读取数据时发现1、0法与补齐法数据可以进行换算,两种数据处理方法得到的遗传系数相同,因此,本研究中仅选择补齐法与比例法进行数据比较分析。
目前,涉及微卫星数据读取方法的文献报道较少,尚未见到使用比例法、补齐法(或1、0法)进行不同数据读取方法比较的报道。微卫星的不同数据读取方法是否会造成分析结果的差异,差异是否显著,这些疑问会影响到对研究结果的认同。本研究中通过计算比较了比例法和补齐法对养殖小体鲟种群的有效等位基因数、期望杂合度、多态信息含量和Hardy-Weinberg平衡遗传偏离指数等数据,经T-test比较分析,这几个遗传多样性指标均无显著性差异(P>0.05)。小体鲟是多倍体鱼类,比二倍体生物纯合子要少,无效等位基因频率低,对试验结果的影响微乎甚微,这可能是造成用两种方法读取结果差异不显著的主要原因。但至少在本研究基础上可以认为,在多倍体微卫星数据处理方面,比例法和补齐法对多倍体遗传分析方面没有太大差别。因此,可以根据分析样品等的实际情况选择读图方法。
3.2 小体鲟养殖群体遗传多样性分析
本试验表明,小体鲟种群表观杂合度较高,该种群在Hardy-Weinberg平衡假设下[16]杂合度的期望值也较高,其原因可能与鲟鱼的高倍性特征有关。群体杂合度越高,反映群体的遗传多样性信息也越多;杂合度越高,说明该位点为群体检测所能提供的遗传多样性信息量越大。有效等位基因数是基因纯合度的倒数[17],它表明了等位基因间的相互影响,等位基因在群体中分布越均匀,有效等位基因就越接近实际检测到的等位基因的绝对数,这有利于改善种群的遗传多样性。本试验中计算出的有效等位基因数与实际检测到的等位基因数差异显著,表明等位基因数在群体中分布不均匀,并且有效等位基因数较高,说明所选的引物能较好地反映群体遗传变异的情况。
多态信息含量是由Botstein等[18]提出的用于度量群体多态程度的指标,当PIC<0.25时为低度多态性,当PIC>0.5时为高度多态性,当0.25<PIC<0.5时为中度多态性。由本试验结果可见,小体鲟种群的平均PIC数值大于0.5,均属于高多态性,说明基因变异较大,具有遗传多样性研究价值。
Hardy-Weinberg平衡遗传偏离指数反映了表观杂合度和期望杂合度两者之间的平衡关系,当d<0时表示杂合子缺失,当d>0时表示杂合子过剩,d值越接近0表示基因型分布越均匀[19]。本研究中,小体鲟种群有7个位点d值大于0,表示均存在杂合子过剩的现象,该现象一般是由群体过小且封闭、群体子代由有限的亲本产生而导致的,一般出现在人工养殖群体中,而在天然群体中较少见[20];在其余6个位点d值均小于0,表明存在杂合子缺失现象,该现象主要由种群退化、近交、无效等位基因、样本太小等所致[21]。
本研究中的小体鲟种群为人工繁殖F2代群体,遗传多样性分析表明,该群体已经存在一定程度的近交现象。鲟鱼怀卵量大,留存的同一批后备亲鱼容易出现近亲,本研究结果进一步证实了此弊端的存在,同时警示养殖业者注意亲鱼管理,提高苗种生产的质量。为此,可通过引进不同来源亲本的方法改善这一情况,以增加养殖种群的遗传多样性丰富度。
参考文献:
[1] Bemis W E,Findeis E K,Grande L.An overview of Acipenseriformes[J].Environmental Biology of Fishes,1997,48(1-4):25-71.
[2] Simeanu C,Simeanu D,Pǎsǎrin B.Research on physic-chemical indices of themeat of the sturgeon species Polyodon spathula[J]. University of Agricultural Sciences and Veterinary Medicine Iasi,2012,57:230-233.
[3] 王静波,曹欢,王小亮,等.高水温对杂交鲟主要免疫指标的影响[J].大连海洋大学学报,2015,29(5):484-488.
[4] Billard R,Lecointre G.Biology and conservation of sturgeon and paddlefish[J].Reviews in Fish Biology and Fisheries,2000,10(4):355-392.
[5] 杨述林,王志刚,樊斌,等.微卫星及其功能研究[J].湖北农业科学,2003(6):91-93.
[6] 刘洋,鲁翠云,李超,等.应用微卫星标记鉴别施氏鲟、达氏鳇及其杂交子代[J].水产学杂志,2015,28(1):18-23.
[7] 向燕,孔杰,周洲,等.鲟鱼养殖亲鱼群体遗传多样性分析[J].西南农业学报,2013,26(5):2112-2115.
[8] 尹洪滨,孙中武,孙大江,等.小体鲟染色体核型及倍性的细胞遗传学研究[J].水产学杂志,2009,22(3):32-35.
[9] 卢迈新,黄樟翰,肖学铮,等.主要养殖鲟鱼的生物学及养殖前景[J].大连水产学院学报,2000,15(4):280-287.
[10] May B,Krueger C C,Kincaid H L.Genetic variation atmicrosatellite loci in sturgeon:primer sequence homology in Acipenser and Scaphirhynchus[J].Canadian Journal of Fisheries and Aquatic Sciences,1997,54(7):1542-1547.
[11] Rodzen JA,May B.Inheritance ofmicrosatellite loci in thewhitesturgeon(Acipenser transmontanus)[J].Genome,2002,45(6):1064-1076.
[12] Dufresne F,StiftM,Vergilino R,et al.Recent progress and challenges in population genetics of polyploid organisms:an overview of current state-of-the-artmolecular and statistical tools[J]. Molecular Ecology,2014,23(1):40-69.
[13] Welsh A,May B.Development and standardization of disomicmicrosatellitemarkers for lake sturgeon genetic studies[J].Journal of Applied Ichthyology,2006,22(5):337-344.
[14] 罗奕龙,杨俊娟,高炜,等.对比两种分析微卫星多态性的电泳方法[J].中华医学遗传学杂志,2004,21(5):526-527.
[15] Weinberger R.The evolution of capillary electrophoresis:past,present,and future[J].American Laboratory,2002,34(10):32-40.
[16] Levene H.On a matching problem arising in genetics[J].The Annals of Mathematical Statistics,1949,20(1):91-94.
[17] Nei M.Molecular Population Genetics and Evolution[M].Amsterdam:North-Holland Publishing Company,1975:2-40.
[18] Botstein D,White R L,Skolnick M,et al.Construction of a genetic linkagemap in man using restriction fragment length polymorphisms[J].American Journal of Human Genetics,1980,32(3):314-331.
[19] 王蕾,张立冬,万玉美,等.牙鲆微卫星标记的筛选及群体遗传结构分析[J].遗传,2010,32(10):1057-1064.
[20] 刘云国,陈松林,李八方.牙鲆养殖群体遗传变异的微卫星标记研究[J].海洋水产研究,2005,26(5):27-33.
[21] Antoro S,Na-Nakorn U,Koedprang W.Study of genetic diversity of orange-spotted grouper,Epinephelus coioides,from Thailand and Indonesia using microsatellite markers[J].Marine Biotechnology,2006,8(1):17-26.
Com parativemethods of reading data derived from genetic diversity analysis by m icrosatellite loci in farmed sterlet Acipenser ruthenus population
Abstract:Thirteen microsatellite lociwere amplified from sterlet Acipenser ruthenus using published cross-species primers for polymorphism analysis.A proportion method and a filling method were comparatively used for reading data derived from microsatellite in order to find the suitablemethod.The results showed that there was no significant difference(P>0.05)between the twomethods so that fillingmethod was chosen for the genetic diversity analysis in the following study.The observed number of alleles per locuswas ranged from 5 to 30,and the levels of observed and expected heterozygosities for polymorphic lociwere changed from 0.089 6 to 0.940 3 and from 0.085 2 to 0.912 6,respectively,with average of0.648 0 and 0.719 3.Therewas average Hardy-Weinberg departure value(d)of-0.098 0 and average polymorphism information content(PIC)of0.702 8,indicating that polymorphic lociwere all suitablemolecularmarkers for genetic analysis of sterlet population.The genetic diversity analysis revealed that the test farmed sterlet population had inbreeding to a certain extent.It is suggested that the genetic diversity of the population be improved by introduction of different broodstocks.
Key words:Acipenser ruthenus;microsatellite;cross-species amplification;genetic diversity
中图分类号:S917.4
文献标志码:A
DOI:10.16535/j.cnki.dlhyxb.2016.05.008
文章编号:2095-1388(2016)05-0516-06
收稿日期:2015-12-11
基金项目:国家“十二五”科技支撑计划项目(2012BAD26B05);北京市鲟鱼、鲑鳟鱼创新团队(SCGWZJ20121102-1);冷水性鱼类科技创新团队(JNKST201611)