花鲈瘦素基因的克隆及低盐度条件下调控表达分析
张沛,温海深,迟美丽,钱焜
(中国海洋大学水产学院,山东青岛266003)
摘要:瘦素 (Leptin,Lep)在动物摄食、生长、生殖、免疫、能量平衡等方面具有重要作用,为研究花鲈Lateolabrax maculatus Lep功能,采用cDNA末端快速扩增法 (RACE)克隆花鲈LepA cDNA全长序列。急性低盐度调控试验设置海水组 (盐度32)、半海水组 (盐度16)和淡水组 (盐度0),盐度调控24、48、96、144、192 h后测定花鲈肝脏中LepA mRNA的表达情况。结果表明:LepA cDNA全长序列为643 bp,其中开放阅读框483 bp,编码161个氨基酸,其氨基酸序列由信号肽和A、B、C、D 4个α螺旋区域组成;花鲈LepA氨基酸序列与其他物种同源性较低,但三级结构较为保守;花鲈LepA主要在肝脏中表达,其次为脑;低盐调控48 h时,半海水组与淡水组LepA表达无显著性差异 (P>0.05),但均显著低于海水组(P<0.05),分别为海水组的33.38%和26.86%。研究表明,急性低盐度调控可降低花鲈LepA表达水平。
关键词:花鲈;瘦素;盐度;基因克隆;基因表达
瘦素 (Leptin,Lep)是肥胖基因 (OB)编码的一种蛋白类激素,它在摄食、生长、生殖、免疫、能量平衡等方面具有重要作用[1]。Lep属于Ⅰ-型螺旋细胞因子家族,其三维结构特征包括4 个α螺旋[2]。从小鼠中首次发现并克隆出Lep基因[1],哺乳动物仅有一种Lep,多数鱼类也只有一种Lep,从部分鱼类如斑马鱼 Danio rerio、青鳉Oryzias latipes、鲤Cyprinus carpio、斜带石斑鱼Epinephelus coioides等中获得了LepA和LepB两种Lep亚型。哺乳动物中Lep主要由脂肪细胞合成[1],而鱼类Lep主要由肝脏分泌[3]。研究表明,物种间Lep氨基酸序列高度不保守,但其三级结构和功能保守性较高[4]。哺乳动物和鱼类中均发现Lep可通过与下丘脑中特异受体结合,进而控制食欲[5],调控能量消耗[6],参与机体的免疫调控[7],促进细胞因子生成,对动物生长繁殖和胚胎发育具有重要作用[8]。目前,Lep在哺乳动物生理、医学等领域的作用研究较为深入,其可通过血液游离或与瘦素结合蛋白结合的方式,与中枢或外周的受体结合发挥作用[9]。有关Lep在鱼类渗透调控中的作用研究,仅见高盐度对罗非鱼Oreochromis mossambicus LepA表达的影响报道,并发现LepA在渗透调控中发挥重要作用[10]。
花鲈Lateolabrax maculatus为东北亚特有种类,主要分布于中国、日本和朝鲜的沿海区域,属广温、广盐性浅海近岸中下层鱼类,为中国重要的网箱与池塘养殖经济鱼类之一[11]。花鲈因其肉质鲜美,颇受消费者喜爱,社会需求日益增加。目前,有关花鲈形态学和养殖技术方面的研究较为普遍,而有关盐度对花鲈生理影响的研究较少,已有报道表明,不同盐度下1龄花鲈的生长特性有差异,最适生长盐度为16~17[12],但有关用盐度调控花鲈生理分子机制的研究尚未见报道。本研究中,通过克隆花鲈LepA基因并对其序列进行分析,探究了各组织中LepA mRNA的表达差异及低盐度调控后的表达水平,旨在为研究花鲈Lep功能奠定基础。
1 材料与方法
1.1 材料
试验用花鲈为山东省青岛市胶南近海网箱养殖的同规格健康花鲈,共78尾,体质量为(803.5± 49.8)g,体长为(40.5±1.4)cm。将花鲈在实验室水族箱中暂养,试验用水为沙滤自然海水 (盐度32),淡水为曝气的自来水,水温为(17.5± 0.4)℃。
1.2 方法
1.2.1 样品的制备 取3尾花鲈,用MS-222麻醉后解剖,取其肠、脾、肌肉、心脏、胃、精巢、肝、鳃、脑、盲肠、头肾、肾、胸腺、垂体14个组织,组织样品用液氮冷冻后于冰箱 (-80℃)中保存,用于LepA mRNA组织荧光定量检测。
1.2.2 急性盐度调控试验 将试验鱼暂养3 d后进行盐度调控试验。首先取6尾鱼作为初始 (0 h)对照,试验分为3组:海水组、半海水组和淡水组,每组设4个平行水族箱(0.6 m×0.8 m×1.0 m),每个水族箱放养6尾鱼。海水组盐度不变,半海水组和淡水组每12 h盐度分别降低4、8,盐度降至16、0后保持稳定。每天9:00和21:00换水3/4,各盐度试验用水以海水和淡水按比例混匀调温后加入水族箱。盐度调控 24、48、96、144、192 h时,分别从每个水族箱取1尾鱼,经MS-222麻醉解剖,取其肝脏用液氮冷冻后于冰箱(-80℃)中保存,用于Lep mRNA荧光定量检测。
1.2.3 总RNA的提取和cDNA第一链合成 取100 mg组织样品,用Trizol法提取总RNA,使用Biodropsis BD-1000核酸仪测定RNA浓度,用10 g/L琼脂糖凝胶电泳检测RNA的完整性。用PrimerScript RT reagent Kit试剂盒反转录成cDNA后,于冰箱 (-80℃)中保存。提取肝脏总RNA,用SmartTMRACE cDNA Amplification Kit试剂盒合成全长克隆cDNA模板。
1.2.4 花鲈LepA cDNA片段的克隆 参照近缘鱼类Lep氨基酸序列,用 CODEHOP设计简并引物[13](表1)。cDNA片段克隆的PCR反应程序:94℃下预变性5 min;94℃下变性30 s,55℃下退火30 s,72℃下延伸1 min,共进行35个循环;最后在72℃下再延伸10 min,用10 g/L琼脂糖凝胶电泳检测PCR产物。用Sagecreation电泳凝胶成像系统分析获得的目的条带,目的片段产物利用TIANgel Midi Purification Kit试剂盒进行凝胶回收,连接pEASY-T1载体,并转化到Trans-T1感受态细胞中,在氨苄抗性固体培养基 (37℃)中过夜培养;用蓝白斑技术筛选目的细菌,用氨苄抗性液体培养基将其扩大培养,菌体用PCR检测,挑选阳性菌液送北京华大基因科技有限公司进行测序。
1.2.5 花鲈LepA cDNA 5′端和3′端克隆 用SMARTTMRACE cDNA Amplification Kit试剂盒扩增5′端和3′端,根据获得的LepA cDNA片段,用Primer 5.0软件设计与试剂盒中 UPM匹配的引物 (表1),PCR反应程序参照试剂盒说明书,RACE PCR产物测序同片段克隆。用DNAMAN拼接基因3′端和5′端序列后得到基因全长。
表1 花鲈瘦素基因克隆与表达的引物
Tab.1 Primers used for cloning and expressing of seabass LepA

引物primers序列sequences(5′-3′)退火温度Tm/℃Fragment-F ATGGACTACACTCTGGCC 55.0 Fragment-R GCAGCAGATTGAGGAACTC 5′RACE LepA-R1 GGAAGGTCCATCCAGATCATCGGCAGG 68.1 LepA-R2 GACTGAGTGTCAGACCAGCAGGGACCAGG 70.5 3′RACE LepA-F1 CTTCCTGGTCCCTGCTGGTCTG 65.7 LepA-F2 GTGGCCGTGCTGGAGGGTTACAA 65.5 LepA-RT-F GAGCAGCTCATGGTCAGG 62.0 LepA -RT-R GGCCACTATGGAGGAAGGT
1.2.6 LepA基因序列分析 用DNAMAN预测开放阅读框并推测氨基酸序列,利用Signal P 3.0软件分析信号肽,用Scratch程序预测二硫键。预测的氨基酸序列在NCBI中进行Blast搜索,用DNAMAN将其与其他物种的Lep氨基酸序列进行多重序列比对。选取部分物种 Lep氨基酸序列,用Clustal X和MEGA 4.0软件以邻接法(Neighbour-Joining)构建系统进化树。用DNAstar中Megalign对鱼类Lep氨基酸进行一致性分析,用SWISSMODEL中Automated Model程序分析蛋白结构。
1.2.7 LepA mRNA的荧光实时定量PCR 根据LepA cDNA全长序列设计表达引物 (表1),以βactin为内参基因,检测花鲈14个组织中LepA mRNA的表达水平。反转录产物用双蒸水进行4倍梯度稀释,共设5个梯度,每个梯度设3个重复,用SYBR Green Premix Ex TaqTM(Tli RNaseH Plus)试剂盒,在ABI Step One Plus实时定量PCR仪上进行荧光定量PCR反应。采用两步法,β-actin、LepA PCR反应程序:94℃下预变性30 s;94℃下变性5 s,退火30 s(β-actin PCR 60℃,LepA PCR 62℃),共进行40个循环;溶解步骤。梯度稀释结果用Excel软件分析扩增效率。试验组肝脏mRNA反转录产物4倍稀释后,做3个重复的荧光定量PCR反应。用2-ΔΔCt法处理荧光定量所得数据。
1.3 数据处理
试验数据均以平均值±标准差 (mean±S.D.)表示,用SPSS 19.0软件进行单因素方差分析,用Duncan法进行多重比较,显著性水平设为0.05。
2 结果与分析
2.1 花鲈LepA cDNA全长序列及其蛋白结构分析
克隆得到花鲈LepA cDNA全长序列为643 bp,其中包括483 bp开放阅读框,共编码161个氨基酸。序列已经提交到 NCBI数据库 (GenBank:KF850511)。花鲈LepA cDNA序列及推测的氨基酸序列如图1所示。LepA氨基酸序列包括信号肽和A、B、C、D 4个α螺旋结构区域,两个保守的半胱氨酸残基 (图2)。对鱼类Lep氨基酸序列进行一致性分析,表明物种间Lep氨基酸序列差异较大,花鲈与斜带石斑鱼一致性较高,为88.9%(表2)。Lep蛋白模拟结果 (图3)表明,物种间Lep基因虽存在差异,但其蛋白三维结构相近。
2.2 LepA蛋白的系统进化分析
基于鱼类及高等物种Lep氨基酸序列的系统进化树结果显示,鲈形目LepA为一分支,其与鲀形目、鳉形目亲缘关系较近,鲑形目为一支,鲤形目为一支,鱼类LepB为一支,高等动物为一支,进化树与传统分类地位相符 (图4)。

图1 花鲈LepA cDNA序列及推测的氨基酸序列
Fig.1 Nucleotide sequence and deduced amino acid sequence of seabass LepA gene

图2 瘦素氨基酸多序列比对
Fig.2 Multiple sequence alignment of Leps from several fishes
注:*表示所有比对物种Lep氨基酸完全一致;虚线表示信号肽;黑框表示A~D α螺旋结构区域;阴影表示两个保守的半胱氨酸残基
Note:*,represents the complete identity of amino acid in all the species;dotted line marks signal peptide;A-D,α-helices are boxed;the cysteine residues are shaded
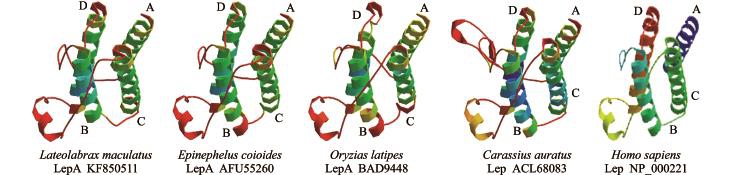
图3 模拟瘦素蛋白结构*
Fig.3 Projected tertiary structure of Leps
表2 鱼类瘦素氨基酸序列的一致性
Tab.2 Amino acid sequence identity of fish %
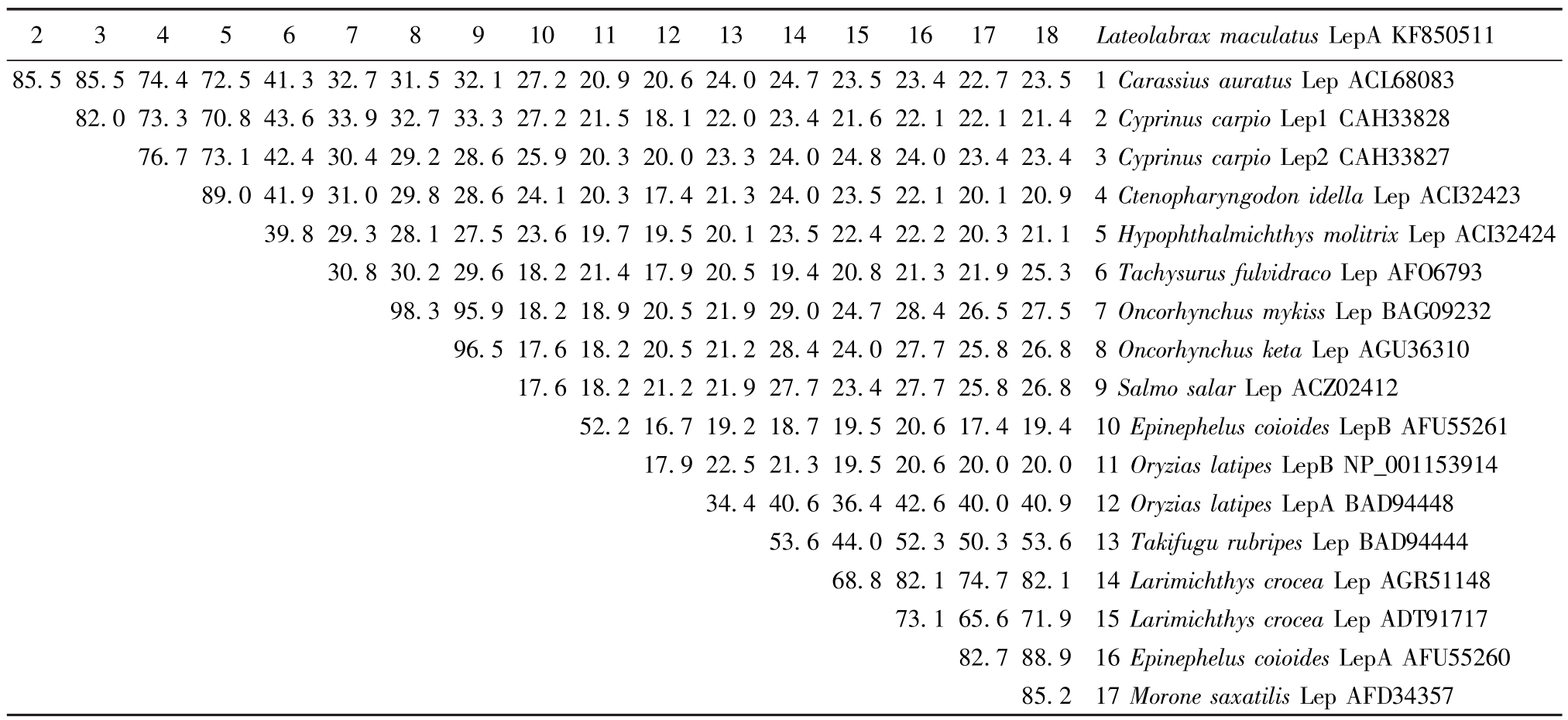
2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 Lateolabrax maculatus LepA KF850511 85.5 85.5 74.4 72.5 41.3 32.7 31.5 32.1 27.2 20.9 20.6 24.0 24.7 23.5 23.4 22.7 23.5 1 Carassius auratus Lep ACL68083 82.0 73.3 70.8 43.6 33.9 32.7 33.3 27.2 21.5 18.1 22.0 23.4 21.6 22.1 22.1 21.4 2 Cyprinus carpio Lep1 CAH33828 76.7 73.1 42.4 30.4 29.2 28.6 25.9 20.3 20.0 23.3 24.0 24.8 24.0 23.4 23.4 3 Cyprinus carpio Lep2 CAH33827 89.0 41.9 31.0 29.8 28.6 24.1 20.3 17.4 21.3 24.0 23.5 22.1 20.1 20.9 4 Ctenopharyngodon idella Lep ACI32423 39.8 29.3 28.1 27.5 23.6 19.7 19.5 20.1 23.5 22.4 22.2 20.3 21.1 5 Hypophthalmichthys molitrix Lep ACI32424 30.8 30.2 29.6 18.2 21.4 17.9 20.5 19.4 20.8 21.3 21.9 25.3 6 Tachysurus fulvidraco Lep AFO6793 98.3 95.9 18.2 18.9 20.5 21.9 29.0 24.7 28.4 26.5 27.5 7 Oncorhynchus mykiss Lep BAG09232 96.5 17.6 18.2 20.5 21.2 28.4 24.0 27.7 25.8 26.8 8 Oncorhynchus keta Lep AGU36310 17.6 18.2 21.2 21.9 27.7 23.4 27.7 25.8 26.8 9 Salmo salar Lep ACZ02412 52.2 16.7 19.2 18.7 19.5 20.6 17.4 19.4 10 Epinephelus coioides LepB AFU55261 17.9 22.5 21.3 19.5 20.6 20.0 20.0 11 Oryzias latipes LepB NP_001153914 34.4 40.6 36.4 42.6 40.0 40.9 12 Oryzias latipes LepA BAD94448 53.6 44.0 52.3 50.3 53.6 13 Takifugu rubripes Lep BAD94444 68.8 82.1 74.7 82.1 14 Larimichthys crocea Lep AGR51148 73.1 65.6 71.9 15 Larimichthys crocea Lep ADT91717 82.7 88.9 16 Epinephelus coioides LepA AFU55260 85.2 17 Morone saxatilis Lep AFD34357
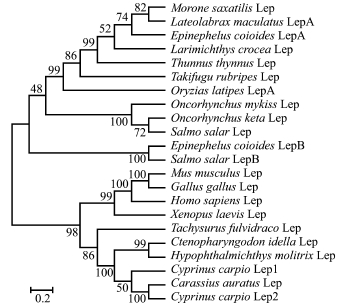
图4 基于瘦素氨基酸序列的N-J系统进化树
Fig.4 Neighbor-Joining tree of the deduced amino acid sequences of the Leps
2.3 LepA mRNA的组织表达模式
以肝脏作为对照因子,计算不同组织 LepA mRNA相对定量表达模式。结果显示:花鲈LepA基因的组织表达十分广泛,肝脏中转录水平最高;脑、垂体、肾脏、盲肠、胃、鳃中表达量次之;其余组织中表达量较低 (图5)。
2.4 LepA mRNA在急性低盐调控后的表达规律
从图6可见:低盐调控48 h,半海水组和淡水组LepA表达量较0 h时显著降低 (P<0.05),且均显著低于海水组 (P<0.05),二者的LepA表达水平分别降至海水组的33.38%和26.86%,但二者间无显著性差异 (P>0.05);调控96 h后,各试验组LepA表达水平较0 h时显著降低 (P<0.05),但各组间均无显著性差异 (P>0.05)。
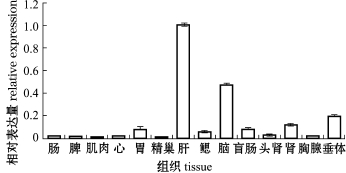
图5 花鲈各组织中LepA mRNA的相对表达量
Fig.5 Relative expression levels of LepA mRNA in tissues of seabass
3 讨论
本研究中首次从花鲈中克隆得到LepA cDNA全长序列。多物种Lep氨基酸序列比对结果表明,花鲈LepA基因符合Lep基因的基本特征,4个α螺旋结构区域内氨基酸序列一致性较高。序列一致性分析表明,物种间Lep cDNA序列和氨基酸序列存在较大差异,这对物种间该基因功能上是否存在差异提出疑问。Lep中有一个由两个半胱氨酸残基形成的二硫键,物种间三维结构较为保守,说明该二硫键在物种进化中对Lep蛋白结构稳定发挥重要作用。鱼类与哺乳动物Lep氨基酸序列的一致性为13%~25%,低一致性可能预示Lep在物种间高度进化后存在结构差异,进而影响其功能。
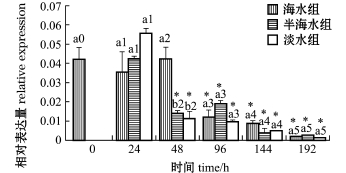
图6 低盐度调控后LepA mRNA的表达规律
Fig.6 mRNA expression pattern of LepA after low salinity regulation
注:标有不同小写字母表示同一时间下不同组间有显著性差异(P<0.05);*表示与对照组 (0 h)有显著性差异(P<0.05)
Note:The means with different letters in different groups are significant differences at the 0.05 probability level at same time;*,significant differences at the 0.05 probability level from control group (0 h)
基于Lep氨基酸序列的系统进化树分析表明,相对近缘物种为一分支,但整体分布与物种分类地位存在差异,高等动物与鲤形目为一个大的分支,而小鼠与鸡的同源性要高于人。这些差异可能是由于物种间Lep氨基酸一级结构低同源性导致的分析误差,同时也表明,物种间Lep结构的改变可能造成功能上的差异,实际情况有待进一步验证。
哺乳动物的Lep基因主要在脂肪组织中表达,不同位置的脂肪组织中表达情况存在差异,但在肝脏中未检测到该基因表达[14]。花鲈LepA基因主要在肝脏中表达,在脑、垂体、肾脏、盲肠、胃中表达量次之,在肠、脾、肌肉、心脏、精巢、头肾、胸腺 中 表 达 量 较 低。对 虹 鳟 Oncorhynchus mykiss[15]、黄颡鱼Pelteobagrus fulvidraco[16]、鳜Siniperca chuatsi[17]、条纹鲈Morone saxatilis[13]的研究均表明,Lep主要在肝脏中表达,而斜带石斑鱼LepA和LepB在肝脏中表达量较低[17]。此外,斜带石斑鱼LepA和LepB均在脑中大量转录,鳜脑中Lep表达也较高,但河豚脑中未检测到该基因表达。同时,虹鳟、斜带石斑鱼、黄颡鱼的卵巢中也检测到Lep基因的较高表达[15-16,18]。花鲈 LepA组织表达分布差异表明,肝脏为花鲈LepA合成分泌的主要场所。脑是鱼类血糖调控的中枢,能够调控机体的代谢水平,花鲈脑中Lep基因表达较高,说明Lep基因可能对糖脂代谢供能存在一定的调控作用。结合其他研究成果可进一步推测,多数鱼类Lep基因主要在肝脏中转录,但在其他组织中的分布情况存在一定差异,这可能与该基因编码较低的氨基酸序列高度不保守有关。Lep结构与组织分布上的差异表明,该基因的功能在物种间可能不同。
本试验中首次研究了低盐度对硬骨鱼类LepA表达的影响,低盐度调控48 h时,半海水组与淡水组LepA表达水平显著低于海水组,表明低盐度调控抑制了LepA基因表达。有研究表明,环境盐度的改变对鱼类代谢率有显著影响,为适应环境渗透压的改变,鱼体需消耗大量能量,以提高机体代谢水平[19]。对蜥蜴Podarcis sicula和罗非鱼研究发现,LepA可动员肝糖原分解,提高血糖水平[20],进而为鱼体适应渗透压改变提供能量。由此推测,随着肝糖原的消耗,鱼体代谢所需能量逐渐转为其他代谢方式提供,减弱了LepA对肝糖原分解作用,进而降低了LepA的转录水平。半海水组和淡水组盐度降低后,花鲈机体耗能增加,进而加速肝糖原分解,促使LepA基因表达降低。对虹鳟[21]的研究表明,禁食可促进Lep转录,但对鲤[22]研究显示,长期进食对Lep表达无显著影响。本试验中,海水组在前3个采样时间点中未检测出显著差异。虹鳟、大西洋鲑与花鲈Lep氨基酸的一致性分别为25.8%和26.8%,Lep氨基酸序列一级结构的高度不保守,可能是造成物种间LepA差异表达的重要原因。此外,有研究指出,鱼类中存在的两种Lep亚型,可能有功能上的分化[3]。多种因素可能共同导致不同研究结果的出现。急性低渗环境可能抑制花鲈的LepA表达,鱼类间LepA基因在盐度调控中的表达模式存在差异,可能是由于物种间LepA氨基酸序列高度不保守造成。鱼类的Lep基因是否参与盐度调控还有待在其他鱼类中进一步研究。
参考文献:
[1] Zhang Y,Proenca R,Maffei M,et al.Positional cloning of the mouse obese gene and its human homologue[J].Nature,1994,372:425-432.
[2] Zhang F,Basinski M B,Beals J M,et al.Crystal structure of the obese protein leptin-E100[J].Nature,1997,387:206-209.
[3] Gorissen M,Bernier N J,Nabuurs S B,et al.Two divergent leptin paralogues in zebra fish(Danio rerio)that originate early in teleoste anevolution[J].Journal of Endocrinology,2009,201:329-339.
[4] Johnson R M,Johnson T M,Londraville R L.Evidence for leptin expression in fishes[J].Journal of Experimental Zoology,2000,286:718-724.
[5] Murashita K,Uji S,Yamamoto T,et al.Production of recombinant leptin and its effects on food intake in rainbow trout(Oncorhynchus mykiss)[J].Comparative Biochemistry and Physiology Part B:Biochemistry and Molecular Biology,2008,150:377-384.
[6] Pelleymounter M A,Cullen M J,Baker M B,et al.Effects of theobese gene product on body weight regulation in ob/ob mice[J]. Science,1995,269:540-543.
[7] Lee E Y,Park H H,Kim Y T,et al.Cloning and sequence analysis of the interleukin-8 gene from flounder(Paralichthys olivaceous)[J].Gene,2001,274:237-243.
[8] Najakshin A M,Mechetina L V,Alabyev B Y,et al.Identification of an IL-8 homolog in lamprey(Lampetra fluviatilis):early evolutionary divergence of chemokines[J].European Journal of Immunology,1999,29:375-382.
[9] Cohen P,Yang G,Yu X X,et al.Induction of leptin receptor expression in the liver by leptin and food deprivation[J].Journal of Biological Chemistry,2005,280(11):10034-10039.
[10] Baltzegar D A,Reading B J,Douros J D,et al.Role for leptin in promoting glucose mobilization during acute hyperosmotic stress in teleost fishes[J].Journal of Endocrinology,2014,220:61-72.
[11] 张美昭,高天翔.花鲈亲鱼人工培育与催产技术研究[J].青岛海洋大学学报,2001,31(2):195-200.
[12] 杜涛,黄洋,覃雪迎,等.不同盐度养殖1龄花鲈(Lateolabrax japonicas)的生长特性差异分析[J].海洋与湖沼,2013,44 (2):337-341.
[13] Eugene T W,David A B,Matthew E P,et al.Cloning and characterization of leptin in a Perciform fish,the striped bass(Morone saxatilis):control of feeding and regulation by nutritional state [J].General and Comparative Endocrinology,2012,178:98-107.
[14] Secombes C J,Wang T,Hong S,et al.Cytokines and innate immunity of fish[J].Developmental and Comparative Immunology,2001,25:713-723.
[15] Laing K J,Zou J J,Wang T H,et al.Identification and analysis of an interleukin 8-like molecule in rainbow trout Oncorhynchus mykiss[J].Developmental and Comparative Immunology,2002,26:433-444.
[16] Gong Y,Luo Z,Zhu Q L,et al.Characterization and tissue distribution of leptin,leptin receptor and leptin receptor overlapping transcript genes in yellow catfish Pelteobagrus fulvidraco[J]. General and Comparative Endocrinology,2013,182:1-6.
[17] He S,Liang X F,Li L,et al.Gene structure and expression of leptin in Chinese perch[J].General and Comparative Endocrinology,2013,194:183-188.
[18] Zhang H X,Chen H P,Zhang Y,et al.Molecular cloning,characterization and expression profiles of multiple leptin genes and a leptin receptor gene in orange-spotted grouper(Epinephelus coioides)[J].General and Comparative Endocrinology,2013,181:295-305.
[19] Boeuf G,Payan P.How should salinity influence fish growth?-review[J].Comparative Biochemistry and Physiology,2001,130c:411-423.
[20] Paolucci M,Buono S,Sciarrillo R,et al.Effects of leptin administration on the endocrine pancreas and liver in the lizard Podarcis sicula[J].Journal of Experimental Zoology Part A:Comparative Experimental Biology,2006,305:383-395.
[21] Kling P,RØnnestad I,Stefansson S O,et al.A homologous salmonid leptin radioimmunoassay indicates elevated plasma leptin levels during fasting of rainbow trout[J].General and Comparative Endocrinology,2009,162(3):307-312.
[22] Huising M O,Geven E J W,Kruiswijk C P,et al.Increased leptin expression in common carp(Cyprinus carpio)after food intake but not after fasting or feeding to satiation[J].Endocrinology,2006,147:5786-5797.
Cloning and expression analysis of Leptin A gene in seabass Lateolabrax maculatus exposed to low salinity
ZHANG Pei,WEN Hai-shen,CHI Mei-li,QIAN Kun
(College of Fisheries,Ocean University of China,Qingdao 266003,China)
Abstract:cDNA sequence of Leptin A(LepA)gene,which plays key roles in food intake,growth,reproduction,immunity and energy balance,was cloned by rapid amplification of cDNA ends(RACE)in seabass Lateolabrax maculatus exposed to acute salinity changes:seawater group(a salinity of 32),semi-seawater group(a salinity of 16)and fresh water group(a salinity of 0).Hepatic LepA mRNA were determined by qPCR 24,48,96,144,and 192 h after salinity exposure.It was found that LepA cDNA was 643 bp in length containing a CDs of 161 amino acid residules,a signal peptide,and four α-helices(A,B,C and D).Deduced amino acid sequencing showed that the sequence of LepA showed low identity to Leps from other fishes,with conservative three-dimensional structure modeling.LepA was primarily expressed in liver,and a little expression of the LepA was observed in brain. There was significantly less expression of LepA mRNA in seabass in semi-seawater group(accounting for 33.38% of seawater group expression)and fresh water group(accounting for 26.86%of seawater group expression)than that in seawater group at 48 h(P<0.05),without significant difference between the semi-seawater group and fresh water group(P>0.05).LepA mRNA expression was induced to be decreased in all groups after 96 h.The findings suggested that expression of LepA mRNA in seabass be decreased under acute low salinity regulation.
Key words:Lateolabrax maculatus;Leptin;salinity;gene clone;gene expression
中图分类号:S917.4
文献标志码:A
DOI:10.16535/j.cnki.dlhyxb.2016.01.003
文章编号:2095-1388(2016)01-0013-06
收稿日期:2015-05-12
基金项目:国家 “十二五”科技支撑计划重大项目 (2011BAD13B03)
作者简介:张沛 (1989—),男,硕士研究生。E-mail:pei325@126.com
通信作者:温海深 (1963—),男,教授。E-mail:wenhaishen@ouc.edu.cn







