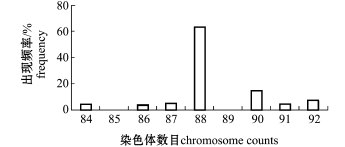
图1 口虾蛄中期分裂相染色体数目统计图
Fig.1 Frequency distribution of chromosome counts recorded in 104 metaphases of mantis shrimp Oratosquilla oratoria
摘要:为了解口虾蛄Oratosquilla oratoria核型组成及DNA含量,以野生口虾蛄为材料,采用秋水仙素内注射法,取其肝胰腺组织经低渗和固定后,获得染色体标本,并对其染色体核型进行了分析,以鸡血细胞DNA为标准,采用流式细胞仪测定口虾蛄不同组织的DNA含量。结果表明:口虾蛄二倍体染色体数目为88,即2n=88,核型组成公式为2n=62m+12sm+14t,染色体总臂数 (NF)为162;口虾蛄不同组织的DNA含量有显著性差异 (P<0.05),各组织DNA含量从大到小依次为肌肉>精巢>卵巢>肝胰腺>血液,肌肉组织含量最高,为10.43 pg/2c,血液含量最低,为8.70 pg/2c,各组织DNA含量平均为9.61 pg/2c;以1 pg=978 Mbp计算,首次成功估算了口虾蛄基因组大小约为9398.58 Mbp。本研究结果可为口虾蛄种质资源保护及未来人工养殖研究提供基础资料。
关键词:口虾蛄;染色体;核型;DNA含量
口虾蛄Oratosquilla oratoria又名虾爬子、皮皮虾等,隶属于节肢动物门Arthropoda、软甲纲Malacostraca、口足目Stomatopoda、虾蛄科Squillidae、口虾蛄属Oratosquilla,为多年生海产经济甲壳类生物,在中国沿海均有分布[1]。其味道鲜美、营养丰富,是经济价值较高的水产品[2]。目前,关于口虾蛄遗传学的研究主要涉及自然种群的遗传结构分析[3]、功能基因的表达调控等方面[4],而对其基础细胞遗传学研究甚少,有关染色体组型及细胞内DNA含量的评估资料缺乏,严重阻碍了口虾蛄种质资源保护及人工养殖研究的进一步发展。
染色体是生物遗传物质的载体,对染色体核型、带型的研究不仅有助于揭示生物的遗传组成[5],还可以从生物进化角度探讨物种间的分类地位及亲缘关系[6],随着基因组学的发展,染色体组型分析作为全基因测序的基础,对了解生物的进化地位及重要功能基因定位有着重要意义[7]。
早在 19世纪,Carnoy就首次报道了褐虾Crangon cataphractus的染色体类型,尽管对十足目染色体研究已有一百余年,但目前已报道过的十足目核型组成也仅占已知十足目种类的0.9%。王青等[8]、相建海[9]、戴继勋等[10]认为:对十足目染色体研究缓慢的原因在于染色体数目多,染色体形态小不易辨认,分裂相不易获得等。就虾蛄科种类而言,染色体数目与核型研究至今尚未见报道。因此,以口虾蛄为研究材料,对其染色体数目与核型进行研究,不仅对口虾蛄的细胞遗传学和人工繁育研究具有重大意义,也可为虾蛄科其他种类的染色体研究提供基础资料。
基因组大小 (DNA含量或C值)是鉴定种质的一个重要内容[11],物种DNA含量的大小可以反映出该物种的进化地位,也是细胞遗传学研究的重要内容之一,因此,研究生物DNA含量具有重要的意义。目前,测定物种基因组大小的方法主要有荧光定量分析法[12]、 孚尔根染色法[13]、 二苯胺[14]、流式细胞术法[15]等。流式细胞术是近30年来发展起来的先进技术,可对单个细胞逐一进行高速准确定量分析并分类[16],该技术具有快速、准确、操作简单、数据量大、测量结果可靠等优点,是测定DNA含量的主流检测手段[17]。
本研究中,采用Giemsa染色法对口虾蛄肝胰腺组织进行了核型分析,并采用流式细胞仪(Patec III,德国)对口虾蛄血液、肌肉、肝胰腺、卵巢、精巢5种不同组织进行DNA含量测定,同时评估其基因组大小,以期为口虾蛄细胞遗传学研究提供基础资料。
1.1 材料
试验用口虾蛄采自辽宁省大连市黑石礁海区,均为性腺发育成熟的野生个体,共78尾,其中雌虾38尾,雄虾40尾,体质量为25~42 g。活体运至实验室后置于25℃循环水槽中暂养。
1.2 方法
1.2.1 染色体的制备 往口虾蛄心孔中慢慢注入质量分数为0.1%的秋水仙素 [剂量为6 μg/g(体质量)],注射后的口虾蛄放在充氧的水槽中暂养2.5 h后解剖。取其肝胰腺并置于生理盐水中经多次漂洗[9,18],加入0.8%的柠檬酸溶液,在室温下低渗40~45 min,低渗后再放入固定液 (V甲醇∶V冰醋酸=9∶1)处理7~10 min,然后转入冰醋酸中放置1 min,经卡诺氏固定液 (V甲醇∶V冰醋酸=3∶1)固定2~3次,每次处理15 min,样品处理完毕后放入冰箱中冷冻过夜。同时采用冷滴片法制片,室内干燥过夜。完全干燥的染色体玻片用10%的Giemsa液染色40 min,自然干燥后镜检。选取图像清晰、染色体分散较好的中期分裂相计数,用以确定染色体数目,根据Levan等[19]的方法进行染色体配对及核型分析,染色体相对长度计算公式为
相对长度=实测长度/全部染色体长度总和×100。1.2.2 DNA含量的测定 鸡红细胞的制备:取雄性健康1龄公鸡,在翅膀下方静脉处采集血液,加入PBS缓冲液,以1500 r/min离心5 min,弃上清液,重复该步骤3次,将最终鸡红细胞悬浊液固定在含有2.5%戊二醛的PBS缓冲液中,并利用血球计数板测得细胞密度为2.8×107cells/mL,将获得的鸡红细胞置于冰箱 (4℃)中保存备用[20]。
取口虾蛄血液、肌肉、肝胰腺、卵巢、精巢组织各20个有效分析样品。将样品分别加入细胞裂解液,经震荡后静置10 min,使用DAPI染色,然后采用流式细胞仪对口虾蛄DNA含量进行测量。以鸡血细胞DNA含量2.5 pg/2c为标准[21],比较口虾蛄各组织样品与鸡血2n峰的相对位置,从而计算口虾蛄不同组织的DNA含量。
1.3 数据处理
试验数据用平均值±标准差表示,用 SPSS 21.0软件进行单因素方差分析,用Duncan法进行多重比较法,显著性水平设为0.05,极显著性水平设为0.01。
2.1 口虾蛄体细胞的染色体数目
本试验中,对104个分散良好、染色体形态清晰的肝胰腺组织中期分裂相进行计数。结果表明,染色体数目为88的分裂相最多,为66个,占总数的63.46% (图1),由此可确定,口虾蛄肝胰腺组织的二倍体染色体数目为88,即2n=88。

图1 口虾蛄中期分裂相染色体数目统计图
Fig.1 Frequency distribution of chromosome counts recorded in 104 metaphases of mantis shrimp Oratosquilla oratoria
2.2 染色体核型
本试验中,口虾蛄有31对中部着丝点染色体(m)、6对亚中部着丝点染色体 (sm)和7对端部着丝点染色体 (t)(图2),染色体臂数 (NF)为162(图3),即口虾蛄染色体核型公式为:2n= 62m+12sm+14t,NF=162。对中期分裂相观察发现,口虾蛄染色体的大小差异较大,最大染色体相对长度为3.999,最小染色体相对长度为1.623,未发现与性别相关的异型染色体。染色体核型分析结果见表1。
2.3 口虾蛄不同组织的DNA含量
以鸡血细胞DNA含量为参比,口虾蛄各个组织DNA相对含量峰值如图4所示,不同组织细胞核DNA相对含量大小依次为肌肉>精巢>卵巢>肝胰腺>血液。口虾蛄不同组织的DNA绝对含量平均值为9.61 pg/2c(表2),此外,以1 pg=978 Mbp计算[22],本研究中发现,口虾蛄基因组平均大小约为9398.58 Mbp。
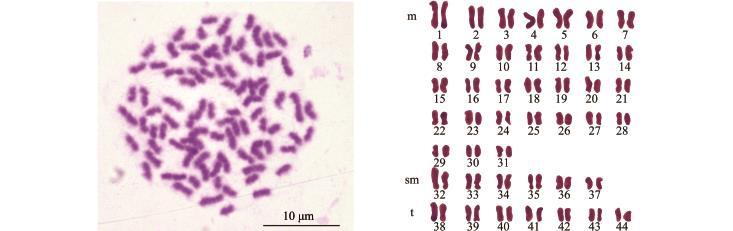
图2 口虾蛄染色体的中期分裂相和核型
Fig.2 Chromosomes metaphase and karyotype in mantis shrimp Oratosquilla oratoria
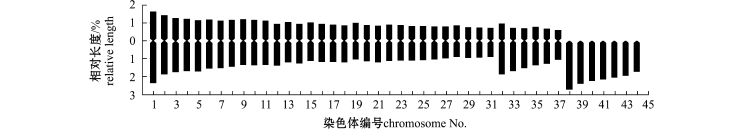
图3 口虾蛄核型模式图
Fig.3 Ideograms showing lengths and shapes of chromosomes in mantis shrimp Oratosquilla oratoria
表1 口虾蛄的核型参数 (平均值±标准差)
Tab.1 Karyotype indices of mantis shrimp Oratosquilla oratoria(mean±S.D.)
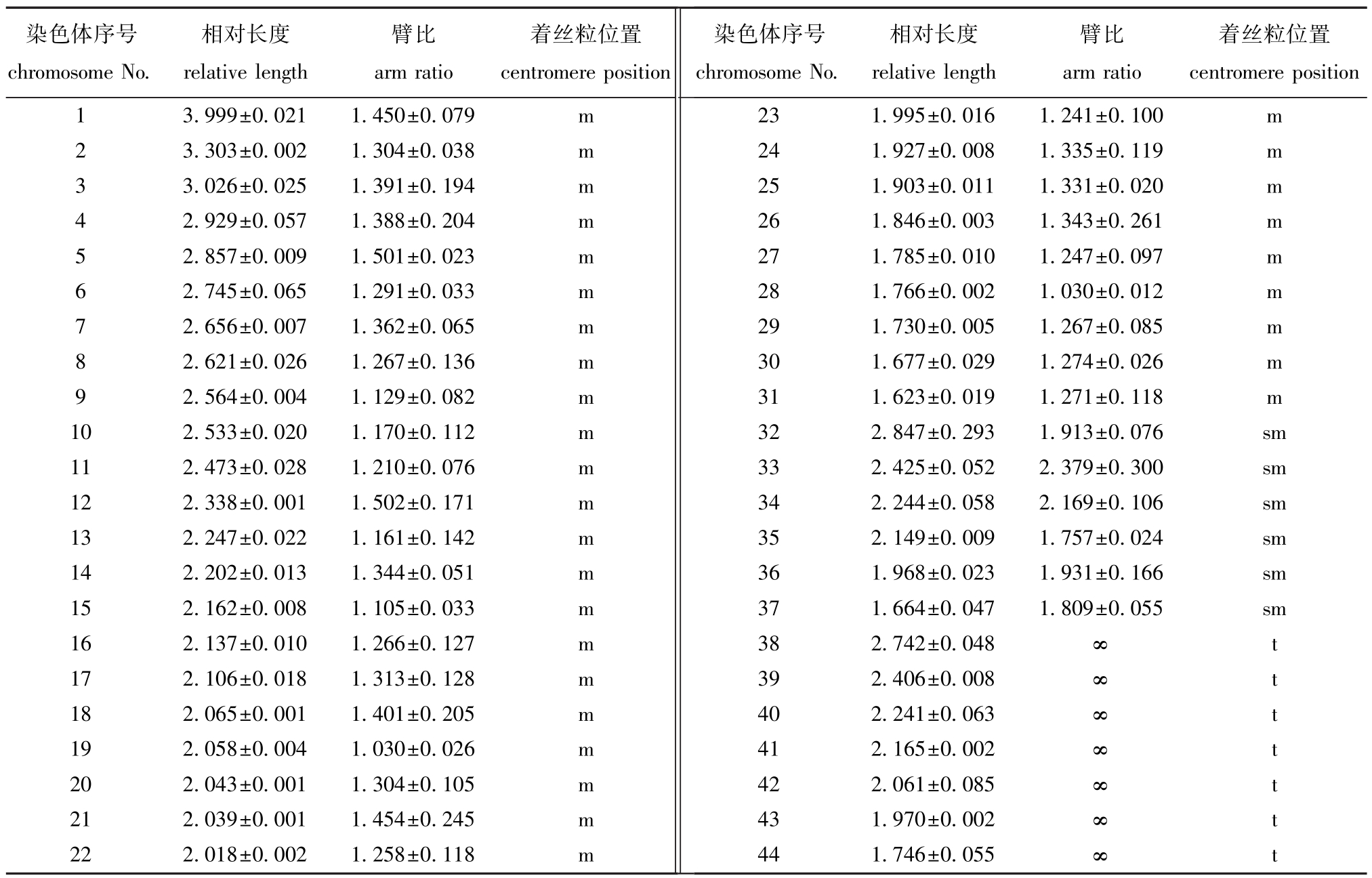
染色体序号chromosome No.相对长度relative length臂比arm ratio着丝粒位置centromere position染色体序号chromosome No.相对长度relative length臂比arm ratio着丝粒位置centromere position 1 3.999±0.021 1.450±0.079 m 2 3.303±0.002 1.304±0.038 m 3 3.026±0.025 1.391±0.194 m 4 2.929±0.057 1.388±0.204 m 5 2.857±0.009 1.501±0.023 m 6 2.745±0.065 1.291±0.033 m 7 2.656±0.007 1.362±0.065 m 8 2.621±0.026 1.267±0.136 m 9 2.564±0.004 1.129±0.082 m 10 2.533±0.020 1.170±0.112 m 11 2.473±0.028 1.210±0.076 m 12 2.338±0.001 1.502±0.171 m 13 2.247±0.022 1.161±0.142 m 14 2.202±0.013 1.344±0.051 m 15 2.162±0.008 1.105±0.033 m 16 2.137±0.010 1.266±0.127 m 17 2.106±0.018 1.313±0.128 m 18 2.065±0.001 1.401±0.205 m 19 2.058±0.004 1.030±0.026 m 20 2.043±0.001 1.304±0.105 m 21 2.039±0.001 1.454±0.245 m 22 2.018±0.002 1.258±0.118 m 23 1.995±0.016 1.241±0.100 m 24 1.927±0.008 1.335±0.119 m 25 1.903±0.011 1.331±0.020 m 26 1.846±0.003 1.343±0.261 m 27 1.785±0.010 1.247±0.097 m 28 1.766±0.002 1.030±0.012 m 29 1.730±0.005 1.267±0.085 m 30 1.677±0.029 1.274±0.026 m 31 1.623±0.019 1.271±0.118 m 32 2.847±0.293 1.913±0.076 sm 33 2.425±0.052 2.379±0.300 sm 34 2.244±0.058 2.169±0.106 sm 35 2.149±0.009 1.757±0.024 sm 36 1.968±0.023 1.931±0.166 sm 37 1.664±0.047 1.809±0.055 sm 38 2.742±0.048 ∞ t 39 2.406±0.008 ∞ t 40 2.241±0.063 ∞ t 41 2.165±0.002 ∞ t 42 2.061±0.085 ∞ t 43 1.970±0.002 ∞ t 44 1.746±0.055 ∞t
通过对口虾蛄5种组织的DNA含量进行测定,结果显示,精巢组织的DNA相对含量直方图具有双峰现象 (图4-E),这可能是由所分析的精巢组织中存在大量单倍体精子、精细胞和次级精母细胞(n),以及少量的二倍体精原细胞和初级精母细胞(2n)所致。5种组织的DNA含量大小依次为肌肉细胞>精巢二倍体细胞>卵巢细胞>肝胰腺细胞>血液细胞 (表2)。经单因素方差分析发现,口虾蛄不同组织的DNA含量有极显著性差异 (P<0.01)。Duncan多重比较结果表明,肌肉和精巢组织的DNA相对含量较高,均显著高于其他组织 (P< 0.05);而血液DNA含量最低,显著低于除肝胰腺组织外的其他组织 (P<0.05)。各组织DNA含量的差异性见表2。试验中还发现,在几种被测组织中,用流式细胞仪测量的结果有一定量的杂峰出现,表明这种组织在制备细胞悬液时破碎细胞较多(图4)。
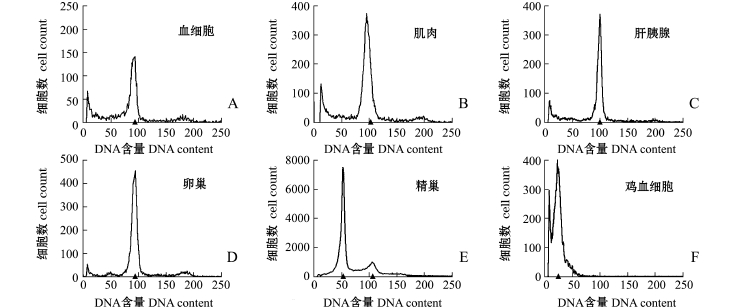
图4 口虾蛄各组织的DNA相对含量直方图
Fig.4 Relative nuclear DNA contents in different tissues in mantis shrimp Oratosquilla oratoria
注:▲为n或2n峰所在位置
Note:▲,the position of n or 2n peak
表2 口虾蛄各组织的DNA含量
Tab.2 DNA contents in different tissues in mantis shrimp Oratosquilla oratoria

注:同列中标有不同小写字母者表示组间有显著性差异 (P<0.05),标有相同小写字母者表示组间无显著性差异 (P>0.05)。精巢细胞DNA测量值为二倍体细胞的测量值
Note:The means with different letters within the same column are significant differences at the 0.05 probability level,and the means with the same letters within the same column are not significant differences.DNA content of testis is measured in diploid cells
组织tissue DNA相对含量relative DNA content各组织与鸡血DNA相对含量比值ratio of the relative DNA content between each tissue and chicken DNA绝对含量/ (pg·2c-1)absolute DNA content基因组大小/ Mbp genome size血液blood 86.02±8.02c3.48 8.70 8508.60肌肉muscle 103.17±5.92a 4.17 10.43 10200.54肝胰腺hepatopancreas 91.51±5.39bc 3.70 9.25 9046.50卵巢ovary 92.75±15.27b 3.75 9.38 9173.64精巢diploid testis 101.74±7.05a 4.12 10.29 10063.62平均值average 95.04±11.02 3.84 9.61 9398.58
本研究发现,口虾蛄肝胰腺组织中染色体数目为88条,与十足目中的对虾科褐美对虾Farfantupenaeus aztecus、桃红美对虾Farfantupenaeus durarum、西方滨对虾Litopenaeus occidental、中国明对虾 Fennempenaeus orimtalis、墨吉明对虾 Fenneropenaeus merguiensis、长毛明对虾Fenneropenaeus penicillatus、斑节对虾Penaeus monodon、近缘新对虾Metapenaeus affinis的染色体数目相同,与对虾科其他物种染色体数目相差较小,而与十足目其他科物种染色体数目差别较大[4]。考虑到染色体数目及组成是亲缘关系远近及物种进化判断的重要依据,本研究结果可能暗示虾蛄科的口虾蛄与对虾科亲缘关系较近。Murofuehi等[23]认为,染色体数目少的核型较为原始,口虾蛄染色体数目比长臂虾科Palaemonidae、海鳌虾科Nephropidae和龙虾科Palinuridae染色体数目少,并且核型组成相似。因此,口虾蛄的进化地位较长臂虾科、海鳌虾科和龙虾科更为原始,有关具体的分类地位,尚待构建进化树进行进一步分析。
本研究中发现,口虾蛄有丝分裂中期,染色体核型大多呈棒状,在其他十足目动物中,上述染色体形态也比较普遍,如中国对虾 Fenneropenaeus chinensis[9-10]、日本沼虾 Macrobrachium nipponense[24]、克氏螯虾 Procambarus clarkii[25],这给染色体分析带来了一定的难度。一般认为,染色体数目出现的百分率>85%的众数为该物种的染色体数[8],考虑到大部分甲壳类染色体众数百分比低于85%,如刀额新对虾metapenaeus ensis[26]、鹰爪虾Trachypenaeus curvirostris[27]、罗氏沼虾Macrobrachium rosenbergii[28],因此,本试验中观察到的染色体众数仅占总数的63.46%,但仍在合理范围内,试验数据可信。Ahmed[29]的核型稳定性假说认为,具有越多中部着丝点的染色体组成,一般有较为稳定的染色体组型。口虾蛄具有31对中部着丝粒染色体,占染色体总数约70.45%,远高于其他多染色体组型的水产种类,如匙吻鲟Polyodon spathula中部着丝粒染色体占染色体总数约36.67%[30],泥鳅Misgurnus anguillicaudatus中部着丝粒染色体占染色体总数的16% ~25%[18],它们皆具有不同倍性以及不稳定的染色体组型,由此也间接证明,口虾蛄也具有稳定的染色体组型。此外,十足目中铠甲虾科Galatheidae和方蟹科Grapsidae中均发现了性染色体[31],但由于口虾蛄染色体数目较多,个体较小,因此,本研究中尚无法确定口虾蛄是否存在性染色体。
一般来说,基因组大小 (DNA含量或C值)代表一个物种的遗传信息,具有种的特征,同一生物个体各个组织的DNA含量是恒定的。但本研究中所检测的口虾蛄5种组织的DNA含量差异显著。不同组织DNA含量的差异性不仅存在于口虾蛄单一物种,也广泛存在于其他水产动物中[32]。本研究中评估到的口虾蛄基因组大小平均约为9398.58 Mbp,远大于其他十足目种类,如棕虾Penaeus aztecus基因组为2386.32 Mbp,凡纳滨对虾Litopenaeus vannamei基因组为 3423.00 Mbp[33]。Koyama等[34]研究发现,包括日本对虾在内的十足目基因组普遍较大,且存在大量重复片段。本试验中也发现,口虾蛄具有庞大的基因组,染色体中可能也存有大量的重复片段,这就可能造成细胞核DNA在复制过程中,出现扩增、重组或流失现象[35],导致不同组织的DNA含量存在显著差异。此外,有报道表明,不同组织细胞结构及功能的特异性也是造成细胞核DNA含量差异的原因[36-37]。
截至目前,已报道的十足目动物核型分析超过100种,但有关虾蛄科种类的染色体组型尚未见报道。本研究中首次报道了十足目、虾蛄科口虾蛄的核型组成,为从亲缘关系及物种进化角度研究虾蛄科在十足目中的系统分类地位提供了数据参考,也为口虾蛄的细胞遗传学和人工繁育研究提供了依据。同时,细胞核DNA含量的大小是生物特征参数之一,准确获得口虾蛄细胞核DNA含量也为开展该物种染色体组工程组学研究提供了基础资料。
参考文献:
[1] 谷德贤,洪星,刘海映.口虾蛄的繁殖行为[J].河北渔业,2008 (1):37-40.
[2] 蒋霞敏,王春琳,赵青松,等.虾蛄的人工养殖技术研究[J].东海海洋,1999,17(2):47-50.
[3] 张代臻,丁鸽,周婷婷,等.黄海海域口虾蛄种群的遗传多样性[J].动物学杂志,2013,48(2):232-240.
[4] 刘海映,刘连为,姜玉声,等.口虾蛄proPO基因全长 cDNA的克隆与组织表达[J].生态学报,2013,33(6):1713-1720.
[5] 乔之怡,董仕,王茜.黑龙江水系二、三倍体鲫鱼遗传组成的比较研究[J].动物科学与动物医学,2004,21(2):61-63.
[6] 陈咏霞,刘静,刘龙.中国鲷科鱼类骨骼系统比较及属种间分类地位探讨[J].水产学报,2014,38(9):1360-1374.
[7] Fanjul-Fernández M,Folgueras A R,Cabrera S,et al.Matrix metalloproteinases:evolution,gene regulation and functional analysis in mouse models[J].Biochimica et Biophysica Acta(BBA)-Molecular Cell Research,2010,1803(1):3-19.
[8] 王青,孔晓瑜,于珊珊,等.十足目染色体研究进展[J].海洋科学,2005,29(6):60-65.
[9] 相建海.中国对虾染色体的研究[J].海洋与湖沼,1988,19 (3):205-209.
[10] 戴继勋,张全启,包振民.中国对虾的核型研究[J].青岛海洋大学学报,1989,19(4):97-104.
[11] 高静,黄晓红,曾华嵩,等.中国6种经济鱼类的基因组大小测定[J].中国水产科学,2010,17(4):689-694.
[12] 朱泽远,杨杰,施用晖,等.SYBR GreenⅠ实时荧光定量PCR法测定中华绒螯蟹基因组大小[J].江苏农业科学,2007(5):164-166.
[13] 赵惠玲.孚尔根整体染色法在动物组织学研究中的应用[J].晋中学院学报,2005,22(3):56-58.
[14] 宋保平,诸葛斌,方慧英,等.产甘油假丝酵母(Candida glycerinogenes)染色体倍性分析[J].微生物学报,2011,51(3):326-331.
[15] 刘江惠,左连富,郭建文,等.流式细胞仪专用鸡红细胞的制备方法[J].河北医科大学学报,2008,29(6):873-875.
[16] 胡凡,王玲.大型流式细胞仪质量控制方法的探讨[J].南京医科大学学报:自然科学版,2005,25(3):211-213.
[17] Hedley D W,Friedlander M L,Taylor I W,et al.Method for analysis of cellular DNA content of paraffin-embedded pathological material using flow cytometry[J].Journal of Histochemistry& Cytochemistry,1983,31(11):1333-1335.
[18] 李雅娟,钱聪,印杰,等.不同倍性泥鳅杂交后代染色体数目组成的研究[J].大连海洋大学学报,2012,27(4):326-332.
[19] Levan A,Fredga K,Sandberg A A.Nomenclature for centromeric position on chromosomes[J].Hereditas,1964,52(2):201-220.
[20] 殷建,赵书涛,武晓东,等.流式细胞仪用鸡红细胞的制备[J].南方医科大学学报:自然科学版,2013,33(1):57-60.
[21] Lamatsch D K,Steinlein C,Schmid M,et al.Noninvasive determination of genome size and ploidy level in fishes by flow cytometry:detection of triploid Poecilia formosa[J].Cytometry,2000,39 (2):91-95.
[22] Gregory T R,Nicol J A,Tamm H,et al.Eukaryotic genome size databases[J].Nucleic Acids Research,2007,35(S1):D332-D338.
[23] Murofushi M,Deguchi Y.Karyotype evolution in decapoda,crustacea[C]//Hirano R,Hanyu I.Proceedings of the Second Asian Fisheries Forum.Tokyo:Asian Fisheries Society,1990:549-553.
[24] 邱高峰,堵南山,赖伟.日本沼虾染色体及其核型的研究[J].海洋与湖沼,1994,25(5):493-498.
[25] 朱越雄,曹广力.克氏螯虾染色体研究[J].水产养殖,1997 (3):12-13.
[26] 周岭华,张晓军,相建海.鹰爪虾染色体数目与核型的研究[J].海洋与湖沼,1999,30(3):250-254.
[27] 张晓军,周岭华,相建海.刀额新对虾染色体核型及细胞核DNA含量[J].海洋与湖沼,2002,33(3):225-231.
[28] 张天澍,王玉凤.罗氏沼虾的染色体研究[J].华中师范大学学报:自然科学版,2003,37(2):231-232.
[29] Ahmed M.Chromosome cytology of peiecypod molluses[J].Journal of Science Karachi,1976,4:77-94.
[30] 薛淑群,尹洪滨.匙吻鲟的细胞遗传学分析[J].水生生物学报,2009,33(6):1078-1082.
[31] Nakamura H K,Machii A,Wada K T,et al.A check list of Decapod chromosomes(Crustacea)[J].Bulletin of the National Research Institute of Aquaculture,1988,13:1-9.
[32] 杨坤,祝东梅,张新辉,等.麦穗鱼不同组织DNA含量及细胞周期的分析研究[J].中国农学通报,2012,28(32):113-118.
[33] Chow S,Dougherty W J,Sandifer P A.Meiotic chromosome complements and nuclear DNA contents of four species of shrimps of the genus Penaeus[J].Journal of Crustacean Biology,1990,10 (1):29-36.
[34] Koyama T,Asakawa S,Katagiri T,et al.Hyper-expansion of large DNA segments in the genome of kuruma shrimp,Marsupenaeus japonicus[J].BMC Genomics,2010,11(1):141.
[35] Price H J.Nuclear DNA content variation within angiosperm species[J].Evolutionary Trends in Plants,1988,2(1):53-60.
[36] Flavell R B.Sequence amplification,deletion and rearrangement:major sources of variation during species divergence[M]//Dover G A,Flavell R B.Genome Evolution.London:Academic Press,1982:301-323.
[37] Michaelson M J,Price H J,Johnston J S,et al.Variation of nuclear DNA content in Helianthus annuus(Asteraceae)[J].American Journal of Botany,1991,78(9):1238-1243.
Chromosome karyotype and nuclear DNA content of mantis shrimp Oratosquilla oratoria
Abstract:The karyotypic pattern was analyzed using colchicine injection method and DNA contents in different tissues were determined using DNA of the chicken blood cell as standard in mantis shrimp Oratosquilla oratoria by a flow cytometer to understand the karyotype,DNA content and genetics content.It was found that there were 88 chromosomes in diploid with 2n=88 in hepatopancreas specimen,with the karyotype formula of 2n=62m+12sm+ 14t,and total chromosome arm number(NF)of 162.The DNA content was significantly different in different tissues(P<0.05),with the order as:muscle>testicle>ovary>hepatopancreas>blood,the maximal value(10.43 pg/2c)in muscle and the minimal(8.70 pg/2c)in blood,with an average of 9.61 pg/2c.Moreover,the average value of the mantis shrimp genome size was estimated to be 9398.58 Mbp as 1 pg=978 Mbp.The findings make a first step towards broodstock conservation and complete aquaculture of the mantis shrimp in the future.
Key words:Oratosquilla oratoria;chromosome;karyotype;DNA content
中图分类号:S917;Q953
文献标志码:A
DOI:10.16535/j.cnki.dlhyxb.2016.01.001
文章编号:2095-1388(2016)01-0001-06
收稿日期:2015-05-04
基金项目:辽宁省科学技术计划项目 (2014203016);教育部留学回国人员科研启动基金资助项目 (2015-311);农业部北方海水增养殖重点实验室资助项目 (2014-MSENC-KF-14);大连海洋大学博士启动基金资助项目 (2014017349)