不同养殖时期刺参肠道内菌群结构的分析
王姣姣,李丹,王轶南,王俊杰,刘艳萍,常亚青
(大连海洋大学农业部北方海水增养殖重点实验室,辽宁大连116023)
摘要:为研究不同养殖时期刺参Apostichopus japonicus肠道内的细菌菌群结构,采用PCR-DGGE技术对DGGE凝胶中的主要条带进行切割、回收、克隆、测序。结果表明:3、4、5、6、9、10月,大连瓦房店海区养殖刺参肠道内细菌16S rDNA-V3可变区序列经PCR扩增及DGGE电泳后分别获得49、49、54、53、52、51个条带;3月与9月的菌群结构相似度最高,为67.9%,4月与10月次之,为63.8%;经条带序列分析得到43个主要条带对应的核酸序列,11个与未培养菌相似,32个与归属为7个细菌类群的细菌序列相似,包括α-变形菌纲、γ-变形菌纲、δ-变形菌纲、黄杆菌纲、蓝藻纲、绿弯菌门、疣微菌门;α-变形菌纲、γ-变形菌纲、黄杆菌纲是不同养殖时期刺参肠道内的主要细菌群落,其中γ-变形菌纲所占比例最高,达25.00%~35.71%,除3月和9月外,黄杆菌纲的相对含量均高于α-变形菌纲。研究表明,春秋两季刺参肠道内菌群结构呈现类似的演替变化规律,α-变形菌纲所占比例下降,而γ-变形菌纲与黄杆菌纲所占比例上升。
关键词:刺参;肠道内含物;变性梯度凝胶电泳;菌群结构
刺参Apostichopusjaponicus是中国重要的海水养殖种类,养殖区域分布于辽宁、山东和河北等地沿海,具有巨大的经济效益和社会效益[1]。动物肠道内的菌群是肠道的重要组成部分,水生动物的肠道菌群与宿主以及所处的水生环境存在相互依赖和相互制约的关系,对营养物质的消化、吸收等方面具有重要作用[2]。刺参营底栖生活并以沉积物为食,细菌在刺参的食物中占据较大的比例[3],刺参肠道内细菌群落组成直接受到养殖环境中菌群变化的影响[4]。目前,有关刺参肠道内细菌群落的研究报道已有不少,张文姬等[5]分析了刺参肠道内可培养微生物的种类,杨志平等[6]利用点种法筛选和鉴定了刺参潜在产酶益生菌。但是由于在自然界中85%~99%的细菌不可分离培养,传统细菌分离方法难以全面反映刺参肠道内细菌群落组成的情况[7],丁君等[8]利用PCR-DGGE技术分析了刺参肠道内的细菌种类。分子技术在刺参肠道菌群研究中的应用,为深入了解刺参肠道内的菌群组成提供了帮助,李彬等[4]运用传统细菌分离培养技术与分子生物学技术相结合的方法,对刺参肠道内的细菌菌群进行了分析。现已开展的研究主要集中在刺参肠道细菌种类的分析,而关于刺参肠道菌群季节性变化的研究却鲜有报道。有关水产养殖动物消化道菌群季节性变化的研究已引起重视[9-10]。本研究中,应用PCR-DGGE技术对不同养殖时期刺参肠道内的细菌群落进行了分析,探讨其演替规律,以期为深入了解刺参肠道菌群结构和刺参肠道微生态研究提供更为详尽的资料。
1 材料与方法
1.1 材料
试验用刺参采自辽宁省大连市瓦房店仙峪湾养殖场。
1.2 方法
1.2.1 样品采集 每次采集9头健康的2龄刺参(约100g),采样时间为2009年6月、2009年9月、2009年10月、2010年3月、2010年4月、2010年5月,置于冰盒中带回实验室,在无菌条件下解剖刺参,取肠道内含物混合后于-20℃下保存[11]。按采样时间将样品编为WI6、WI9、WI10、WI3、WI4、WI5号。
1.2.2 DNA提取 将刺参肠道内含物样品与Tris-HCl buffer、超纯水和Al2(SO4)3涡旋混匀后,用NaOH调pH至8,离心,弃上清,置于冰箱 (-20℃)中保存备用。利用溶菌酶法[12]进行粗提,再利用苯酚抽提法[13]进行提取,作为PCR反应的模板,于-20℃下保存备用。
1.2.3 细菌16S rDNA-V3可变区域的PCR扩增
采用特异性引物:F341(5′-CCTACGGGAGGCAGCAG)和R518(5′-ATTACCGCGGCTGCTGG),并在F341的5′端加入一段长为40 bp的富含GC的片段(CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGG)[14-15],对细菌16S rDNA基因V3可变区PCR后[12],产物于4℃下保存备用。
1.2.4 变性梯度凝胶电泳及分析 采用D-Code System(Bio-Rad)按下列条件进行:8%的聚丙烯酰胺,变性剂浓度为30% ~60%,电压为75 V,温度为60℃,电泳时间为12 h。用SYBR-Green I染色,经UVP凝胶系统成像后,采用凝胶分析软件Quantity One对DGGE图谱各样品条带的迁移率、灰度和数量进行数字化处理,然后进行多样性和相似性分析[16]。
1.2.5 DGGE条带的测序分析 在紫外灯下对DGGE凝胶中主要条带 (优势条带、共有条带等具有代表性的特殊条带)切割、回收并扩增。经纯化后的PCR产物连接到载体pMD19-T中,采用T载体通用引物M13进行菌落PCR,PCR产物经10g/L琼脂糖凝胶电泳检测是否为阳性克隆,将阳性克隆对应的白色转化子于含有Amp的LB液体培养基中过夜培养(37℃),进行质粒提取。取上述质粒5μL经10g/L琼脂糖凝胶电泳,检测是否含有质粒DNA。克隆过程中每个条带选择多个转化子,确保每个条带达到3~5个阳性克隆。将回收质粒交由宝生物工程(大连)有限公司测序。测序获得的序列在NCBI的GenBank数据库中进行Blast同源性比较(http://www.ncbi.nlm.nih.gov/Blast/)。
2 结果与分析

图1 刺参肠道菌群16S rDNA-V3 可变区PCR-DGGE 图谱
Fig. 1 PCR-DGGE profiles of the 16S rDNA V3 region of the bacterial flora in intestines of sea cucumber Apostichopusjaponicus
注:A为变性梯度凝胶电泳图谱;B为模式图;C为主要条带示意图
Note: A, denaturant gradient gel electrophoresis profiles; B, schematic chart; C, the schematic diagram of the main bands
2.1 不同养殖时期刺参肠道内菌群DGGE图谱的构建与分析
不同养殖时期刺参肠道内菌群的16SrDNAV3可变区片段的变性梯度凝胶电泳指纹图谱见图1-A,3月(WI3)、4月(WI4)、5月(WI5)、6月(WI6)、9月(WI9)、10月(WI10)的条带数目分别为49、49、54、53、52、51。运用Quantity One软件,以5月份为标准构建模式图谱 (图1-B),分析出3月、4月、5月、6月、9月和10月6个不同时期刺参肠道内菌群结构的相似性结果(表1)。从表1可见,WI3和WI9的菌群结构相似度最高,为 67.9%,WI4与 WI10次之,为63.8%,WI3与WI6的菌群结构相似度最小,为21.6%。刺参肠道各样品的多样性指数均在2.0左右,均匀度为0.20~0.24,差异不明显 (表2)。经UPGMA聚类分析 (图2)表明,WI3和WI9聚为一支,WI4和WI10聚为另一支。
表1 刺参肠道内菌群的相似性矩阵图
Tab 1 Similarity matrix of DGGE patterns in bacterial florain gut contents of sea cucumber Apostichopus japonicus%
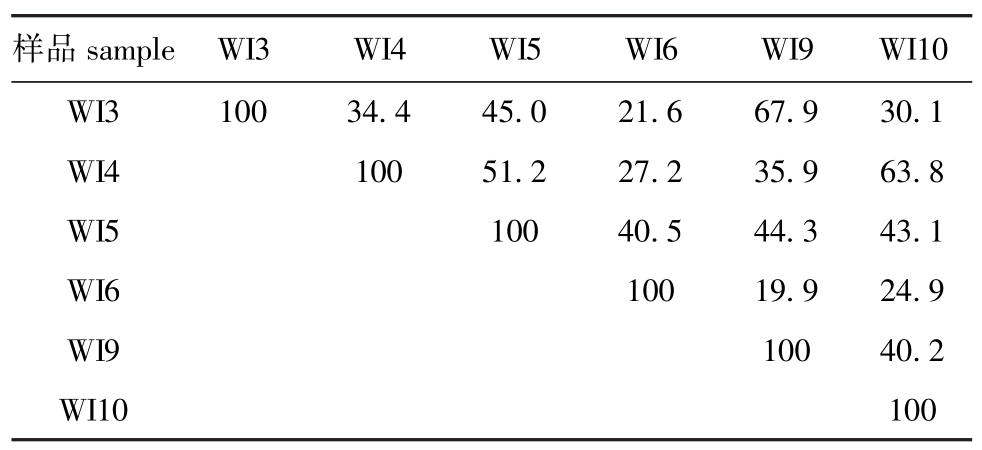
样品sample WI3 WI4 WI5 WI6 WI9 WI10 WI3 100 34.4 45.0 21.6 67.9 30.1 WI4 100 51.2 27.2 35.9 63.8 WI5 100 40.5 44.3 43.1 WI6 100 19.9 24.9 WI9 100 40.2 WI10100
表2 刺参肠道内菌群的多样性指数、均匀度和丰富度
Tab2 Shannon-Wienerindex(H),evenness(EH)and richness(S)ofthebacterialfloraingutcontents ofseacucumberApostichopusjaponicus
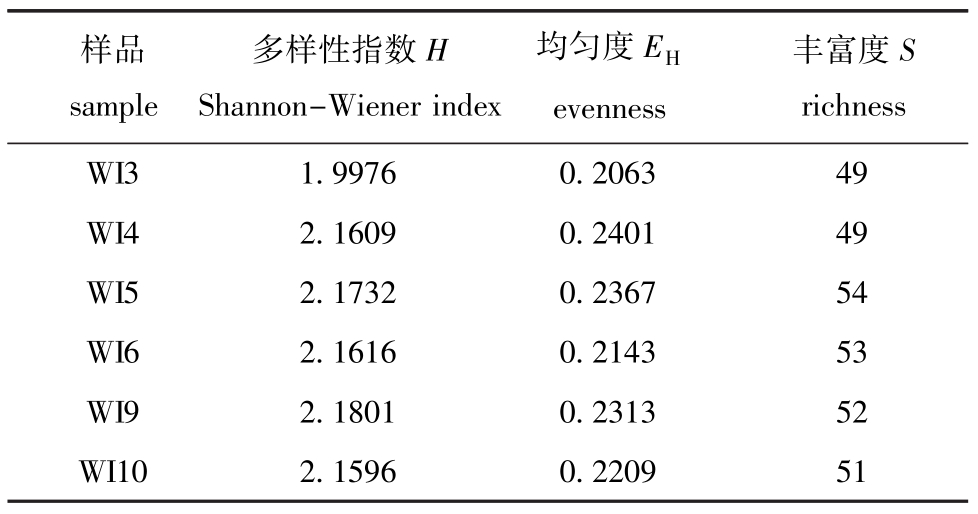
样品sample多样性指数H Shannon-Wienerindex均匀度EHevenness丰富度S richness WI3 1.9976 0.2063 49 WI4 2.1609 0.2401 49 WI5 2.1732 0.2367 54 WI6 2.1616 0.2143 53 WI9 2.1801 0.2313 52 WI10 2.1596 0.2209 51
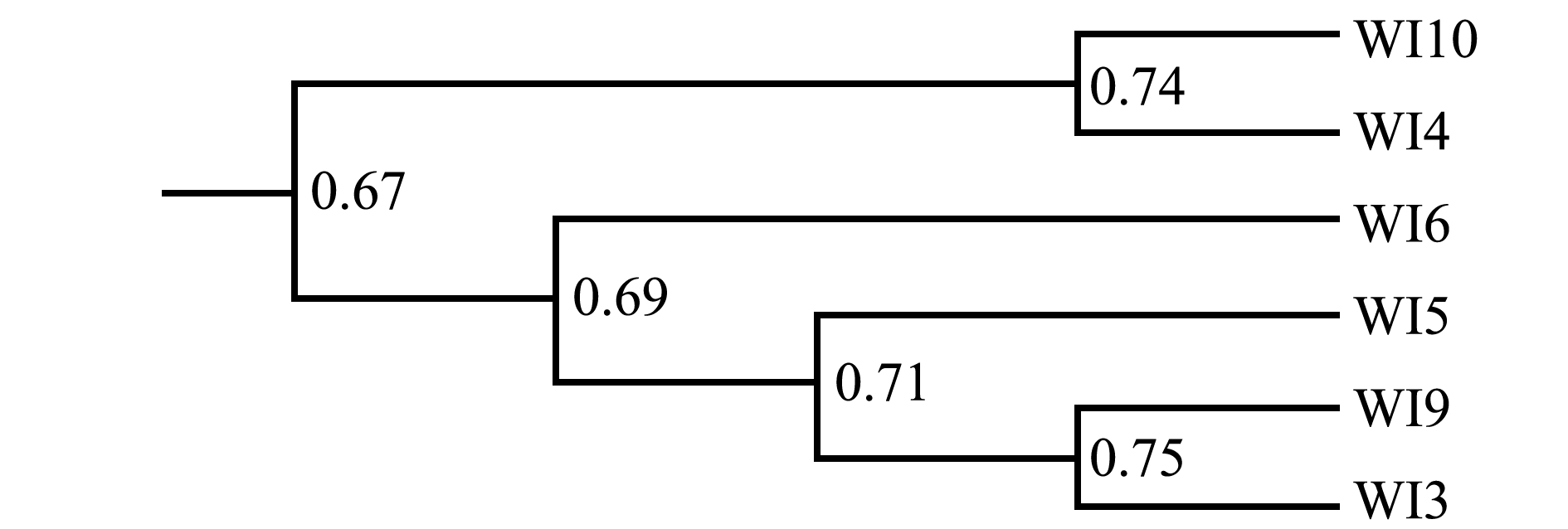
图2 PCR-DGGE图谱聚类分析
Fig.2 ClusteranalysisofPCR-DGGE
2.2 刺参肠道内菌群的结构及变化
对不同位置刺参肠道内含物图谱条带进行切胶回收,由主要条带示意图 (图1-C)可知,3月、4月、5月、6月、9月、10月样品的可切胶回收条带数分别为18、17、20、14、16、14条。序列通过NCBI中BLAST进行同源性比对分析 (表3), 11个条带与未培养菌同源性达到98%以上,其余32个条带明确分类的菌种与7个细菌类群相似,其中 7条序列 (AgcW20、AgcW23、AgcW28、AgcW31、AgcW34、AgcW37、AgcW42)与 α-变形菌纲Alpha proteobacteria相似,18条序列 (Agc W02、 AgcW03、 AgcW04、 AgcW08、 AgcW09、AgcW10、AgcW12、AgcW13、AgcW14、AgcW16、AgcW19、AgcW21、AgcW30、AgcW32、AgcW33、AgcW35、AgcW36、AgcW41)与γ-变形菌纲Gamma proteobacteria相似,1条序列 (AgcW17)与δ-变形菌纲 Delta proteobacteria相似,3条序列(AgcW15、AgcW26、AgcW39)与黄杆菌纲 Flavobacteria相似,1条序列 (AgcW01)与蓝藻纲Cyanobacteria相似,1条序列 (AgcW05)与绿弯菌门Chloroflexi相似,1条序列 (AgcW06)与疣微菌门Verrucomicrobia相似。各时期存在一些共有条带,如AgcW05、AgcW25、AgcW26、AgcW27、AgcW29、AgcW30、AgcW39、AgcW40、AgcW42、AgcW43。其 中,AgcW25、 AgcW27、 AgcW29、AgcW40、AgcW43号条带为未培养细菌。
对克隆得到的序列进行统计分析,得到不同月份刺参肠道内细菌的群落组成 (图3)。不同月份刺参肠道内细菌群落均包含α-变形菌纲、γ-变形菌纲、黄杆菌纲,但相对含量各不相同。在3月份时,γ-变形菌纲 (27.78%) >α-变形菌纲(22.22%)>黄杆菌纲 (11.11%);在4月份时, γ-变形菌纲 (29.41%)>黄杆菌纲 (11.76%)= α-变形菌纲 (11.76%);在5月份时,γ-变形菌纲 (30.00%)>α-变形菌纲 (10.00%)=黄杆菌纲 (10.00%);在 6月份时,γ-变形菌纲(35.71%)>黄杆菌纲 (21.42%)>α-变形菌纲(7.14%);在9月份时,γ-变形菌纲(25.00%)>α-变形菌纲 (18.75%)>黄杆菌纲 (12.50%);在10月份时,γ-变形菌纲 (35.71%)>黄杆菌纲(14.28%)=α-变形菌纲 (14.28%)。不同月份刺参肠道内的优势菌群均为γ-变形菌纲。
3 讨论
刺参营底栖生活,摄食无选择性[17],多以沉积物为食,摄入大量的底泥、有机物质和细菌,肠道的菌群结构与底泥较为相近[18],并且刺参的肠道中存在定植菌群辅助消化[19]。与此一致,不同
时期刺参肠道内细菌16SrDNA-V3序列DGGE图谱包含50个左右的可见条带,表明刺参肠道内均存在丰富菌群。高菲等[20]利用PCR-DGGE技术研究了刺参肠道内的细菌群落组成,共得到13条序列,将其归类于α-变形菌纲、γ-变形菌纲、δ-变形菌纲、拟杆菌纲和柔膜菌纲等5大类群,γ-变形菌纲为其优势菌群。李彬等[4]所认定的刺参肠道内8株优势菌中,有7株菌属于变形菌门的γ-变形菌纲。Huaide等[21]研究显示,中国对虾Fen+ neropenaeus chinensis肠道内的优势菌也属于γ-变形菌纲。本研究中,在不同养殖时期共获得43个主要的DGGE条带序列,获得的条带数量较多,可较全面地反映刺参肠道内的菌群结构。除未培养菌外,获得的条带序列可归属为α-变形菌纲、γ-变形菌纲、δ-变形菌纲、黄杆菌纲、蓝藻纲、绿弯菌门、疣微菌门7个细菌类群,其中优势菌群为γ-变形菌纲,与以上研究学者得出的结论相同。然而,本研究中获得了与黄杆菌纲、蓝藻纲、绿弯菌门、疣微菌门等菌种相似的条带,与高菲等[20]的研究存在差异,这可能与样品来源不同有关。
表3 刺参肠道内细菌PCR-DGGE代表条带的测序结果
Tab3 SequencingresultsofPCR-DGGEbandsingutcontentsofseacucumberApostichopusjaponicus
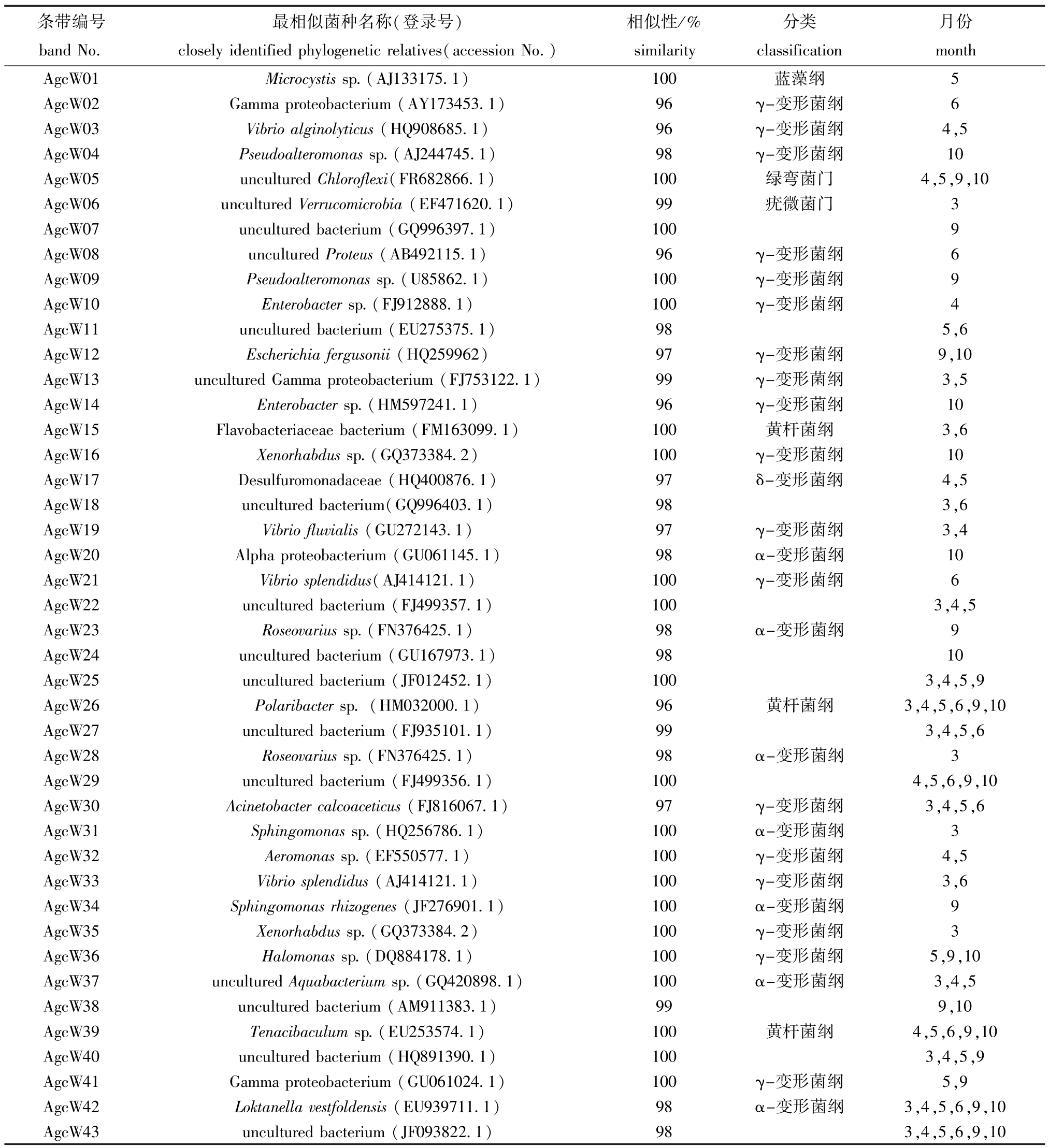
条带编号bandNo.月份month AgcW01 Microcystissp.(AJ133175.1) 100 蓝藻纲最相似菌种名称(登录号) closelyidentifiedphylogeneticrelatives(accessionNo.)相似性/% similarity分类classification 5 AgcW02 Gammaproteobacterium(AY173453.1) 96 γ-变形菌纲 6 AgcW03 Vibrioalginolyticus(HQ908685.1) 96 γ-变形菌纲 4,5 AgcW04 Pseudoalteromonassp.(AJ244745.1) 98 γ-变形菌纲 10 AgcW05 unculturedChloroflexi(FR682866.1) 100 绿弯菌门 4,5,9,10 AgcW06 unculturedVerrucomicrobia(EF471620.1) 99 疣微菌门 3 AgcW07 unculturedbacterium(GQ996397.1) 100 9 AgcW08 unculturedProteus(AB492115.1) 96 γ-变形菌纲 6 AgcW09 Pseudoalteromonassp.(U85862.1) 100 γ-变形菌纲 9 AgcW10 Enterobactersp.(FJ912888.1) 100 γ-变形菌纲 4 AgcW11 unculturedbacterium(EU275375.1) 98 5,6 AgcW12 Escherichiafergusonii(HQ259962) 97 γ-变形菌纲 9,10 AgcW13 unculturedGammaproteobacterium(FJ753122.1) 99 γ-变形菌纲 3,5 AgcW14 Enterobactersp.(HM597241.1) 96 γ-变形菌纲 10 AgcW15 Flavobacteriaceaebacterium(FM163099.1) 100 黄杆菌纲 3,6 AgcW16 Xenorhabdussp.(GQ373384.2) 100 γ-变形菌纲 10 AgcW17 Desulfuromonadaceae(HQ400876.1) 97 δ-变形菌纲 4,5 AgcW18 unculturedbacterium(GQ996403.1) 98 3,6 AgcW19 Vibriofluvialis(GU272143.1) 97 γ-变形菌纲 3,4 AgcW20 Alphaproteobacterium(GU061145.1) 98 α-变形菌纲 10 AgcW21 Vibriosplendidus(AJ414121.1) 100 γ-变形菌纲 6 AgcW22 unculturedbacterium(FJ499357.1) 100 3,4,5 AgcW23 Roseovariussp.(FN376425.1) 98 α-变形菌纲 9 AgcW24 unculturedbacterium(GU167973.1) 98 10 AgcW25 unculturedbacterium(JF012452.1) 100 3,4,5,9 AgcW26 Polaribactersp.(HM032000.1) 96 黄杆菌纲 3,4,5,6,9,10 AgcW27 unculturedbacterium(FJ935101.1) 99 3,4,5,6 AgcW28 Roseovariussp.(FN376425.1) 98 α-变形菌纲 3 AgcW29 unculturedbacterium(FJ499356.1) 100 4,5,6,9,10 AgcW30 Acinetobactercalcoaceticus(FJ816067.1) 97 γ-变形菌纲 3,4,5,6 AgcW31 Sphingomonassp.(HQ256786.1) 100 α-变形菌纲 3 AgcW32 Aeromonassp.(EF550577.1) 100 γ-变形菌纲 4,5 AgcW33 Vibriosplendidus(AJ414121.1) 100 γ-变形菌纲 3,6 AgcW34 Sphingomonasrhizogenes(JF276901.1) 100 α-变形菌纲 9 AgcW35 Xenorhabdussp.(GQ373384.2) 100 γ-变形菌纲 3 AgcW36 Halomonassp.(DQ884178.1) 100 γ-变形菌纲 5,9,10 AgcW37 unculturedAquabacteriumsp.(GQ420898.1) 100 α-变形菌纲 3,4,5 AgcW38 unculturedbacterium(AM911383.1) 99 9,10 AgcW39 Tenacibaculumsp.(EU253574.1) 100 黄杆菌纲 4,5,6,9,10 AgcW40 unculturedbacterium(HQ891390.1) 100 3,4,5,9 AgcW41 Gammaproteobacterium(GU061024.1) 100 γ-变形菌纲 5,9 AgcW42 Loktanellavestfoldensis(EU939711.1) 98 α-变形菌纲 3,4,5,6,9,10 AgcW43 unculturedbacterium(JF093822.1) 98 3,4,5,6,9,10
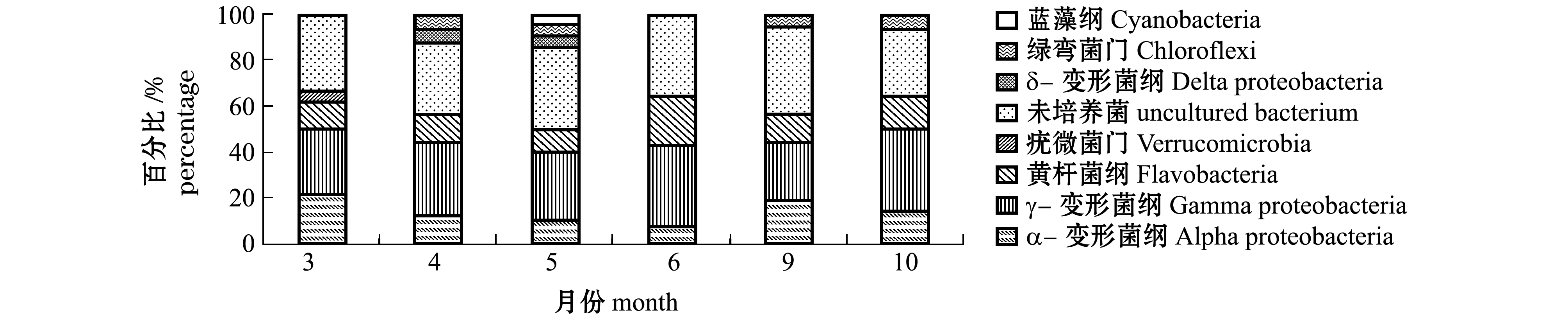
图3 不同养殖月份刺参肠道内细菌群落组成
Fig.3 Bacterial flora in gut contents of sea cucumber Apostichopus japonicus in different culturemonths
受水温影响,刺参的摄食和生理活动在周年养殖期内存在2个升降周期[22]。冬季冰封期,水温较低,刺参摄食量减少,处于半休眠状态;春季随水温回升,刺参摄食量增加进入快速生长期;进入6月下旬,海水温度上升,刺参活动减弱,摄食量又下降,当水温超过21℃时刺参则进入夏眠,夏眠期间,刺参的肠道衰竭、停止摄食、基础代谢下降,黄、渤海区刺参的夏眠期大约可持续100d左右[3];秋季水温下降后,刺参结束夏眠,生长出新的肠道并重新运动和觅食,进入第二个生长期。本研究中,采集养殖刺参的肠道内容物时间段为3月—6月和9月—10月,基本涵盖了刺参两个摄食旺盛的时期。本研究中结果显示,不同时期刺参肠道内菌群中均存在α-变形菌纲、γ-变形菌纲和黄杆菌纲,但不同时期其相对含量不同,除3月和9月外,黄杆菌纲的相对含量均高于α-变形菌纲。3月至4月,α-变形菌纲所占比例急剧下降,由22.22%下降至11.76%,之后两个月均未超过10%。与此同时,γ-变形菌纲所占比例逐渐上升,由27.78%(3月)上升至35.71%(6月);黄杆菌纲所占比例前期较稳定,6月明显提高。表明3月至6月刺参肠道内菌群结构变化较大,且呈现出明显的演替变化规律,即α-变形菌纲下降,而γ-变形菌纲与黄杆菌纲比例上升。9月至10月期间菌群结构发生了与此相似的变化趋势,其中γ-变形菌纲所占比例由25.00%升至35.71%。现有研究发现,栉孔扇贝[9]、草鱼[10]等消化道内细菌的群落组成相对稳定,细菌多样性季节变化并不明显。本研究中,得出的结果与之不同。推测刺参肠道内菌群结构的季节性变化可能与刺参的摄食和生理活动特点有关,在刺参由摄食停止或较弱状态发展至摄食旺盛状态的过程中,刺参肠道内菌群结构呈规律性演替,并且在两个摄食旺盛生长快速的时期都表现出类似的过程。春秋两季刺参肠道内菌群结构经历类似的变化,聚类分析显示,3月和9月的菌群结构相似,而4月和10月的菌群结构相似,结果也与此一致。此外,刺参肠道细菌菌群平衡破坏易引发疾病[23],肠道菌群结构的这种变化也可能是春秋季节疾病多发的原因之一。
刺参肠道内存在一定量的潜在致病菌,其与肠道内的其他细菌形成平衡的菌群系统,仅在特定条件下大量增殖并使刺参患病。李彬等[4]研究发现,正常刺参肠道中存在可引起刺参腐皮综合征的灿烂弧菌,而刺参肠道内还存在对致病灿烂弧菌具有拮抗作用的微生物[24]。本研究中,3月和6月在刺参肠道中亦发现灿烂弧菌,除3月外,不同时期肠道内均存在Tenacibaculum sp.,且为优势条带。根据张莹等[25]的发现,腐皮综合征刺参病灶处存在优势菌Tenacibaculum sp.,推测其可能亦为刺参肠道内的潜在致病菌。
参考文献:
[1]王轶南,穆晓虎,封妮莎,等.仿刺参体腔液中酚氧化酶活性的分析[J].大连海洋大学学报,2013,28(4):319-322.
[2]冷晓飞,姜海峰,刘小林,等.皱纹盘鲍肠道菌群组成及PCRDGGE指纹图谱分析[J].海洋科学,2013,37(5):10-14.
[3]常亚青,丁君,宋坚,等.海参、海胆生物学研究与养殖[M].北京:海洋出版社,2004:113-192.
[4]李彬,荣小军,廖梅杰,等.刺参肠道与养殖池塘环境中异养细菌和弧菌数量周年变化[J].海洋科学,2012,36(4):63-67.
[5]张文姬,侯红漫,张公亮,等.仿刺参肠道可培养微生物多样性研究[J].食品工业科技,2011(9):149-151.
[6]杨志平,孙飞雪,刘志明,等.刺参潜在产酶益生菌的筛选和鉴定[J].大连海洋大学学报,2013,28(1):17-20.
[7] Amann R I,Ludwig W,Schlefier K H.Phylogenetic identification and in situ detection of individualmicrobial cells without cultivation[J].Microbio1 Rev,1995,59(1):143-169.
[8] 丁君,李娇,王姮,等.利用16S rDNA方法检测刺参消化道细菌种类[J].海洋环境科学,2010(2):250-254.
[9] 杨彩霞,李赟,张婧宇,等.栉孔扇贝消化盲囊细菌群落组成的季节变化分析[J].水产学报,2012,36(10):1579-1584.
[10] 倪加加,余育和.不同月份养殖草鱼幼鱼消化道微生物群落动态变化研究[J].水产学报,2013,37(10):1558-1563.
[11] 王轶南,王俊杰,王姣姣,等.用16S rDNA克隆文库法分析患病刺参幼苗的菌群结构[J].大连海洋大学学报,2014,29 (3):205-211.
[12] 何夙旭,周志刚,姚斌,等.3种DNA提取方法对养殖池塘不同生境菌群PCR-DGGE分析的影响[J].中国农业科技导报,2009(1):73-79.
[13] 李丹,潘迎捷,赵勇,等.3种提取河豚鱼肠道微生物总DNA的方法比较[J].食品科学,2012(9):154-157.
[14] Li Z Y,He L M,Mao X L.Cultivable bacterial community from South China sea sponge as revealed by DGGE fingerprinting and 16S rDNA phylogenetic analysis[J].Curr Microbiol,2007,55: 465-472.
[15] Holben W E,Feris K P,Kettunen A,et al.GC fractionation enhancesmicrobial community diversity assessment and detection of minority populations of bacteria by denaturing gradient gel electrophoresis[J].Applied and Environmental Microbiology,2004, 70(4):2263-2270.
[16] 刘新春,吴成强,张昱,等.PCR-DGGE法用于活性污泥系统中微生物群落结构变化的解析[J].生态学报,2005,25(4): 842-847.
[17] 于东祥,孙慧玲,陈四清,等.海参健康养殖技术[M].北京:海洋出版社,2005:152-153.
[18] 王轶南,朱世伟,常亚青.刺参肠道及养殖池塘菌群组成的PCR-DGGE指纹图谱分析[J].渔业科学进展,2010,31(3): 119-121.
[19] Clifford C,Walsh J,Reidy N,etal.Digestive enzymesand subcellular localization of disaccharidases in somemarine sciences[J]. Comp Biochem Physiol,1982,71B:105-110.
[20] 高菲,孙慧玲,许强,等.刺参消化道内含物细菌群落组成的PCR-DGGE分析[J].中国水产科学,2010,17(4):671-680.
[21] Huaide L,LeiW,Mei L,et al.The intestinalmicrobial diversity in Chinese shrimp(Fenneropenaeus chinensis)as determined by PCR-DGGE and clone library analyses[J].Aquaculture,2011, 317(1):32-36.
[22] 周玮,徐浩然,林长松,等.池塘养殖仿刺参生理状态周年变化研究[J].大连水产学院学报,2009,24(S1):30-34.
[23] 张春云,王印庚,荣小军.养殖刺参腐皮综合征病原菌的分离与鉴定[J].水产学报,2006,30(1):118-122.
[24] 李虹宇,张公亮,侯红漫,等.仿刺参相关微生物对致病灿烂弧菌的拮抗及机理研究[J].食品工业,2012(9):117-120.
[25] 张莹,姜国良,刘云,等.仿刺参“腐皮综合症”病灶处优势菌的分离鉴定及AHLs信号分子的检测[J].微生学通报,2009, 36(11):1664-1669.
Bacterial flora in gut contents of sea cucumber Apostichopus japonicus at different culture periods
WANG Jiao-jiao,LIDan,WANG Yi-nan,WANG Jun-jie,LIU Yan-ping,CHANG Ya-qing
(Key Laboratory of Mariculture&Stock Enhancement in North China's Sea,Ministry of Agriculture,Dalian Ocean University,Dalian 116023,China)
Abstract:The bacterial flora in gut contentswas studied in cultured sea cucumber Apostichopus japonicus collected on June,September and October of2009,and March,April and May of2010 by PCR-DGGE in which variable regional fragments of 16S rDNA-V3 were amplified by specific primers of bacteria.There were 49,49,54,53,52 and 51 bands in the samples collected on March,April,May,June,September and October,respectively,in the DGGE gelswhich were cut,recovered,cloned,and sequenced.Themaximal similarity(67.9%)in bacterial flora was observed in the samples collected on March and September,followed by the samples(63.8%)collected on between April and October.The sequencing analysis revealed that11 nucleic acid sequenceswere similar to uncultured bacteria,and 32 sequenceswere similar to the sequences of 7 groups of bacterial includingα-proteobacteria,γ-proteobacteria,δ-proteobacteria,Flavobacteria,Cyanobacteria,Chloroflexi and Verrucomicrobia in the 43 major bands.The dominantgroups in the bacterial flora in different culture periodswere found to beα-proteobacteria,γ-proteobacteria and Flavobacteria.It was found thatγ-proteobacteria had the maximal proportion,varying from 25.00%to 35.71%.There was higher relative count in Flavobacterium thanα-proteobacteria in most samples except that on March and September.The findings indicated that similar flora succession was observed in spring and autumn,the proportion ofα-proteobacteria being declined while that ofγ-proteobacteria and Flavobacterium elevated.
Keywords:Apostichopus japonicus;gut content;PCR-DGGE;flora
中图分类号:Q959;S94
文献标志码:A
DOI:10.16535/j.cnki.dlhyxb.2015.04.001
文章编号:2095-1388(2015)04-0345-06
收稿日期:2014-09-12
基金项目:辽宁省科技攻关重大项目 (2015203003);国家海洋局海洋公益项目 (201405003);国家 “863”计划项目 (2012AA10A412)
作者简介:王姣姣 (1989—),女,硕士研究生。E-mail:wangjj.0311@163.com
通信作者:常亚青 (1967—),男,博士,教授。E-mail:yqchang@dlou.edu.cn





