泥鳅MSAP技术反应体系的建立及优化
周贺,徐雯,李雅娟,秦艳杰,李霞,麻天宇,肖逸啸,马海艳
(大连海洋大学 辽宁省海洋生物资源恢复与生境修复重点实验室, 辽宁 大连 116023)
摘要:为获得不同倍性泥鳅基因组DNA甲基化水平及模式,以泥鳅Misgurnusanguillicaudatus为研究对象,利用正交试验建立了甲基化敏感扩增多态性(MSAP)方法的反应体系,并利用该方法对二倍体泥鳅鳍基因组DNA进行了MSAP分析。结果表明:最佳双酶切反应体系为800 ng的泥鳅基因组DNA,用EcoR I、HpaII和MspI各10 U,在37 ℃ 下反应8 h后即可酶切;最佳预扩增反应体系为模板4 μL、预扩增引物0.8 μL、 0.2 mmol/L dNTPs、1.5 mmol/L Mg2+和Taq1 U;最佳选择性扩增反应体系为预扩增产物稀释20倍的模板2 μL、 引物1.5 μL、 0.375 mmol/L dNTPs、1.5 mmol/L Mg2+和Taq1 U;二倍体泥鳅全甲基化率分别为24.1%、20.8%、22.3%,半甲基化率分别41.4%、20.8%、33.3%,总甲基化率分别为65.5%、41.6%、55.6%。研究表明,该反应体系稳定、可靠、重复性好,为MSAP技术在多倍体泥鳅相关研究中的应用奠定了基础。
关键词:泥鳅;DNA甲基化;甲基化敏感扩增多态性(MSAP);反应体系
表观遗传(Epigenetics)是指在基因的DNA序列未发生改变的情况下,基因功能发生可遗传的变化,并最终导致表型的变化[1]。DNA甲基化(DNA methylation)作为表观遗传学研究的重要内容,是目前表观遗传学研究的热点之一。甲基化敏感扩增多态性(methylation sensitive amplification polymorphism,MSAP)技术,是一种简单、有效、可靠且能在全基因组水平检测胞嘧啶甲基化模式及其变化的方法[2],在生物的DNA甲基化研究中应用广泛。
多倍化在许多植物和动物中普遍存在[3]。泥鳅Misgurnusanguillicaudatus存在天然多倍体现象。研究发现,日本的泥鳅除了存在正常有性生殖二倍体外,还存在无性生殖二倍体和杂种生殖三倍体,但未发现天然四倍体[4-7]。中国的泥鳅存在二倍体、三倍体和四倍体3种不同倍性[8-11]。近年来,本研究室人员对中国特有的天然四倍体泥鳅的分布[11]、体细胞染色体核型分析[12]、染色体带型与FISH研究[13]、减数分裂染色体行为[14]、泥鳅细胞培养及染色体标本制备[15]等方面进行了系统地研究。从细胞及分子遗传学角度证实了天然四倍体泥鳅是含有四套染色体组的同源四倍体,因此,泥鳅是探究同源多倍化机制的理想试材。已有研究证明,自然或人工合成的异源多倍体在其发生与形成后的稳定过程中都常常伴随着快速且有时在进化上保守的表观遗传变化,包括DNA甲基化、组蛋白修饰、RNA干扰等[16]。而且,表观遗传修饰在多倍体形成后的二倍化过程中也发挥了重要作用[17]。相对于动、植物异源多倍化现象,对同源多倍化过程中遗传及表观遗传变化特征的研究较少。本研究中,作者以泥鳅为研究对象,利用正交试验建立MSAP方法的最佳反应体系,并利用该方法对二倍体泥鳅基因组DNA进行MSAP分析,旨在为探究同源四倍体泥鳅多倍化过程中基于DNA甲基化的表观遗传变化特征,为鱼类同源多倍化机制的研究提供新线索。
1材料与方法
1.1材料
试验用二倍体泥鳅购自大连市农贸市场,暂养在辽宁省海洋生物资源恢复与生境修复重点实验室水族箱中,暂养温度为(22±1)℃。二倍体泥鳅的倍性经血红细胞核测量及流式细胞仪检测确定。
试验用主要试剂有限制性内切酶EcoR I、HpaII、MspI(Fermentas),TaqDNA聚合酶(TaKaRa公司),氯仿-异戊醇(体积比为24∶1)、EDTA、Tris、尿素、TEMED[生工生物工程(上海)股份有限公司], 引物、接头[生工生物工程(上海)股份有限公司]。
1.2方法
1.2.1 基因组DNA的提取 采用尿素法[18]提取基因组DNA,取适量鱼鳍经尿素缓冲液消化后,使用氯仿-异戊醇等抽提2次后,经100% (体积分数,下同) 无水乙醇沉淀,再用70%无水乙醇清洗后获得DNA。
1.2.2 DNA浓度与质量的检测 用Eppendorf D30核酸蛋白测定仪测定基因组DNA的浓度并计算260 nm与280 nm 处OD值的比值,以确定DNA的纯度;用8 g/L琼脂糖凝胶电泳检测基因组DNA(180 V,电泳20 min)的质量。
1.2.3 MSAP双酶切体系的优化 选取EcoR I/HpaII和EcoR I/MspI 两组内切酶分别对基因组DNA进行酶切。20 μL体系中包括:800 ng基因组DNA,10×buffer Tango缓冲液4 μL,EcoR I、HpaII和MspI各10 U,混匀后用PCR仪温育酶切,将酶切时间分别设定为4、6、8 h,完成后用10 g/L琼脂糖凝胶检测酶切产物的质量。
1.2.4 MSAP连接体系 连接体系为30 μL,将接头等加入酶切液中,于22 ℃下连接2 h,接头在加入酶切液之前先合为双链。双酶切剩余量为17 μL,加入5×T4 buffer 6 μL,混匀后放入PCR仪中进行连接,引物及接头设计参照曹哲明等[19]的方法,本试验中研究所用的接头、引物详见表1。
表1接头和引物序列
Tab.1SequencesofadaptorsandprimersusedforMSAP
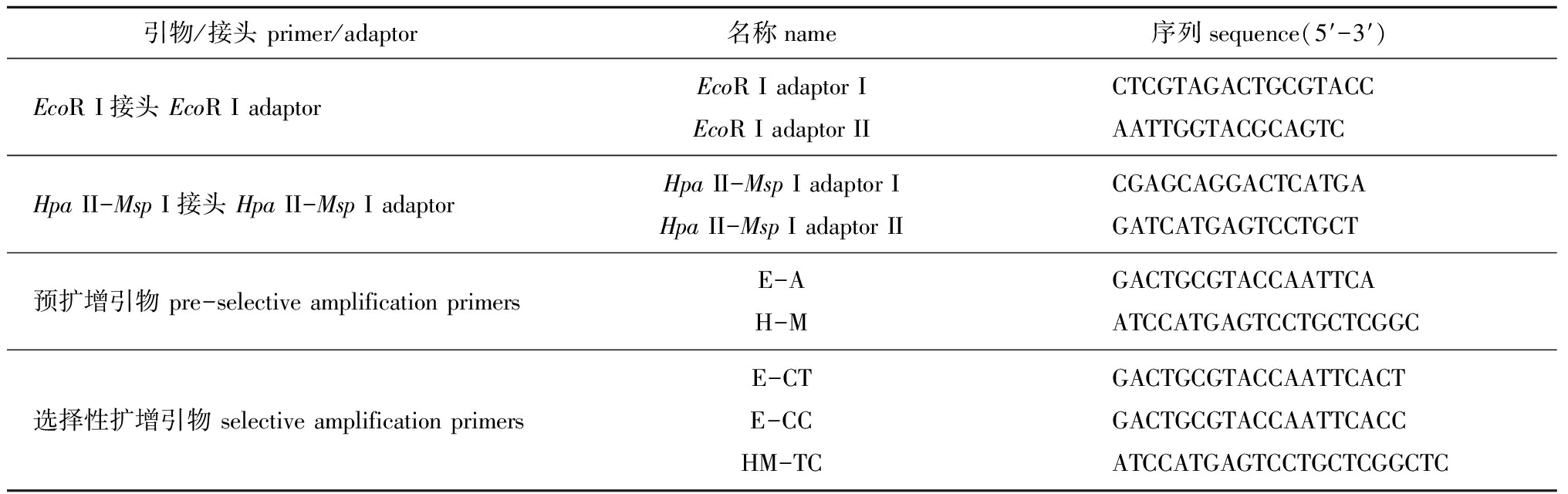
引物/接头primer/adaptor名称name序列sequence(5′-3′) EcoRI接头EcoRIadaptorEcoRIadaptorIEcoRIadaptorIICTCGTAGACTGCGTACCAATTGGTACGCAGTCHpaII-MspI接头HpaII-MspIadaptorHpaII-MspIadaptorIHpaII-MspIadaptorIICGAGCAGGACTCATGAGATCATGAGTCCTGCT预扩增引物pre-selectiveamplificationprimersE-AH-MGACTGCGTACCAATTCAATCCATGAGTCCTGCTCGGC选择性扩增引物selectiveamplificationprimersE-CTE-CCHM-TCGACTGCGTACCAATTCACTGACTGCGTACCAATTCACCATCCATGAGTCCTGCTCGGCTC
1.2.5 MSAP预扩增体系 采用E-A和H-M引物组合(表1),反应总体积为20 μL。为了确定MSAP预扩增体系中4个因素(预扩模板、引物、dNTPs、Mg2+浓度)的最佳反应浓度,选用L9(34)正交表[20]进行4因素3水平的正交试验设计(表2)。引物浓度及PCR反应程序参照秦艳杰[21]的方法。PCR反应程序:94 ℃下预变性2 min;94 ℃ 下变性30 s, 56 ℃下退火40 s, 72 ℃下延伸 60 s, 共进行30个循环;最后在72 ℃下再延伸 5 min,4 ℃下保存。用琼脂糖凝胶电泳检测扩增产物的质量,将预扩增产物稀释20倍后用作选择性扩增模板,剩余产物于冰箱(-20 ℃)中保存备用。试验重复2次。
1.2.6 选择性扩增体系 选择性扩增选取E-CC、HM-TC引物组合(表1),反应总体积为20 μL。为了确定MSAP选择性扩增体系中4个因素(预扩模板、引物、dNTPs、Mg2+浓度)的最佳反应浓度,选用L9(34)正交表[20]进行4因素3水平的正交试验设计(表3)。引物浓度、模板稀释倍数和PCR反应程序参照秦艳杰[21]的方法。PCR反应程序:94 ℃下预变性2 min;94 ℃下循环变性40 s, 65~56 ℃(每循环下降0.7 ℃) 下退火40 s,在72 ℃下延伸60 s,共进行12个循环;94 ℃下循环变性40 s,56 ℃下退火40 s,72 ℃下延伸60 s,共进行30个循环;最后在72 ℃下再延伸5 min,4 ℃下保存。
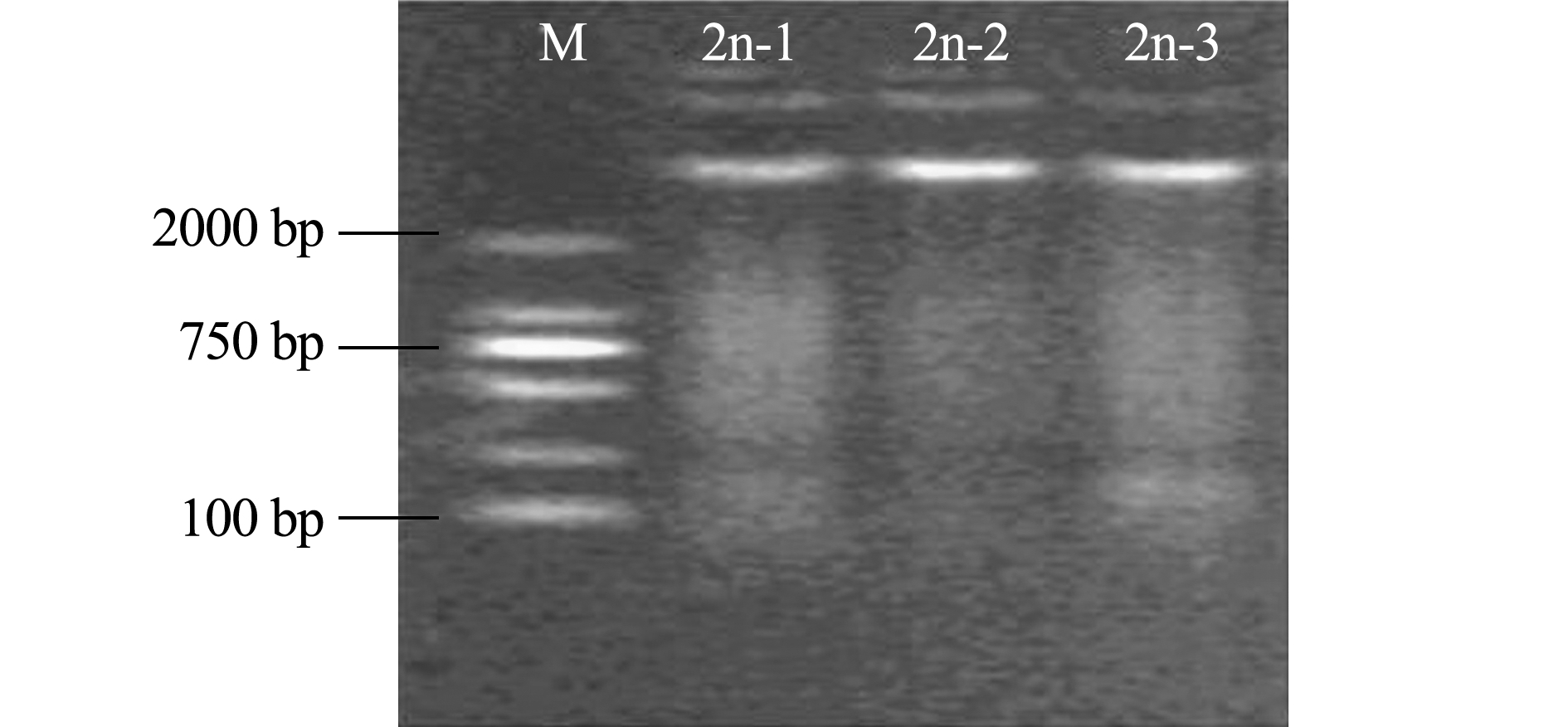
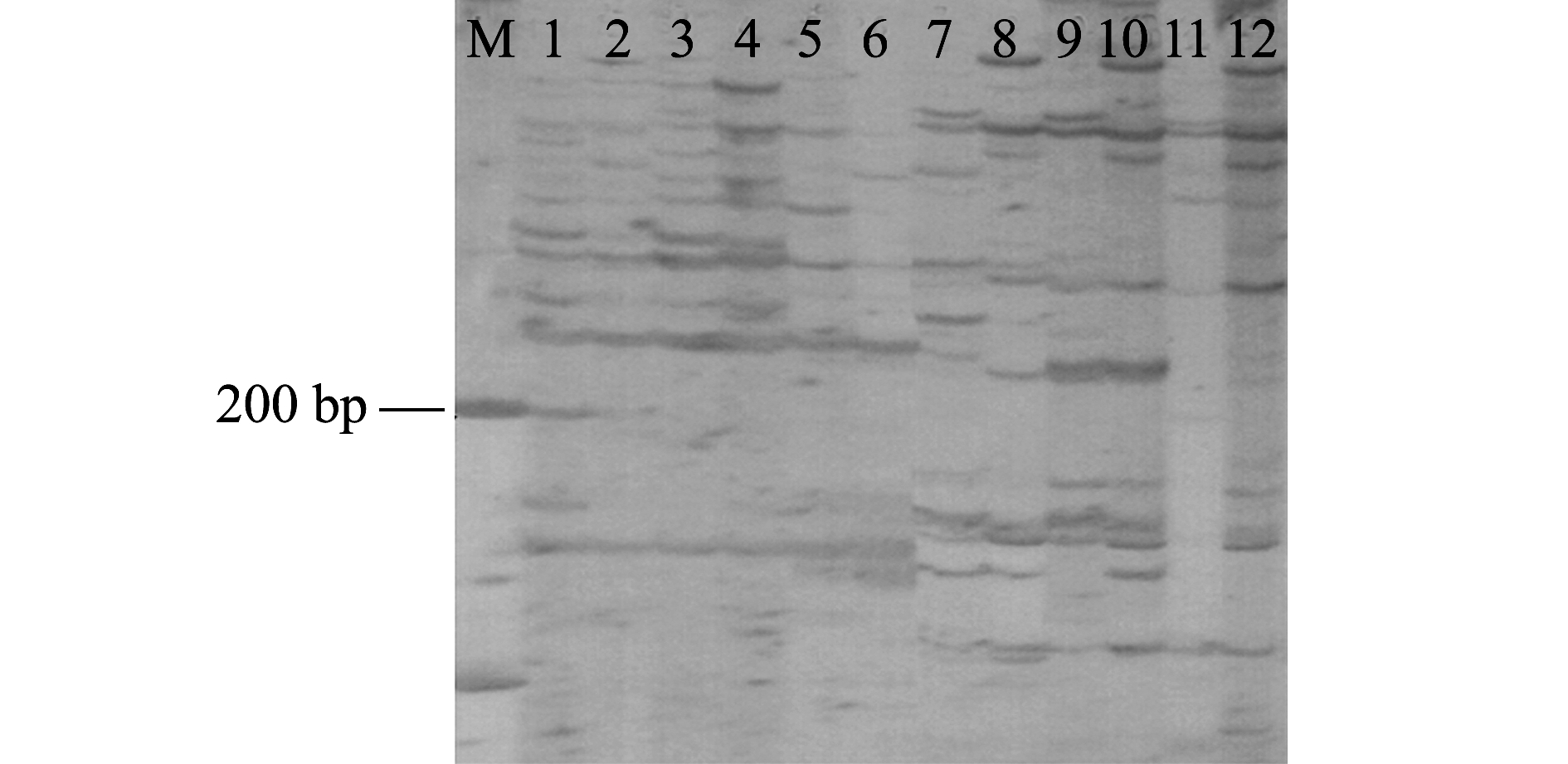
1.2.7 泥鳅基因组DNA甲基化模式分析 选用E-CT/HM-TC和E-CC/HM-TC两对选择性扩增引物(表1),分别对3个二倍体泥鳅的基因组做MSAP分析,选择性扩增产物变性后,用6%变性聚丙烯酰胺凝胶电泳检测,银染后获得图谱,分析其甲基化率。在MSAP电泳图上,每个样品对应两个泳道,即H泳道和M泳道,H泳道是指利用EcoR I/HpaII这组酶处理的样品,而M泳道是指利用EcoR I/MspI这组酶处理的样品。每个样品的基因组甲基化主要分为:非甲基化,即条带在两个泳道同时出现;全甲基化,即条带在M泳道出现而H泳道缺失;半甲基化,即条带在H泳道出现而M泳道缺失,通过统计甲基化率来确定样品的甲基化程度。
表2预扩增反应体系各成分的因素-水平正交设计表
Tab.2Factors-levelsofthecomponentsforpre-selectiveamplificationorthogonalarray
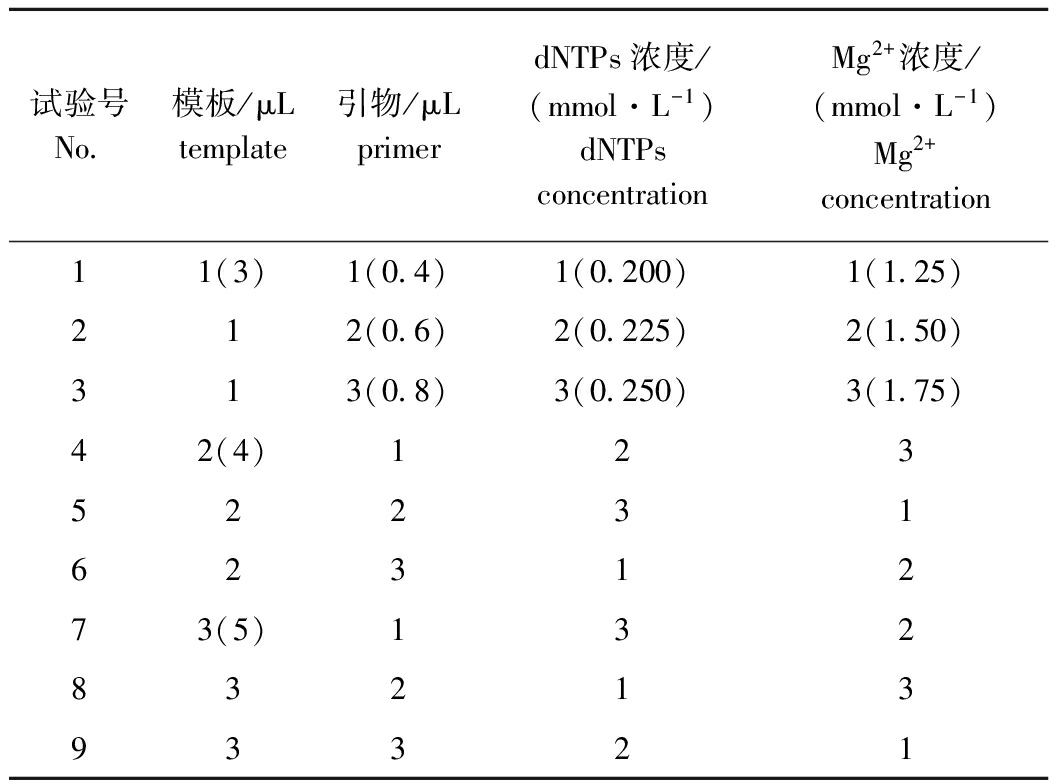
试验号No 模板/μLtemplate引物/μLprimerdNTPs浓度/(mmol·L-1)dNTPsconcentrationMg2+浓度/(mmol·L-1)Mg2+concentration11(3)1(0 4)1(0 200)1(1 25)212(0 6)2(0 225)2(1 50)313(0 8)3(0 250)3(1 75)42(4)123522316231273(5)1328321393321
表3选择性扩增反应体系各成分的因素-水平正交设计表
Tab.3Factors-levelsofthecomponentsforselectiveamplification-orthogonalarray
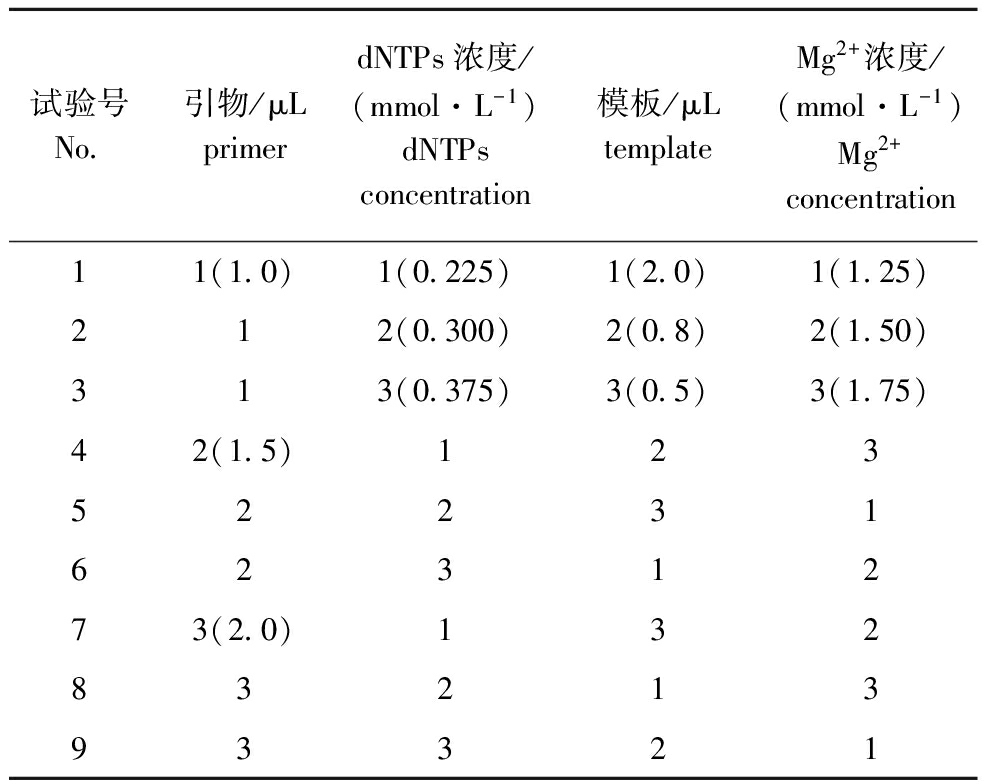
试验号No 引物/μLprimerdNTPs浓度/(mmol·L-1)dNTPsconcentration模板/μLtemplateMg2+浓度/(mmol·L-1)Mg2+concentration11(1 0)1(0 225)1(2 0)1(1 25)212(0 300)2(0 8)2(1 50)313(0 375)3(0 5)3(1 75)42(1 5)123522316231273(2 0)1328321393321
2结果与分析
2.1泥鳅基因组DNA质量的检测
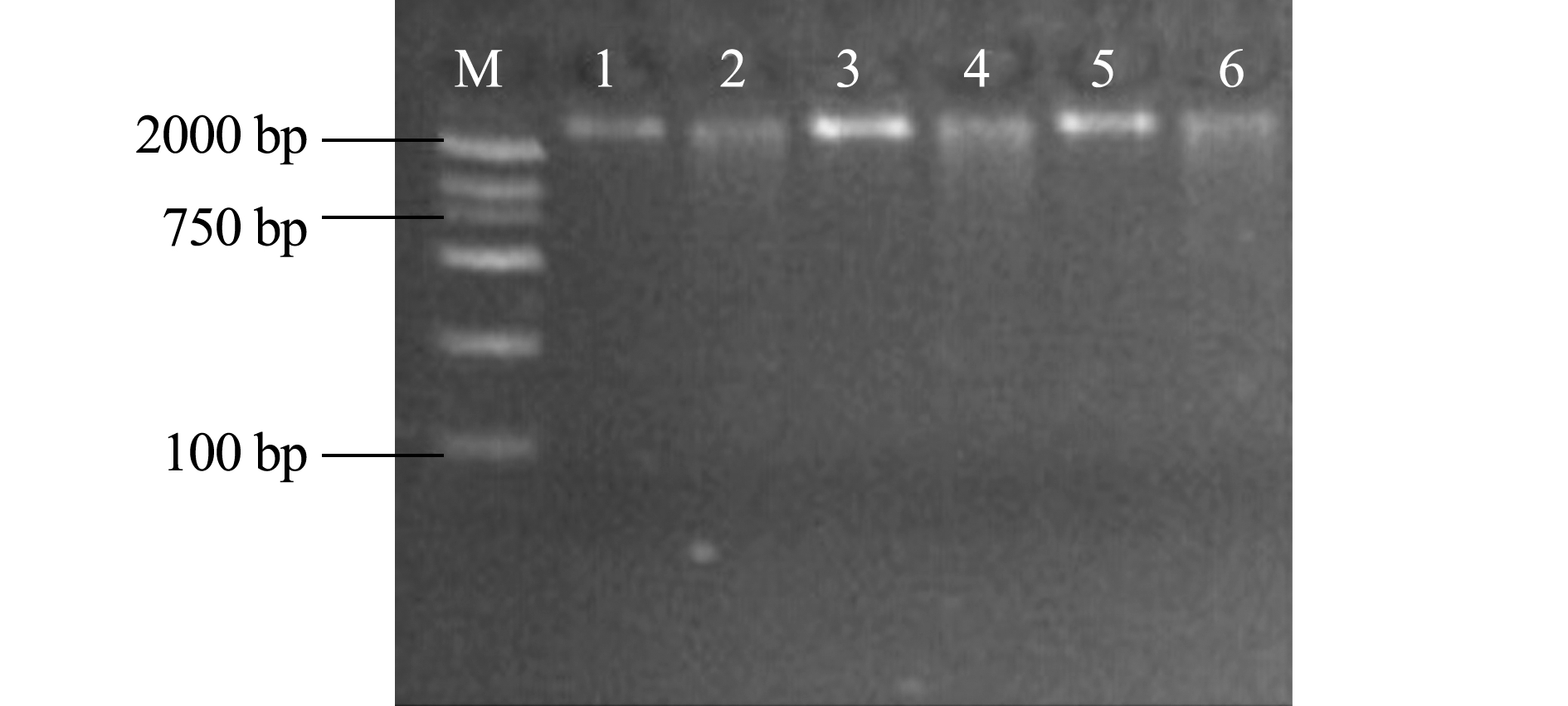
由于MSAP方法对泥鳅基因DNA的质量要求较高,因此,提取的泥鳅基因组DNA条带清晰,带型完整,点样孔无残留,无RNA(图1)。使用Eppendorf D30核酸蛋白测定仪测量DNA浓度,OD260 nm/OD280 nm的值为1.8~2.0,表明该DNA完全符合后续试验要求。
2.2MSAP双酶切体系
使用EcoR I/HpaII和EcoR I/MspI 两组限制性内切酶分别对基因组DNA进行酶切,于37 ℃下分别保温4、6、8 h,用10 g/L琼脂糖凝胶检测酶切产物。由图2可看出,酶切8 h的产物相对酶切4、6 h较完全,并呈弥散状态。后续试验也表明,酶切8 h的产物符合后续试验要求。
2.3MSAP预扩增
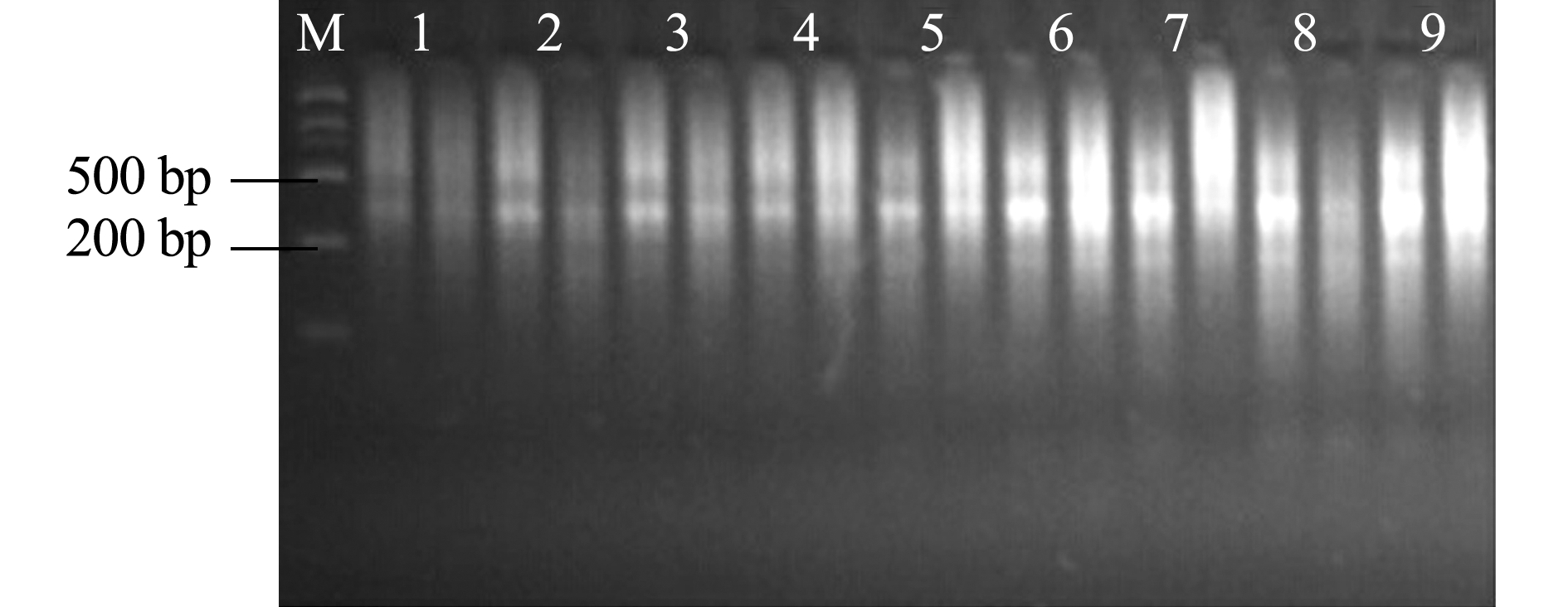
根据预扩增产物片段范围应集中在500 bp左右且呈弥散状分布的原则,对电泳图中电泳条带的大小、强弱等进行直观分析。从图3可见,在不同的处理间由于预扩增模板、预扩引物、Mg2+、dNTPs加入的量不同,扩增结果存在明显差异。其中,1、2、3、4、5、8泳道条带较暗,扩增产物较低;7、9泳道非特异性扩增较多;组合6的扩增条带明显,且在200~700 bp均匀弥散,在500 bp左右最亮。因此,确定组合6为最佳预扩增体系,即模板为4 μL、引物0.8 μL、0.2 mmol/L dNTPs、1.5 mmol/L Mg2+、TaqDNA酶1 U。
2.4MSAP选择性扩增
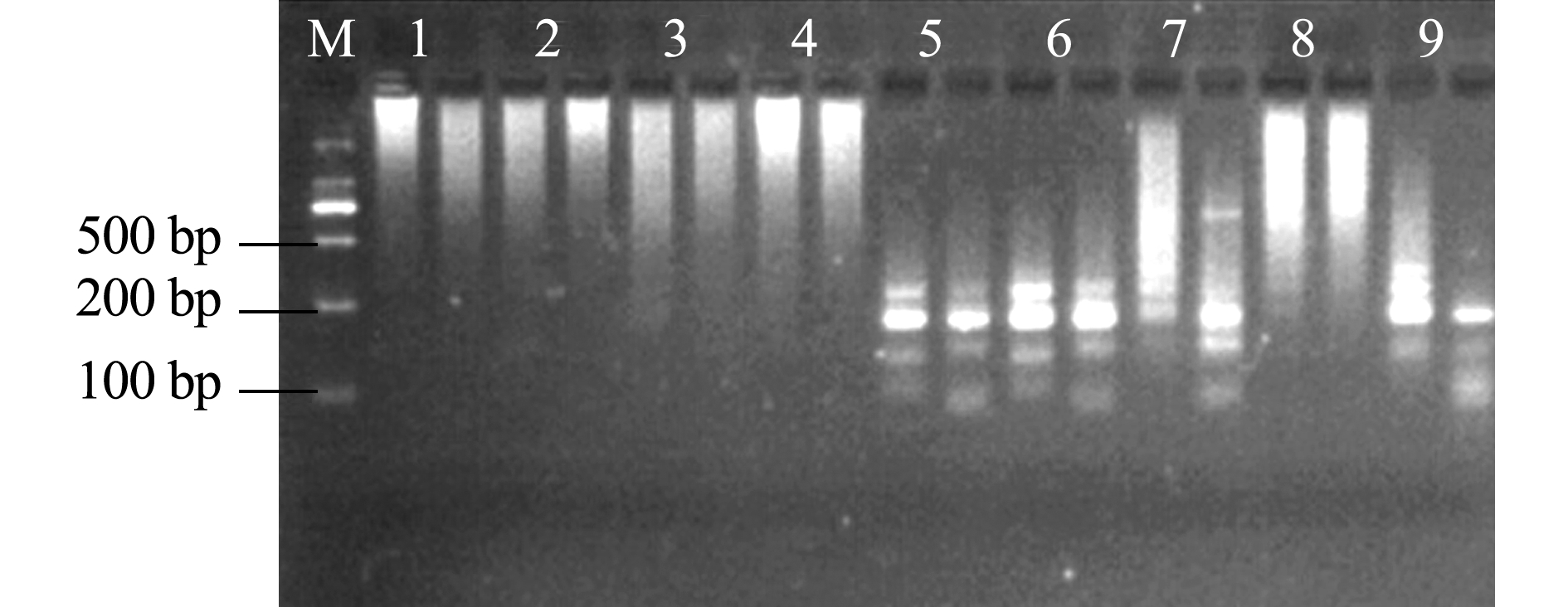
根据预试验结果初步筛选用E-CT/HM-TC引物进行选择性扩增。从图4可见,在这9个正交组合中,1、2、3、4、7和8泳道产生大量非特异性条带;第9泳道相比于第6泳道,有非特异性条带产生;第5泳道相比于第6泳道,亮度较暗,产物较低。因此,确定组合6为最佳选择性扩增体系,即稀释20倍的模板2 μL、引物1.5 μL、0.375 mmol/L dNTPs、1.5 mmol/L Mg2+、TaqDNA酶1 U。
2.5最适MSAP反应体系的验证与应用
为验证获得的最佳泥鳅MSAP反应体系,选取3个二倍体泥鳅鳍基因组DNA样品在该反应体系下进行MSAP分析。扩增产物变性后在6%变性聚丙烯酰胺凝胶上电泳,银染后获得了清晰、多态性好的指纹图谱(图5)。由表4数据可算出:3个样品的全甲基化率分别为24.1%、20.8%、22.3%,半甲基化率分别41.4%、20.8%、33.3%,总甲基化率分别为65.5%、41.6%、55.6%;3个样品的非甲基化率分别为34.5%、58.4%、44.4%。证明该体系能够应用于泥鳅基因组DNA甲基化模式分析。
3讨论
20世纪以来, 表观遗传学快速发展, 已成为生物学、医学和农学等研究领域的前沿。DNA甲基化是表观遗传修饰的主要方式之一。检测DNA 甲基化的方法有重亚硫酸法、高效液相色谱法(HPLC)、甲基化特异性PCR、基因芯片法、质谱或色谱法、甲基化敏感性扩增多态性(MSAP)和基因测序法等[22],其中MSAP技术由于其具有简便、高效、重复性好、需样量小、多态性丰富等优点,已被广泛用于检测动、植物基因组胞嘧啶甲基化水平和模式的研究中[23-25]。近年来,在栉孔扇贝Chlamysfarreri[26]、海湾扇贝Argopectenirradians[26-27]、虾夷扇贝Patinopectenyessoensis[26]、草鱼Ctenopharyngodonidellus[19]、刺参Apostichopusjaponicas[28]、中国明对虾Fenneropenaeuschinensis[29]等多种水产动物的基因组甲基化研究中已得到应用。
表4二倍体泥鳅基因组DNA的甲基化水平
Tab.4GenomicDNAmethylationlevelsofdiploidloach

样品samples总位点数totalsites非甲基化的CCGG位点none-methylatedCCGGsites甲基化的CCGG位点MethylatedCCGGsites全甲基化位点fully-methylatedsites半甲基化位点hemi-methylatedsites总计total2n-12910712192n-2241455102n-31884610
MSAP是基于AFLP的改良技术,涉及DNA提取、酶切、扩增、产物电泳检测等多个环节。在影响MSAP分析的各个因素中,基因组DNA的酶切产物质量是决定MSAP成功的关键。酶切时间过短,酶切不完全,扩增中往往出现高分子量的片段,不能真实反映基因组甲基化信息;酶切时间过长,可能会导致酶对DNA的非特异性切割,影响试验的准确性。因此,适当的酶切时间是保证MSAP试验结果准确的前提。为了使模板DNA得到充分酶切,一般会加大酶的使用量,但加大酶量将增加试验成本。因此,本研究中重点优化了内切酶的酶切时间,结果表明,酶切时间为8 h,酶切反应充分,避免出现大分子量片段多而密的情况,获得了清晰可辨的多态性条带,便于数据统计。
预扩增和选择性扩增也是影响MSAP的关键步骤。扩增的结果是由扩增反应中各个因素综合作用的结果,多数学者认为,引物用量、模板用量、Mg2+浓度、dNTPs浓度是影响扩增结果的主要因素。因为引物用量过低,则产物的量明显较低,相反,则会产生非特异性条带;模板量过低,则条带较暗,产物量较低;Mg2+浓度对扩增的结果影响也较大,这在选择性扩增中表现的最为明显,Mg2+过量易产生大量非特异性条带,相反,不足则易使产量降低;dNTPs可以为反应提供能量,dNTPs 浓度过低会降低产物的产量,而且dNTPs浓度与Mg2+浓度要相适应,否则会直接影响PCR结果。因此,本试验中着重对泥鳅MSAP反应体系中预扩增和选择性扩增进行优化。结果表明:预扩增最佳反应体系为模板4 μL、引物0.8 μL、0.2 mmol/L dNTPs、1.5 mmol/L Mg2+;选择性扩增最佳反应体系为稀释20倍的模板2 μL、引物1.5 μL、0.375 mmol/L dNTPs、1.5 mmol/L Mg2+。本试验结果与多数研究报道一致[30-32]。用该体系对泥鳅全基因组DNA进行甲基化水平和模式分析,经统计分析表明,3个二倍体泥鳅个体之间的甲基化程度存在差异,其原因有待于进一步研究。
综上所述,通过本试验优化后得到了稳定、重复性好的MSAP体系,为今后探究同源四倍体泥鳅多倍化过程中基于DNA甲基化的表观遗传变化特征奠定了基础。
参考文献:
[1] Jablonka E,Lamb M J.The changing concept of epigenetics[J].Annals of the New York Academy of Sciences,2002,981:82-96.
[2] Xu M L,Li X Q,Korban S S.AFLP-based detection of DNA methylation[J].Plant Molecular Biology Reporter,2000,18:361-368.
[3] 李智慧,逯翔,高云,等.物种多倍化与表观遗传学[J].科学通报,2010,55(35):3358-3366.
[4] Morishima K,Horie S,Yamaha E,et al.A cryptic clonal line of the loachMisgurnusanguillicaudatus(Teleostei:Cobitidae) evidenced by induced gynogenesis,interspecific hybridization,microsatellite genotyping and multilocus DNA fingerprinting[J].Zoological Science,2002,19:565-575.
[5] Arai K,Matsubara K,Suzuki R.Karyotype and erythrocyte size of spontaneous tetraploidy and triploidy in the loachMisgurnusanguillicaudatus[J].Nippon Suisan Gakkaishi,1991,57:2167-2172.
[6] Zhang Quanqi,Arai K.Distribution and reproductive capacity of natural triploid individuals and occurrence of unreduced eggs as a cause of polyploidization in the loach,Misgurnusanguillicaudatus[J].Ichthyological Research,1999,46:153-161.
[7] 糸納正樹,荒井克俊.北海道蓴菜沼産ドジョウにおける非還元二倍性卵の形成[J].水産育種,2004,33:107-113.
[8] 李康,李渝成,周暾.两种泥鳅染色体组型的比较研究[J].动物学研究,1983,4(1):75-80.
[9] 李渝成,李康.马口鱼和泥鳅的核型研究,兼论鱼类染色体数目多态与分类的关系[J].武汉大学学报:自然科学版,1987,1:107-111.
[10] 印杰,赵振山,陈小奇,等.二倍体和四倍体泥鳅染色体组型比较[J].水生生物学报,2005,29(4):469-472.
[11] 李雅娟,印傑,王嘉博,等.中国におけるドジョウ倍数体の分布に関する研究[J].日本水産学会誌,2008,74(2):177-182.
[12] 李雅娟,田萍萍,李莹,等.中国洪湖不同倍性泥鳅的染色体组型及形态特征比较分析[J].大连水产学院学报,2009,24(3):236-241.
[13] Li Y J,Tian Y,Zhang M Z,et al.Chromosome banding and FISH with rDNA probe in the diploid and tetraploid loachMisgurnusanguillicaudatus[J].Ichthyological Research,2010,57(4):358-366.
[14] Li Y J,Yu Z,Zhang M Z,et al.The origin of natural tetraploid loachMisgurnusanguillicaudatus(Teleostei:Cobitidae) inferred from meiotic chromosome configurations[J].Genetica,2011,139:805-811.
[15] 刘博,李雅娟,李霞,等.不同倍性泥鳅鳍细胞培养及染色体标本制备方法的研究[J].大连海洋大学学报,2013,28(6):557-562.
[16] Liu B,Wendel J F.Epigenetic phenomena and the evolution of plant allopolyploids[J].Molecular Phylogenetics and Evolution,2003,29:365-379.
[17] Ma X F,Gustafson J P.Genome evolution of allopolyploids:a process of cytological and genetic diploidization[J].Cytogenetic and Genome Research,2005,109(1/3):236-249.
[18] Fujikawa N,Nakayama I,Onozato H.Genetic inactivation of crucian carp eggs using a simple X-ray irradiation apparatus[J]. Bull Natl Res Inst Aquaculture,1993,22:11-19.
[19] 曹哲明,丁炜东,俞菊华,等.草鱼全同胞鱼苗不同个体甲基化位点的差异[J].动物学报,2007,53(6):1083-1088.
[20] 潘承毅,何迎晖.数理统计的原理与方法[M].上海:同济大学出版社,1993:292-293.
[21] 秦艳杰.海湾扇贝遗传图谱构建及壳色基因、生长相关QTL的定位研究[D].青岛:中国科学院研究生院(海洋研究所),2006:42-44.
[22] Dahl C,Guldberg P.DNA methylation analysis techniques[J].Biogerontology,2003,4(4):233-250.
[23] Cervera M T,Ruiz-García L,Martínez-Zapater J M.Analysis of DNA methylation inArabiodopsisthalianabased on methylation-sensitive AFLP markers[J].Molecular Genetics and Genomics,2002,268(4):543-552.
[24] 万亚琴.DNA甲基化与肉牛杂种优势关系的初步研究[D].重庆:西南大学,2008:30-35.
[25] 蒋曹德.猪DNA甲基化与杂种表现的关系及遗传多样性分析[D].武汉:华中农业大学,2004:64-66.
[26] 于涛,杨爱国,吴彪,等.栉孔扇贝、虾夷扇贝及其杂交子代的MSAP分析[J].水产学报,2010,34(9):1335-1341.
[27] 吕佳,侯睿,李宁,等.应用MSAP技术研究扇贝全基因组DNA甲基化水平[J].中国海洋大学学报,2013,43(10):48-53.
[28] 郭婷婷,孙国华,杨建敏,等.刺参 (Apostichopusjaponicus) 不同组织基因组甲基化状态MSAP分析[J].海洋与湖沼,2013,44(1):77-82.
[29] 杜盈,何玉英,李健,等.野生和“黄海1号”中国明对虾不同组织基因组DNA的MSAP分析[J].中国水产科学,2013,20(3):536-543.
[30] 高寰,张铮,周婷,等.三叶木通MSAP反应体系的优化及引物筛选[J].中草药,2012,43(3):572-576.
[31] 吕晓婷,赵春梅,王爱华,等.苹果MSAP技术体系的优化及其应用[J].中国农学通报,2012,28(22):287-292.
[32] 曹喜兵,赵改丽,范国强.泡桐MSAP体系建立及引物筛选[J].河南农业大学学报,2012,46(5):535-541.
EstablishmentandoptimizationofMSAPreactionsystemforloachMisgurnusanguillicaudatus
ZHOU He, XU Wen, LI Ya-juan, QIN Yan-jie, LI Xia, MA Tian-yu, XIAO Yi-xiao, MA Hai-yan
(Key Laboratory of Marine Bio-resources Restoration and Habitat Reparation in Liaoning Province, Dalian Ocean University, Dalian 116023, China)
Abstract:A reaction system of MSAP was established by orthogonal experiment in order to obtain genomic DNA methylation level and pattern of different ploidy of loachMisgurnusanguillicaudatusand used for MSAP analysis of diploid loach. The results showed that the best double digestion systems for MSAP including 10 UEcoR I, 10 UHpaII, and 10 UMspI were observed for 8 h at 37 ℃; the best pre-selective amplification mixture contained 0.2 mmol/L dNTPs, 1.5 mmol/L Mg2+,Taq1 U and selective primers 0.8 μL in total 20 μL; the best selective amplification mixture contained 0.375 mmol/L dNTPs, 1.5 mmol/L Mg2+,Taq1 U and selective primers 1.5 μL in total 20 μL. The atlas showed that full methylation rate of three samples were 24.1%, 20.8% and 22.3%, respectively; hemi-methylation rates were 41.4%, 20.8% and 33.3%, respectively; and total methylation rate was 65.5%, 41.6% and 55.6%, respectively. The findings indicated that this MSAP reaction system was stable, reliable and reproducible, providing the foundation for the MSAP technology with loach-related research.
Key words:Misgurnusanguillicaudatus; DNA methylation; methylation sensitive amplification polymorphism(MSAP); reaction system
DOI:10.3969/J.ISSN.2095-1388.2014.06.002
文章编号:2095-1388(2014)06-0550-06
收稿日期:2014-08-29
基金项目:国家自然科学基金资助项目(31272650);大连海洋大学博士启动基金资助项目(017208)
作者简介:周贺(1985—), 女, 讲师。 E-mail:zhouhe@dlou.edu.cn
通信作者:李雅娟(1961—), 女, 教授。 E-mail: liyajuan@dlou.edu.cn
中图分类号:S965.1
文献标志码::A