表1引物序列
Tab.1Sequencesoftheprimers
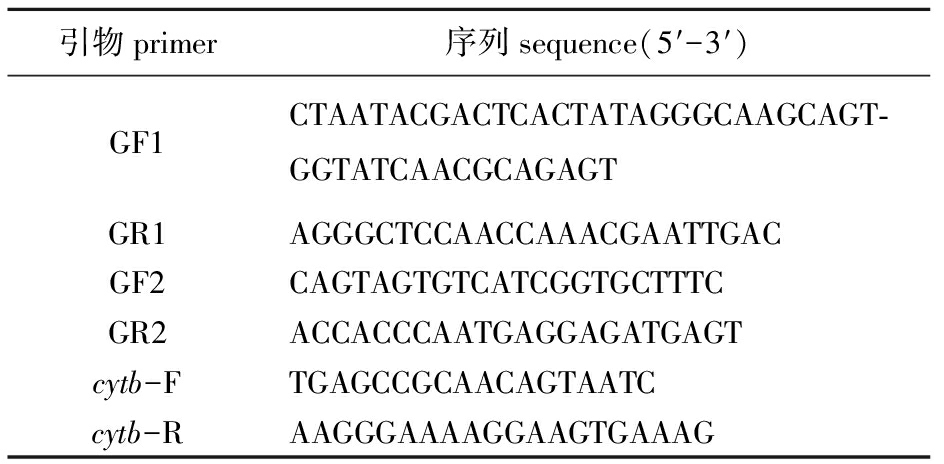
引物primer序列sequence(5′-3′)GF1CTAATACGACTCACTATAGGGCAAGCAGT⁃GGTATCAACGCAGAGTGR1AGGGCTCCAACCAAACGAATTGACGF2CAGTAGTGTCATCGGTGCTTTCGR2ACCACCCAATGAGGAGATGAGTcytb-FTGAGCCGCAACAGTAATCcytb-RAAGGGAAAAGGAAGTGAAAG
摘要:根据GenBank中登录的仿刺参Apostichopusjaponicusghitm基因的EST片段,采用RACE扩增法克隆了仿刺参ghitm基因的cDNA全长序列,并利用qRT-PCR技术检测了经LPS诱导后仿刺参ghitm的表达变化情况。结果表明:仿刺参ghitm基因(Aj-ghitm)的cDNA序列全长为1325 bp,包含一个1005 bp编码334个氨基酸的开放阅读框,序列两端分别为140 bp的5′UTR和180 bp的3′UTR。序列分析结果显示,Aj-ghitm编码的蛋白含有一个8次跨膜结构域,与GenBank中登录的紫色球海胆GHITM蛋白的氨基酸序列相似度为65.4%,且在系统发育树中聚为一支;qRT-PCR检测结果表明,经LPS刺激可诱导仿刺参体腔细胞Aj-ghitmmRNA的表达且呈先下降后升高最后回复到接近正常水平的趋势,在24 h时上升到最高,随后下降。本研究是有关棘皮动物ghitm基因的首次报道,其结果可为进一步探讨ghitm在仿刺参抗细菌感染以及生长发育等方面的功能及作用机制提供参考。
关键词:仿刺参;仿刺参ghitm;基因克隆;基因表达
生长激素诱导的跨膜蛋白(Growth-hormone inducible transmembrane protein,GHITM),是在研究表达生长激素拮抗物的转基因小鼠(Growth-hormone antagonist,GHA)的差异表达基因时被首次发现[1]。该基因主要存在于试验小鼠的脂肪组织中,其表达量显著高于对照组。随后Yoshida等[2]研究发现,在小鼠胚胎和分化组织以及哺乳动物细胞系中也存在ghitm基因的表达。Knapp等[3]研究也发现,在相同的摄食量下,GHA小鼠的体质量增加明显超过对照小鼠,且主要为身体脂肪量的增加,暗示ghitm与动物的生长调节有关。2000年,Zou等[4]研究证实,GHITM蛋白在氧自由基对抗衰老的机制中发挥作用,与动物的年龄、生长相关。除受生长激素(Growth hormone,GH)诱导与调节外,GHITM蛋白的另一个特征是具有6~7个跨膜结构域,是一个典型的跨膜蛋白,因而得名生长激素诱导的跨膜蛋白。
Reimers等[5]在分析GHITM蛋白的氨基酸序列特征时发现,其C末端具有特殊结构域(UPF0005)。该结构域为BI-1样超家族(BI-1-like superfamily )的特征结构域,因此,将其归类为BI-1样超家族的新成员。除GHITM外,BI-1样超家族还包括BI-1(Bax inhibitor-1)、RECS1(Responsive to centrifugal force and shear stress gene 1)、GAAP(Golgi anti-apoptotic protein)和LFG(Lifeguard)等4个家族成员。这些家族成员在植物到哺乳动物中保守存在,都是定位于细胞膜上且具有6~7个跨膜结构域的高疏水性蛋白。研究发现,该家族成员有促进BCL-2或抑制BAX的作用,从而抑制细胞凋亡[6-9]。RECS1还可抑制肿瘤坏死因子受体介导的NF-κB转录激活,在免疫、炎症、发育和肿瘤的发生发展中发挥重要作用[10]。GHITM蛋白受到GH的诱导与调控,与动物的生长、发育、肿瘤形成等相关,但具体生物学功能及作用机制目前尚不十分明确,有待进一步研究。
仿刺参Apostichopusjaponicus是中国北方海水养殖业中主要的养殖品种之一,也是辽宁乃至北方地区海水养殖业的主要养殖品种和支柱产业。随着仿刺参养殖业的不断发展,各种病害问题日益突出。有关仿刺参病害防控机制及分子免疫学的研究已成为近年来的研究热点领域之一,并有望为仿刺参病害防控提供新的理念和技术方法。在前期的研究中,作者分析了LPS诱导后仿刺参体腔细胞表达谱测序结果,从中获得了差异表达基因ghitm的部分序列。本研究中,采用RACE扩增法获得该基因的全长序列,并采用qRT-PCR技术检测LPS诱导后ghitm基因的表达情况,旨在为进一步探讨仿刺参ghitm基因的生物学功能及作用机制提供理论基础。
1.1材料
试验用仿刺参取自大连湾某养殖场,体质量为34.9~58.6 g,取回后在农业部北方海水增养殖重点实验室水槽(200 L)中暂养7 d后用于试验。暂养期间,每天换水量为1/2,并投喂人工饵料。暂养用水为沙滤海水,水温为17~19 ℃。
总RNA提取试剂盒、UNIQ-10柱式胶回收试剂盒购自天根生化科技有限公司;氨苄青霉素、LPS购自Sigma公司;SMARTTMRACE cDNA扩增试剂盒由Clontech公司提供;PCR扩增用Taq酶、pMDl8-T载体、M-MLV反转录酶及SYBR©Premix TaqTM定量PCR试剂盒等购自TaKaRa公司。核酸测序以及各类引物的合成分别由华大基因科技有限公司和生工生物工程(上海)股份有限公司完成。
1.2方法
1.2.1 总RNA的提取及cDNA的合成 取健康仿刺参,用注射器抽取体腔液,在4 ℃下以1000g低速离心2 min,收集体腔细胞,采用RNAsimple总RNA提取试剂盒提取仿刺参体腔细胞总RNA,用琼脂糖凝胶电泳及紫外分光光度仪检测其完整性与纯度,于-80 ℃下保存备用。采用M-MLV RT-PCR试剂盒将所得总RNA反转录为cDNA。
1.2.2 RACE扩增 参考表达谱测序中获得的仿刺参ghitm基因部分序列,利用Primer Premier 5.0软件设计5′RACE特异性引物GR1,UPM长引物GF1由试剂盒提供,引物序列见表1。3′端已包括终止密码子和非编码区序列,不需要进一步扩增。使用SMARTTMRACE cDNA扩增试剂盒进行5′RACE扩增,反应体系(共25 μL):10×ExTaqBuffer (Mg2+Plus) 2.5 μL,cDNA 2.5 μL,引物GF1 2.5 μL,引物GR1 0.5 μL,dNTP Mixture(各2.5 mmol/L) 2.0 μL,Taq酶(5 U/μL) 0.5 μL,ddH2O 14.5 μL。5′RACE扩增程序:94 ℃下预变性5 min;94 ℃下变性30 s,72 ℃下退火3 min,进行5个循环;94 ℃下变性30 s,70 ℃下退火30 s,72 ℃下 延伸3 min,进行5个循环; 94 ℃下变性30 s, 65 ℃下退火30 s,72 ℃下延伸3 min,进行35个循环;最后在72 ℃下延伸7 min,终止反应。扩增产物经琼脂糖凝胶电泳检测,用UNIQ-10柱式胶回收试剂盒回收纯化后,连接到pMDl8-T载体,转化至E.coliDH5α感受态细胞中。挑取阳性克隆送华大基因科技有限公司进行测序,并将所得片段与原仿刺参ghitm基因部分序列拼接后获得仿刺参ghitm基因全长cDNA序列。
表1引物序列
Tab.1Sequencesoftheprimers

引物primer序列sequence(5′-3′)GF1CTAATACGACTCACTATAGGGCAAGCAGT⁃GGTATCAACGCAGAGTGR1AGGGCTCCAACCAAACGAATTGACGF2CAGTAGTGTCATCGGTGCTTTCGR2ACCACCCAATGAGGAGATGAGTcytb-FTGAGCCGCAACAGTAATCcytb-RAAGGGAAAAGGAAGTGAAAG
1.2.3 生物信息学分析 利用DNAStar软件包中的SeqMan程序对测序结果中载体序列进行去除以及与原序列进行拼接;用ClustalX 1.83软件、NCBI的Blast(http://blast.ncbi.nlm.nih.gov/Blast. cgi)和EBI的EMBOSS Needle(http://www.ebi.ac. uk/Tools/psa/emboss_needle/)进行序列比对和同源性分析。采用NCBI的ORF Finder(http://www. ncbi.nlm.nih.gov/gorf/gorf.html)和CD Search(http://www.ncbi.nlm.nih.gov/structure/cdd/wrpsb. cgi)分别进行开放阅读框预测和保守结构域分析。使用EXPASY网站的Prosite(http://web.expasy. org/cgi-bin/protparam/protparam)和TMpred网站的TMpred在线分析工具(http://www.ch.embnet. org/software/TMPRED_form.html)分别进行序列编码蛋白的理化性质与跨膜结构预测在线分析。采用Mega 4.0的Neighbor-joining法构建系统进化树(bootstrap=1000)。
1.2.4 LPS诱导后仿刺参ghitm基因的表达分析 分别配制PBS缓冲液(0.01 mol/L,pH 7.4)和0.1 mg/mL LPS储备液(将0.1 mg LPS溶于1 mL PBS缓冲液),经0.22 μm滤膜过滤除菌后备用。选取30只健康仿刺参,随机分成两组,分别暂养在两个相同的水槽(200 L)中。其中一组用1 mL无菌注射器给每只刺参体腔注射LPS 100 μL,另一组注射等量PBS。分别于注射后0、12、24、48 h抽取体腔液,按照“1.2.1”节方法,抽提总RNA并反转录制备cDNA模板。根据已获得的仿刺参ghitm编码序列设计qRT-PCR引物GF2和GR2(表1),根据杨爱馥等[11]对仿刺参看家基因表达稳定性的比较,选取细胞色素b(cyto-chrome b,cytb)作为内参基因(表1)。
按照SYBR©Premix TaqTM(Perfect Real-time)试剂盒操作步骤,并作适当修改后进行qRT-PCR反应。反应体系(共20 μL):SYBR Premix ExTaq(2×) 10 μL, 10 μmol/L 上、下游引物各0.5 μL,cDNA模板1.0 μL,ddH2O 8.0 μL。反应条件:95 ℃下预变性5 min;95 ℃下变性30 s,60 ℃下退火32 s,共进行40个循环。不同处理的每个时间点分别取3个重复样本。扩增反应在ABI 7500 Real-time PCR system (Applied Biosystems)上进行,反应结束后进行融解曲线的绘制,并使用定量PCR仪自带软件包进行溶解曲线分析[12]。所得数据应用相对CT法(2-△△CT)处理,在此基础上进行ghitm基因的表达变化分析[13]。在使用CT方法前,确定目的基因与看家基因扩增效率基本一致。
1.3数据处理
试验数据均用平均值±标准误表示,采用SPSS 17.0软件对试验数据进行统计分析,用单因素方差分析法(One-way ANOVA)进行方差分析,用Duncan 法进行多重比较,显著性水平设为0.05。
2.1Aj-ghitm全长cDNA及编码氨基酸的序列结构分析
以总RNA逆转录得到的5′RACE cDNA为模板,利用设计好的巢式引物进行5′RACE扩增,获得3条扩增条带,其中最长片段接近750 bp,且亮度最高(图1)。该条带经切胶、回收,克隆测序,去载体接头序列后,获得条带大小为603 bp的目的片段,利用Seqman软件与仿刺参ghitm的EST序列(1089 bp)进行拼接后,获得cDNA全长序列(图2)。将拼接获得的序列命名为Aj-ghitm,并提交至GenBank,登录号为KC886718。该cDNA序列全长为1325 bp,两端分别为140 bp的5′UTR和180 bp的3′UTR,开放阅读框为1005 bp,编码了长度为334个氨基酸多肽。预测编码蛋白相对分子量为35 410,理论等电点为9.70。进一步分析发现,Aj-ghitm编码蛋白脂溶性指数为95.87,疏水性指数为0.403,属于疏水性蛋白。
基于CD Search的结构域分析显示,Aj-ghitm编码蛋白包含一个保守的GHITM结构域(cd10431)。基于编码蛋白氨基酸的疏水性进行拓扑结构预测,结果显示,Aj-GHITM蛋白具有8个可能的跨膜区域:69~88、117~135、150~168、186~205、207~227、 239~256、266~282、308~331氨基酸残基处(图2)。一般认为,跨膜结构预测分数在500以上即为可信,此8处区域跨膜结构预测的分数在777~2175,表明Aj-ghitm基因编码了一个具有8个跨膜结构的蛋白。其中69~88、150~168、207~227和266~282跨膜区域为由外向内跨膜(用“---“表示), 117~135、186~205、239~256和308~331跨膜区域为由内向外跨膜(用“——”表示)(图3)。
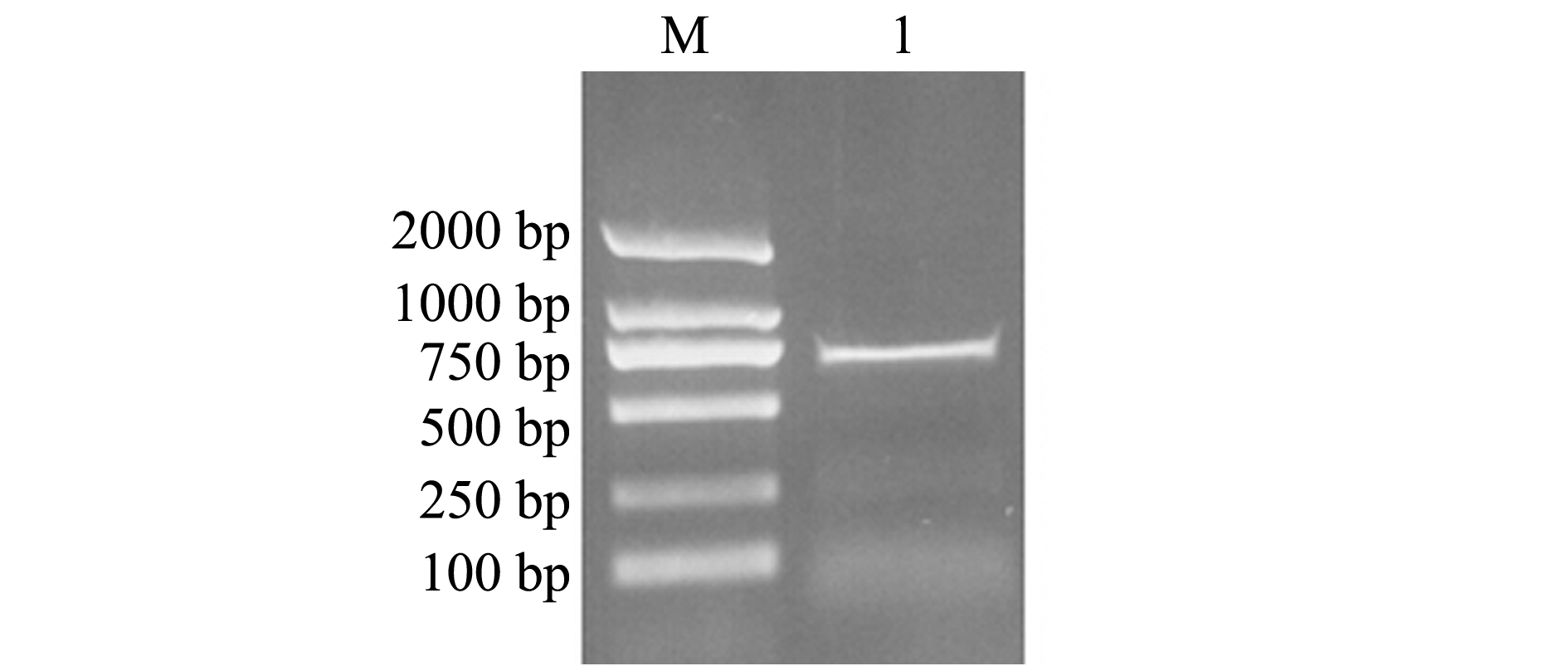
注:M为DNA Marker DL2000;1为5′RACE扩增产物
Note: M, DNA Marker DL2000; 1, 5′RACE amplification products
图1 5′RACE扩增产物检测结果
Fig.1 5′RACE amplification results
2.2Aj-GHITM氨基酸序列的同源性比较和系统进化分析
BlastX搜索和序列比对分析显示,Aj-GHITM蛋白的氨基酸序列与GenBank中登录的GHITM氨基酸序列具有较高的相似度和一致性。基于ClustalX 1.83软件的氨基酸序列同源性分析显示,Aj-GHITM氨基酸序列的保守性较高,与海葵、太平洋牡蛎、黑腹果蝇、紫色球海胆、斑马鱼、非洲爪蟾和人的GHITM一致性和相似度分别为46.7%~64.3%和66.2%~78.8%(表2、图4)。在这些序列中,Aj-GHITM与紫海胆GHITM的一致性和相似度最高,分别为64.3%和78.8%;与人GHITM的一致性和相似度最低,分别为46.7%和66.2%。
根据氨基酸序列比对结果,利用Mega 4.0软件构建基于Neighbor-joining法的系统进化树 (图5)。结果显示,Aj-GHITM与紫海胆GHITM聚为一支,亲缘关系最近。此外,基于更大范围的相似序列构建的系统进化树显示,Aj-GHITM被聚类在GHITM分支中,与其他动物BI-1、GAAP、LFG和RECS蛋白等一起构成了BI-1样超家族的5大分支,即Aj-GHITM属于BI-1样超家族。

注:起始密码子ATG和终止密码子TAA用方框标出,灰色阴影为推测的GHITM结构域(cd10431),跨膜区域用下划线标出
Note: The initiation codon (ATG) and stop codon (TAA) are boxed;GHITM domain (cd10431) is shaded;The amino acids of the transmembrane region are underlined
图2 ghitm cDNA序列与预测的编码氨基酸序列
Fig.2 cDNA sequence and the deduced amino acids of ghitm
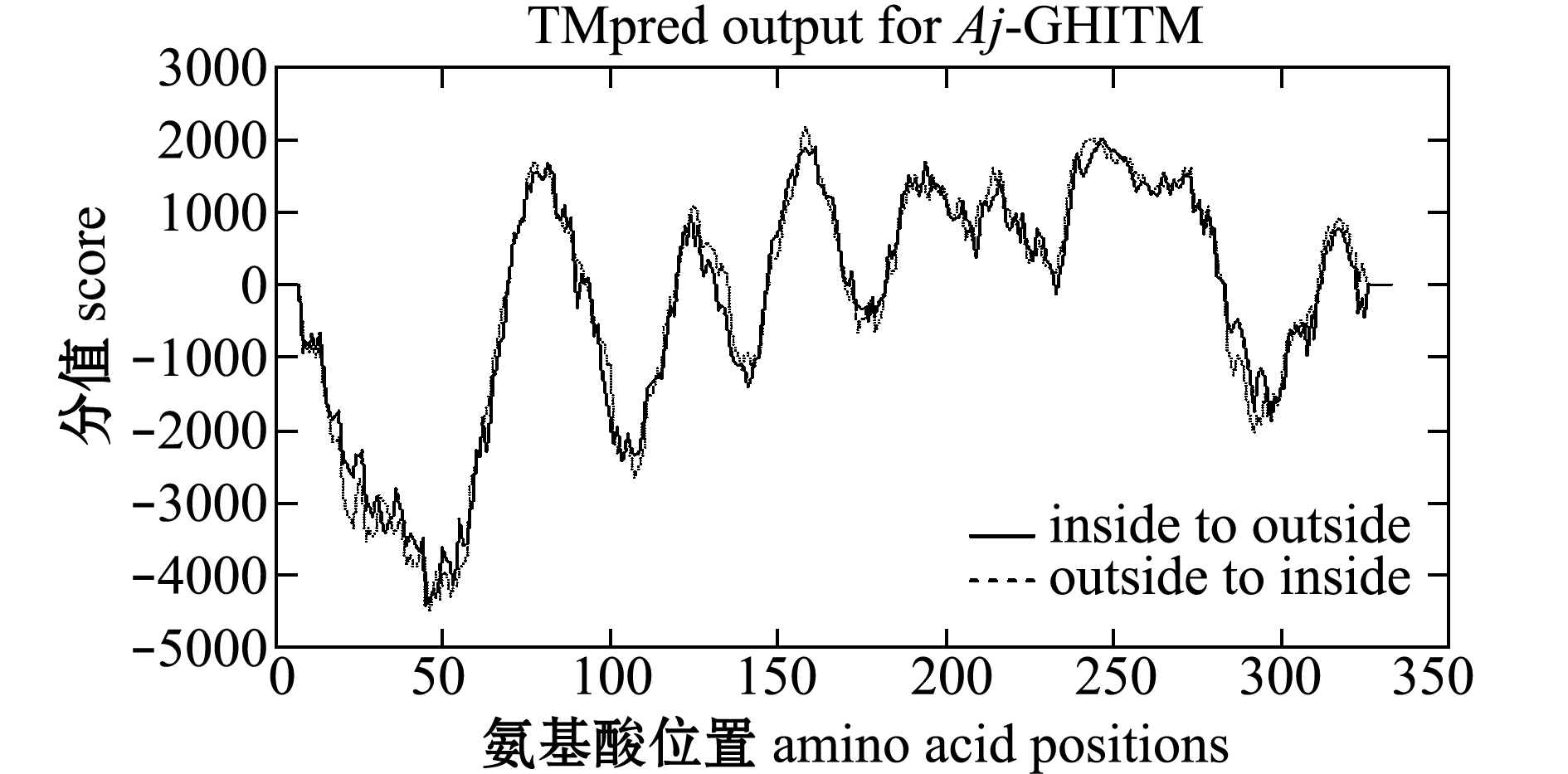
图3 Aj-GHITM跨膜拓扑结构预测
Fig.3 Predicted membrane topology of Aj-GHITM
2.3LPS刺激后Aj-ghitmmRNA表达的变化
为了研究Aj-ghitm在仿刺参抗感染中的作用,检测了经LPS刺激后仿刺参Aj-ghitmmRNA表达量的变化。基于cytb内参基因的qRT-PCR检测结果显示(图6),Aj-ghitmmRNA的表达水平经LPS刺激后,呈先下降后升高最后回复到接近正常水平的变化趋势。在LPS刺激后12 h,Aj-ghitmmRNA的表达水平较PBS对照组下降了55.5%,在24 h时显著升高(P<0.05),为对照组的3.78倍,随后在48 h时又显著下降(P<0.05),为对照组的1.29倍。在LPS刺激后的24 h,表达量显著高于其他时间点的表达水平 (P<0.05)。
表2用于同源性比较及进化树构建的氨基酸序列及其与仿刺参GHITM的氨基酸相似度
Tab.2AminoacidsequencesforconstructionofphylogenicanalysisandthesimilaritywithAj-GHITM

物种speciesGenBank登陆号GenBankaccessionNo 一致性identity/%相似度similarity/%仿刺参Apostichopusjaponicus紫色球海胆Strongylocentrotuspurpuratus海葵Nasoniavitripennis太平洋牡蛎Crassostreagigas非洲爪蟾Xenopuslaevis斑马鱼Daniorerio黑腹果蝇Drosophilamelanogaster人HomosapiensKC886718XP_791078 1XP_001608127 1EKC33390 1NP_001080095 1NP_956885 1AHN59577BAG60581 1—64 350 353 850 048 150 046 7—78 870 972 170 869 466 866 2

注: 阴影表示保守氨基酸,黑色、灰色和浅灰色分别表示保守性为100%、[75%~100%)和[50%~75%)的氨基酸
Note: Conserved amino acids are shaded. Black, grey and light-grey shades represent the amino acids conservation of 100%,[75%-100%), and[50%-75%), respectively
图4 仿刺参与其他动物GHTIM氨基酸序列的比较分析
Fig.4 Multiple sequence alignment of GHITM between sea cucumber and other animals
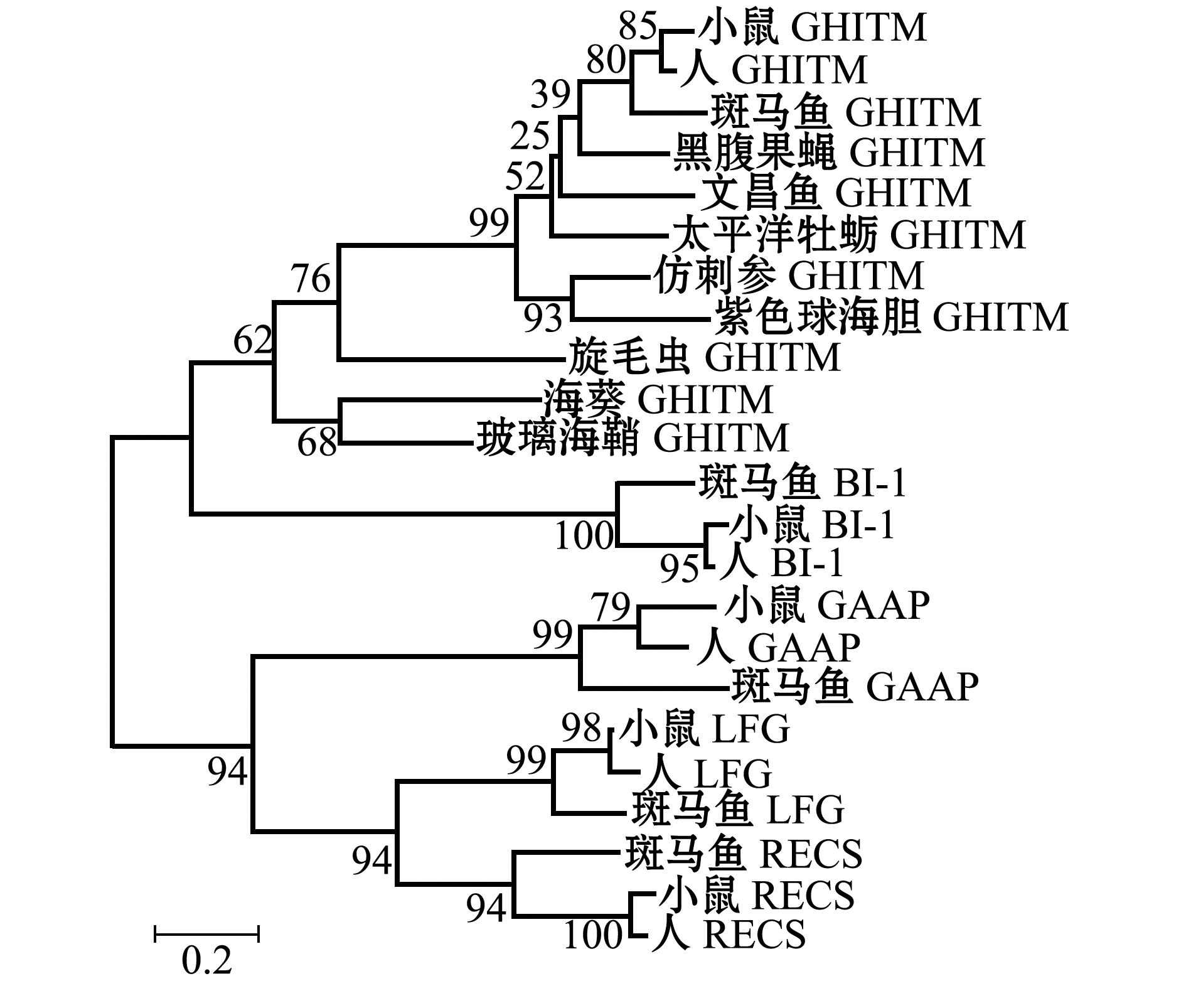
图5 仿刺参GHITM的系统进化树
Fig.5 Phylogenetic tree of Aj-GHITM
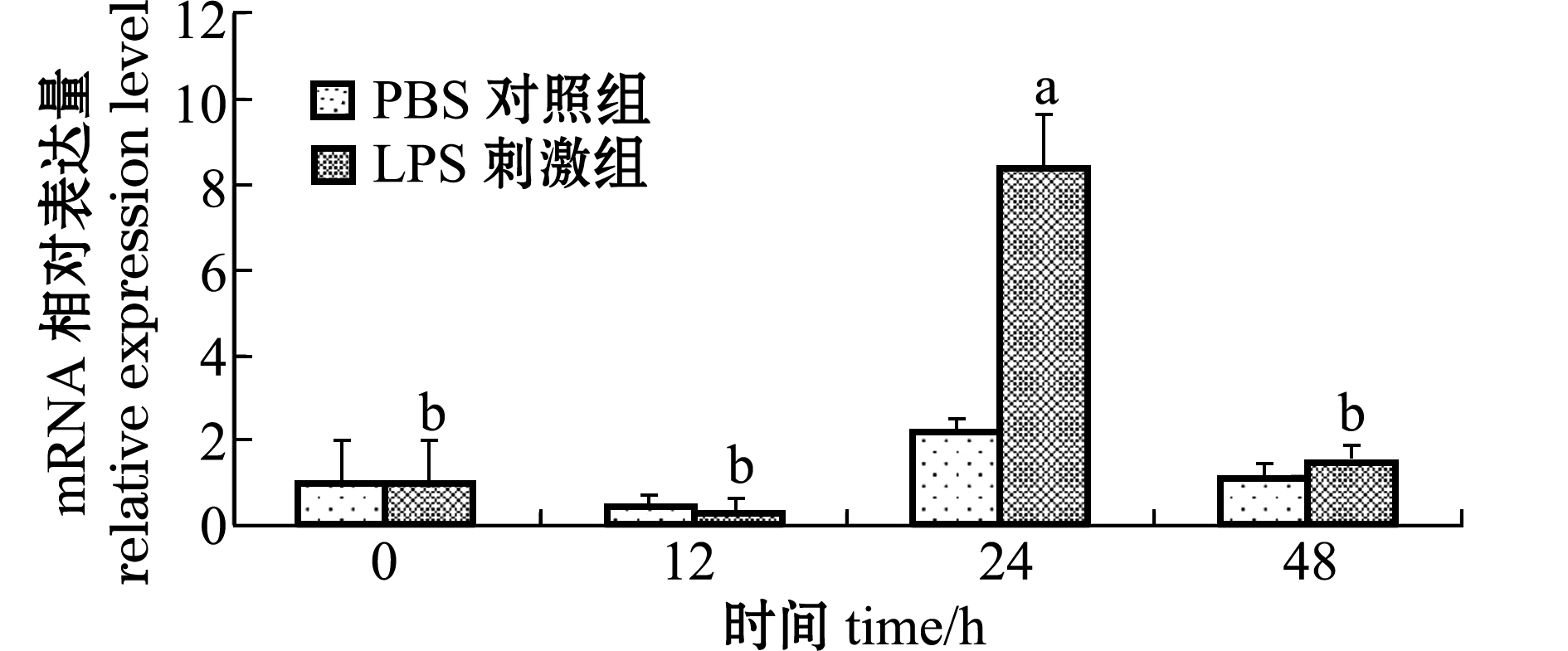
注:标有不同字母表示试验组间有显著性差异(P<0.05)
Note: The means with different letters are significant differences at the 0.05 probability level in the test groups
图6 qRT-PCR检测Aj-ghitm mRNA表达的变化
Fig.6 mRNA expression levels of Aj-ghitm detected by qRT-PCR
GHITM是一种受GH诱导的跨膜蛋白,自Li等[1]2001年首先在小鼠体组织发现以来,在人、兔、鸡和斑马鱼等脊椎动物物种中也发现了它的存在。但在无脊椎动物中,除太平洋牡蛎、紫海胆和海葵等模式生物的大规模测序结果中发现有相关基因存在[14-16]外,仅Yoshida等[2]报道了家蚕的GHITM,认为其是家蚕前胸腺源受体(PGDR)的同源物,目前尚未见有关其他无脊椎动物ghitm基因的克隆与其蛋白功能研究的报道。本研究中成功地克隆到了棘皮动物仿刺参ghitm的全长序列(Aj-ghitm),预测编码蛋白Aj-GHTIM包含了一个典型的GHITM结构域(cd10341),与其他动物GHITM氨基酸的相似度较高,具有较高的同源性。基于不同物种GHITM构建的系统进化树分析显示,Aj-GHTIM与同属棘皮动物门的紫海胆GHITM聚为一支,与海葵、人和小鼠分别形成不同分支。在系统进化过程中,通常物种分类地位越接近,同源物的氨基酸相似度越高,系统发育地位越接近,Aj-ghitm的系统发育地位也符合这一规律。本研究是仿刺参ghitmcDNA全长克隆与序列分析的首次报道,也是国际上有关棘皮动物ghitm的首次报道。
基于相似序列构建的系统进化树分析还显示,Aj-GHITM所在的GHITM家族属于BI-1样超家族,与BI-1、GAAP、LFG和RECS等其他几种多跨膜结构蛋白家族一起,分别形成了该样超家族的5大分支,这与Reimers等[5]对BI-1样超家族的系统发育分析结果一致。BI-1样超家族是一类古老的凋亡抑制蛋白,通过促进BCL-2或抑制BAX的作用,而抑制细胞凋亡的发生,在细胞凋亡和肿瘤发生过程中发挥重要作用[9]。生物信息学分析发现,BI-1样超家族成员都具有一个保守的跨膜结构域,以及复杂多样的跨膜区蛋白的N-末端与C-末端[17]。值得注意的是,TMpred在线分析结果显示,Aj-GHTIM有8个跨膜区域,这与Reimers等[5]认为GHITM为6个跨膜蛋白的结论存在一定差异。这种差异可能是由于棘皮动物与脊椎动物之间的亲缘关系较远造成的。保守跨膜结构域的存在证明其对GHITM生物学功能的重要性,而不同物种GHITM跨膜次数的差异则反映了其生物学功能的多样性。
GHITM最早发现于患侏儒症的小鼠脂肪组织中,因其表达受GH诱导而得名[1]。后来发现,GHITM介导了生长激素/胰岛素样生长因子1(GH/IGF1)轴对动物中枢神经系统的神经保护作用[18]。Nagel等[19]研究发现,ghitm在禽类的T淋巴细胞中有分布,其mRNA表达受趋化因子的诱导调控。Koskela等[20]在研究鸡免疫系统发育时发现,ghitm有调控生长的作用,是B淋巴细胞的特异性基因之一。这些研究表明,GHITM有着复杂的生物学功能,在脊椎动物的生长发育、免疫系统调节等方面都有重要作用。
在无脊椎动物方面,家蚕的GHITM同源物PGDR参与了家蚕的蜕皮和变态发育过程,黑腹果蝇ghitmmRNA表达则受其衰老状态的激活调控[2]。熊涛等[21]研究发现,日本血吸虫肺期童虫ghitm表达较高,可能在日本血吸虫的生长发育中起关键作用。可见,无脊椎动物GHITM仍与动物生长调控密切相关。但由于无脊椎动物仿刺参不具备像脊椎动物那样复杂的内分泌系统,也未发现生长激素或类似物存在,无脊椎动物GHITM可能不是通过GH的诱导来发挥作用,而是通过调控细胞凋亡来发挥作用,但其具体作用机制尚待进一步研究。Henke等[22]研究表明,GHITM可稳定线粒体细胞色素C,抑制后者的凋亡释放,从而维持线粒体的正常结构。Kim等[17]采用细胞转染方法研究了BI-1样超家族成员的抗凋亡作用,发现包括GHITM在内的所有家族成员都可抑制抗肿瘤药物依托泊苷或应激诱导的细胞凋亡,证实了BI-1样超家族成员抗凋亡功能的保守性[5]。本研究中发现,LPS刺激可诱导仿刺参体腔细胞ghitmmRNA的表达变化,表达量在24 h时升到最高,48 h时回复到正常水平附近。LPS是革兰氏阴性细菌细胞壁的组成成分,是一种重要的病原相关模式分子,能够引发免疫细胞形态、功能和基因表达的变化。Aj-ghitm的表达变化暗示其参与了仿刺参抗细菌感染和天然免疫防御反应。笔者推测,仿刺参可能通过Aj-ghitm启动某种信号传导途径,激活免疫应答反应,或者通过某种途径启动Aj-ghitm的表达变化,改变其抗凋亡活性,从而调控细胞凋亡的发生,实现抗感染作用。Aj-ghitm的具体作用与调控机制将是下一步研究的方向。
参考文献:
[1] Li Y,Kelder B,Kopchick J J.Identification,isolation,and cloning of growth hormone (GH)-inducible interscapular brown adipose complementary deoxyribonucleic acid from GH antagonist mice[J].Endocrinology,2001,142(7):2937-2945.
[2] Yoshida T,Nagata S,Kataoka H.Ghitmis an ortholog of theBombyxmoriprothoracic gland-derived receptor that is ubiquitously expressed in mammalian cells and requires an N-terminal signal sequence for expression[J].Biochemical and Biophysical Research Communications,2006,341(1):13-18.
[3] Knapp J R,Chen W Y,Turner N D,et al.Growth patterns and body composition of transgenic mice expressing mutated bovine somatotropin genes[J].Journal of Animal Science,1994,72(11):2812-2819.
[4] Zou S,Meadows S,Sharp L,et al.Genome-wide study of aging and oxidative stress response inDrosophilamelanogaster[J].Proceedings of the National Academy of Sciences,2000,97(25):13726-13731.
[5] Reimers K,Choi C Y U,Bucan V,et al.The growth-hormone inducible transmembrane protein (Ghitm) belongs to the Bax inhibitory protein-like family[J].International Journal of Biological Sciences,2007,3(7):471-476.
[6] Xu Q,Reed J C.Bax inhibitor-1,a mammalian apoptosis suppressor identified by functional screening in yeast[J].Molecular Cell,1998,1(3):337-346.
[7] Holzerlandt R,Orengo C,Kellam P,et al.Identification of new herpesvirus gene homologs in the human genome[J].Genome Research,2002,12(11):1739-1748.
[8] Somia N V,Schmitt M J,Vetter D E,et al.LFG:an anti-apoptotic gene that provides protection from Fas-mediated cell death[J].Proceedings of the National Academy of Sciences,1999,96(22):12667-12672.
[9] Reimers K,Choi C Y U,Bucan V,et al.The Bax inhibitor-1 (BI-1) family in apoptosis and tumorigenesis[J].Current Molecular Medicine,2008,8(2):148-156.
[10] 蔡慈峰,吴明江,廖志勇,等.RECS1负调控肿瘤坏死因子受体 2 介导的 NF-κB 激活[J].中国生物化学与分子生物学报,2010(1):36-42.
[11] 杨爱馥,周遵春,董颖,等.仿刺参 cytb 和β-actin基因表达稳定性比较[J].中国农业科技导报,2010,12(1):79-84.
[12] 孙国华,吉成龙,杨建敏,等.刺参 (Apostichopusjaponicus) 高温胁迫应答基因应激表达特性的分析[J].海洋与湖沼,2013 (1):182-188.
[13] Livak K J,Schmittgen T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2-△△CTMethod[J].Methods,2001,25(4):402-408.
[14] Zhang G,Fang X,Guo X,et al.The oyster genome reveals stress adaptation and complexity of shell formation[J].Nature,2012,490:49-54.
[15] Hibino T,Loza-Coll M,Messier C,et al.The immune gene repertoire encoded in the purple sea urchin genome[J].Developmental Biology,2006,300(1):349-365.
[16] Putnam N H,Srivastava M,Hellsten U,et al.Sea anemone genome reveals ancestral eumetazoan gene repertoire and genomic organization[J].Science,2007,317:86-94.
[17] Kim J H,Lee E R,Jeon K,et al.Role of BI-1 (TEGT)-mediated ERK1/2 activation in mitochondria-mediated apoptosis and splenomegaly in BI-1 transgenic mice[J].Biochimica Biophysica Acta (BBA)-Molecular Cell Research,2012,1823(4):876-888.
[18] Harvey S,Baudet M L.Proteomic actions of growth hormone in the nervous system[M]//Genomics,Proteomics,and the Nervous System.New York: Springer,2011:137-158.
[19] Nagel J E,Smith R J,Shaw L,et al.Identification of genes differentially expressed in T cells following stimulation with the chemokines CXCL12 and CXCL10[J].BMC Immunology,2004,5(1):17-30.
[20] Koskela K,Kohonen P,Nieminen P,et al.Insight into lymphoid development by gene expression profiling of avian B cells[J].Immunogenetics,2003,55(6):412-422.
[21] 熊涛,刘镕,赵琴平,等.日本血吸虫生长激素诱导的跨膜蛋白(GHITM)基因克隆与表达[J].微循环学杂志,2012(4):66.
[22] Henke N,Lisak D A,Schneider L,et al.The ancient cell death suppressor BAX inhibitor-1[J].Cell Calcium,2011,50(3):251-260.
Abstract:The full-length cDNA sequence ofghitmwere firstly cloned by RACE technique from coelomocytes of sea cucumberApostichopusjaponicusaccording to the EST sequence previously obtained from Genbank, and we named it asAj-ghitm(GenBank:KC886718). The full-length cDNA ofAj-ghitmwas 1325 bp, containing a 5′UTR of 140 bp and a 3′UTR of 180 bp, with an open reading frame of 1005 bp encoding 334 amino acids. SMART analysis showed that the deduced protein has an eight-transmembrane domain. Multiple alignment and phylogenic analysis showed thatAj-GHITM and the sea urchin GHITM were closely clustered in one group with the highest similarity of 65.4%. Quantitative real-time PCR revealed thatAj-ghitmmRNA expression level was decreased first and then increased, finally recovery to the normal level after LPS-induced, with the maximum expression 24 h followed by a lower expression. The findings give a reference for further study of the role of GHITM in the development and anti-bacterial infection in sea cucumber.
Key words:Apostichopusjaponicus;Aj-ghitm; gene cloning; gene expression
DOI:10.3969/J.ISSN.2095-1388.2014.06.001
文章编号:2095-1388(2014)06-0543-07
收稿日期:2014-03-18
基金项目:国家“十二五”科技支撑计划项目(2011BAD13B03);海洋公益性行业科研专项(201405003)
中图分类号:Q954.4
文献标志码::A