基于线粒体控制区部分序列的大青鲨种群遗传结构研究
郑真真1,许强华1、2,戴小杰1、2,朱江峰1、2
(1.上海海洋大学 海洋科学学院,上海 201306;2.大洋渔业资源可持续开发省部共建教育部重点实验室,农业部大洋渔业资源环境科学观测实验站,国家远洋渔业工程技术研究中心,上海 201306)
摘要:基于线粒体DNA(mtDNA)控制区部分序列对采集自太平洋、大西洋、印度洋的5个大青鲨Prionaceglauca群体165尾成鱼样本进行遗传多样性与遗传结构分析。结果表明:165尾大青鲨的mtDNA控制区片段长为694 bp,共得到110个变异位点,定义了145个单倍型,碱基组成中A为33.46%,T为36.40%,C为18.61%,G为11.53%,G+C含量为30.14%;单倍型多样性指数(Hd)在各个采样点都较高,平均值为0.997 3±0.001 4,核苷酸多样性指数(π)为 0.015 10±0.000 91,这表明大青鲨群体的遗传多样性水平很高,遗传资源丰富;分子方差分析表明,99.28%的遗传变异出现在种群内,0.72%的遗传变异来自于种群间;采用邻接法构建的系统发育树表明,5个群体间未形成显著的遗传结构,群体间的高基因交流值(Nm)和低遗传分化指数(Fst)揭示了三大洋的大青鲨群体间基因交流频繁,不存在显著的遗传分化。
关键词:大青鲨;种群遗传结构;线粒体DNA;控制区
大青鲨Prionaceglauca是一种大型中上层鲨鱼,隶属于真鲨目Carcharhiniformes、真鲨科Carcharhinidae、大青鲨属PrionaceCantor,是3种最丰富的大洋性鲨鱼之一[1]。作为一种大型的软骨鱼类,大青鲨广泛分布于太平洋、大西洋、印度洋(简称三大洋)海域,其分布范围最北可达挪威,最南可达智利。大青鲨在太平洋主要分布于20°N~20°S,在大西洋主要分布于从纽芬兰到阿根廷的东部海域,以及从挪威到南非的西部海域[2-3]。根据大西洋金枪鱼养护委员会(ICCAT)1990—2010年的统计数据,大青鲨在金枪鱼的兼捕种类中占的比重最大,大青鲨数量正在急剧下降,已引起了ICCAT的重点关注[4]。结合当前的捕捞情况和资源现状,保护大青鲨种群已成为渔业管理中一项重要任务。
线粒体DNA(mitochondrial DNA,mtDNA)呈共价闭合的环状双链,具有分子小、结构简单、严格遵守母系遗传的特点,能有效地揭示群体内外的遗传变异,现已成为鱼类分子生态学、遗传多样性研究的重要标记[5-6]。线粒体控制区(D-loop)具有较高的突变和累积,是研究种内遗传分化的重要工具,被广泛应用于各物种的遗传分化和遗传结构研究[7]。陈骁等[8]利用线粒体控制区对中国南部海域条纹斑竹鲨Chiloscylliumplagiosum的遗传多态性进行了研究,结果表明,条纹斑竹鲨的基因流主要在浅海近岸水域扩散,遗传变异程度受地理结构和距离隔离的影响;Mendonca等[9]采用COⅠ基因部分序列和D-loop部分序列评估大西洋西部斜锯牙鲨Rhizoprionodonacutus种群核苷酸的差异,结果表明,大西洋斜锯牙鲨和加勒比斜锯牙鲨两个不同物种之间存在较高水平的遗传差异;Keeney等[10]利用线粒体控制区对大西洋墨西哥湾169个黑鳍真鲨Carcharhinuslimbatus幼体样本的遗传异质性进行了相关研究,结果表明,该海域的黑鳍真鲨并没有出现明显的种群遗传结构。
近年来,不少学者对大青鲨的繁殖生物学特征[11]、资源状况[12-15]、行为特性[16-18]、栖息环境[19]和生长[4]等进行了研究,为大青鲨群体的资源保护和可持续发展提供了科学资料。戴小杰等[20]对东太平洋热带海域大青鲨繁殖生物学特征的研究表明,大青鲨是繁殖能力很强的大型鲨鱼之一。大青鲨为季节性的高度洄游性种类,对其的标记回捕记录和渔获数据表明,南北大西洋大青鲨为同一种群。利用线粒体DNA非编码区序列和微卫星标记分析显示,北大西洋东西部的大青鲨种群也无显著性差异[21-23]。但迄今为止,全球范围内对大青鲨的遗传多样性以及群体遗传结构等方面的研究尚未见报道。本研究中,以三大洋中大青鲨的5个采样群体为研究对象,通过分析其mtDNA的D-loop区部分序列,分析其遗传多样性和种群遗传结构,并对其地理分布进行初步探讨,以期为全球大青鲨资源的合理开发、管理和保护提供科学依据。
1材料与方法
1.1材料
试验用165尾大青鲨成鱼的肌肉组织样本取自2010年9月—2012年2月中国科学观察员在三大洋公海海域金枪鱼延绳钓渔船的渔获物。该船作业期间在赤道附近海域有5个采样位点,分别为中东太平洋(Central East Pacific,CEP)、中西太平洋(Central West Pacific,CWP)、西南大西洋(Southwest Atlantic,SWA)、中东大西洋(Central East Atlantic,CEA)和印度洋(Indian Ocean,INO),采样位点及采样时间见表1。剪取大青鲨肌肉组织(约20 g)浸入无水乙醇中,于冰箱(-20 ℃)中保存,带回实验室后保存于冰箱(-80 ℃)中备用。
1.2方法
1.2.1 基因组DNA的提取 取每尾大青鲨50~100 mg肌肉组织剪碎,使用组织基因组DNA快速提取试剂盒(北京艾德莱生物科技有限公司)提取大青鲨基因组DNA。用10 g/L琼脂糖凝胶电泳检测基因组DNA的完整性,用紫外分光光度计检测提取的DNA浓度和纯度。
表1大青鲨的采样位点、采样时间
Tab.1ThesamplinglocationsanddateforbluesharkPrionaceglauca
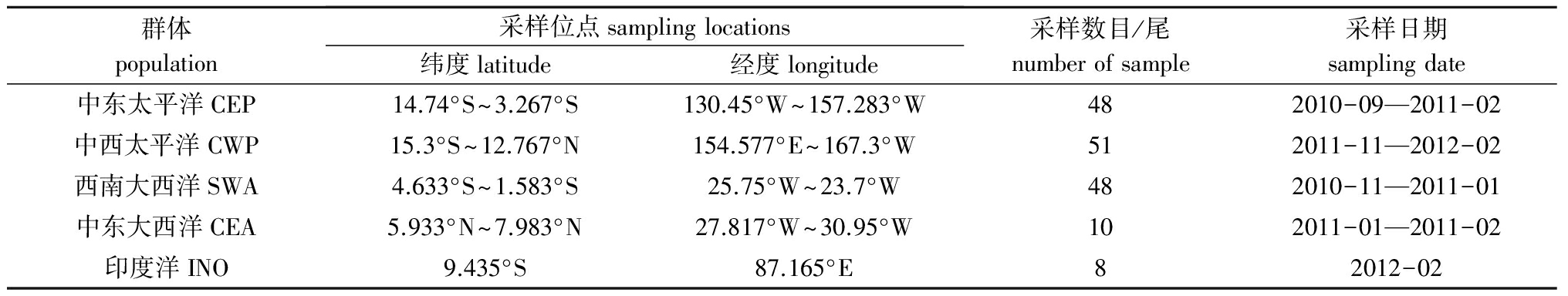
群体population采样位点samplinglocations纬度latitude经度longitude采样数目/尾numberofsample采样日期samplingdate中东太平洋CEP14.74°S~3.267°S130.45°W~157.283°W482010-09—2011-02中西太平洋CWP15.3°S~12.767°N154.577°E~167.3°W512011-11—2012-02西南大西洋SWA4.633°S~1.583°S25.75°W~23.7°W482010-11—2011-01中东大西洋CEA5.933°N~7.983°N27.817°W~30.95°W102011-01—2011-02印度洋INO9.435°S87.165°E82012-02
1.2.2 D-loop区基因的 PCR扩增和测序 以大青鲨基因组DNA为模板,随机抽取3个大青鲨样本直接采用引物5′ TTGGCTCCCAAAGCCAARATTCTG 3′(正向)、5′GTGGCTTAGCAAGARGTCTTG 3′(反向)(上海杰李生物技术有限公司),扩增出的D-loop区基因片段长约1100 bp,PCR扩增体系(共20 μL):Taq聚合酶0.2 μL,10×buffer 2.0 μL,dNTP 1.6 μL,正反向引物各1 μL(10 pmol/μL),模板1.0 μL,用双蒸水ddH2O补足至20 μL。PCR反应条件:95 ℃下预变性7 min;95 ℃下变性45 s,56 ℃下退火60 s,72 ℃下延伸90 s,共进行35个循环;最后在72 ℃下再延伸7 min,4 ℃下保存。PCR扩增产物用10 g/L琼脂糖凝胶电泳分离,得到目的片段,送交生工生物工程(上海)股份有限公司测序。根据3个样本的测序结果,自行设计另外一对引物:PGCR-F,5′ TGCACGTTAGTACCACGCTAA 3′;PGCR-R,5′ AACTGGCCCTACATAACCTGC 3′。扩增出的D-loop基因片段长约700 bp,PCR扩增体系同上。PCR反应条件:95 ℃下预变性3 min;95 ℃下变性30 s,55 ℃下退火30 s,72 ℃下延伸60 s,共进行30个循环;最后在72 ℃下再延伸10 min,4 ℃下保存。送交生工生物工程(上海)股份有限公司测序,得到165个大青鲨样本的序列片段。
1.2.3 数据分析 测序结果先经Blast程序进行序列比对检索,结合DNA Star软件检测其同源性,使用BioEdit软件进行控制区序列的对位排列(alignment)[24],并对序列辅以人工校对、编辑和比对,确保序列无误。采用Mega 4.0软件[25]进行序列分析,包括序列长度、碱基组成和亲缘关系树的构建,大青鲨单倍型系统发育树的构建采用邻接法(Neighbor-joining,NJ)[26-27],序列间的遗传距离以Kimura双参数法(Kimura 2-parameter)[28]进行估计。系统发育树分支的置信度采用自引导法(bootstrap analysis,BP)[29]重复检测(1000次重复)。应用DnaSP 4.0软件[30]计算单倍型多样性指数(Hd)与核苷酸多样性指数(π)。应用Arlequin 3.0 软件[31]进行分子方差分析(AMOVA),其中,采用单倍型频率计算Fst值,根据公式M=1/4(1/Fst-1)计算基因流Nm。
2结果与分析
2.1mtDNAD-loop区基因序列特征
经PCR扩增后,获得165个长度为694 bp的线粒体控制区(D-loop区)的碱基序列,序列中各碱基在DNA双链中平均所占的比例:A(腺嘌呤)为33.46%,T(胸腺嘧啶)为36.40%,C(胞嘧啶)为18.61%,G(鸟嘌呤)为11.53%,G+C含量为30.14%,仅占A+T含量的二分之一,大青鲨mtDNA D-loop区基因片段中G含量都比较低,平均含量为11.53%(表2),明显低于其他3种碱基的含量,表现出显著的碱基组成偏向性。
表2大青鲨mtDNAD-loop区基因序列的碱基组成
Tab.2NucleotidecompositionsofcontrolregionsequenceofmitochondrialDNAinblueshark
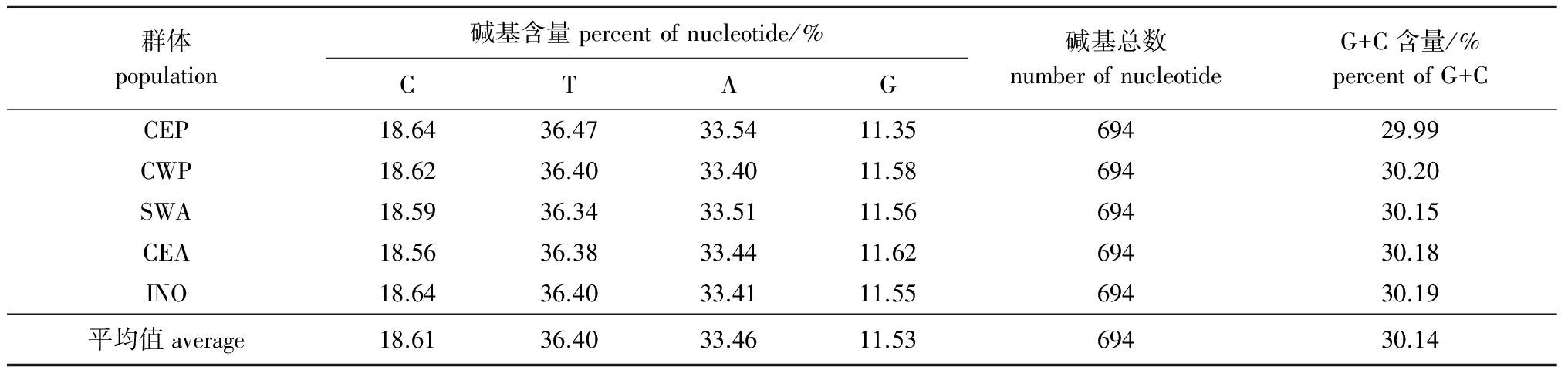
群体population碱基含量percentofnucleotide/%CTAG碱基总数numberofnucleotideG+C含量/%percentofG+CCEP18.6436.4733.5411.3569429.99CWP18.6236.4033.4011.5869430.20SWA18.5936.3433.5111.5669430.15CEA18.5636.3833.4411.6269430.18INO18.6436.4033.4111.5569430.19平均值average18.6136.4033.4611.5369430.14
2.2遗传多样性和单倍型分布
在165尾大青鲨mtDNA D-loop区域的694 bp序列中共检测到110个变异位点,变异率为15.85%,定义145个单倍型,分别为H1~H145。5个海域的大青鲨群体mtDNA D-loop统计结果显示,165尾个体的单倍型多样性指数为0.997 3±0.001 4,核苷酸多样性指数为0.015 10±0.000 91(表3)。165尾大青鲨个体中共检测到145种单倍型。除了CEA群体拥有自己特有的单倍型,其他群体都拥有共享单倍型。SWA和CWP群体同时拥有单倍型H24,而单倍型H66、H71、H76、H78、H79同时出现在CWP和CEP群体,单倍型H79、H92同时出现在CEP和INO群体(表4)。这些大量存在的共享单倍型表明,全球大青鲨群体间存在较强的基因交流。
表3大青鲨群体线粒体DNA控制区序列的遗传多样性参数
Tab.3GeneticdiversityparametersofbluesharkbasedoncontrolregionsequencedataofmitochondrialDNA

群体population样本数number/尾单倍型数haplotypenumber/尾单倍型多样性haplotypediversity核苷酸多样性nucleotidediversitySWA48460 998±0 0050 01602±0 00152CEA10101 000±0 0450 01654±0 00194CEP48430 995±0 0060 01333±0 00150CWP51470 997±0 0050 01693±0 00214INO881 000±0 0630 01688±0 00459合计total1651450 9973±0 00140 01510±0 00091
表4各采样位点的大青鲨单倍型分布
Tab.4Haplotypedistributionofbluesharksampledatvariouslocalities

群体population采样点单倍型分布haplotypedistributionamongsamplinglocalitiesSWAH1~H46CEAH47~H56CEPH57~H98CWPH24、H66、H71、H76、H78、H79、H99~H139INOH79、H92、H140~H145
2.3系统发育树
对大青鲨145个单倍型采用邻接法构建系统发育树。结果显示:各单倍型广泛分布在单倍型邻接树上,未形成明显的分支,不存在显著的谱系结构。基于线粒体DNA D-loop区构建的单倍型间系谱关系显示:145种单倍型只形成单一的网络,各单倍型间呈现高水平的平行演化(由于145个单倍型未形成明显分支,不存在显著谱系结构,故图略)。
2.4群体遗传结构分析
用分子方差分析(AMOVA)法对群体间的遗传结构进行分析,结果显示,99.28%的遗传变异出现在种群内,而0.72%的遗传变异出现在种群间;群体间的遗传分化指数Fst为0.007 21(表5),说明群体间不存在显著的遗传分化。
表5大青鲨种群的分子方差分析
Tab.5AMOVAanalysisofbluesharkpopulations
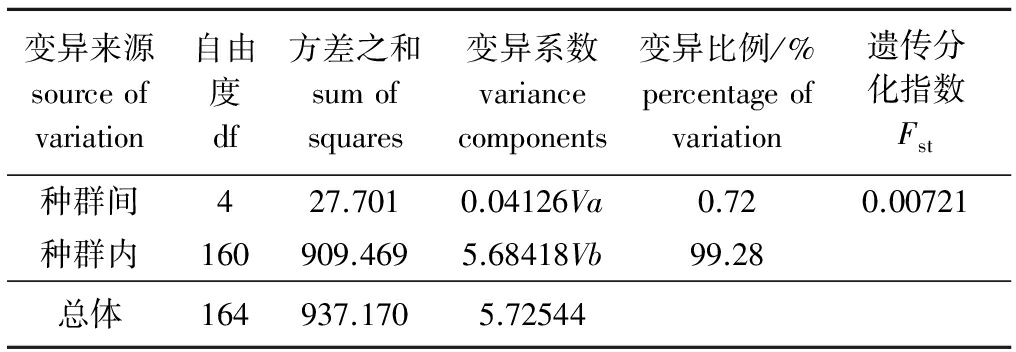
变异来源sourceofvariation自由度df方差之和sumofsquares变异系数variancecomponents变异比例/%percentageofvariation遗传分化指数Fst种群间427.7010.04126Va0.720.00721种群内160909.4695.68418Vb99.28总体164937.1705.72544
利用Fst进一步检验大青鲨群体间的遗传结构,根据两两群体间的Fst值可以发现,所有的Fst值均较小,除SWA群体与CEP群体间统计检验极显著(P=0.009<0.01)外,其他任何两个群体间的统计检验均不显著(P>0.05)。本研究结果表明,除西南大西洋与中东太平洋的大青鲨群体间存在着显著的遗传分化外,其他采样区域内的大青鲨群体均不存在显著的遗传结构。除此之外,5个采样点间大青鲨的基因交流(Nm)也比较频繁,CWP群体与CEA群体间Nm最小,为11.83(表6)。
表6采样区域大青鲨种群间的遗传分化分析及基因流Nm
Tab.6FstanalysisandNmvaluesofbluesharkpopulationsinvarioussamplingregions
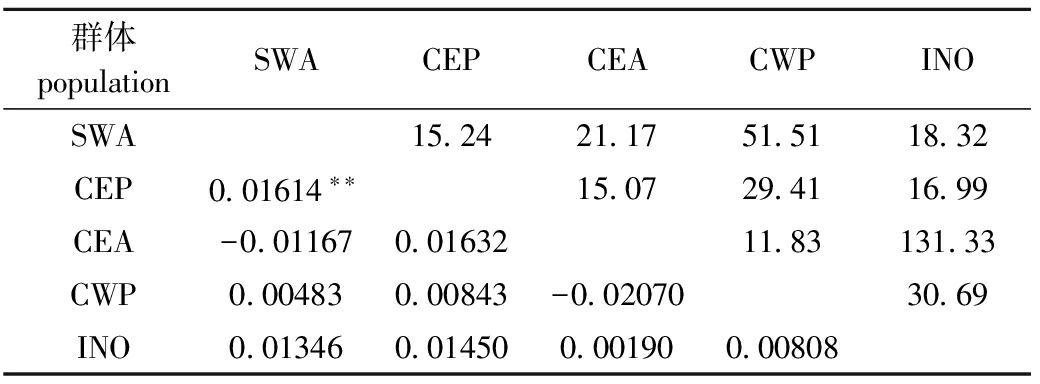
群体populationSWACEPCEACWPINOSWA15 2421 1751 5118 32CEP0 01614∗∗15 0729 4116 99CEA-0 011670 0163211 83131 33CWP0 004830 00843-0 0207030 69INO0 013460 014500 001900 00808
注:下三角为Fst值, 上三角为Nm值;**表示有极显著性差异(P<0.01);P值采用1000次重复来进行估计
Note:The lower triangle givesFstvalues,and the triangle showsNmvalues;** denotes very significant difference(P<0.01);Pvalues are estimated by 1000 repetitions
3讨论
大青鲨是分布最为广泛、种群数量最多的一类大洋性鲨鱼[32],尽管大洋交界处会有不同地理种群的个体混合,但标志重捕研究表明,大青鲨的运动范围局限在其本身所在的海域,各海域间大青鲨的基因交流很少[33]。一般认为:群体之间的Nm<1时,说明群体可能由于遗传漂变而发生了分化;而Nm>1时,则表明群体间的基因流水平较高,群体间遗传分化较小;当Nm>4时,表明种群间的基因交流更为充分,遗传分化更小[34]。本研究中得到的Nm值均比4大得多,这说明大青鲨作为一种高度洄游的物种,它们之间的基因交流非常频繁。根据ICCAT的研究结果[35],大西洋的大青鲨至少存在3个不同的群体,分别为北大西洋、南大西洋和地中海群体。但Kohler等[21]、Shivji等[22]、Lessa等[23]根据线粒体DNA控制区序列及微卫星标记分析得出,采样区域内北大西洋的东部和西部大青鲨种群不存在遗传分化,这与本研究结果相符。对北大西洋和南大西洋大青鲨种群进行的微卫星分析,也证实了北大西洋和南大西洋种群之间存在的遗传差异[36]。由于受到北美洲大陆的地理性隔离,北大西洋种群和北太平洋种群被认为是两种不同的群体。对北大西洋大青鲨的标志重捕研究,显示了大青鲨的季节性纬度运动特点[37-38],释放点的大青鲨向东、西两个方向上的迁移都很常见,但跨越赤道的运动却极少,结果为“北大西洋大青鲨为单一种群”这一假说提供了证据。此外, Karl等[39]采用线粒体控制区测定技术和微卫星技术对大西洋西部低鳍真鲨种群的遗传关系进行了相关研究,表明相毗邻的种群之间没有显著的遗传差异,且遗传多样性水平较低;Hoelzel等[40]检测了采集自大西洋西北部、大西洋东北部、地中海、印度洋和西太平洋姥鲨Cetorhinusmaximus的mtDNA控制区的多样性,发现其遗传变异度相当低,不同海域间没有太大的区别。本研究结果也显示,大青鲨种群的遗传差异不显著,其原因可能是由于软骨鱼类的DNA进化速率比其他鱼类慢造成的,同时大青鲨具有生长速度慢、性成熟晚、繁殖周期长、繁殖率低等生物特征,而且大青鲨在大西洋中又是金枪鱼延绳钓的主要兼捕对象,所以捕捞压力对于大青鲨种群的遗传分化造成了较大的影响。
遗传多样性是生物多样性的重要组成部分,是保护生物学研究的核心之一,对遗传多样性的研究具有重要的理论和实践意义[41]。单倍型多样性和核苷酸多样性是衡量一个物种或种群线粒体DNA变异程度的两个重要指标[42]。Martin[43]在比较了鲨鱼和哺乳动物线粒体序列后发现,鲨鱼的线粒体进化速率远低于哺乳动物和许多高等脊椎动物,即使是公认进化速率最快的控制区,其进化速率也比其他脊椎动物低。Duncan等[44]对采自全球各大洋20个海域的27l尾路氏双髻鲨Sphyrnalewini线粒体控制区序列进行分析时,仅发现22个多态位点,24个单倍型,表明路氏双髻鲨线粒体DNA变异程度较低。但本研究中对三大洋5个海域的165尾大青鲨线粒体D-loop区序列进行分析时,发现了110个多态位点,145个单倍型,分子方差分析得到Fst值为0.007 21。当0<Fst<0.05时,表明群体遗传分化水平较弱,因此,作者认为,大青鲨的线粒体DNA(D-loop区)具有较高的变异水平,只是由于不同群体之间强烈的基因交流,导致其遗传分化水平较低。
迄今为止,对大青鲨种群遗传结构的研究多数是基于标记重捕技术开展的。利用线粒体DNA标记等分子手段对大青鲨种群进行的遗传结构研究很少,并且仅集中在大西洋。国内外利用线粒体DNA标记对印度洋和太平洋大青鲨种群遗传结构的研究尚处于空白,这在很大程度上限制了对全球大青鲨种群遗传结构的全面认识,从而影响到对大青鲨种群的有效保护和管理。本研究是首次利用线粒体D-loop分子标记对三大洋的大青鲨群体遗传结构进行的研究,为大青鲨资源的管理和保护提供了一定的理论依据。但本研究中5个海域的采样样本数量相对不足,需要在今后的研究中进一步增加采样点和样本数,以期获得更准确的数据。
参考文献:
[1] Mejuto J,García-Cortés B.Reproductive and distribution parameters of the blue sharkPrionaceglauca,on the basis of on-board observations at sea in the Atlantic,Indian and Pacific oceans[J].Collect Vol Sci Pap ICCAT,2005,58(3):974-1000.
[2] Nakano H.Age,reproduction and migration of blue shark in the North Pacific Ocean[J].Bulletin-National Research Institute of Far Seas Fisheries,1994,31:141-190.
[3] Tavares R,Ortiz M,Arocha F.Population structure,distribution and relative abundance of the blue shark(Prionaceglauca)in the Caribbean Sea and adjacent waters of the North Atlantic[J].Fisheries Research,2012,129:137-152.
[4] 高春霞,田思泉,戴小杰,等.热带中东大西洋拟锥齿鲨生物学的初步研究[J].上海海洋大学学报,2013,22(2):289-294.
[5] 郭新红,刘少军,刘巧,等.鱼类线粒体 DNA 研究新进展[J].遗传学报,2004,31(9):983-1000.
[6] 闫杰,许强华,陈新军,等.东太平洋公海茎柔鱼种群遗传结构初步研究[J].水产学报,2012,35(11):1617-1623.
[7] 谢振宇,杜继曾,陈学群.线粒体控制区在鱼类种内遗传分化中的意义[J].遗传,2006,28(3):362-368.
[8] 陈骁,杨圣云,潘聪.我国南部海域条纹斑竹鲨线粒体 DNA 控制区遗传多态性研究[J].海洋学报,2009,30(6):115-121.
[9] Mendonça F F,Ussami L H F,Hashimoto D T,et al.Identification and characterization of polymorphic microsatellite loci in the blue sharkPrionaceglauca,and cross-amplification in other shark species[J].Journal of Fish Biology,2012,80(7):2643-2646.
[10] Keeney D B,Heupel M R,Hueter R E,et al.Microsatellite and mitochondrial DNA analyses of the genetic structure of blacktip shark(Carcharhinuslimbatus)nurseries in the northwestern Atlantic,Gulf of Mexico,and Caribbean Sea[J].Molecular Ecology,2005,14(7):1911-1923.
[11] 吴峰,戴小杰,姜润林.热带中东大西洋海域大青鲨繁殖生物学研究[J].海洋湖沼通报,2012(3):30-36.
[12] Lessa R P.Distribution and relative abundance of the blue shark,Prionaceglauca,in the southwestern equatorial Atlantic Ocean[J].Fishery Bulletin,1994,92:474-480.
[13] Montealegre-Quijano S,Vooren C M.Distribution and abundance of the life stages of the blue shark(Prionaceglauca)in the Southwest Atlantic[J].Fisheries Research,2010,101(3):168-179.
[14] Vas P.The abundance of the blue shark,Prionaceglauca,in the western English Channel[J].Environmental Biology of Fishes,1990,29(3):209-225.
[15] Zhu Jiangfeng,Dai Xiaojie,Xu Liuxiong,et al.Reproductive biology of female blue sharkPrionaceglaucain the southeastern Pacific Ocean[J].Environmental Biology of Fishes,2011,91(1):95-102.
[16] Sciarrotta T,Nelson D.Diel behavior of the blue shark,Prionaceglauca,near Santa Catalina Island[J].California Fish Bull,1977,75(3):519-528.
[17] Queiroz N,Lima F P,Maia A,et al.Movement of blue shark,Prionaceglauca,in the north-east Atlantic based on mark-recapture data[J].Journal of the Marine Biological Association of the United Kingdom,2005,85(5):1107-1112.
[18] Stevens,J D,Bradford R W,West G L.Satellite tagging of blue sharks(Prionaceglauca)and other pelagic sharks off eastern Australia:depth behaviour,temperature experience and movements[J].Marine Biology,2010,157(3):575-591.
[19] 宋利明,胡振新.马绍尔群岛海域大青鲨栖息地综合指数[J].水产学报,2011,35(8):1208-1216.
[20] 戴小杰,许柳雄.东太平洋热带海域大青鲨繁殖生物学特征[J].水产学报,2005,29(4):565-569.
[21] Kohler N E,Turner P A,Hoey J J,et al.Tag and recapture data for three pelagic shark species:blue shark(Prionaceglauca),shortfinmako(Isurusoxyrinchus),and porbeagle(Lamnanasus)in the north Atlantic ocean[J].Collect Vol Sci Pap ICCAT,2002,54(4):1231-1260.
[22] Shivji M,Clarke S,Pank M,et al.Genetic identification of pelagic shark body parts for conservation and trade monitoring[J].Conservation Biology,2002,16(4):1036-1047.
[23] Lessa R,Santana F M,Hazin F H.Age and growth of the blue shark(Prionaceglauca)(Linnaeus,1758)off northeastern Brazil[J].Fisheries Research,2004,66(1):19-30.
[24] Thompson J D,Gibson T J,Plewniak F,et al.The CLUSTAL_X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Research,1997,25(24):4876-4882.
[25] Kumar S,Tamura K,Nei M.MEGA3:integrated software for molecular evolutionary genetics analysis and sequence alignment[J].Briefings in Bioinformatics,2004,5(2):150-163.
[26] Ma B,Xin L,Zhang K Z.A new quartet approach for reconstructing phylogenetic trees:quartet joining method[J].Journal of Combinatorial Optimization,2008,16(3):293-306.
[27] Zhang W,Sun Z R.Random local neighbor joining:a new method for reconstructing phylogenetic trees[J].Molecular Phylogenetics and Evolution,2008,47(1):117-128.
[28] Kimura M A.Simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J].Journal of Molecular Evolution,1980,16(2):111-120.
[29] Felsenstein J.Confidence limits on phylogenies:an approach using the bootstrap[J].Evolution,1985:783-791.
[30] Rozas J,Juan C S,Messeguer X,et al.DnaSP,DNA polymorphism analyses by the coalescent and other methods[J].Bioinformatics,2003,19(18):2496-2497.
[31] Excoffier L,Smouse P E,Quattro J M.Analysis of molecular variance inferred from metric distances among DNA haplotypes:application to human mitochondrial DNA restriction data[J].Genetics,1992,131(2):479-491.
[32] Kubodera T,Watanabe H,Ichii T.Feeding habits of the blue shark,Prionaceglauca,and salmon shark,Lamnaditropis,in the transition region of the Western North Pacific[J].Reviews in Fish Biology and Fisheries,2007,17(2/3):111-124.
[33] Mandelman J W,Cooper P W,Werner T B,et al.Shark bycatch and depredation in the U.S.Atlantic pelagic longline fishery[J].Reviews in Fish Biology and Fisheries,2008,18(4):427-442.
[34] 乐小亮,赵爽,刘海林,等.基于线粒体细胞色素 b 基因全序列的海南长臀鮠南渡江种群遗传变异分析[J].生态科学,2010,29(3):247-250.
[35] Campana S E,Joyce W,Manning M J.Bycatch and discard mortality in commercially caught blue sharksPrionaceglaucaassessed using archival satellite pop-up tags[J].Marine Ecology Progress Series,2009,387:241-253.
[36] Fitzpatrick S,Shivji M S,Chapman D D,et al.Development and characterization of 10 polymorphic microsatellite loci for the blue shark,Prionaceglauca,and their cross shark-species amplification[J].Conservation Genetics Resources,2011,3(3):523-527.
[37] Stevens J.First results of shark tagging in the north-east Atlantic,1972-1975[J].Mar Biol Assoc UK,1976,56(4):929-937.
[38] Kohler N E,Turner P A.Shark tagging:a review of conventional methods and studies[J].Environmental Biology of Fishes,2001,60(1/3):191-224.
[39] Karl S A,Castro A L F,Lopez J A,et al.Phylogeography and conservation of the bull shark(Carcharhinusleucas)inferred from mitochondrial and microsatellite DNA[J].Conservation Genetics,2011,12(2):371-382.
[40] Hoelzel A R,Shivji M S,Magnussen J,et al.Low worldwide genetic diversity in the basking shark(Cetorhinusmaximus)[J].Biology Letters,2006,2(4):639-642.
[41] 尚占环,姚爱兴.生物遗传多样性研究方法及其保护措施[J].宁夏农学院学报,2002,23(1):66-69.
[42] Nei M.Molecular evolutionary genetics[J].Journal of Human Evolution,1989,18(8):808-810.
[43] Martin A P,Naylor G J,Palumbi S R.Rates of mitochondrial DNA evolution in sharks are slow compared with mammals[J].Nature,1992,357:153-155.
[44] Duncan K M,Martin A P,Bowen B W,et al.Global phylogeography of the scalloped hammerhead shark(Sphyrnalewini)[J].Molecular Ecology,2006,15(8):2239-2251.
PopulationgeneticstructureofbluesharkPrionaceglaucabasedonpartialcontrolregionsequenceinmitochondrialDNA
ZHENG Zhen-zhen1,XU Qiang-hua1,2,DAI Xiao-jie1,2,ZHU Jiang-feng1,2
(1.College of Marine Sciences,Shanghai Ocean University,Shanghai 201306,China;2.Key Laboratory of Sustainable Exploitation of Oceanic Fisheries Resources,Ministry of Education,Scientific Observing and Experimental Station of Oceanic Fishery Resources,Ministry of Agriculture,National Engineering Research Center for Oceanic Fisheries,Shanghai 201306,China)
Abstract:The sequence variations and population genetic structures were studied by partial fragments of mtDNA control region in 165 blue sharkPrionaceglaucasamples collected from 5 locations in Pacific, Atlantic and Indian oceans. A total of 145 mtDNA haplotypes and 110 variable sites were detected in the 694 bp fragments of the mitochondrial control region, with nucleotide composition of A: 33.46%, T:36.40%, C:18.61%, G:11.53%. The haplotype diversities were found to be high and the mean haplotype diversity was 0.997 3±0.001 4,while the nucleotide diversity was found to be 0.015 10±0.000 91.The AMOVA test revealed that there was 99.28% of the genetic variation within populations and 0.72% of the genetic variation among populations. The neighbor-joining tree revealed no significant genetic differentiation among five sampling locations. LowFstvalues and highNmvalues from this investigation indicated that there were no significant genetic differentiations among the five localities due to the high rates of gene flow between the sampled populations.
Key words:Prionaceglauca; population genetic structure; mitochondrial DNA; control region
DOI:10.3969/J.ISSN.2095-1388.2014.05.008
文章编号:2095-1388(2014)05-0463-06
收稿日期:2014-01-26
基金项目:国家自然科学基金重大研究计划培育项目(91131006);上海市教委科研创新项目曙光计划(13SG51)
作者简介:郑真真(1987—), 女, 硕士研究生。E-mail:zzzheng2013@126.com
通信作者:许强华(1974—), 女, 博士, 教授。E-mail:qhxu@shou.edu.cn
中图分类号:S917.4
文献标志码::A





