虾夷马粪海胆NLR家族基因片段的克隆及表达特征分析
陈亚东,刘晓飞,常亚青,仇雪梅,王秀利,刘洋
(大连海洋大学 农业部北方海水增养殖重点实验室,辽宁 大连 116023)
摘要:采用同源克隆和半定量RT-PCR方法,对虾夷马粪海胆StrongylocentrotusintermediusNLR家族基因片段进行了克隆和组织表达特征分析。将紫球海胆S.purpuratus的9组NLR家族聚类的编码区序列进行比对后,根据保守区域设计引物扩增虾夷马粪海胆中相应的基因,通过测序获得了9组不同NLR聚类基因片段,每组各10个测序结果,通过基因序列及氨基酸序列比对分析,发现存在长度及序列结果上的差异,这说明每组NLR家族聚类都存在多个家族成员。同时选取虾夷马粪海胆的不同组织提取RNA,反转录后使用同样的引物进行半定量RT-PCR分析,结果表明,NLR基因在虾夷马粪海胆的管足、肠、围口膜、体腔细胞、性腺中存在高效表达,且在围口膜及性腺中表达量最高,表明NLR基因在虾夷马粪海胆中普遍表达,并存在表达差异。研究表明,NLR基因在海胆免疫过程中起着重要作用。
关键词:虾夷马粪海胆;NLR家族;半定量RT-PCR
NLR家族(the nucleotide-binding domain and leucine-rich repeat containing gene family)属于模式识别受体[1-2]的重要一员,同源于植物抗病R蛋白,主要存在于细胞质内,识别进入细胞内的抗原物质,包括细菌组分、肽聚糖和核酸等[3]。目前已经有多种NLR分子被发现,它们都具有保守的结构域:N端可变区域、中间保守的NBD(nucleotide-binding domain)结构域和C端的LRR(leucine-rich repeats)结构域。LRR结构域的主要作用是识别包括细菌及真菌的一些细胞组分、病毒的核酸等[4-5]。N端可变区域主要作用是和其他蛋白结合,传递接收到的抗原信号,是效应区,但有一些NLR分子没有明显的N端结构域[6]。N端可变区域的不同变化对于NLR蛋白激活不同后续免疫过程具有重要意义。NBD结构域是NLR分子多聚化所需要的结构。
根据N端结构域的不同,NLR家族可分为五类:NLRA、NLRB、NLRC、NLRP 和NLRX[7]。这些NLR 家族蛋白分子位于细胞质内,识别特异的微生物组分,最终激活NF-κB信号通路,再调控表达IL-8、COX-2、IL-1β和抗菌肽等基因,进行免疫反应,以达到保护机体不受外来病原菌侵扰的目的[8]。紫球海胆Strongylocentrotuspurpuratus基因组内存在208个NLR家族基因,种类比人类NLR家族(20种)丰富得多[9]。Hibino等[10]对紫球海胆的 NLR 家族基因进行了聚类分析,并进一步划分出9组亚类,也进行了初步的实时定量表达分析。虾夷马粪海胆与紫球海胆基因组具有高度的同源性,有关紫球海胆的NLR家族基因的研究结果可为研究虾夷马粪海胆NLR家族基因提供参考。
虾夷马粪海胆Strongylocentrotusintermedius原产于日本北海道及以北沿海,在俄罗斯远东地区的萨哈林岛等地也有分布。大连海洋大学于1989年将其从日本引入中国,现已成为中国北方沿海特别是辽东和山东半岛重要的养殖种类, 养殖规模越来越大。海胆作为一种模式生物已经被广泛应用于基础生物学研究。目前,对海胆生物学的研究主要集中在繁殖育苗[11-12]、饵料、疾病和营养等应用研究,也有关于细胞学[13]、分子标记[14-16]和功能基因克隆与分析[17-18]等基础研究,但对虾夷马粪海胆免疫系统分子水平的相关研究在国内尚未见报道。NLR家族作为重要的免疫基因家族,其研究结果可以促进虾夷马粪海胆免疫系统分子机制的研究。本研究中克隆了虾夷马粪海胆NLR家族基因编码区的保守序列,分析了核酸和氨基酸序列的多样性,采用半定量RT-PCR技术检测了NLR基因在不同组织中的表达,以期为研究虾夷马粪海胆天然免疫系统中NLR分子功能奠定基础。
1材料与方法
1.1材料
试验用2龄虾夷马粪海胆取自大连海洋大学农业部北方海水增养殖重点实验室,海胆饲养于16~25 ℃的海水中,每日投喂新鲜海带。抽取体腔细胞前一日停止投喂。
1.2方法
1.2.1 RNA的制备及cDNA的合成 选取8枚健康海胆(雌、雄各4枚),分别使用注射器抽取虾夷马粪海胆的体腔液,在4 ℃下以5000 r/min离心10 min,收集体腔细胞,活体解剖虾夷马粪海胆,取其围口膜、肠、管足、雌雄性腺等组织,液氮冻存,于冰箱(-80 ℃)中保存备用。
按照EasyPureTMRNA Kit试剂盒(北京全式金生物技术有限公司)的方法提取各组织的总RNA,用凝胶电泳检测总RNA的质量,-80 ℃下保存备用。为了减少个体差异及性别对试验结果的影响,各组织均使用4枚海胆的RNA等量混合后用于反转录,其中精巢、卵巢各使用4枚雄性或雌性海胆,其他组织均使用2枚雌性和2枚雄性海胆,混合后分别使用cDNA第一链合成试剂盒(北京全式金生物技术有限公司)合成cDNA第一链。反转录产物于冰箱(-20 ℃)中保存,用于后续基因克隆及半定量分析。
1.2.2 引物设计 根据已报道的紫球海胆NLR家族基因的编码区序列[10],比对分析同一聚类的编码区的同源序列,根据保守区序列,应用Primer Premier 5.0软件设计引物11对,用于NLR基因部分序列的克隆和半定量RT-PCR分析。除NLR5、NLR6和内参18S rRNA引物为特异性引物外,其他为简并引物,其中,内参基因的引物根据GenBank中虾夷马粪海胆的18S rRNA序列设计(Accession:D14365)。所有引物由上海生工生物有限公司合成(表1)。
1.2.3 NLR部分基因片段的扩增及回收测序 虾夷马粪海胆NLR基因片段扩增的PCR反应体系(50 μL):ddH2O 36.5 μL;dNTP(各2.5 mmol/L)4 μL;10×Taq buffer 5 μL;上、下游引物(10 μmol/L)各1 μL;cDNA模板2 μL;Taq酶0.5 μL。PCR反应程序:94 ℃下预变性5 min; 94 ℃下变性30 s,退火30 s,72 ℃下延伸1 min,共进行35个循环;最后在72 ℃下再延伸10 min,4 ℃下保存。用10 g/L琼脂糖凝胶电泳检测PCR产物质量。最后用凝胶回收试剂盒(北京全式金生物技术有限公司)纯化回收目的条带,将纯化后的目的片段连接至PMD19-T载体中,然后将连接产物转化至DH5α感受态细胞中,在附加氨苄青霉素、IPTG、X-gal的LB固体培养基中培养(37 ℃)12~16 h,挑取白色单菌落小量培养,使用PMD19-T通用引物进行菌液PCR扩增,扩增体系和程序与上述相同,各选取10个有目的基因插入的阳性单克隆菌液送样测序。
1.2.4 生物信息学分析 将测序结果使用BlastX(http://blast.ncbi.nlm.nih.gov/Blast.cgi)软件进行比对,用NCBI的Conserved Domains(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)搜索并预测蛋白的保守区域,按照正确的阅读框进行翻译,对氨基酸序列进行比对,由Clustal X程序完成氨基酸序列多重比对分析。使用DNAStar软件进行基因及氨基酸序列相似性的比对计算。
1.2.5 半定量RT-PCR分析 对扩增得到基因片段进行比对,确认为虾夷马粪海胆中NLR家族的基因后,使用表1中引物,采用半定量RT-PCR法检测各组NLR基因的表达情况。选取虾夷马粪海胆18S rRNA作为内参基因。
根据预试验结果,PCR扩增基因片段时基因及内参基因的引物都可以在55 ℃下获得较好的效果,因此,半定量的退火温度统一选用了55 ℃;预试验中,内参基因选取了18、19、20、21、22共5个循环数,目的基因选取了26、28、30、32共4个循环数,以求获得达到较好扩增效果时的循环数,最终发现,内参基因在18个循环、目的基因在30个循环数时扩增效果最好。
以根据内参基因调整浓度一致后的6种组织的cDNA为模板进行PCR扩增。PCR扩增程序:94 ℃下预变性5 min;94 ℃下变性30 s,55 ℃下退火30 s,72 ℃下再延伸1 min,进行18或30个循环;最后在72 ℃下再延伸10 min,4 ℃下保存。用10 g/L琼脂糖凝胶进行电泳检测PCR产物的质量。
表1NLR基因克隆引物
Tab.1PrimersusedforcloningofNLRgenes
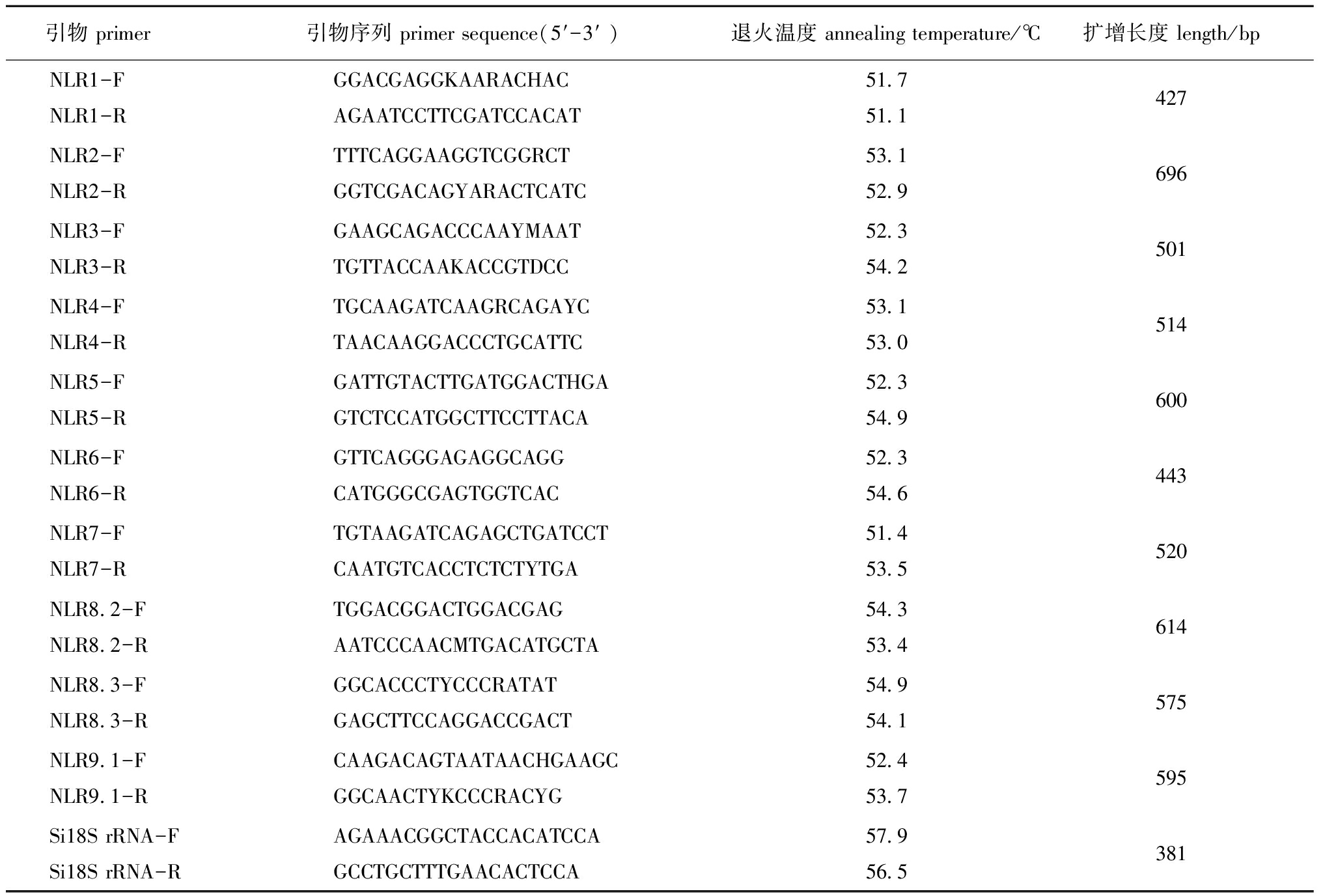
引物primer 引物序列primersequence(5′-3′) 退火温度annealingtemperature/℃扩增长度length/bpNLR1-FNLR1-RGGACGAGGKAARACHACAGAATCCTTCGATCCACAT51 751 1427NLR2-FNLR2-RTTTCAGGAAGGTCGGRCTGGTCGACAGYARACTCATC53 152 9696NLR3-FNLR3-RGAAGCAGACCCAAYMAATTGTTACCAAKACCGTDCC52 354 2501NLR4-FNLR4-RTGCAAGATCAAGRCAGAYCTAACAAGGACCCTGCATTC53 153 0514NLR5-FNLR5-RGATTGTACTTGATGGACTHGAGTCTCCATGGCTTCCTTACA52 354 9600NLR6-FNLR6-RGTTCAGGGAGAGGCAGGCATGGGCGAGTGGTCAC52 354 6443NLR7-FNLR7-RTGTAAGATCAGAGCTGATCCTCAATGTCACCTCTCTYTGA51 453 5520NLR8 2-FNLR8 2-RTGGACGGACTGGACGAGAATCCCAACMTGACATGCTA54 353 4614NLR8 3-FNLR8 3-RGGCACCCTYCCCRATATGAGCTTCCAGGACCGACT54 954 1575NLR9 1-FNLR9 1-RCAAGACAGTAATAACHGAAGCGGCAACTYKCCCRACYG52 453 7595Si18SrRNA-FSi18SrRNA-RAGAAACGGCTACCACATCCAGCCTGCTTTGAACACTCCA57 956 5381
注:字符R、Y、M、K和H代表简并碱基;R=A/G,Y=C/T,M=A/C,K=G/T, H=A/C/T
Note:R,Y,M,K and H represent degenerate bases;R=A/G,Y=C/T,M=A/C,K=G/T, H=A/C/T
2结果与分析
2.1NLR部分基因片段的克隆
经电泳检测获得了预期中的9组NLR基因聚类:NLR1、NLR3、NLR4、NLR5、NLR6、NLR7、NLR8.2、NLR8.3、NLR9。NLR2组基因扩增未得到结果。为探索各组NLR基因的序列多样性,本研究中各选取10个单克隆进行测序,获得各组NLR基因的部分序列。
2.2虾夷马粪海胆NLR基因部分序列的生物信息学分析
经过BlastX比对发现:克隆得到9组基因均含有NACHT和LRR and PYD domains-containing protein类似蛋白,保守区域均为NACHT结构;与紫球海胆NLR基因的同源性最高,与设计引物所使用的模板基因均达到70%以上的序列相似度。 将NLR基因核酸序列按照正确的阅读框进行翻译,获得了编码的氨基酸序列,并使用Clustal X 1.8软件对氨基酸序列进行序列比对,同时使用DNAStar软件分别进行核酸和氨基酸序列比对计算,相似性为100%。
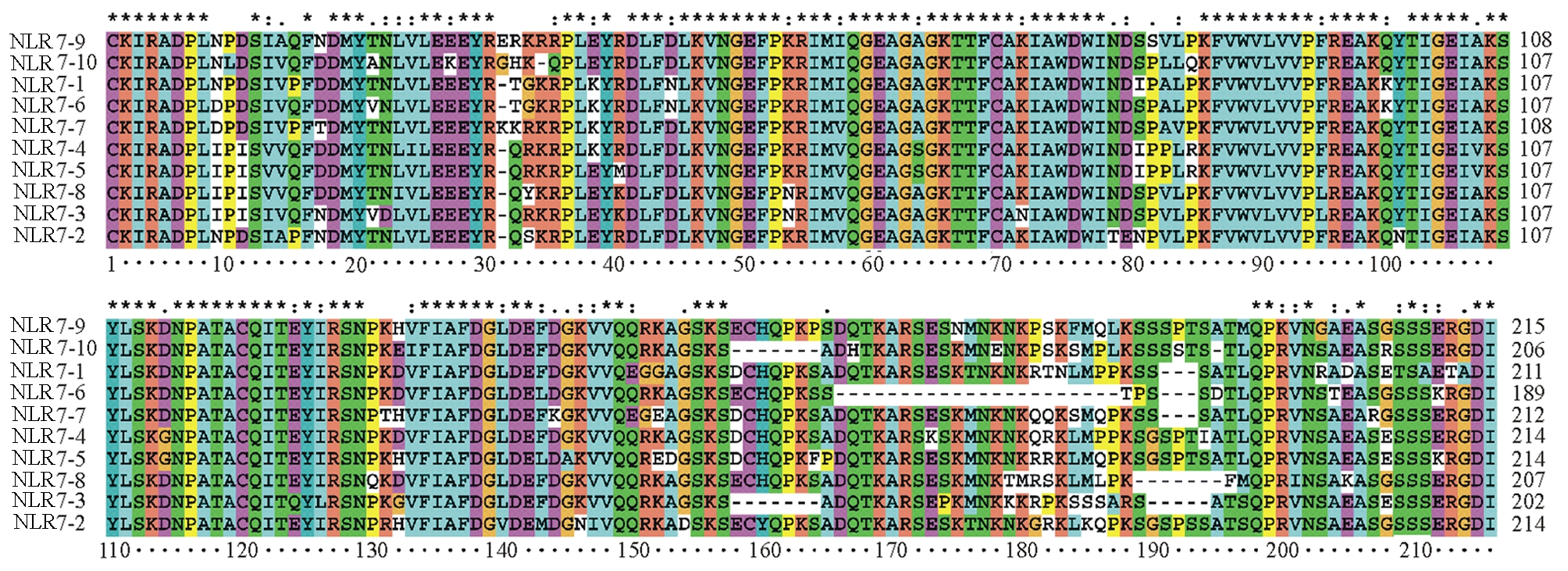
对测序结果分析发现,NLR4、NLR6、NLR8.3、NLR9.1组基因在10个测序结果中存在完全一致的序列,其余5组NLR基因未出现完全一致的序列。同组内的NLR基因序列也存在核酸和氨基酸序列长度和组成差异,其中NLR7组基因的多样性最高,10个测序结果中存在8种不同的序列长度,其次为NLR8.2及NLR5组,分别存在5种和4种不同长度的序列,其余NLR基因组存在2~3种不同长度的序列(表2)。
此外,通过Clustal X序列比对发现,同一聚类的氨基酸序列存在多种差异,存在序列变化最多的是NLR7组,有些序列存在较长的氨基酸序列缺失,如NLR7-6在160~190号氨基酸之间比其他序列缺失22个氨基酸(图1)。其余组的NLR基因也都存在类似的差异。本试验中总共获得了有差异的NLR家族基因序列80条,可以看出,NLR家族存在许多成员,尤其以NLR7组的基因序列差异最大。
表2NLR基因cDNA及氨基酸序列多样性分析
Tab.2AnalysisofdiversityandaminoacidsequenceofNLRcDNA
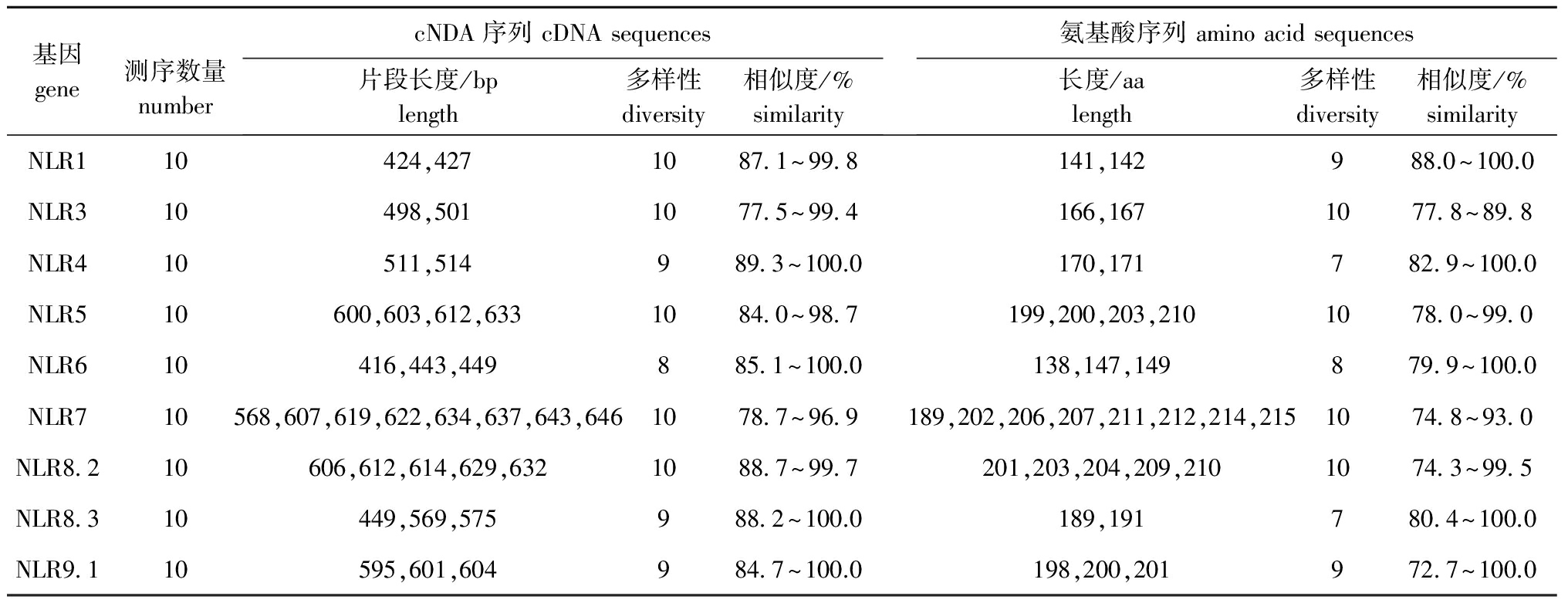
基因gene测序数量numbercNDA序列cDNAsequences片段长度/bplength多样性diversity相似度/%similarity氨基酸序列aminoacidsequences长度/aalength多样性diversity相似度/%similarityNLR110424,4271087 1~99 8141,142988.0~100.0NLR310498,5011077 5~99 4166,1671077 8~89 8NLR410511,514989 3~100.0170,171782 9~100.0NLR510600,603,612,6331084 0~98 7199,200,203,2101078 0~99 0NLR610416,443,449885 1~100.0138,147,149879 9~100.0NLR710568,607,619,622,634,637,643,6461078 7~96 9189,202,206,207,211,212,214,2151074 8~93 0NLR8 210606,612,614,629,6321088 7~99 7201,203,204,209,2101074 3~99 5NLR8 310449,569,575988 2~100.0189,191780 4~100.0NLR9 110595,601,604984 7~100.0198,200,201972 7~100.0
2.3半定量RT-PCR分析
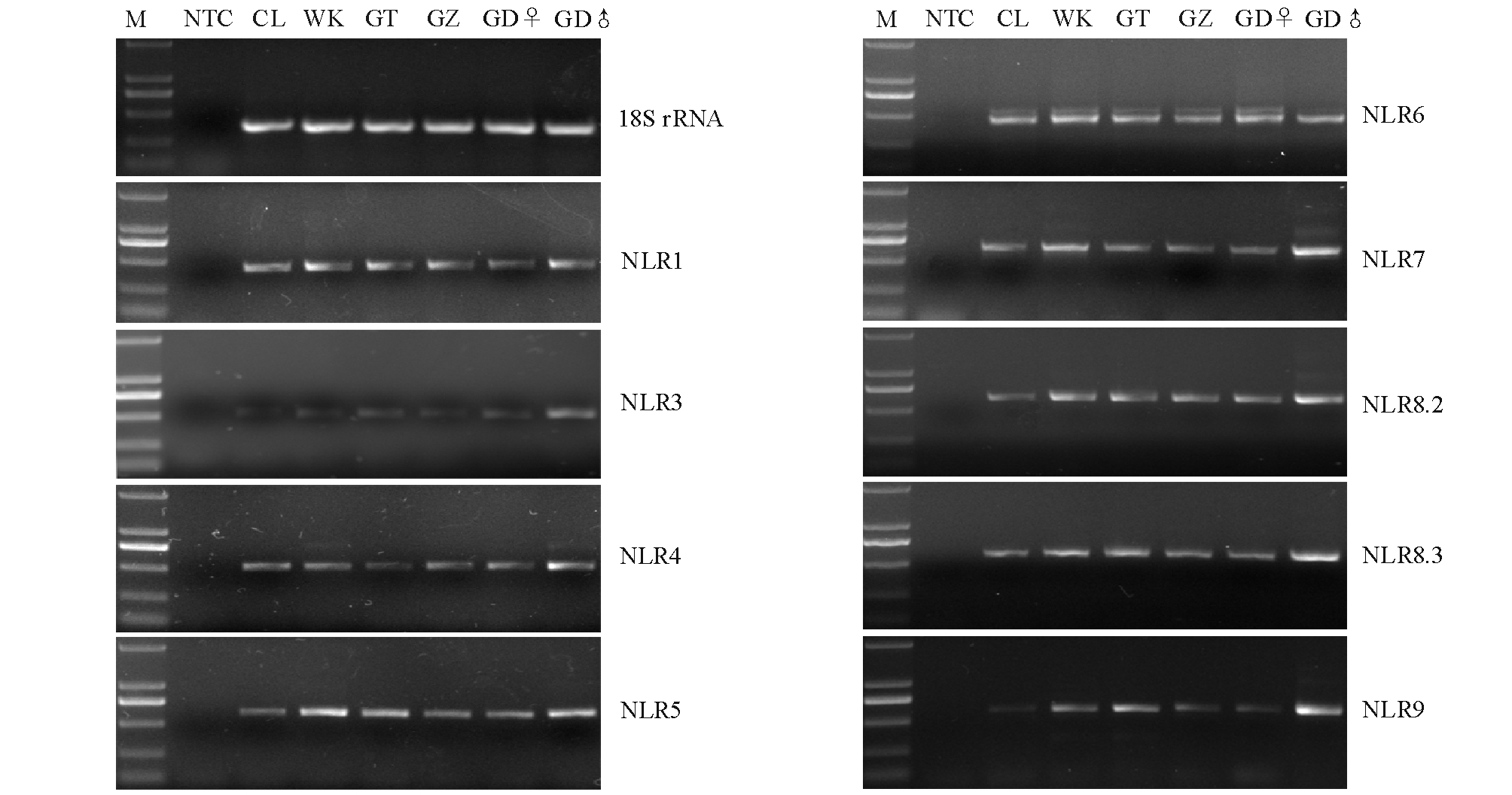
提取虾夷马粪海胆体腔细胞、围口膜、肠、管足、雄性性腺、雌性性腺组织的总RNA,反转录后在扩增效果好的循环数及退火温度下对NLR家族基因进行半定量RT-PCR分析,PCR产物经电泳检测可看出,不同分组的NLR基因在不同组织中的表达情况表现出差异(图2)。
从不同组织的扩增结果可以看出,NLR基因在6种组织中均有扩增,从产物浓度来看,除NLR9组在雄性性腺及肠中表达量最高并与其他组织存在明显差异外,其余基因在6种组织中的表达量未见有明显差异。NLR3组整体表达量较低,仅在精巢中表达量达到一定水平,在其余组织中的表达量微弱。NLR家族各基因在围口膜、性腺、肠中均有较高水平表达,在体腔细胞和管足中表达水平也较高,各家族间基因表达量存在部分差异,但未有较大浮动(图2)。
总体看来,NLR1、NLR3、NLR4组基因处于较低的表达水平,而NLR5、NLR6、NLR7、NLR8.2、NLR8.3、NLR9.1组基因处于较高的表达水平,比较基因家族序列结果来看,基因和氨基酸序列差异越大的基因家族聚类,在各种组织中的表达量就越高。对比不同家族的表达量发现,序列差异最大的家族聚类NLR7、NLR8.2,表达量都处于较高水平。这可能与这两组基因的功能相关,推测这两组基因在虾夷马粪海胆的免疫反应中起着重要的作用。
3讨论
NLR家族作为重要的固有免疫体系中的一员,表达的多样性极大地影响虾夷马粪海胆抗病力的强弱,其他物种中也存在着较多的NLR分子,Huang等[19]研究发现,文昌鱼基因组中有118个NLR分子。紫球海胆基因组测序完成后,通过基因预测发现,其存在208个NLR 家族基因,种类比人类NLR家族(20种)丰富得多[9]。通过对这3种物种的对比发现,越是低等的物种中NLR基因家族越丰富,NLR基因在低等动物的免疫反应中起着重要的作用。研究NLR家族基因可以有效了解低等动物体内的免疫反应基本机制。本研究中,利用设计的NLR引物进行PCR扩增获得了虾夷马粪海胆的9组NLR家族聚类基因片段,证明了NLR家族基因在虾夷马粪海胆各组织中都存在表达,而且通过对同一组基因的大量测序结果比对分析发现,每一个聚类都存在多个成员,在cDNA长度上、基因序列上都存在差异。翻译后的氨基酸序列同样存在长度和氨基酸序列的变化。本研究中,共克隆出80条虾夷马粪海胆NLR家族基因的部分序列,并且具有序列差异,可以看出,NLR家族基因的多样性极高。NLR是细胞内的模式识别受体,主要功能是识别外来抗原,正是因为NLR家族基因具有如此的多样性,才能够满足类别多样的抗原识别要求。
从半定量RT-PCR检测结果来看,NLR基因作为重要的免疫基因,在虾夷马粪海胆各组织中都存在表达,在不同组织中都处于高效表达的状态,但表达量存在些许差异。肠道及性腺表达量普遍较高,这符合已有研究中发现的NLR家族在肠道中的表达量最高的结果[6]。
结合测序结果及半定量分析结果分析发现,同一NLR家族聚类在DNA序列及氨基酸序列上均存在差异,有些组差异较大,而且序列差异越大表达量就越高,这对于海胆免疫外界病原体的入侵以及对入侵后病原体的及时处理可能起着至关重要的作用。海胆体内只存在先天性免疫系统抵抗外界侵扰,成年海胆体腔内的体腔细胞主要承担对外界病原侵扰的免疫应答反应,并通过对外来物质的吞噬和封装功能来实现。海胆体腔内含有多种体腔细胞,各自发挥不同的免疫功能[20-21]。本研究中,肠及围口膜的高效表达可能与长期接触外来病原体有关。本研究中获得了虾夷马粪海胆的部分NLR家族基因序列,并对其不同组织的表达多样性进行了研究,为后续NLR基因作用机制的探究及优良抗病海胆家系的筛选奠定了基础。
参考文献:
[1] Medzhitov R,Janeway C A.Innate immunity: the virtues of a nonclonal system of recognition[J].Cell,1997,91(3):295-298.
[2] Medzhitov R,Janeway C A.Decoding the patterns of self and oneself by the innate immune system[J].Science,2002,296:298-300.
[3] Medzhitov R.Recognition of microorganisms and activation of the immune response[J].Nature,2007,449:819-826.
[4] Nimchuk Z,Eulgem T,Holt B F,et al.Recognition and response in the plant immune system[J].Annual Review of Genetics,2003,37(1):579-609.
[5] Nürnberger T,Brunner F,Kemmerling B,et al.Innate immunity in plants and animals:striking similarities and obvious differences[J].Immunological Reviews,2004,198(1):249-266.
[6] Ting J P Y,Davis B K.CATERPILLER:a novel gene family important in immunity,cell death,and diseases[J].Annu Rev Immunol,2005,23:387-414.
[7] Ting J P Y,Lovering R C,Alnemri E S P D,et al.The NLR gene family:an official nomenclature[J].Immunity,2008,28(3):285.
[8] Kufer T A,Fritz J H,Philpott D J.NACHT-LRR proteins (NLRs) in bacterial infection and immunity[J].Trends in Microbiology,2005,13(8):381-388.
[9] Sodergren E,Weinstock G M,Davidson E H,et al.The genome of the sea urchinStrongylocentrotuspurpuratus[J].Science,2006,314:941-952.
[10] Hibino T,Loza-Coll M,Messier C,et al.The immune gene repertoire encoded in the purple sea urchin genome[J].Developmental Biology,2006,300(1):349-365.
[11] 常亚青,王子臣,宋坚,等.四种海胆杂交的可行性及子代的早期发育[J].水产学报,2000,24(3):211-216.
[12] 李世英,张明,高绪生,等.大连近海虾夷马粪海胆生殖周期的初步研究[J].大连水产学院学报,2000,15(1):60-64.
[13] 丁君,常亚青,王长海,等.不同种海胆体腔细胞类型及体液中的酶活力[J].中国水产科学,2006,13(1):33-38.
[14] 仇雪梅,吴领知,郭晓黎,等.虾夷马粪海胆CYP51基因cDNA的克隆及其SNPs分析[J].大连海洋大学学报,2012,27(4):294-299.
[15] Wang Xiuli,Feng Teng,Yang Lei,et al.dbEST-derived SSR markers in sea urchin (Hemicentrotuspulcherrimus)[J].Conservation Genetics,2009,10(3):729-731.
[16] Wang Xiuli, Qiu Xuemei,Meng Xiangying.Preliminary study on polymorphism analysis of SpRunt-1 gene by PCR-SSCP inStrongylocentrotusintermediusand its association with growth traits[J].Molecular Biology Reports,2010,37(1):411-415.
[17] 丁君,孙巍,常亚青.虾夷马粪海胆性腺cDNA文库构建及部分EST序列分析[J].中国水产科学,2011,18(1):222-229.
[18] 丁君,孙巍,常亚青.虾夷马粪海胆硬脂酰辅酶 A 去饱和酶和蛋白酪氨酸磷酸酶基因的克隆与表达[J].大连海洋大学学报,2012,27(6):489-494.
[19] Huang S,Yuan S,Guo L,et al.Genomic analysis of the immune gene repertoire of amphioxus reveals extraordinary innate complexity and diversity[J].Genome Research,2008,18(7):1112-1126.
[20] Smith L C,Rast J P,Brockton V,et al.The sea urchin immune system[J].Invertebrate Survival Journal,2006(3):25-39.
[21] Smith L C.Diversification of innate immune genes:lessons from the purple sea urchin[J].Disease Models & Mechanisms,2010,3(5/6):274-279.
CloningofpartialsequenceandexpressionofNLRfamilyinseaurchinStrongylocentrotusintermedius
CHEN Ya-dong, LIU Xiao-fei, CHANG Ya-qing, QIU Xue-mei, WANG Xiu-li, LIU Yang
(Key Laboratory of Mariculture & Stock Enhancement in North China’s Sea, Ministry of Agriculture,Dalian Ocean University, Dalian 116023, China)
Abstract:In this study, homologous cloning and semi-quantitative RT-PCR technologies were used to clone the fragments of NLR genes and to detect their expression patterns in tissues of sea urchinStrongylocentrotusintermedius. The primers were designed according to the homology alignment of NLR genes from purple sea urchinS.purpuratus, and 9 fragment groups of NLR genes were obtained from the sea urchin, by sequencing of 10 clones for each group. Then bioinformatic analysis of these genes was conducted by the method of Blast, finding the diversity of cDNA sequences and amino acid sequences of these NLR genes. Also, the RNA was extracted from different tissues in the sea urchin, and the expression of NLR genes was analyzed by semi-quantitative PCR in different tissues of the sea urchin after reverse transcription. The results showed that 9 fragment groups of NLR genes were all expressed in coelomocytes (CL), membrana peristomialis (WK), intestines (GT), tube-feet (GZ), and gonad (GD) at a high level, the maximum in membrana peristomialis and gonad, indicating that NLR gene family plays an important role in the congenital immune system of the sea urchin, with different expression. The findings will provide the foundation for further study of NLR genes in the sea urchin.
Key words:Strongylocentrotusintermedius; NLR family; semi-quantitative RT-PCR
DOI:10.3969/J.ISSN.2095-1388.2014.04.003
文章编号:2095-1388(2014)04-0336-06
收稿日期:2013-11-10
基金项目:国家自然科学基金资助项目(31101923)
作者简介:陈亚东(1988—), 男,硕士研究生。E-mail:chenyadong3@163.com
通信作者:刘洋(1978—), 男,博士,副教授。E-mail:liuyang@dlou.edu.cn
中图分类号:S917
文献标志码::A