两种乌鲂科鱼类线粒体DNA片段序列的比较分析
刘连为1,陈新军1、2,许强华1、2,陆化杰1、2,方舟1
(1.上海海洋大学 海洋科学学院,上海 201306;2.上海海洋大学 大洋生物资源开发和利用上海市高校重点实验室,大洋渔业资 源可持续开发省部共建教育部重点实验室,上海 201306)
摘要:为检测日本乌鲂Bramajaponica与斯氏长鳍乌鲂Taractichthyssteindachneri的种间遗传差异,对线粒体16S rRNA、细胞色素b(Cytb)和细胞色素氧化酶Ⅰ(COⅠ)基因片段序列进行了测定。经比对获得同源片段序列的长度分别为491、478、558 bp,其中16S rRNA基因片段上存在2处碱基插入/缺失。核苷酸组成分析结果表明:日本乌鲂与斯氏长鳍乌鲂在这3个基因片段上的G+C含量差异不显著(P>0.05)(平均为47.27%、46.59%),G含量均偏低,特别是在Cytb和COⅠ这两个基因的第三密码子位点上表现尤为突出(平均为9.80%、12.31%)。两种鱼类在线粒体基因组不同片段上的遗传差异比较明显,在总长度为1529 bp的核苷酸序列中,两种间共检测到204处核苷酸替换,两个蛋白质编码基因上的核苷酸替换主要发生在第三密码子位点上;两种鱼类在3个基因片段上的种间净遗传距离依次为0.112 7(16S rRNA)、0.166 4(Cytb)和0.135 3(COⅠ),遗传差异水平均达到属间差异水平。NJ系统树显示,斯氏长鳍乌鲂与小鳞异鲂Xenobramamicrolepis的亲缘关系较近,而与日本乌鲂的亲缘关系较远。根据Cytb基因片段序列分析,日本乌鲂与斯氏长鳍乌鲂的分歧时间约为832万年,两种间分化事件发生在中新世。
关键词:日本乌鲂;斯氏长鳍乌鲂;线粒体DNA;序列分析;分歧时间
日本乌鲂Bramajaponica与斯氏长鳍乌鲂Taractichthyssteindachneri隶属于鲈形目、乌鲂科,为大洋性底栖鱼类[1]。日本乌鲂主要分布在北太平洋亚热带至亚寒带海域,曾是流刺网渔业的主要种类[2],而斯氏长鳍乌鲂主要分布在印度洋至太平洋海域,二者均为延绳钓渔业的兼捕对象,可作为潜在的商业性种类进行规模性开发和利用[3-4]。日本乌鲂与斯氏长鳍乌鲂外部形态和个体大小差别不大,在长期的冷冻、运输和加工过程中其外部形态组织有可能受到损伤,从而导致两个种类难以判别[5]。国外学者对日本乌鲂与斯氏长鳍乌鲂的生物学特性进行了研究[6-7],但二者在分子水平上的遗传差异目前尚未见报道。近年来,基于线粒体DNA片段序列分析的DNA条形码已成功应用于鱼类及其制品的鉴定和商品鱼的保护与管理中[8-11]。本研究中,测定了日本乌鲂与斯氏长鳍乌鲂的线粒体16S rRNA、细胞色素b(Cytb)和细胞色素氧化酶Ⅰ(COⅠ)基因片段序列,比较分析了两种鱼类种间遗传分化程度,为准确鉴定两种鱼及其制品提供技术支持,同时探讨了两种间的亲缘关系及分化年代,以期为乌鲂科鱼类群体遗传学及分子系统发育研究提供基础材料。
1材料与方法
1.1材料
试验用日本乌鲂、斯氏长鳍乌鲂均于2012年6月采自北太平洋中部,日本乌鲂19尾,斯氏长鳍乌鲂2尾。
1.2方法
1.2.1 基因组DNA的提取 取两种鱼的肌肉组织用体积分数为95%的乙醇固定,于-20 ℃下保存备用。采用Genomic DNA Isolation Kit试剂盒(成都福际生物技术有限公司)提取基因组DNA,用12 g/L琼脂糖凝胶电泳检测DNA质量,用紫外分光光度计检测DNA浓度。
1.2.2 PCR扩增及测序 参照GenBank上登录的日本乌鲂3个基因片段及斯氏长鳍乌鲂COⅠ基因片段序列自行设计引物,引物序列见表1。PCR反应总体积为25 μL,包括10×PCR Buffer 2.5 μL、Taq DNA polymerase(5 U/μL)0.2 μL、dNTP(2.5 mmol/L)2 μL、上下游引物(10 μmol/L)各0.6 μL、DNA模板20 ng,用ddH2O补足25 μL。PCR扩增在Eppendorf PCR仪上进行,反应条件为:94 ℃下预变性2 min;94 ℃下变性30 s,57 ℃下退火45 s,72 ℃下延伸45 s,共进行35个循环;最后在72 ℃下延伸2 min。PCR产物经12 g/L琼脂糖凝胶电泳分离,经Biospin Gel Extraction Kit(杭州博日科技有限公司)纯化后送至生工生物工程(上海)股份有限公司进行双向测序。
表13个基因片段PCR扩增引物序列
Tab.1Primersequencesofthethreegenefragments
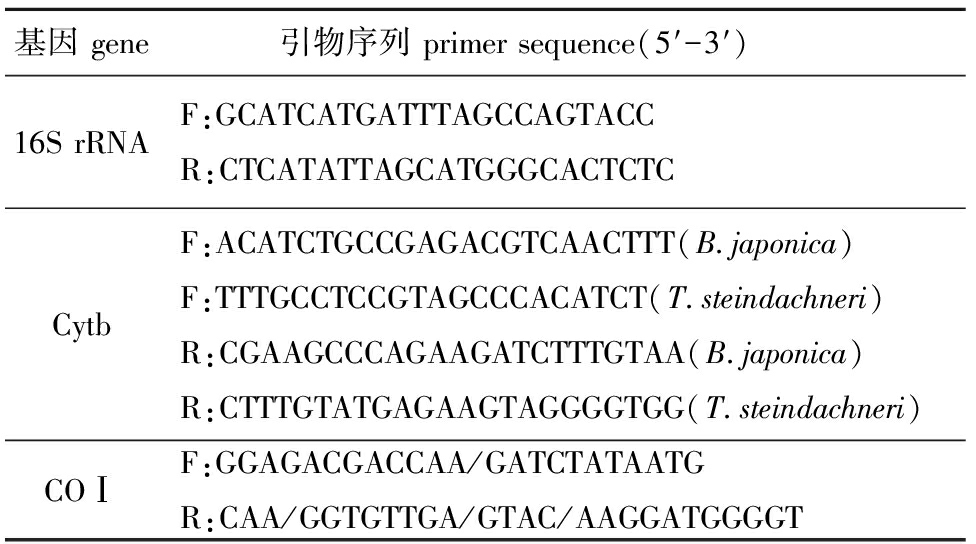
基因gene 引物序列primersequence(5′-3′)16SrRNAF:GCATCATGATTTAGCCAGTACCR:CTCATATTAGCATGGGCACTCTCCytbF:ACATCTGCCGAGACGTCAACTTT(B japonica)F:TTTGCCTCCGTAGCCCACATCT(T steindachneri)R:CGAAGCCCAGAAGATCTTTGTAA(B japonica)R:CTTTGTATGAGAAGTAGGGGTGG(T steindachneri)COⅠF:GGAGACGACCAA/GATCTATAATGR:CAA/GGTGTTGA/GTAC/AAGGATGGGGT
1.3数据处理
所得序列采用ClustalX 1.83软件[12]进行编辑、校对和排序。采用Mega 4.0软件[13]中的Statistics统计DNA序列碱基组成。采用邻接法[14](Neighbor-Joining,NJ)基于Kimura双参数模型构建系统树,分析两种乌鲂科鱼类的亲缘关系,系统树中节点的自举置信水平应用Bootstrap(重复次数1000,分节点分支支持率小于50%的省略)估计,并将蛋白质编码基因核苷酸序列转化为氨基酸序列。单倍型数、多态位点数、简约信息位点数采用DnaSP 4.10软件[15]计算。
2结果与分析
2.1基因片段的序列分析
经比对排序得到同源片段序列长度分别为491 bp(16S rRNA)、478 bp(Cytb)和558 bp(COⅠ),获得的3个基因片段序列在GenBank上的登录号为KF546805~KF546828。日本乌鲂在线粒体16S rRNA、Cytb和COⅠ基因片段上分别检测到3、9、5个多态位点;斯氏长鳍乌鲂在线粒体16S rRNA、Cytb基因片段上分别检测到2、5个多态位点,所测两个COⅠ基因片段序列与GenBank上登陆的该基因序列(登陆号:EF609477.1)完全相同。核苷酸组成分析结果表明,日本乌鲂与斯氏长鳍乌鲂在3个基因片段上的核苷酸组成差异不显著(P>0.05),G含量均偏低,特别是在Cytb和COⅠ这两个蛋白质编码基因的第三密码子位点上的含量更低(平均为9.80%、12.31%),A+T含量均高于G+C含量(表2)。
表2日本乌鲂与斯氏长鳍乌鲂线粒体16SrRNA、Cytb和COⅠ基因片段的核苷酸组成
Tab.2Thenucleotidecompositionsof16SrRNA,CytbandCOⅠgenefragmentsinPacificpomfreBramajaponicaandTaractichthyssteindachneri
%
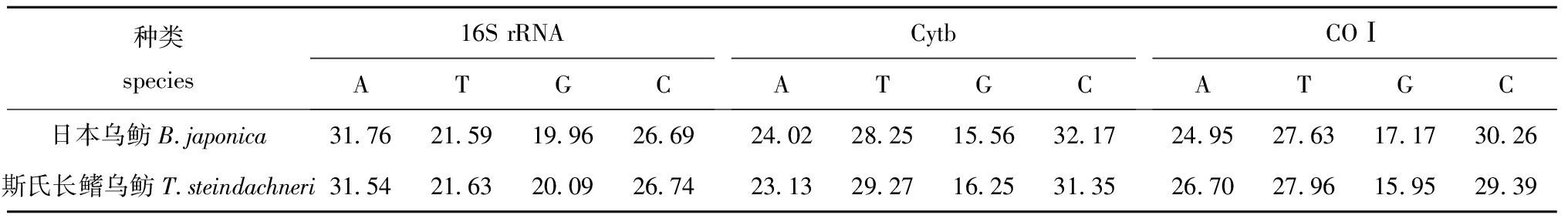
种类species16SrRNAATGCCytbATGCCOⅠATGC日本乌鲂B japonica31 7621 5919 9626 6924 0228 2515 5632 1724 9527 6317 1730 26斯氏长鳍乌鲂T steindachneri31 5421 6320 0926 7423 1329 2716 2531 3526 7027 9615 9529 39
日本乌鲂与斯氏长鳍乌鲂在3个基因片段序列上存在不同程度的遗传分化(表3)。在16S rRNA基因片段上存在2处碱基插入/缺失,两种间检测到的多态位点数及其定义的单倍型数最少。在Cytb基因片段上,两种间检测到的多态位点数及其定义的单倍型数最多。78处突变中有73处位于密码子第三位点上(58处为转换),5处位于密码子第一位点上(3处为转换)(图1)。在其编码的氨基酸序列上,两种间检测到7处氨基酸替代,4处是由第三密码子位点上的核苷酸替代引起,3处是由第一密码子位点上的核苷酸替代引起。在COⅠ基因片段上,71处突变中有66处位于密码子第三位点上(52处为转换),5处转换位于密码子第一位点上(图2)。在其编码的氨基酸序列上,两种间检测到3处氨基酸替代,均由第三密码子位点上的核苷酸替代引起。两种鱼类在3个基因片段上的序列差异百分比分别为11.20%(16S rRNA)、16.25%(Cytb)、12.72%(COⅠ),编码的氨基酸序列差异百分比分别为4.38%(Cytb)、1.61%(COⅠ),基于Kimura双参数法计算得到的两种间净遗传距离分别为0.112 7(16S rRNA)、0.166 4(Cytb)、0.135 3(COⅠ)。
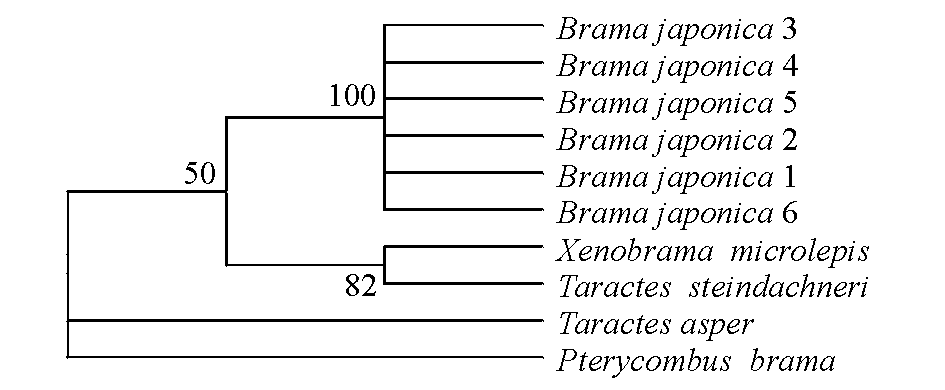
从GenBank上检索并下载乌鲂科高鳍鲂Pterycombusbrama(GenBank:GU225016.1)、粗棱鲂Taractesasper(GenBank:GU440550.1)、小鳞异鲂Xenobramamicrolepis(GenBank:EF609495.1)的COⅠ基因序列作比较,利用Kimura双参数模型构建NJ系统树(图3)。可以看出,斯氏长鳍乌鲂与小鳞异鲂首先聚在一起,然后与日本乌鲂聚在一起。这表明,斯氏长鳍乌鲂较之日本乌鲂与小鳞异鲂亲缘关系更近。
表33个基因片段序列的多态性
Tab.3Sequencevariabilityofthreegenefragments

基因gene片段长度/bpalignedlength插入/缺失位点gappedsites多态位点variablesites简约信息位点parsimonyinformativesites单倍型数No ofhaplotype转换/颠换transition/transversion16SrRNA49125551632/23Cytb480078731261/17COⅠ55807168757/14
2.2种间分化时间
基于Kimura双参数法计算得到日本乌鲂与斯氏长鳍乌鲂在Cytb基因片段上的种间净遗传距离为0.166 4。将每百万年2%的核苷酸分歧速率应用于两种鱼类Cytb基因片段上,得到日本乌鲂与斯氏长鳍乌鲂的分歧时间约为832万年,两种间分化事件发生在中新世(Micene)。
3讨论
乌鲂科鱼类中不少种类具有较高的食用价值,但其渔获量并不高,是一种未充分利用的渔业资源,具有较大的开发潜力[5]。乌鲂科部分种类在生长过程中外部形态经历着明显的变化,通常很难找到确定的形态特征来区分不同生长时期的种类,而且不同种类在某些外部分类特征上存在重叠,这些均给乌鲂科鱼类的鉴定带来一定困难[5]。分子遗传标记由于具有多态性高、不受外界环境限制等优点,已广泛应用于水产生物的遗传学研究中[16]。其中,线粒体DNA因具有结构简单、母系遗传、进化速率快、几乎不发生重组等特点,在鱼类遗传学研究中得以广泛应用[17-19]。本研究中,对日本乌鲂与斯氏长鳍乌鲂的线粒体DNA部分片段序列进行比较分析,二者在线粒体3个基因片段上的遗传差异比较明显,在总长度为1529 bp的核苷酸序列中,两种间共检测到204处核苷酸替换。其中,在长度为1038 bp的两个蛋白质编码基因片段中检测到149处核苷酸替换。两种鱼类在3个基因片段上的序列差异百分比及种间净遗传距离依次均为Cytb>COⅠ>16S rRNA;16S rRNA基因序列比较保守,适用于种以上阶元间的遗传差异及系统进化分析,而Cytb和COⅠ基因进化速率适中,适用于种及种以上阶元间的遗传学研究[20-21]。日本乌鲂与斯氏长鳍乌鲂分别属于乌鲂属和长鳍乌鲂属,通过与其他鱼类近缘种间遗传差异水平比较,认为二者在3个基因片段上的遗传差异水平均达到属间差异水平[22-24]。
目前,COⅠ基因常作为DNA条形码在鱼类及其制品的鉴定、分类地位检测、系统进化方面得以广泛应用[25-27]。Zhang等[28]基于该基因的DNA条形码对日本近海158种海洋鱼类进行鉴定,NJ系统树显示,所有序列以种为单元分布,其中包括日本乌鲂和红棱鲂Taractesrubescens。本研究中,利用该基因片段序列研究乌鲂科5个不同属种类的系统进化,NJ系统树显示,斯氏长鳍乌鲂与小鳞异鲂组成的分支,依次与日本乌鲂、粗棱鲂聚在一起,最后与高鳍鲂聚在一起。前者在分类地位上属于乌鲂亚科(Braminae),后者属于高鳍鲂亚科(Pteraclinae),种间亲缘关系与形态特征一致。DNA分子结构上的变化可以用来估计进化过程中涉及时期的方法,并因此为种系发生设置一个时间尺度[29]。不同基因由于承受的选择压力不同,从而导致进化速率差异较大。本研究中,采用硬骨鱼类常用的Cytb基因核苷酸分歧速率来估算两种间的分化年代,分化时间约为832万年前。
致谢:本研究得到国家远洋渔业工程技术研究中心和农业部大洋渔业资源环境科学观测实验站的资助,上海海洋大学伍汉霖教授、台湾中央研究院生物多样性研究中心邵广昭研究员、台湾中央研究院生物多样性研究博物馆林沛立经理在物种鉴定方面给予了帮助和指导,在此一并表示感谢!
参考文献:
[1] 刘爱民,刘刚.中国乌鲂科两个新记录种[J].水产科学,1989,8(2):30-31.
[2] Watanabe H,Kubodera T,Kawahara S.Feeding habits of Pacific pomfretBramajaponicain the transition zone of the central North Pacific[J].Fisheries Science,2003,69(2):269-276.
[3] Polovina J J,Abecassis M,Howell E A,et al.Increases in the relative abundance of mid-trophic level fishes concurrent with declines in apex predators in the subtropical North Pacific,1996-2006[J].Fishery Bulletin,2009,107(4):523-531.
[4] Beverly S,Curran D,Musyl M,et al.Effects of eliminating shallow hooks from tuna longline sets on target and non-target species in the Hawaii-based pelagic tuna fishery[J].Fisheries Research,2009,96(2/3):281-288.
[5] Last P R,Moteki M.Bramidae[M]//Carpenter K E,Neim V H.FAO species identification guide for fishery purpose:the living marine resources of the western central Pacific.Rome:FAO,2001:2824-2830.
[6] Pearcy W G,Fisher J P,Yoklavich M M.Biology of the Pacific pomfret (Bramajaponica) in the North Pacific Ocean[J].Canadian Journal of Fisheries and Aquatic Sciences,1993,50(12):2608-2625.
[7] Hawn D R,Seki M P,Nishimoto R.An estimation of the life history and ecology of opah and monchong in the North Pacific[C]// 15thMeeting of the Standing Committee on Tuna and Billfish SCTB15 Working Paper.Honolulu:HI,2002:1-10.
[8] Ardura A,Linde A R,Moreira J C,et al.DNA barcoding for conservation and management of Amazonian commercial fish[J].Biological Conservation,2010,143(6):1438-1443.
[9] Cawthorn D,Steinman H A,Witthuhn R C.DNA barcoding reveals a high incidence of fish species misrepresentation and substitution on the South African market[J].Food Research International,2012,46(1):30-40.
[10] 胡嵘,杜鹤,崔丽娜,等.海马、海龙基于COⅠ条形码的DNA分子鉴定[J].吉林中医药,2012,32(3):272-276.
[11] 李新光,王璐,赵峰,等.DNA条形码技术在鱼肉及其制品鉴别中的应用[J].食品科学,2013,34(18):337-342.
[12] Thompson J D,Gibson T J,Plewniak F,et al.The Clustal X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Research,1997,25(24):4876-4882.
[13] Tamura K,Dudley J,Nei M,et al.MEGA 4:molecular evolutionary genetics analysis (MEGA) software version 4.0[J].Molecular Biology and Evolution,2007,24(8):1596-1599.
[14] Saitou N,Nei M.The neighbor-joining method:a new method for reconstructing phylogenetic trees[J].Molecular Biology and Evolution,1987,4(4):406-425.
[15] Rozas J,Sanche-DelBarrio J C,Messenguer X,et al.DNA polymorphism analyses by the coalescent and other methods[J].Bioinformatics,2003,19(18):2496-2497.
[16] 刘云国,刘贤德,高焕,等.水产生物DNA分子标记技术[M].北京:科学出版社,2009:59-62.
[17] 黄立群,仪慧兰,崔松林,等.历山中国大鲵线粒体片段序列的测定及其遗传差异研究[J].大连海洋大学学报,2012,27(6):513-517.
[18] 薛泰强,杜宁,高天翔.基于线粒体COⅠ及Cytb基因的4种鱚科鱼类系统发育研究[J].中国海洋大学学报:自然科学版,2010,40(增刊):91-98.
[19] 徐钢春,魏广莲,李建林,等.基于线粒体DNA D-loop序列分析养殖刀鲚与湖鲚的遗传多样性[J].大连海洋大学学报,2012,27(5):448-452.
[20] 孙鹏,尹飞,彭士明,等.条石鲷线粒体COⅠ和Cytb序列的遗传变异分析[J].水产学报,2011,35(3):327-333.
[21] 毕潇潇,高天翔,肖永双,等.4种鳕鱼线粒体16S rRNA、COⅠ和Cytb基因片段序列的比较分析[J].南方水产,2009,5(3):46-52.
[22] Ma H Y,Ma C Y,Ma L B.Molecular identification of genusScylla(Decapoda: Portunidae) based on DNA barcoding and polymerase chain reaction[J].Biochemical Systematics and Ecology,2012,41:41-47.
[23] 肖志忠,肖永双,马道远,等.基于线粒体DNA部分片段探讨条石鲷与斑石鲷的亲缘关系[J].海洋学报,2011,33(5):115-123.
[24] 任桂静,刘奇,高天翔,等.基于线粒体DNA序列探讨斑头鱼分类地位[J].动物分类学报,2011,36(2):332-340.
[25] 杨帆,何利军,雷光春,等.中国东南沿海弹涂鱼科常见鱼类的遗传多样性和DNA条形码[J].生态学杂志,2012,31(3):676-683.
[26] 柳淑芳,陈亮亮,戴芳群,等.基于线粒体COⅠ基因的DNA条形码在石首鱼科(Sciaenidae)鱼类系统分类中的应用[J].海洋与湖沼,2010,41(2):223-232.
[27] 孟玮,杨天燕,海萨,等.基于线粒体COⅠ基因序列的亚东鲑DNA条形码研究[J].水产学杂志,2010,23(1):6-10.
[28] Zhang J B,Hanner B.DNA barcoding is a useful tool for the identification of marine fishes from Japan[J].Biochemical Systematics and Ecology,2011,39(1):31-42.
[29] Cox C B,Moore P D.Biogeography:an ecological and evolutionary approach[M].7th ed.Oxford:Blackwell Publishing,2005:330 331.
331.
ComparativesequenceanalysisofmitochondriaDNAfragmentsbetweentwomembersinBramidae
LIU Lian-wei1, CHEN Xin-jun1,2, XU Qiang-hua1,2, LU Hua-jie1,2, FANG Zhou1
(1.College of Marine Sciences,Shanghai Ocean University, Shanghai 201306,China;2.Key Laboratory of Shanghai Education Commission for Oceanic Fisheries Resources Exploitation, Key Laboratory of Sustainable Exploitation of Oceanic Fisheries Resources, Ministry of Education, Shanghai Ocean University, Shanghai 201306, China)
Abstract:Partial sequences of mitochondrial 16S rRNA, Cytb and COⅠ genes were amplified and sequenced in Pacific pomfretBramajaponicaand pomfretTaractichthyssteindachneriin order to evaluate the genetic variance in the two species. Three homologous fragments of 491 bp for 16S rRNA, 478 bp for Cytb and 558 bp for COⅠ genes were obtained by sequences aligning, and two insertion or deletion of sites were found in the 16S rRNA gene fragment. The nucleotide composition analysis indicated that there was no significant difference in G+C content in the three fragments in the two species (47.27% in Pacific pomfret and 46.59% in pomfret), a strong bias against guanine(G), especially in the third codon positions of Cytb and COⅠ genes(9.80% in Pacific pomfret and 12.31% in pomfret). Significant difference in genetic variance between the two species was observed in the three mtDNA fragments in which 204 nucleotide substitutions were checked in the total 1529 bp nucleotides analyzed, and nucleotide substitutions in the two protein coding genes were mainly found at the third codon positions. The net genetic distances were 0.112 7 in 16S rRNA, 0.166 4 in Cytb, and 0.135 3 in COⅠ gene fragments between the two species, showing intergenus differentiation within family. NJ phylogenetic tree revealed that the relationship between pomfret andXenobramamicrolepiswas closer than that between pomfret and Pacific pomfret. It is speculated based on sequence analysis of partial Cytb gene that the divergence between Pacific pomfret and pomfret is derived about 8.32 million years ago, in Miocene.
Key words:Bramajaponica;Taractichthyssteindachneri; mitochondrial DNA; sequence analysis; divergence time
DOI:10.3969/J.ISSN.2095-1388.2014.03.004
文章编号:2095-1388(2014)03-0221-06
收稿日期:2013-09-30
基金项目:国家自然科学基金资助项目(41276156);上海市科委重大项目(12231203900);国家发改委产业化专项(2159999)
作者简介:刘连为(1983—), 男, 博士研究生。E-mail:liulianwei1@163.com
通信作者:陈新军(1967—), 男, 博士,教授。E-mail:xjchen@shou.edu.cn
中图分类号:S917
文献标志码::A




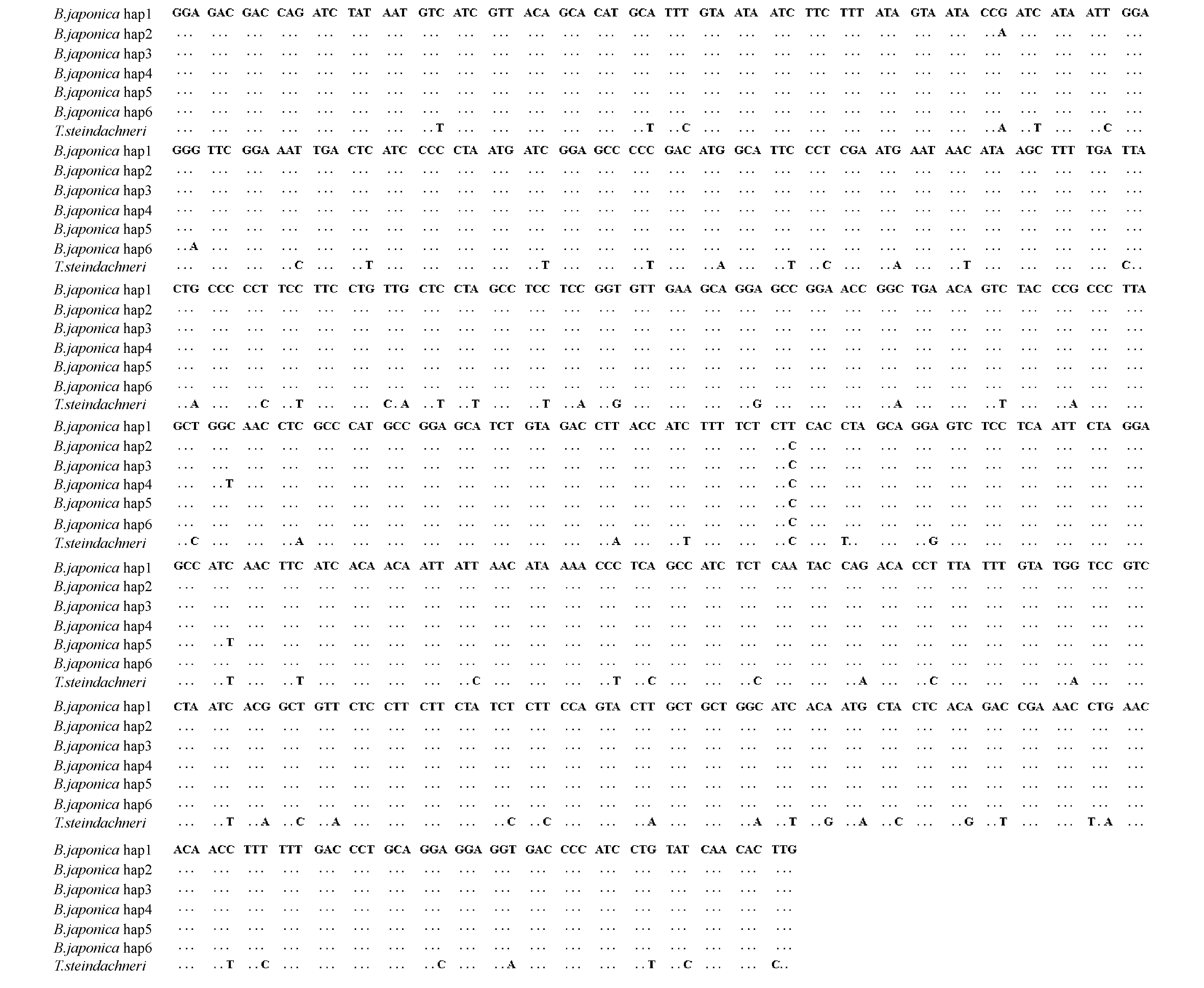
 331.
331.