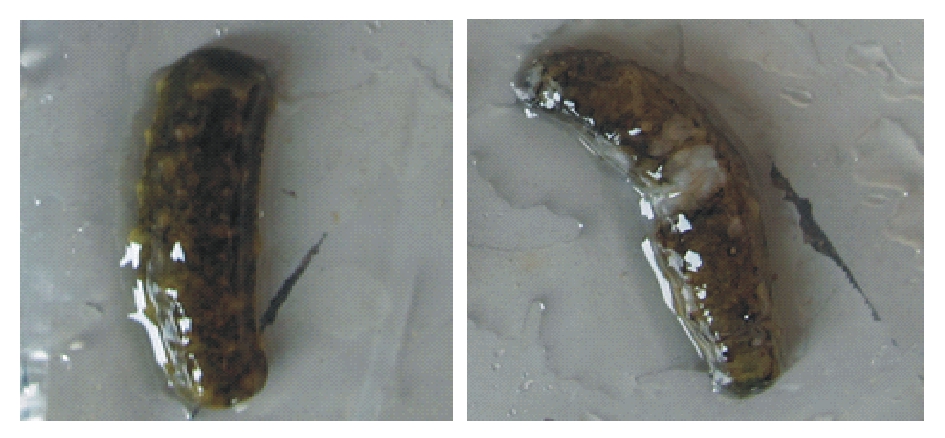
注:A 未化皮刺参幼苗;B 化皮刺参幼苗
Note:A,juvenile sea cucumber without lesion;B,diseased juvenile sea cucumber with lesion
图1 刺参幼苗
Fig.1 The juvenile sea cucumber
摘要:采用16S rDNA克隆文库法对某刺参养殖场患病刺参Apostichopusjaponicus幼苗(体长为3~5 cm)表皮的菌群结构进行分析。结果表明:化皮参苗病灶组织和未明显化皮参苗表皮菌群均由γ-变形菌纲、β-变形菌纲和α-变形菌纲组成,优势菌为γ-变形菌纲,主要包括志贺氏菌Shigellasp.、不动杆菌Acinetobactersp.和假单胞菌Pseudomonassp.;化皮参苗病灶组织中志贺氏菌、不动杆菌和假单胞菌比例分别为49%、17%和5%,未明显化皮参苗表皮中3类菌的比例分别为20%、26%、9%。将该养殖场育苗池海水作为对照并进行菌群结构分析,结果显示,水中菌群同样由γ-变形菌纲、β-变形菌纲和α-变形菌纲组成,优势菌也为γ-变形菌纲,主要包含弧菌Vibriosp.(75%)与志贺氏菌(12%)。此外,对化皮参苗病灶处进行细菌分离培养,得到一株优势菌,经16S rDNA初步鉴定为弧菌。研究表明,采用16S rDNA克隆文库法所得结果与传统的病原分离培养法存在差异。
关键词:刺参;化皮;菌落结构;16S rDNA克隆文库
刺参Apostichopusjaponicus病害是目前限制刺参养殖产业发展的一个主要问题[1-2]。刺参的化皮病又称“腐皮综合症”“烂皮病”等,该病发病快,一旦发病很快就会蔓延全池,死亡率高达90%以上,且幼参的感染率、发病率和死亡率均高于成参。现有研究表明,细菌是刺参疾病的主要病原,并且同一病症往往存在多种病原,弧菌Vibriosp.、气单胞菌Aeromonassp.、假交替单胞菌Pseudoalteromonassp.、希瓦氏菌Shewanellasp.等都可导致刺参患病[3-8]。杨嘉龙等[3]认为,溶藻弧菌Vibrioalginolyticus为刺参主要致病菌。王印庚等[4]研究显示,导致保苗期刺参“腐皮综合症”的致病菌是假交替单胞菌Pseudoalteromonasnigrifaciens。马悦欣等[5]在对大连地区刺参幼苗溃烂病的研究中得到的主要致病菌为弧菌。王高学等[6]研究发现,杀鲑气单胞菌Aeromonassalmonicida能够感染刺参。Li等[7]从患病刺参中发现3种刺参致病菌,其致病力依次为希瓦氏菌>灿烂弧菌>河豚毒素假交替单胞菌。Deng等[8]从患皮肤溃疡海参的室内越冬池塘和室外池塘中分离出具有致病力的6种弧菌。
刺参病原菌的多样化表明,病原菌为条件致病菌,在不同病例中由于环境条件的不同,引起刺参发病的细菌也不同。另一方面,传统的细菌分离培养方法对于病原研究也具有一定的局限性,培养条件的差异可能导致不同的分离结果,培养条件的不足甚至可能造成真正病原菌的丢失。借助不依赖分离培养的分子技术,可为病原研究提供新的依据[9]。细菌16S rDNA克隆文库法是分子生态学研究中用来调查细菌物种组成的常用方法[10]。克隆的方法不需要进行环境中细菌的纯培养,测序信息量大,所得序列信息可用来比较不同样品中微生物的丰度及多样性情况,从系统发育树获得的物种信息可指导培养试验,同时还可为其他分子生物学方法提供探针和引物信息。该方法已广泛用于土壤、海洋、湖泊、动物肠道等多种生态系统细菌多样性的调查中,揭示了环境中前所未知的微生物多样性[11-14]。
2011年11月,辽宁省瓦房店市海源水产养殖场育苗期间刺参幼苗发生大量化皮现象,作者采用16S rDNA克隆文库方法对患病刺参幼苗(以下简称参苗)的菌群结构进行了研究,以期为刺参病原菌的研究以及刺参的科学养殖提供参考。
1.1材料
选择10个育苗池,从每个池选取未化皮(图1-A)与化皮刺参参苗(图1-B)各5头(体长为3~5 cm)装入无菌袋中,置于冰盒中带回实验室。同时用无菌瓶采集池塘水面下10 cm左右处的海水样品500 mL带回。
1.2方法
1.2.1 样品的制作与处理 在室验室条件下,用无菌海水反复冲洗参苗表皮,无菌条件下剪取化皮刺参病灶组织与未化皮刺参表皮,分别装入1.5 mL离心管中于-20 ℃下保存备用。记录样品名称分别为W1(海水)、W2(化皮参苗病灶组织)和W3(未化皮参苗表皮)。
海水样品用800~1250目筛绢过滤后,再经0.22 μm滤膜过滤,将过滤后的滤膜剪碎放入1.5 mL离心管中,并加入1.0 mL SET液涡旋混匀,于-20 ℃下保存备用。
1.2.2 总DNA的提取 将样品在室温下解冻进行DNA粗提取,并采用苯酚抽提法纯化DNA[15]。
1.2.3 细菌16S rDNA克隆文库的构建 对上述样品进行细菌16S rDNA序列扩增,测序后与NCBI中相似序列进行Blast比对,根据比对结果对细菌进行鉴定。扩增16S rDNA的正向引物为27F:5′ AGAGTTTGATC(C/A)TGGCTCAG 3′,反向引物为1492R:5′ TACGG(C/T)TACCTTG TTACGACTT 3′。PCR反应体系(共25.0 μL):MasterMix 12.5 μL,引物 (10 μmol/L)F、R各1.0 μL,模板 1.0 μL,灭菌ddH2O 9.5 μL。PCR反应扩增程序:95 ℃下预变性2 min;94 ℃下变性30 s,52 ℃下退火30 s,72 ℃下延伸90 s,共进行30个循环;最后在72 ℃下再延伸5 min,于4 ℃下保存。经10 g/L琼脂糖凝胶电泳检测确定目的条带后,切胶回收DNA。经纯化后的PCR产物连接到载体pMD19-T中,于16 ℃下连接30 min。连接体系(共10 μL):PMD19-T 1.0 μL,回收DNA 4.0 μL,Solution 5.0 μL。转化过程:将10 μL连接产物加到50 μL感受态细胞E.coliDH5α中,轻轻混匀后冰浴30 min,于42 ℃下放置45 s,冰浴1 min,加入890 μL不含氨苄青霉素(Amp)的液体LB培养基, 于37 ℃下以100 r/min离心60 min,取150~200 μL涂布于含有X-gal、IPTG和Amp的LB培养基中,于37 ℃下培养12~16 h后,选择具有氨苄青霉素抗性的白色转化子(蓝白斑筛选)。每份样品随机挑选100个左右的白色克隆子,送交Invitrogen上海贸易有限公司进行测序。
基因序列使用Clustal X软件进行多序列比对,相似性高于97%的序列归为同一个操作分类单元(operational taxonomic unit,OTU),每一OTU中的代表性克隆子与其最相似菌株的16S rDNA序列通过Clustal X软件进行完全比对[16],并使用Mega 4.0软件,采用邻位相连法获得系统发育树。
16S rDNA克隆文库覆盖率(C)计算公式为
C=[1-(n1/N)]×100%,
其中,N代表阳性克隆子总数,n1代表仅含单个克隆子的数量。
1.2.4 病灶处细菌的分离培养及16S rDNA鉴定 无菌条件下用接种针从化皮刺参病灶组织处取样,于Zobell 2216E培养基平板上进行划线分离培养,置28 ℃下恒温培养24~48 h。从平板上挑取优势单菌落进行多次划线培养,挑取优势菌进行分离纯化直到获得纯菌落(记为BZ10),并与体积分数为15%的甘油生理盐水制成菌悬液,于-80 ℃下冻存。采用煮沸法提取菌株BZ10的DNA,扩增其16S rDNA序列,并进行初步鉴定,方法同“1.2.3”。
2.1化皮刺参幼苗的观察
化皮参苗患病初期,其活动能力和附着力变弱,摄食量减少,身体收缩、僵直,疣足尖端开始溃烂,口部肿大,不能收缩、闭合。随着病情的加重,病参溃疡处增多,全身溃烂,最后死亡(图1)。此外,附着物上留有一白色印痕。
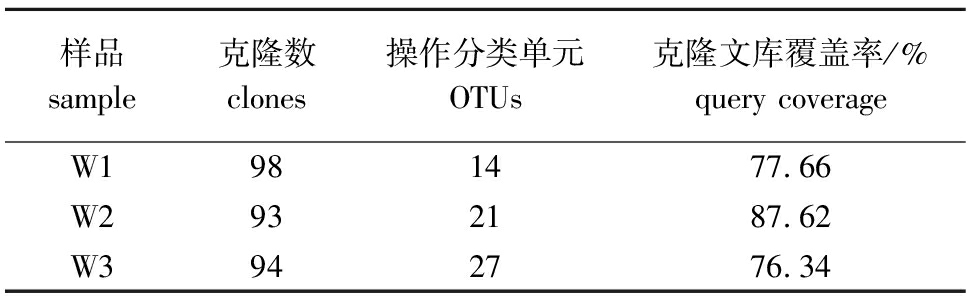
2.2细菌16S rDNA克隆文库覆盖率的检测
对随机挑取的样品克隆子进行测序,通过软件分析得到不同的OTU。根据覆盖率公式计算所构建的化皮参苗病灶组织、未化皮参苗表皮和海水细菌16S rDNA克隆文库的覆盖率分别为87.62%、76.34%和77.66%,均大于70%(表1),说明所构建的文库达到标准。

注:A 未化皮刺参幼苗;B 化皮刺参幼苗
Note:A,juvenile sea cucumber without lesion;B,diseased juvenile sea cucumber with lesion
图1 刺参幼苗
Fig.1 The juvenile sea cucumber
表1样品覆盖率
Tab.1Coverageofsamples
样品sample克隆数clones操作分类单元OTUs克隆文库覆盖率/%querycoverageW1981477 66W2932187 62W3942776 34
2.3刺参幼苗表皮的菌群结构
对化皮参苗病灶样品所得OTU中的克隆子数量及其相似菌株进行Blast和Fasta比对得到如下结果:克隆子与GenBank数据库中已知细菌的16S rDNA序列相似性最高为99%,最低为94%,此文库所测得γ-变形菌纲、β-变形菌纲和α-变形菌纲的比例分别为76%、12%和4%,此外还有8%的菌株分类地位不确定。71个克隆子与来自γ-变形菌纲的菌株序列相似,其中,45个克隆子与志贺氏菌Shigellasp.序列相似,7个克隆子与假单胞菌Pseudomonassp.序列相似,15个克隆子与不动杆菌属Acinetobactersp.序列相似,3个克隆子与嗜麦芽寡养单胞菌Stenotrophomonasmaltophilia序列相似,1个克隆子与假交替单胞菌序列相似;11个克隆子与来自β-变形菌纲的菌株序列相似,其中,3个克隆子与草螺菌属Herbaspirillumsp.序列相似,2个克隆子与内生霉菌伯克霍尔德氏菌Burkholderiafungorum序列相似,2个克隆子与争论产碱菌Variovoraxparadoxus序列相似,1个克隆子与Curvibactersp.菌序列相似,1个克隆子与罗尔斯顿菌Ralstoniasp.序列相似;4个克隆子与来自α-变形菌纲的菌株序列相似,分别与慢生根瘤菌属Bradyrhizobiumsp.、uncultured alpha proteobacteria和鞘氨醇单胞菌属Sphingomonassp.序列相似(表2)。可见,刺参幼苗病灶组织克隆文库中γ-变形菌纲占绝大部分,其中主要为志贺氏菌(49%)、不动杆菌(17%)和假单胞菌(5%)(图2-A)。
对未化皮参苗样品所得OTU中的克隆子数量及其相似菌株进行Blast和Fasta比对得到如下结果:克隆子与GenBank数据库中已知细菌的16S rDNA序列相似性最高为99%,最低为90%,此文库所测得γ-变形菌纲、β-变形菌纲和α-变形菌纲的比例分别为60%、17%和6%。56个克隆子与来自γ-变形菌纲的菌株序列相似,其中,19个克隆子与志贺氏菌序列相似,24个克隆子与不动杆菌序列相似,8个克隆子与假单胞菌序列相似,其余5个克隆子与纤维弧菌Cellvibriosp.、肠杆菌Enterobactersp.、嗜冷杆菌Psychrobactersp.、寡养单胞菌Stenotrophomonassp.和未培养菌序列相似;16个克隆子与来自β-变形菌纲的菌株序列相似,分别与贪噬菌属、草螺菌属、青枯菌属Ralstoniasp.和Curvibacter菌序列相似;6个克隆子与来自α-变形菌纲的菌株序列相似,主要包括鞘氨醇单胞菌属细菌(表3)。可见,未化皮参苗表皮克隆文库的优势菌纲亦为γ-变形菌纲,其中主要为志贺氏菌(20%)、不动杆菌(26%)和假单胞菌(9%)(图2-B)。
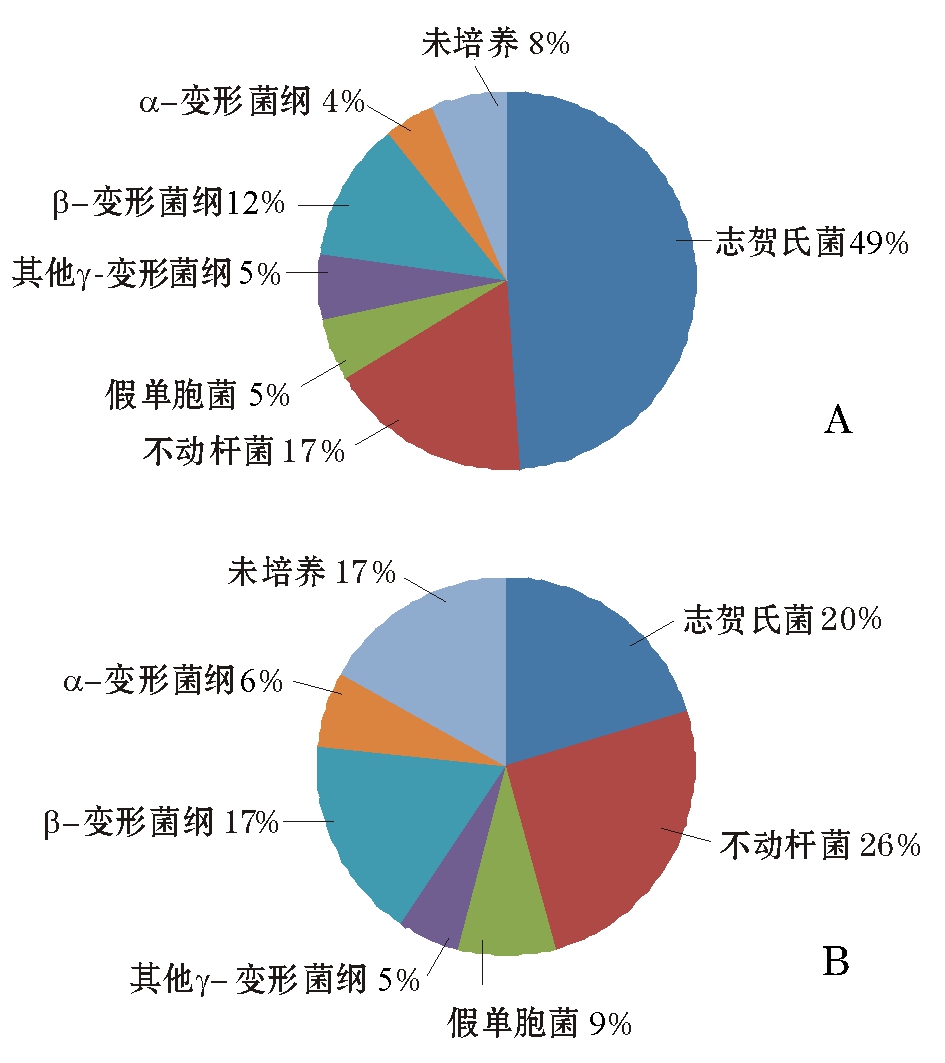
注:A 化皮刺参幼苗病灶;B 未化皮刺参幼苗表皮
Note:A, diseased juvenile sea cucumber with lesion;B, juvenile sea cucumber without lesion
图2 化皮参苗和未化皮参苗菌群结构的比较
Fig.2 Comparison of the flora in diseased juvenile sea cucumber with lesion and without lesion
表2化皮参苗病灶组织细菌16SrDNA克隆文库序列分析结果
Tab.2Blastsearchingfloraof16SrDNAclonesfromthediseasedjuvenileseacucumberwithlesions
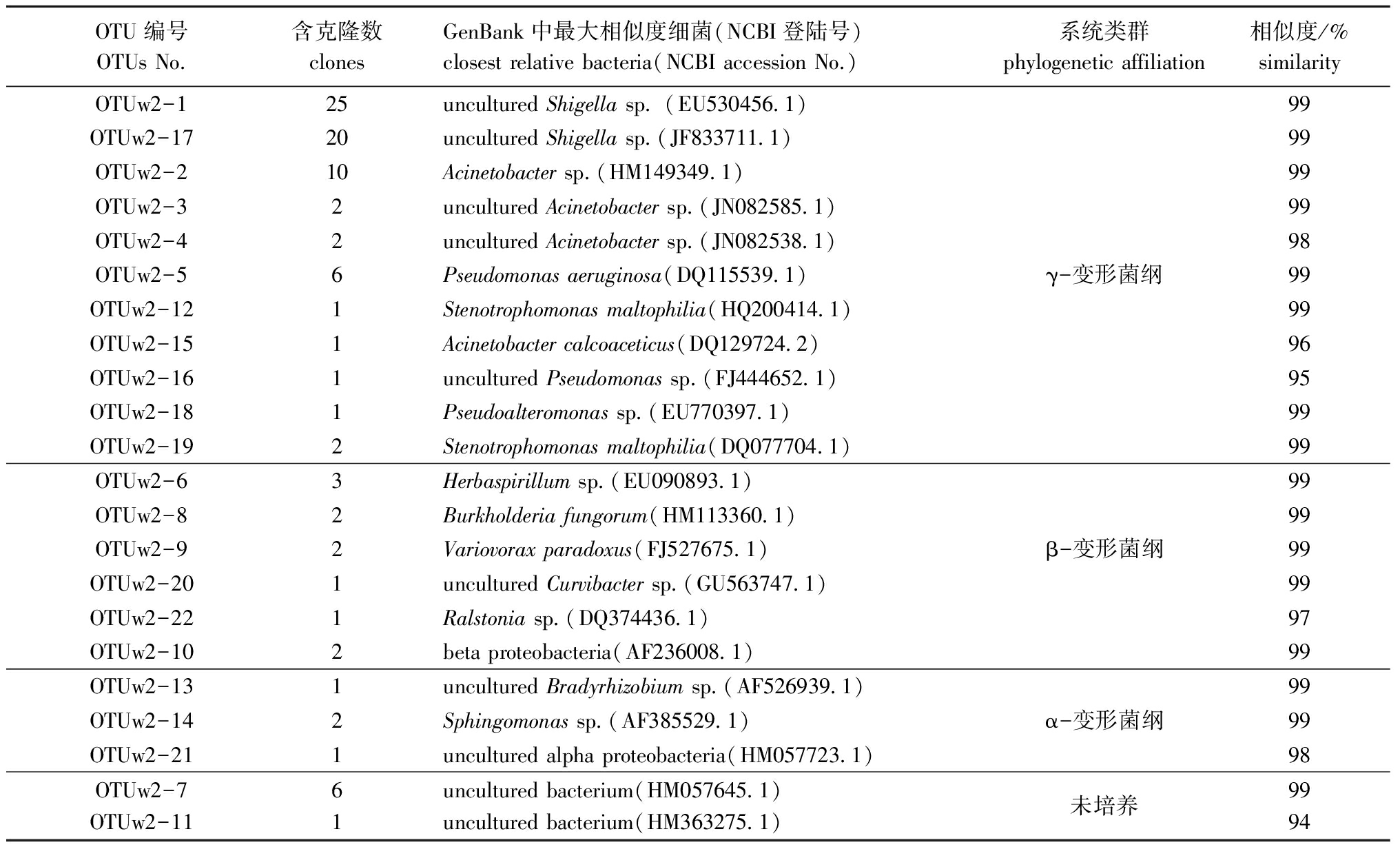
OTU编号OTUsNo.含克隆数clonesGenBank中最大相似度细菌(NCBI登陆号)closestrelativebacteria(NCBIaccessionNo.)系统类群phylogeneticaffiliation相似度/%similarityOTUw2-125unculturedShigellasp (EU530456 1)99OTUw2-1720unculturedShigellasp (JF833711 1)99OTUw2-210Acinetobactersp (HM149349 1)99OTUw2-32unculturedAcinetobactersp (JN082585 1)99OTUw2-42unculturedAcinetobactersp (JN082538 1)98OTUw2-56Pseudomonasaeruginosa(DQ115539 1)γ-变形菌纲99OTUw2-121Stenotrophomonasmaltophilia(HQ200414 1)99OTUw2-151Acinetobactercalcoaceticus(DQ129724 2)96OTUw2-161unculturedPseudomonassp (FJ444652 1)95OTUw2-181Pseudoalteromonassp (EU770397 1)99OTUw2-192Stenotrophomonasmaltophilia(DQ077704 1)99OTUw2-63Herbaspirillumsp (EU090893 1)99OTUw2-82Burkholderiafungorum(HM113360 1)99OTUw2-92Variovoraxparadoxus(FJ527675 1)β-变形菌纲99OTUw2-201unculturedCurvibactersp (GU563747 1)99OTUw2-221Ralstoniasp (DQ374436 1)97OTUw2-102betaproteobacteria(AF236008 1)99OTUw2-131unculturedBradyrhizobiumsp (AF526939 1)99OTUw2-142Sphingomonassp (AF385529 1)α-变形菌纲99OTUw2-211unculturedalphaproteobacteria(HM057723 1)98OTUw2-7OTUw2-1161unculturedbacterium(HM057645 1)unculturedbacterium(HM363275 1)未培养9994
表3未化皮参苗表皮细菌16SrDNA克隆文库序列分析结果
Tab.3Blastsearchingfloraof16SrDNAclonesfromthejuvenileseacucumberwithoutlesions
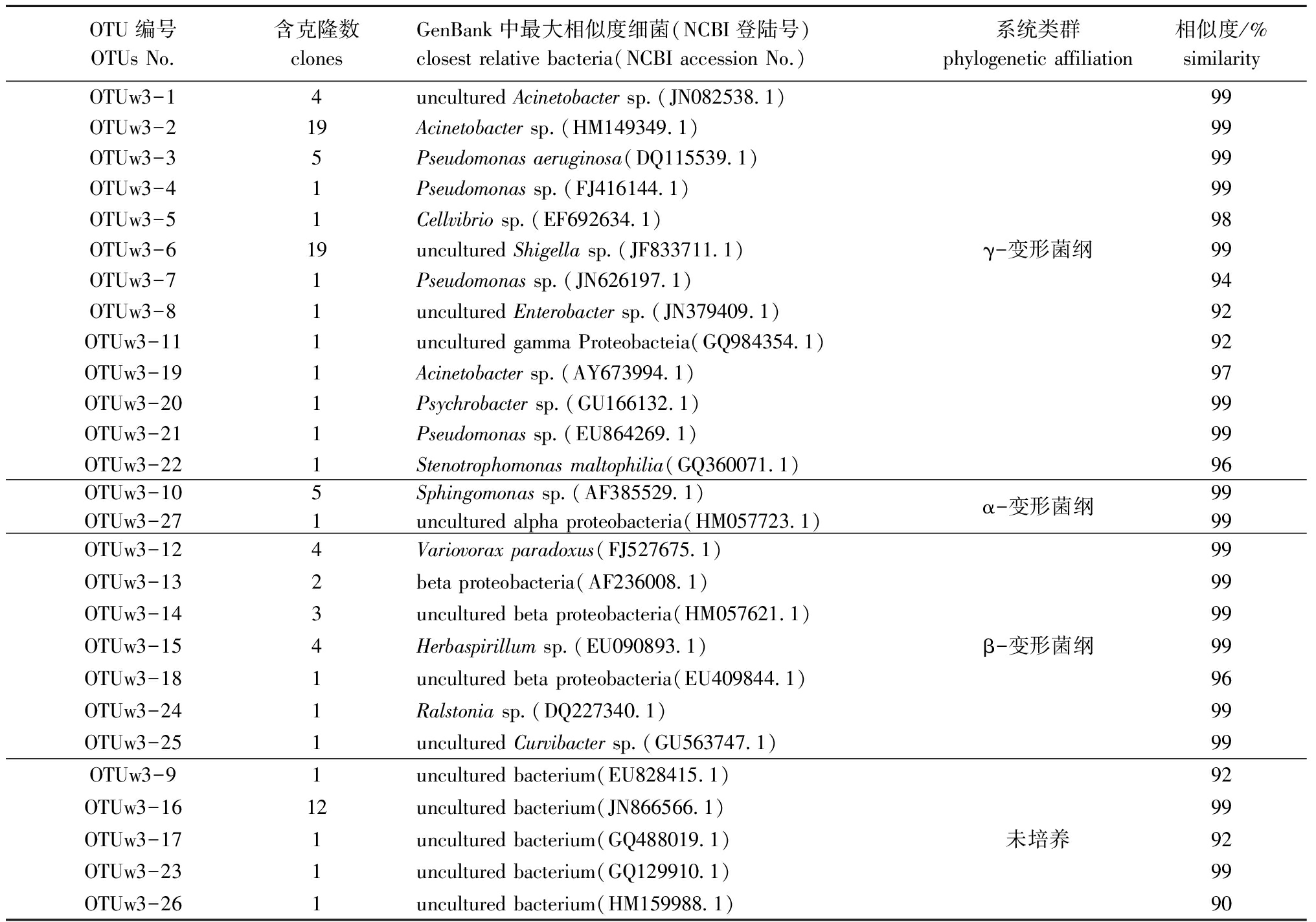
OTU编号OTUsNo.含克隆数clonesGenBank中最大相似度细菌(NCBI登陆号)closestrelativebacteria(NCBIaccessionNo.)系统类群phylogeneticaffiliation相似度/%similarityOTUw3-14unculturedAcinetobactersp (JN082538 1)99OTUw3-219Acinetobactersp (HM149349 1)99OTUw3-35Pseudomonasaeruginosa(DQ115539 1)99OTUw3-41Pseudomonassp (FJ416144 1)99OTUw3-51Cellvibriosp (EF692634 1)98OTUw3-619unculturedShigellasp (JF833711 1)γ-变形菌纲99OTUw3-71Pseudomonassp (JN626197 1)94OTUw3-81unculturedEnterobactersp (JN379409 1)92OTUw3-111unculturedgammaProteobacteia(GQ984354 1)92OTUw3-191Acinetobactersp (AY673994 1)97OTUw3-201Psychrobactersp (GU166132 1)99OTUw3-211Pseudomonassp (EU864269 1)99OTUw3-221Stenotrophomonasmaltophilia(GQ360071 1)96OTUw3-10OTUw3-2751Sphingomonassp (AF385529 1)unculturedalphaproteobacteria(HM057723 1)α-变形菌纲9999OTUw3-124Variovoraxparadoxus(FJ527675 1)99OTUw3-132betaproteobacteria(AF236008 1)99OTUw3-143unculturedbetaproteobacteria(HM057621 1)99OTUw3-154Herbaspirillumsp (EU090893 1)β-变形菌纲99OTUw3-181unculturedbetaproteobacteria(EU409844 1)96OTUw3-241Ralstoniasp (DQ227340 1)99OTUw3-251unculturedCurvibactersp (GU563747 1)99OTUw3-91unculturedbacterium(EU828415 1)92OTUw3-1612unculturedbacterium(JN866566 1)99OTUw3-171unculturedbacterium(GQ488019 1)未培养92OTUw3-231unculturedbacterium(GQ129910 1)99OTUw3-261unculturedbacterium(HM159988 1)90
2.4育苗池海水的菌群结构
对海水样品所得的OTU进行分析,得到所含克隆子数量及其相似菌株见表4,这些克隆子与GenBank数据库中已知细菌的16S rDNA序列相似性最高为99%,最低为94%,此文库所测得γ-变形菌纲、β-变形菌纲和α-变形菌纲的比例分别为93%、5%和2%。91个克隆子与来自γ-变形菌纲的菌株序列较为相似,其中,74个克隆子(OTUw1-1)与副溶血性弧菌Vibrioparahaemolyticus序列相似,相似度为99%,12个克隆子(OTUw1-2、3、12)与志贺氏菌相似,相似度均在96%以上,其余5个克隆子分别与创伤弧菌Vibriovulnificus、塔式弧菌Vibriotasmaniensis、铜绿假单胞菌Pseudomonasaeruginosa、弧菌属细菌Vibriosp.和盐单胞菌属细菌Halomonassp.序列相似;5个克隆子与来自β-变形菌纲的序列相似,分别与草螺菌属Herbaspirillumsp.、内生霉菌伯克霍尔德氏菌和Curvibactersp.菌属序列相似;2个克隆子与来自α-变形菌纲的菌株序列相似,分别与玫瑰杆菌属Roseobacter和鞘氨醇单胞菌属序列相似。海水克隆文库与患病组织的优势菌纲一样,为γ-变形菌纲,其中主要为弧菌(76%)和志贺氏菌(12%)。
表4海水细菌16SrDNA克隆文库序列分析结果
Tab.4Blastsearchingfloraof16SrDNAclonesfromtheseawater
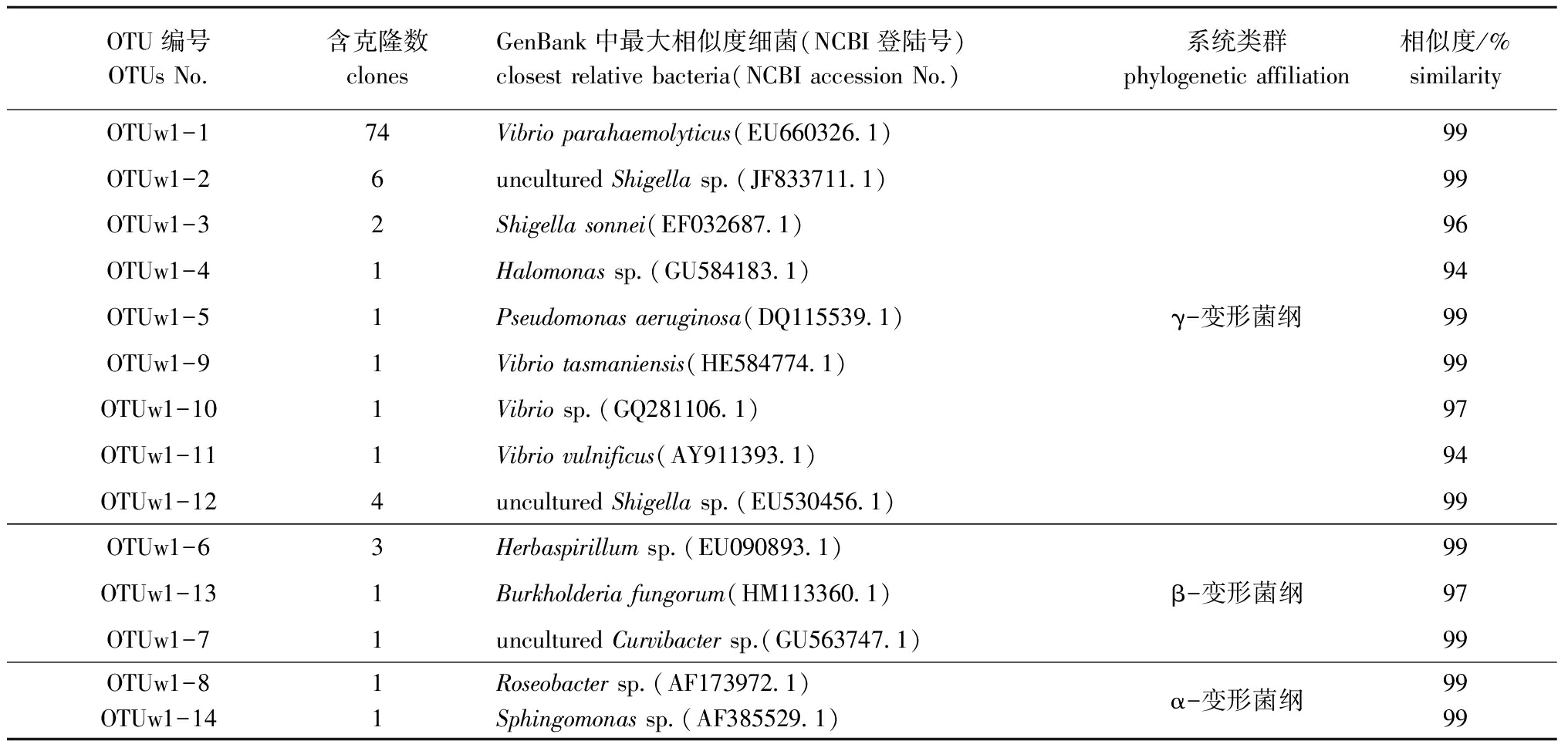
OTU编号OTUsNo.含克隆数clonesGenBank中最大相似度细菌(NCBI登陆号)closestrelativebacteria(NCBIaccessionNo.)系统类群phylogeneticaffiliation相似度/%similarityOTUw1-174Vibrioparahaemolyticus(EU660326 1)99OTUw1-26unculturedShigellasp (JF833711 1)99OTUw1-32Shigellasonnei(EF032687 1)96OTUw1-41Halomonassp (GU584183 1)94OTUw1-51Pseudomonasaeruginosa(DQ115539 1)γ-变形菌纲99OTUw1-91Vibriotasmaniensis(HE584774 1)99OTUw1-101Vibriosp (GQ281106 1)97OTUw1-111Vibriovulnificus(AY911393 1)94OTUw1-124unculturedShigellasp (EU530456 1)99OTUw1-63Herbaspirillumsp (EU090893 1)99OTUw1-131Burkholderiafungorum(HM113360 1)β-变形菌纲97OTUw1-71unculturedCurvibactersp.(GU563747 1)99OTUw1-8OTUw1-1411Roseobactersp (AF173972 1)Sphingomonassp (AF385529 1)α-变形菌纲9999
2.5病灶组织的细菌分离及其初步鉴定
分离获得的优势菌株BZ10,其菌落为白色,圆形,直径为0.05~0.10 mm,不透明,边缘整齐光滑,不黏稠。BZ10经 16S rDNA引物扩增得到长度为1426 bp的序列,经比对其与弧菌属细菌序列相似,且与菌株Vibriosp.(登陆号:EF187013.1)和溶藻弧菌(登录号:JN188406.1)的相似率均为100%。系统发育树显示,BZ10与溶藻弧菌亲缘关系较近,但置信度仅为20,不能鉴定到种(图3)。

图3 病灶组织优势菌株BZ10 16S rDNA系统发育树
Fig.3 Phylogenetic tree of the bacterial 16S rDNA from the BZ10
关于刺参养殖池塘养殖水环境中优势菌群的研究较多。关晓燕等[17]采用PCR-DGGE基因指纹技术对辽宁省普兰店刺参养殖池塘养殖水环境的菌群多样性进行了分析,结果表明,高温季节刺参养殖水环境中细菌大多数属于变形菌门,月优势菌群分别为假单胞菌、β-变形菌纲、绿藻门和γ-变形菌纲。李彬等[18]运用传统细菌分离培养与分子生物学技术相结合的方法对冬季养殖池塘环境的菌群进行了分析,结果显示,水体中以假单胞菌和芽孢杆菌为主。初航[19]对养殖池塘水体可培养微生物的多样性进行研究,结果表明,养殖水环境中涵盖菌株数最多的为芽孢菌属、弧菌属、盐芽孢杆菌和寡养单胞菌。本研究中,育苗池海水的菌群结构为γ-变形菌纲、α-变形菌纲和β-变形菌纲,其中γ-变形菌纲占为优势菌群,与相关研究基本一致。但是,在其γ-变形菌纲中,弧菌占绝大部分,还有一定量的志贺氏菌,表明用于参苗养殖的海水水质出现了一定程度的恶化。
志贺氏菌主要通过消化道途径传播,是引起人类传染性疾病的主要病原体之一,可引起细菌性痢疾[20]。有关水产动物体内志贺氏菌的研究多基于食品安全的角度。志贺氏菌可以在鱼、贝类体内生长繁殖,有造成水产品污染的可能[21]。对罗非鱼体中致病菌污染情况的研究发现,志贺氏菌阳性率达39.7%[22]。石晓辉等[23]在对某发病刺参的研究中,从溃烂刺参体壁组织中分离得到5株菌,其中包括1株痢疾志贺氏菌Shigelladysenteria,此外未见其他志贺氏菌对水产动物致病的报道。虽然石晓辉等[23]并未证实其对刺参的致病性,但发现这株痢疾志贺氏菌对蛋白具有较强的分解能力,认为其可分解刺参的体壁组织并导致刺参的腐皮综合症。本研究中,化皮参苗病灶组织和未化皮参苗表皮菌群类型基本一致,γ-变形菌纲为其中优势菌群,两者均含有大量的志贺氏菌、不动杆菌和假单胞菌,同时在海水中也检测到了一定量的志贺氏菌,显示了该养殖池及参苗微生态结构的特殊性。同时选取化皮参苗与未化皮参苗进行菌群结构研究,其中未化皮参苗可能未感染病菌,也可能已感染但尚未表现出症状。化皮刺参组织中的志贺氏菌量明显比未化皮参苗组织中的多,提示此次病例与志贺氏菌密切相关,志贺氏菌可能为此次病例的致病菌,也可能是刺参患病后才在病灶处大量滋生的次生细菌。虽然这仍有待于进一步研究证实,但可以明确,病灶处大量存在的志贺氏菌至少可通过对刺参体壁的分解破坏而加重病情。
本研究中,采用传统方法从刺参病灶处进行了细菌的分离培养,但是采集到的优势菌种却为弧菌,与16S rDNA克隆文库法所得优势菌为志贺氏菌的结果截然不同,其原因可能是培养条件所致。本研究中采用2216E培养基,而志贺氏菌属肠杆菌,兼性厌氧,王迎庆等[24]通过对SS 培养基、Mac 培养基、HE 培养基和MT 培养基进行比较,发现SS 培养基对志贺氏菌的检出率最高。在病原的分离培养研究中,培养基是否适宜影响研究结果的可靠性。自然界中大部分微生物从其原位环境中被带走时就会萎缩、凋谢,环境中发生的极其微小的变化都可能使其丧失生态位和生活力[25-26],从而难以在人工条件下完全复苏或降低可培养性。传统分离方法往往加入了浓度远高于自然状况的营养物质,其结果是在新的选择压力下群落结构通常会发生变化,适应富营养条件的菌种成为优势种,取代了自然条件下的优势种,使得传统方法分析结果可能具有片面性。不过,由于采用非培养手段进行病原研究的相关借鉴很少,研究方法造成的差异仍需进一步探讨。
参考文献:
[1] 杨志平,孙飞雪,刘志明,等.刺参肠道潜在产酶益生菌的筛选和鉴定[J].大连海洋大学学报,2013,28(1):17-20.
[2] 王轶南,穆晓虎,封妮莎,等.仿刺参体腔液中酚氧化酶活性的分析[J].大连海洋大学学报,2013,28(4):319-322.
[3] 杨嘉龙,周丽,绳秀珍,等.养殖刺参溃疡病病原菌RH2的鉴定及其生物学特性分析[J].水产学报,2007,31(4):504-511.
[4] 王印庚,方波,张春云,等.养殖刺参保苗期重大疾病“腐皮综合症”病原及其感染源分析[J].中国水产科学,2006,13(4):610-616.
[5] 马悦欣,徐高蓉,常亚青,等.大连地区刺参幼参溃烂病细菌性病原的初步研究[J].大连水产学院学报,2006,21(1):13-17.
[6] 王高学,原居林,赵云奎,等.刺参表皮溃烂病病原菌的分离鉴定与药敏试验[J].西北农林科技大学学报:自然科学版,2007,35(8):87-90.
[7] Li H, Qiao G,Gu J Q,et al.Phenotypic and genetic characterization of bacteria isolated from diseased cultured sea cucumberApostichopusjaponicusin northeastern China[J].Disease of Aquatic Organisms,2010,91:223-235.
[8] Deng H,He C B,Zhou Z C,et al.Isolation and pathogenicity of pathogens from skin ulceration disease and viscera ejection syndrome of the sea cucumberApostichopusjaponicus[J].Aquaculture,2009,287(1/2):18-27.
[9] 王轶南,刘艳萍,常亚青.患病与健康虾夷马粪海胆体腔液菌群的PCR-DGGE分析比较[J].中国农业科技导报,2011(1):111-116.
[10] Giovannoni S J,Britschgi T B,Moyer C L,et al.Genetic diversity in Sargasso Sea bacterioplankton[J].Nature,1990,345:60-63.
[11] Amann R I,Ludwig W,Schleifer K H.Phylogenetic identification and in situ detection of individual microbial cells with OTUs cultivation[J].Microbial Rev,1995,59:143-169.
[12] Kemp P F,Aller J Y.Bacterial diversity in aquatic and other environments:what 16S rDNA libraries can tell us[J].FEMS Micriobiol Ecol,2004,47:161-177.
[13] Hugenholtz P,Goebel B M,Pace N R.Impact of culture-independent studies on the emerging phylogenetic view of bacterial diversity[J].J Bacteriol,1998,180:4765-4774.
[14] 王大珍.微生物生态学的发展及应用[J].科学,1993,45(2):18-20.
[15] Irma N,Rivera L,Erin K.Method of DNA extraction and application of multiplex polymerase chain reaction to detect toxigenicVibriocholeraeO1 and O139 from aquatic ecosystems[J].Environ Microbiol,2003,5(7):599-606.
[16] Saitou N, Nei M.The neighbor-joining method:a new method for reconstructing phylogenetic trees[J].Mol Bio Ecol,1987,4:406-425.
[17] 关晓燕,周遵春,陈仲,等.应用PCR-DGGE指纹技术分析高温季节仿刺参养殖水环境中菌群多样性[J].海洋湖沼通报,2010(1):82-88.
[18] 李彬,荣小军,廖梅杰,等.冬季刺参养殖环境与肠道内细菌菌群的研究[J].海洋科学,2010,34(1):64-69.
[19] 初航.三疣梭子蟹养殖塘水体可培养微生物多样性研究[D].大连:辽宁师范大学,2008.
[20] Parsot C.Shigellatype Ⅲ secretion effectors:how,where,when,for what purposes[J].Current Opinion in Microbiology,2009,12(1):110-116.
[21] Shimotori S,Kojima F,Harushima H,et al.Evaluation of recovery methods ofShigellaspecies from fresh marine fish and shellfishes[J].Kansenshogaku Zasshi,1990,64(10):1337-1344.
[22] Onyango D M,Wandili S,Kakai R,et al.Isolation ofSalmonellaandShigellafrom fish harvested from the Winam Gulf of Lake Victoria,Kenya[J].J Infect Dev Ctries,2009,3(2):99-104.
[23] 石晓辉,李太武,陈燕,等.发病仿刺参细菌的分离鉴定和生长特性的研究[J].水产科学,2009,28(8):435-438.
[24] 王迎庆,蒋力,苏德茂.四种培养基对志贺氏菌属检出效果的实验比较[J].中国卫生检验杂志,1993,3(4):236-237.
[25] Kaeberlein T,Lewis K,Epstein S S.Isolating “uncultivable” microorganisms in pure culture in a simulated natural environment[J].Science,2002,296:1127-1129.
[26] Simu K,Hagstrom A.Oligotrophic bacterioplankton with a novel single-cell life strategy[J].Appl Environ Microbiol,2004,70:2445-2451.
Abstract:Flora on eipthelia of diseased juvenile sea cucumberApostichopusjaponicuswith body length of 3-5 cm showing body wall lesions was studied by a method of 16S rDNA cloning library. The results showed that the bacteria were composed of Alphaproteo bacteria, Betaproteo bacteria and Gammaproteo bacteria, with the predominant flora ofShigellasp.,Acinetobactersp. andPseudomonassp. on the lesions of the diseased sea cucumber as well as sea cucumber without obvious symptom. There was 49% ofShigellasp., 17% ofAcinetobactersp. and 5% ofPseudomonassp. in the diseased sea cucumber, while there was 20% ofShigellasp., 26% ofAcinetobactersp. and 9% ofPseudomonassp. in those without lesions. The members of Alphaproteo bacteria, Betaproteo bacteria and Gammaproteo bacteria were found in the seawater from the culture ponds as a control group, with predominant phaproteobacteria includingVibriosp.(75%) andShigellasp.(12%).Also, a predominant bacterium strain was isolated from the diseased cultured sea cucumber by a traditional method based on bacterial culture, and identified asVibriosp. by 16S rDNA cloning library. The findings indicate that the predominant flora is varied with the identifying method including a traditional method and 16S rDNA cloning library.
Key words:Apostichopusjaponicus; body wall lesion; flora; 16S rDNA cloning library
DOI:10.3969/J.ISSN.2095-1388.2014.03.001
文章编号:2095-1388(2014)03-0205-07
收稿日期:2013-09-12
基金项目:国家海洋局海洋公益性行业科研专项(201105007);辽宁省高校杰出青年学者成长计划项目(LjQ2011073); 国家“星火计划”项目(2012GA651002);辽宁省重大科技项目(2011203003);辽宁省教育厅科研项目(L2012263)
中图分类号:S947.9
文献标志码::A