表1引物序列
Tab.1Thesequenceoftheprimers
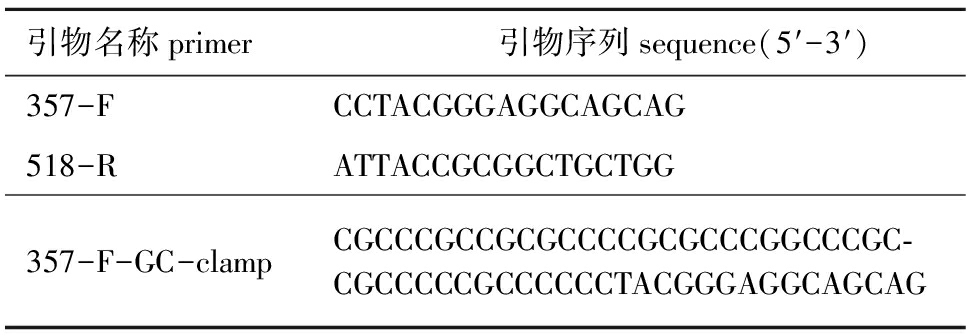
引物名称primer 引物序列sequence(5′-3′)357-FCCTACGGGAGGCAGCAG518-RATTACCGCGGCTGCTGG357-F-GC-clampCGCCCGCCGCGCCCCGCGCCCGGCCCGC⁃CGCCCCCGCCCCCCTACGGGAGGCAGCAG
摘要:采用PCR-DGGE技术,分析了鲢Hypophthalmichthysmolitrix、鳙Aristichthysnobilis肠道微生物的群落结构及其多样性。PCR-DGGE指纹图谱显示:鲢样品的平均条带数为16,个体间差异较小;鳙样品的平均条带数为14,个体间差异较大。基于PCR-DGGE指纹谱带的UPGMA聚类分析和相似性分析显示:鲢样品的平均相似系数为0.569,属于中等相似水平;鳙样品的平均相似系数为0.623,也属于中等相似水平。测序结果表明:鲢肠道微生物主要有拟杆菌门、 厚壁菌门、变形菌门和疣微菌门;鳙肠道微生物主要有拟杆菌门、变形菌门、蓝细菌门和厚壁菌门。
关键词:鲢;鳙;PCR-DGGE;肠道微生物;群落结构
肠道微生物是鱼类肠道的重要组成部分,其具有营养、防御和免疫调节等生理功能,并且对鱼类的生长发育具有重要影响[1-3]。鱼类肠道微生物与所处的肠道环境及鱼类生活的水体环境紧密相关,所以更容易受到食物供给、环境变化等的影响,且鱼类的生长发育也会对肠道微生物产生影响[4-5]。目前,已有学者研究了鲢Hypophthalmichthysmolitrix、鳙Aristichthysnobilis肠道微生物的群落结构和功能[6-7]。由于大部分肠道微生物处于不可培养状态,使得通过传统培养分离法研究环境微生物的群落结构比较困难。变性梯度凝胶电泳(DGGE)具有突变检出率高、检测片段小、重复性好、操作简便等优点,已被广泛应用于分析自然环境、肠道微生物的生物多样性和群落动态等的研究中[8-11]。本研究中,使用聚合酶链式反应-变性梯度凝胶电泳(PCR-DGGE)方法,研究了南太湖湖州水域鲢、鳙的肠道微生物,建立了肠道微生物的DGGE指纹图谱,分析其群落结构和多样性,以期为鱼类肠道微生物的鉴定、定植益生菌的开发和养殖环境的改善等提供依据。
1.1材料
于2011年11月在南太湖湖州水域采集野生鲢、鳙,每尾鱼的体质量均为4 kg左右。
1.2方法
在无菌环境下,取鱼肠道内含物,置于无菌离心管中,每种鱼设3个重复,并将样品于冰箱(-80 ℃)中保存备用。
1.2.1 肠道微生物总基因组DNA的提取 在无菌状态下,将鲢、鳙肠道内含物研磨成糊状,分别用粪便基因组DNA提取试剂盒(天根生化公司)提取鱼类肠道内含物的微生物总基因组,然后置于冰箱(-80 ℃)中保存备用。
1.2.2 16S rDNA的PCR扩增 采用原核微生物16S rDNA通用引物[12]357-F、518-R扩增样品肠道微生物基因组DNA的V3区,再将PCR产物用引物518-R、357-F-GC-clamp进行扩增,引物序列如表1所示。PCR反应体系(20 μL):10 μL 2×TaqPCR MasterMix(天根生化公司),2 μL引物(357-F 1 μL、518-R 1 μL),1 μL DNA,7 μL ddH2O。 PCR反应程序:95 ℃下预变性5 min;94 ℃下变性30 s,55 ℃下退火30 s,72 ℃下延伸30 s,共进行32个循环;最后在72 ℃下延伸10 min,于4 ℃下保温30 min。PCR产物经过GoldView染色后用10 g/L琼脂糖凝胶电泳进行检测。
1.2.3 PCR-DGGE指纹图谱 将扩增到的16SrDNA 基因V3区进行DGGE分离,其中变性剂梯度范围为30%~60%,电压设置为100 V,电泳12 h,用银染法染色,使用Imager EP(美国ALPHA)凝胶成像系统获取PCR-DGGE指纹图谱。
表1引物序列
Tab.1Thesequenceoftheprimers

引物名称primer 引物序列sequence(5′-3′)357-FCCTACGGGAGGCAGCAG518-RATTACCGCGGCTGCTGG357-F-GC-clampCGCCCGCCGCGCCCCGCGCCCGGCCCGC⁃CGCCCCCGCCCCCCTACGGGAGGCAGCAG
使用Quantity One(BIO-RAD Laboratories, CA, USA)软件对PCR-DGGE指纹图谱进行分析,得到DGGE图谱的相似系数,对DGGE图谱进行非加权配对算术平均法聚类分析(UPGMA)。
1.2.4 酶连接转化 将PCR产物用DNA片段凝胶回收试剂盒(北京三博远志生物技术有限公司)进行回收,并连接到PGM-T载体(天根生化公司)中,然后转入至大肠杆菌感受态E.coliDH5α中,将转化后的大肠杆菌感受态均匀涂布于LB(含氨苄青霉素100 μg/mL、IPTG、X-Gal)的固体培养基上,于37 ℃下恒温培养12 h。
1.2.5 测序 选择鉴定后的阳性克隆送交上海杰李生物技术有限公司进行测序。将测到的微生物序列在GeneBank里进行比对(http://blast.ncbi. nlm.nih.gov/),寻找并下载基因库中与测序结果最为相似的16S rDNA基因序列。
2.1鲢、鳙肠道菌群的PCR-DGGE指纹图谱
图1为太湖鲢、鳙肠道微生物的DGGE指纹图谱,L1~L3和Y1~Y3泳道分别代表鲢和鳙的3个样品,不同位置的条带代表不同的微生物,条带的明暗程度代表微生物的相对数量。图2为DGGE指纹图谱分析示意图,其中鲢L1~L3样品的条带数分别为18、18、12,平均条带数为16,鳙Y1~Y3样品的条带数分别为17、12、13,平均条带数为14。DGGE图谱分析表明,鲢肠道微生物种类多于鳙,但鳙个体之间的差异较鲢大,鲢、鳙肠道微生物存在明显差异,且各有不同的优势菌条带。
2.2指纹图谱的聚类分析和相似系数分析
从表2可见:鲢L1~L3样品的相似系数分别为0.745、0.576、0.386,平均为0.569;鳙Y1~Y3样品的相似系数分别为0.504、0.500、0.866,平均为0.623。相似系数分析表明,该批样品中鳙个体间肠道微生物群落结构的相似性高于鲢,说明采集的鲢、鳙样品之间肠道微生物有较大的差异。
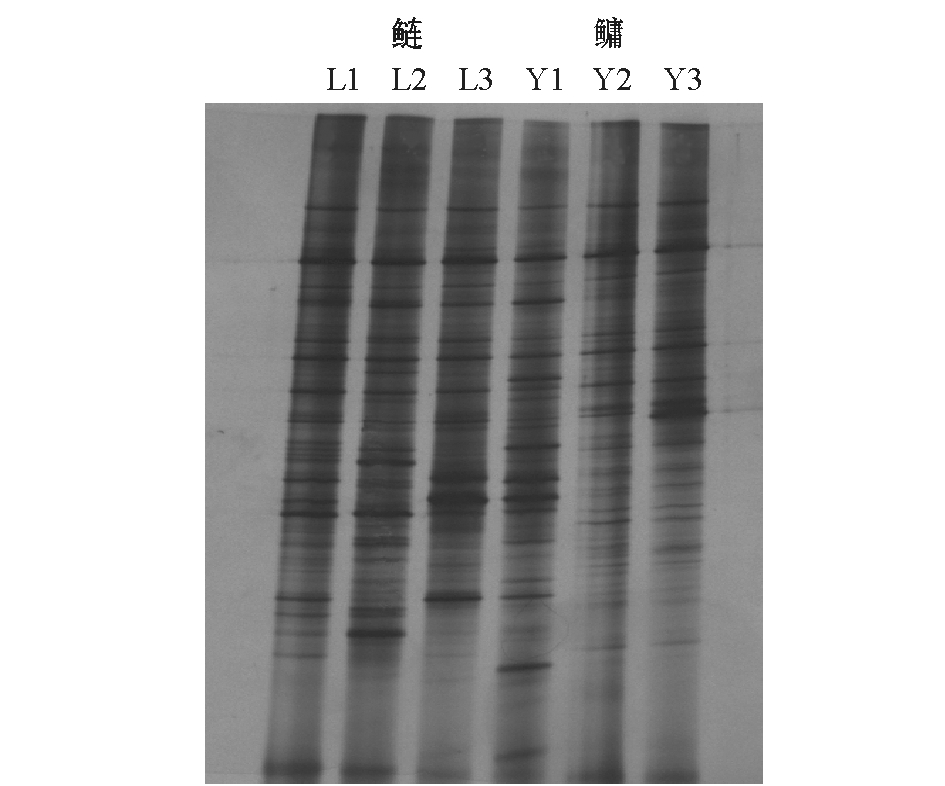
图1 鲢、鳙肠道微生物16S rDNA变性梯度凝胶电泳(DGGE)指纹图谱
Fig.1 Denaturing gradient gel electrophoresis(DGGE) fingerprinting of 16S rDNA intestinal microbials from silver carp Hypophthalmichthys molitrix and bighead carp Aristichthys nobilis
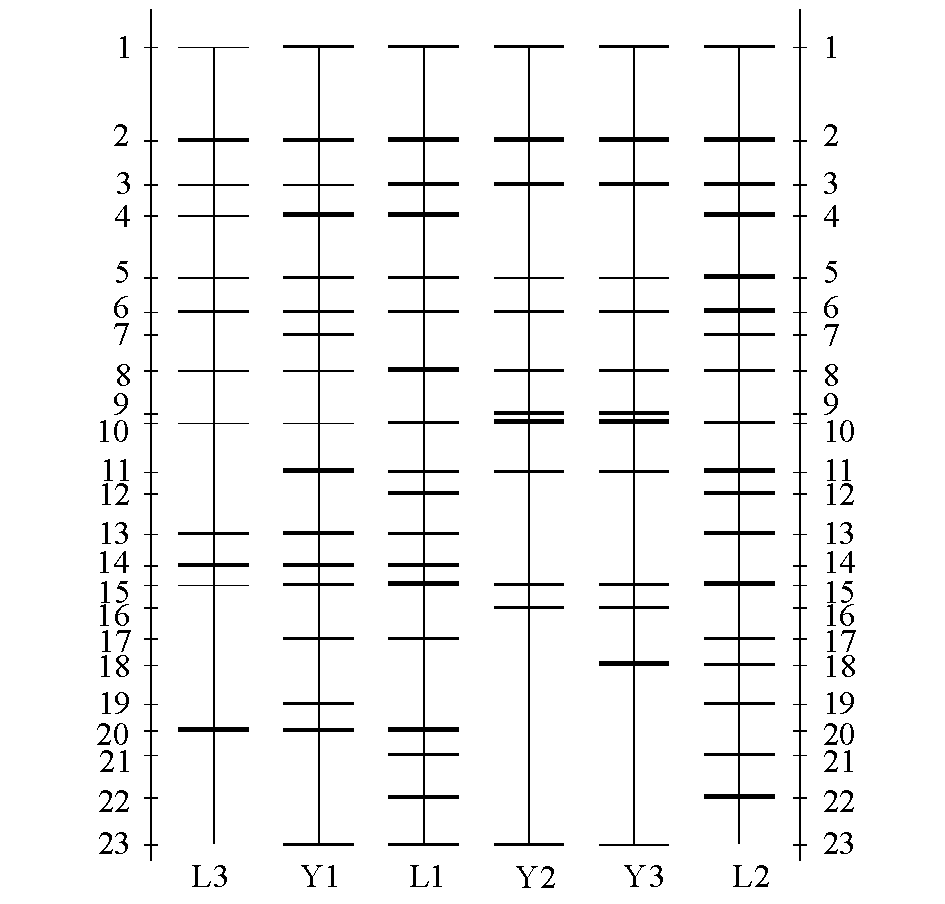
图2 鲢、鳙肠道微生物16S rDNA变性梯度凝胶电泳(DGGE)指纹图谱分析示意图
Fig.2 DGGE fingerprinting sketching map of 16S rDNA intestinal microbials from silver carp H.molitrix and bighead carp A.nobilis
基于鲢、鳙肠道菌群PCR-DGGE指纹图谱进行的UPGMA 聚类分析,结果如图3所示。其中,鲢L1、L2、L3与鳙Y1聚为一个大枝,鳙Y2、Y3聚为另一个大枝。
表2鲢、鳙肠道微生物DGGE图谱的相似系数
Tab.2SimilarityofDGGEfingerprintingforintestinalmicrobialsinsilvercarpH.molitrixandbigheadcarpA.nobilis
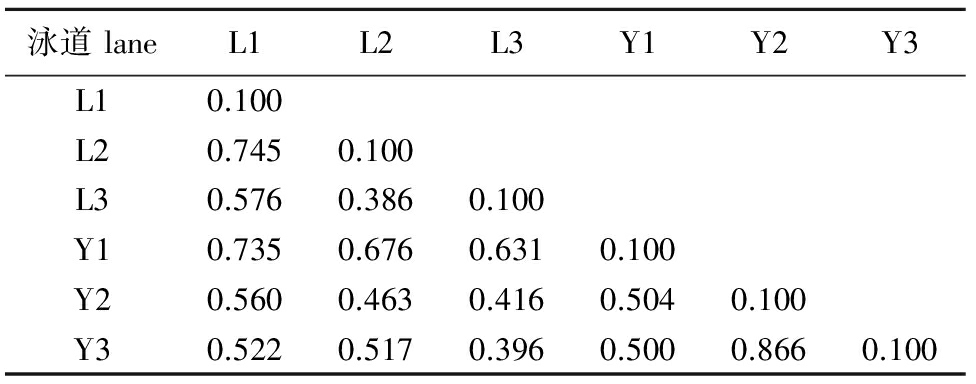
泳道laneL1L2L3Y1Y2Y3L10.100L20.7450.100L30.5760.3860.100Y10.7350.6760.6310.100Y20.5600.4630.4160.5040.100Y30.5220.5170.3960.5000.8660.100
2.3鲢、鳙肠道微生物的群落结构
对鲢、鳙肠道微生物进行测序,共检测了33个样品,成功测序样品27个,失败样品6个,其中鲢共检测到14个微生物序列,鳙共检测到13个微生物序列,测序结果如表3所示。鲢肠道微生物中检测到假单胞菌属、芽孢杆菌属、梭杆菌属、突柄杆菌属、乳球菌属和红细菌属等,鳙肠道微生物中检测到假单胞菌属、金黄杆菌属、气单胞菌属、芽孢杆菌属、肠杆菌属和蓝藻属的一些细菌等。分析表明,鲢肠道微生物主要有拟杆菌门、 厚壁菌门、变形菌门、梭杆菌门、疣微菌门,分别占测序结果的14.3%、28.6%、35.8%、14.3%和7.0%;鳙肠道微生物主要有拟杆菌门、厚壁菌门、变形菌门、蓝细菌门,分别占测序结果的15.4%、23.1%、53.8%和7.7%。
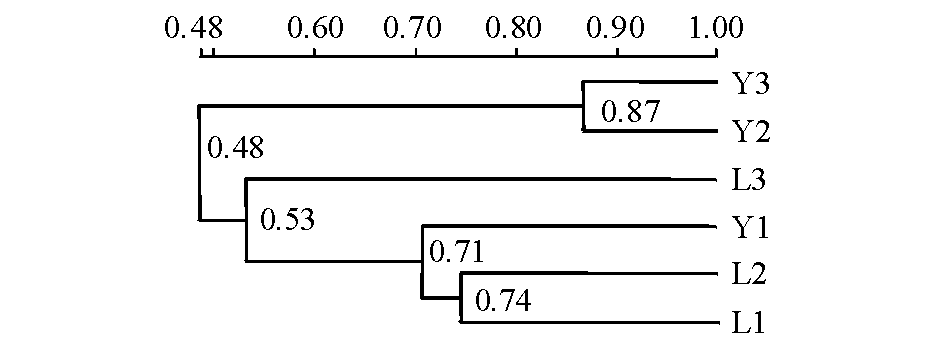
图3 鲢、鳙肠道微生物DGGE图谱的聚类分析
Fig.3 Cluster analysis of DGGE fingerprinting for intestinal microbials in silver carp H.molitrix and bighead carp A.nobilis
表3鲢、鳙肠道微生物16S rDNA特征片段序列分析结果
Tab.3Sequencinganalysisof16SrDNAintestinalmicrobialsfromsilvercarpH.molitrixandbigheadcarpA.nobilis
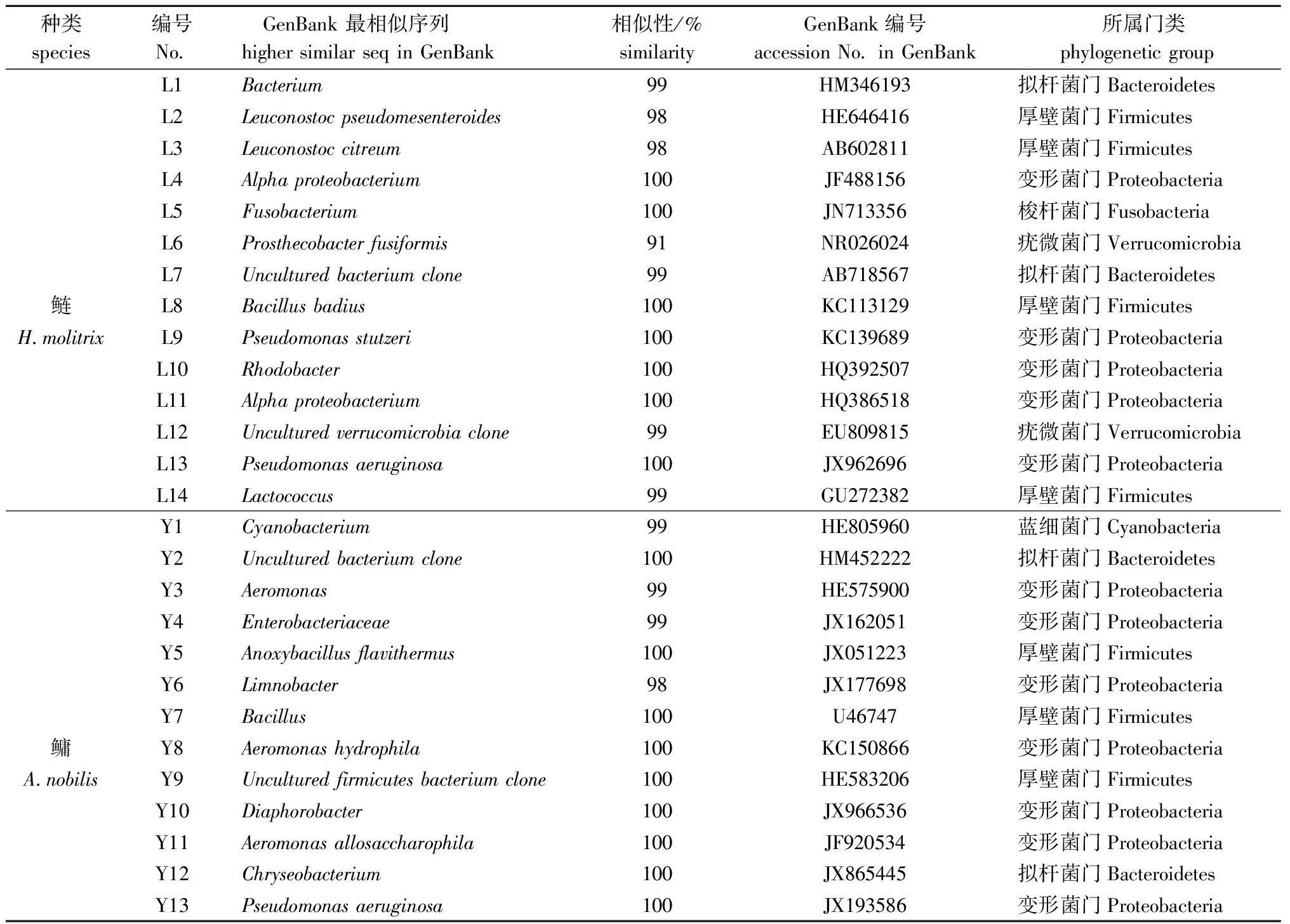
种类species编号No GenBank最相似序列highersimilarseqinGenBank相似性/%similarityGenBank编号accessionNo inGenBank 所属门类 phylogeneticgroupL1Bacterium99HM346193拟杆菌门BacteroidetesL2Leuconostocpseudomesenteroides98HE646416厚壁菌门FirmicutesL3Leuconostoccitreum98AB602811厚壁菌门FirmicutesL4Alphaproteobacterium100JF488156变形菌门ProteobacteriaL5Fusobacterium100JN713356梭杆菌门FusobacteriaL6Prosthecobacterfusiformis91NR026024疣微菌门VerrucomicrobiaL7Unculturedbacteriumclone99AB718567拟杆菌门Bacteroidetes鲢L8Bacillusbadius100KC113129厚壁菌门FirmicutesH molitrixL9Pseudomonasstutzeri100KC139689变形菌门ProteobacteriaL10Rhodobacter100HQ392507变形菌门ProteobacteriaL11Alphaproteobacterium100HQ386518变形菌门ProteobacteriaL12Unculturedverrucomicrobiaclone99EU809815疣微菌门VerrucomicrobiaL13Pseudomonasaeruginosa100JX962696变形菌门ProteobacteriaL14Lactococcus99GU272382厚壁菌门FirmicutesY1Cyanobacterium99HE805960蓝细菌门CyanobacteriaY2Unculturedbacteriumclone100HM452222拟杆菌门BacteroidetesY3Aeromonas99HE575900变形菌门ProteobacteriaY4Enterobacteriaceae99JX162051变形菌门ProteobacteriaY5Anoxybacillusflavithermus100JX051223厚壁菌门FirmicutesY6Limnobacter98JX177698变形菌门ProteobacteriaY7Bacillus100U46747厚壁菌门Firmicutes鳙Y8Aeromonashydrophila100KC150866变形菌门ProteobacteriaA nobilisY9Unculturedfirmicutesbacteriumclone100HE583206厚壁菌门FirmicutesY10Diaphorobacter100JX966536变形菌门ProteobacteriaY11Aeromonasallosaccharophila100JF920534变形菌门ProteobacteriaY12Chryseobacterium100JX865445拟杆菌门BacteroidetesY13Pseudomonasaeruginosa100JX193586变形菌门Proteobacteria
肠道为多种细菌提供了良好的生存环境,肠道细菌在宿主体内生存和增殖,对维持宿主的正常生命活动起着十分重要的作用。基于鱼类肠道内含物基因组的PCR-DGGE图谱及测定的序列能够反映鱼类肠道微生物的群落结构。刘淮德等[13]和颜庆云等[14]研究表明,PCR-DGGE技术是研究水产动物肠道微生物的有效方法,李可俊等[15]使用PCR-DGGE方法研究了长江口8种野生鱼类的肠道菌群,结果发现,鱼类肠道菌群多样性的差异与鱼类的食性相关, 且食性差异大的鱼类之间肠道菌群差异也更为明显。本研究中,使用PCR-DGGE技术建立了太湖鲢、鳙肠道微生物群落结构的DGGE图谱,鲢肠道微生物多样性略高于鳙,鲢、鳙肠道微生物存在明显差异,且各有不同的优势菌条带;基于PCR-DGGE指纹图谱的聚类分析和相似系数分析表明,鳙肠道微生物群落结构相似性高于鲢。
周玉法等[6]分析了常见的4中淡水鱼肠道菌群,结果发现,气单胞菌属在鲢肠道微生物菌群中占优势地位,且未检测出葡萄球菌属。李学梅等[16]对3种室内养殖鱼类的肠道微生物进行研究,结果发现,斑点叉尾鲴肠道中的菌群主要为变形杆菌,而银鲫和异育银鲫肠道中的菌群主要为梭杆菌属和气单胞菌属等。本研究中,在鲢肠道微生物中检测到假单胞菌属、芽孢杆菌属、梭杆菌属、突柄杆菌属、乳球菌属、红细菌属等,在鳙肠道微生物中检测到假单胞菌属、金黄杆菌属、气单胞菌属、芽孢杆菌属、肠杆菌属和蓝藻属的一些细菌等。在鲢、鳙肠道中都检测到芽孢杆菌属和假单胞菌属的微生物,此结果与Zhou等[17]和赵庆新[18]在鲤科鱼肠道菌群中未检测到芽孢杆菌属的研究结果并不一致。芽孢杆菌是较常用的水体改良剂和饲料添加剂,不仅可以有效地抑制病原菌等有害微生物的生长,而且可以分解有机物质、有机硫化物等,改善水体环境;芽孢杆菌还能产生多种消化酶,提高鱼类对营养物质的吸收等。而假单胞菌是常见的条件致病菌,在鲢、鳙肠道中均有检出,虽然检出率不高,但也可以反映出,此时期南太湖水域的水质不理想。而与其他报道出现的差异有可能是采样区域不同或是试验方法不同所致。
Feng等[19]、Kim等[20]研究表明,变形菌门的微生物是鱼类肠道微生物的优势菌群。本试验结果表明:鲢肠道微生物群落组成有拟杆菌门、 厚壁菌门、变形菌门、疣微菌门,优势种群为变形菌门和厚壁菌门,占测序总数的35.8%和28.6%;鳙肠道微生物群落组成有拟杆菌门、变形菌门、蓝细菌门、厚壁菌门,其中变形菌门和厚壁菌门的微生物占有明显的优势,占测序总数的53.8%和23.1%。该结果与Feng等[19]、Kim等[20]的研究结果一致。本试验中,在鲢肠道内还检测到疣微菌门,这在其他鱼类肠道微生物的研究报道中很少提及,因为疣微菌门是被新分类出不久的一类细菌,目前只识别了少数几个种类,主要发现于水体、土壤环境和人类粪便中,其作用目前尚不清楚。在鳙肠道中还检测到了蓝藻,控制蓝藻是治理太湖水域环境的一个重要工作,蓝藻的爆发会严重影响太湖的水质安全与环境,在该批样品中只检测到一个蓝藻序列,表明此时期南太湖湖州水域中蓝藻相对较少,说明对太湖的治理工作有了一定的效果。
鱼类肠道微生物的数量和群落结构与其生境、发育、摄食饵料等密切相关。Yoshimizu等[21]研究表明,鱼类肠道微生物的群落结构具有季节性差异,即微生物数量在夏季最多,而在冬季最少;尹军霞等[22]研究表明,南美白对虾肠道厌氧菌总数、双歧杆菌随水温的升高而显著增加;Ward等[23]研究表明,杂食性鱼类肠道微生物的多样性比肉食性鱼类高;尹军霞等[24]研究表明,肉食性鱼类肠道中厌氧菌和双歧杆菌比草食性多;周文豪等[25]研究表明,摄食不同饵料会对草鱼肠道菌群产生影响。本研究中的样品全部为同一时期采自南太湖湖州水域,并且均取3个个体进行同种鱼个体之间的比较,降低了环境等因素对肠道微生物群落结构的影响,虽然鲢、鳙个体之间肠道菌群的PCR-DGGE图谱并不完全一致,但聚类分析发现,同种鱼的不同个体之间的相似性较高。
对鱼类肠道微生物群落结构进行研究,可为开发饲料添加剂提供科学依据。张梁等[26]用添加光合细菌的饲料喂养草鱼, 能够显著加快草鱼的生长发育,降低饵料系数,在一定范围内,超氧化物歧化酶的活性可随添加水平的增加而上升。由于国内养殖废水很少进行处理,直接排放到自然环境中,对环境造成了很大影响,研究鱼类肠道微生物,可以指导合理使用微生物制剂,减少对环境的污染,从而改善鱼类的生长性能和开发定植益生菌等。
参考文献:
[1] 宋增福,吴天星.鱼类肠道正常菌群研究进展[J].水产科学,2007,26(8):471-474.
[2] 吕欣荣,肖克宇.鱼类肠道菌群的研究现状[J].江西水产科技,2008(2):12-18.
[3] 赵明晓,贺永亮.肠道细菌与宿主共生的研究进展[J].重庆文理学院学报:自然科学版,2008,27(2):56-59.
[4] Spanggaard B,Huber I,Nielsen J,et al.The microflora of rainbow trout intestine:a comparison of traditional and molecular identification[J].Aquaculture,2000,182(1):1-15.
[5] Savas S,Kubilay A,Basmaz N.Effect of bacterial load in feeds on intestinal microflora of seabream (Sparusaurata) larvae and juveniles[J].The Israeli Journal of Aquaculture-Bamidgeh,2005,57(1):3-9.
[6] 周玉法,李代军,刘东燕,等.4 种常见淡水鱼肠道菌群的分析[J].中国畜牧兽医,2012,39(8):220-223.
[7] 王琴,熊邦喜,朱玉婷,等.主养草鱼池塘三种混养模式下鱼类肠道菌群PCR-DGGE比较[J].农业生物技术学报,2012,20(3):308-315.
[8] 刘上峰,傅俊江,李麓芸.变性梯度凝胶电泳的原理、应用及其进展[J].国外医学:遗传学分册,2002,25(2):74-76.
[9] 马悦欣,Holmstrm C,Webb J,et al.变性梯度凝胶电泳(DGGE)在微生物生态学中的应用[J].生态学报,2003,23(8):1561-1569.
[10] 常亚青,封妮莎,王轶南,等.虾夷马粪海胆致病菌强壮弧菌的PCR检测方法[J].大连海洋大学学报,2012,27(4):289-293.
[11] 马悦欣,王颖,杨凤,等.应用PCR-DGGE技术研究四角蛤蜊的细菌多样性[J].大连海洋大学学报,2011,26(3):243-246.
[12] 张明月.DGGE用于两种不同类型生境的微生物群落结构的研究[D].上海:上海海洋大学,2012.
[13] 刘淮德,王雷,王宝杰,等.应用PCR-DGGE分析南美白对虾肠道微生物多样性[J].饲料工业,2008,29(20):55-58.
[14] 颜庆云,余育和,张堂林,等.滤食性鲢、鳙肠含物PCR-DGGE指纹分析[J].水产学报,2009,33(6):972-979.
[15] 李可俊,管卫兵,徐晋麟,等.PCR-DGGE对长江河口八种野生鱼类肠道菌群多样性的比较研究[J].中国微生态学杂志,2007,19(3):268-272.
[16] 李学梅,余育和,解绶启,等.三种室内饲养鱼类肠道微生物群落PCR-DGGE指纹分析[J].水生生物学报,2011,35(3):423-429.
[17] Zhou Z,Liu Y,Shi P,et al.Molecular characterization of the autochthonous microbiota in the gastrointestinal tract of adult yellow grouper (Epinephelusawoara) cultured in cages[J].Aquaculture,2009,286(3):184-189.
[18] 赵庆新.鲤科(Cyprinidae)鱼肠道菌群分析[J].微生物学杂志,2001,21(2):18-20.
[19] Feng J B,Hu C Q,Luo P,et al.Microbiota of yellow grouper (EpinephelusawooraTemminck & Schlegel,1842) fed two different diets[J].Aquaculture Research,2010,41(12):1778-1790.
[20] Kim D H,Brunt J,Austin B.Microbial diversity of intestinal contents and mucus in rainbow trout (Oncorhynchusmykiss)[J].Journal of Applied Microbiology,2007,102(6):1654-1664.
[21] Yoshimizu M,Kimura T,Sakai M.Studies on the intestinal microflora of salmonids:Ⅰ.The intestinal microflora of fish reared in fresh water and sea water[J].Bulletin of the Japanese Society of Scientific Fisheries,1976,42:1281-1290.
[22] 尹军霞,沈文英,郦萍.水温对南美白对虾肠道菌群影响的研究[J].海洋科学,2004,28(5):33-36.
[23] Ward N L,Steven B,Penn K,et al.Characterization of the intestinal microbiota of two Antarctic notothenioid fish species[J].Extremophiles,2009,13(4):679-685.
[24] 尹军霞,沈文英,张建龙,等.不同食性鱼肠道壁菌群的研究[J].水利渔业,2003,23(5):7-8.
[25] 周文豪,陈孝煊.摄食不同饵料对草鱼肠道菌群影响的研究[J].华中农业大学学报,1998,17(3):252-256.
[26] 张梁,张伟,任小丽. 光合细菌对草鱼生长及免疫相关酶活性的影响[J].粮食与饲料工业,2007(2):37-38.
Abstract:The intestinal microbial community structure and diversity were studied in silver carpHypophthalmichthysmolitrixand bighead carpAristichthysnobilisby PCR-DGGE (Polymerase chain reaction-denaturing gradient gel electrophoresis) technique. PCR-DGGE fingerprinting showed that the silver carp had an average band of 16, with few differences between individuals, and the bighead carp had 14 bands on an average, with large differences between individuals. UPGMA cluster analysis and similarity analysis of PCR-DGGE fingerprint bands revealed that the silver carp had average similarity coefficient of 0.569,at a medium similar level, and the bighead carp had average similarity coefficient of 0.623,also at a medium similar level. The sequencing results showed that the main intestinal microbials in silver carp were consisted of Bacteroidetes, Firmicutes, Proteobacteria and Verrucomicrobia, and Bacteroidetes, Proteobacteria, Cyanobacteria and Firmicutes were found in the main intestinal microbials in the bighead carp.
Key words:Hypophthalmichthysmolitrix;Aristichthysnobilis; PCR-DGGE; intestinal microbial flora; community structure
DOI:10.3969/J.ISSN.2095-1388.2014.01.005
文章编号:2095-1388(2014)01-0022-05
收稿日期:2013-05-19
基金项目:国家水体污染控制与治理科技重大专项(2008ZX07101-006-09)
中图分类号:S917.1
文献标志码::A