基于微卫星标记的刺参群体遗传结构分析及与经济性状的相关性研究
李云霞,李娇,丁君,常亚青
(大连海洋大学农业部北方海水增养殖重点开放实验室,辽宁大连116023)
摘要:采用微卫星分子标记技术对7个刺参Apostichopus japonicus(Selenka)群体:辽宁葫芦岛养殖群体、山东烟台野生群体、山东莱州养殖群体、大连旅顺底播群体、丹东东港养殖群体、大连广鹿岛底播群体、大连黑石礁野生群体的遗传多样性进行分析,并随机抽取7个群体中的180头刺参进行体长、体宽、体积、体质量和总棘数与17个微卫星标记之间的相关性研究。结果表明:用12个微卫星标记共扩增获得83个等位基因,每个位点的等位基因数为3~12个,长度为102~368 bp。刺参群体位点多样性分析显示,各刺参群体等位基因数为5.083 3~5.416 7,平均值为5.261 9;有效等位基因数为3.205 0~3.701 1,平均值为3.446 8;观测杂合度为0.547 5~0.686 9,平均值为0.614 9;期望杂合度为0.649 1~0.697 1,平均值为0.674 1;多态性信息含量为0.598 9~0.638 6,平均值为0.620 0。对各群体的Hardy-Weinber平衡检测显示,7个刺参群体均存在不同程度的平衡偏离。刺参群体遗传分化与基因流分析显示,刺参群体间遗传分化系数平均值为0.078 2,变异主要来源于群体内;基因流平均值为5.301 9,刺参群体间存在一定程度的基因交流。刺参群体间遗传相似性系数为0.731 8~0.886 0,UPGMA聚类显示,7个群体中山东烟台野生群体单独为一类,其他6个群体聚为一类。刺参微卫星标记与经济性状相关性分析显示,HC312、EAJ07位点与刺参体长、体积、体质量显著相关 (P<0.05);IS45、EAJ03、EAJ04位点与刺参体长、体积、体宽、体质显著相关 (P<0.05);SJ04位点仅与刺参总棘数显著相关 (P<0.05);SJ09位点与刺参体积、体宽、体质量显著相关 (P<0.05);SJ18位点仅与刺参体宽显著相关 (P<0.05);SJ19位点与刺参体积、体宽、体质量、总棘数显著相关 (P<0.05);SP072位点与刺参体积、体质量显著相关 (P<0.05)。本研究结果可为刺参群体结构优化、经济性状的QTL定位、养殖和选育提供参考。
关键词:刺参;微卫星;遗传多样性;经济性状
刺参Apostichopus japonicus(Selenka)自然分布于中国北方沿海、朝鲜半岛、日本和俄罗斯远东地区。近年来刺参养殖在中国北方沿海蓬勃开展, 2011年产量达到13.7万t,刺参养殖已成为具有北方特色的海水养殖支柱产业之一,但随着养殖规模的不断扩大,刺参良种的缺乏和养殖环境的恶化和病害的频发等均影响着刺参产业的发展[1],其中刺参良种的缺乏是制约刺参养殖业发展的瓶颈。进行刺参种质改良首先应对其种质资源、遗传结构特征进行分析;此外,在水产经济动物育种中,由于对目标性状缺乏准确可靠的早期预选手段,育种盲目性大、效率低也是急需解决的问题。DNA分子标记技术是进行物种群体结构分析和资源调查的有效手段,与经济性状相关的DNA标记可实现水产经济动物育种目标性状的早期选择。
微卫星标记技术 (SSR)广泛应用于物种鉴定与遗传变异分析、群体结构与亲缘关系分析、分子标记辅助选择等领域[2]。目前,海洋生物微卫星标记日益丰富[3-6],但有关刺参微卫星标记的报道有限[7-9],Kang等[10]、 Kim 等[11]、 Chang等[12]分别用微卫星标记对不同刺参群体进行了遗传差异、遗传结构和遗传多样性的分析。本研究中应用微卫星标记对山东烟台、莱州,以及辽宁葫芦岛、大连、丹东的7个刺参群体进行遗传多样性分析及微卫星标记与刺参主要经济性状的相关性研究,以期为刺参的养殖及选育提供理论基础。
1 材料与方法
1.1 材料
7个刺参群体分别为山东烟台野生群体(SDYS)、大连黑石礁野生群体 (DLYS)、丹东东港养殖群体 (DD)、葫芦岛养殖群体 (HLD)、旅顺底播群体 (LS)、山东莱州养殖群体 (SDYZ)、广鹿岛底播群体 (GLD)。每个群体取30头,分别取其管足存放于冰箱 (-80℃)中保存备用。随机取其中180头刺参,测量其体长、体宽、体积、体质量和总棘数5个经济性状,用于微卫星与刺参经济性状的相关性研究。
1.2 方法
1.2.1 基因组DNA的提取 刺参基因组DNA的提取参照文献 [13],用10 g/L琼脂糖凝胶电泳进行检测。
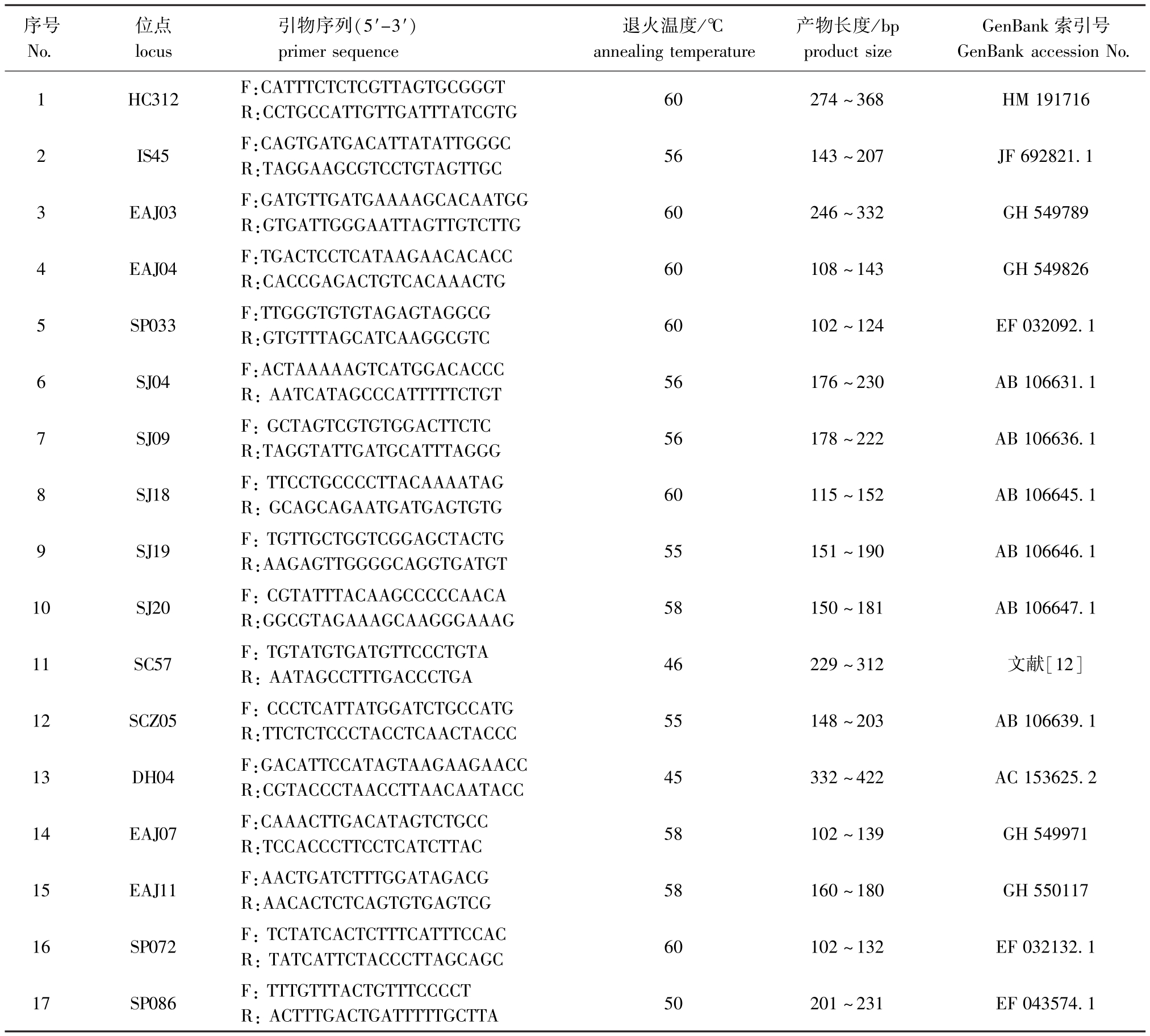
1.2.2 PCR扩增 微卫星引物序列和退火温度见表1,引物由上海生工生物技术有限公司合成。微卫星反应体系共20μL,包括DNA模板3μL,10× Buffer 2μL,上、下游引物各0.8μL,2.5 mmol/L dNTP 1.6μL,25 mmol/L MgCl21.2μL,5 U/μLTaq酶0.1μL,用双蒸水补至20μL。反应程序: 94℃下预变性5 min;94℃下变性30 s,退火30 s,72℃下延伸30 s,共进行30个循环;最后在72℃下再延伸10min。PCR扩增产物采用8%的非变性聚丙烯酰胺凝胶电泳,银染检测。
表1 刺参微卫星引物信息
Tab.1 Information ofm icrostaellitemarkers used in sea cucumber Apostichopus japonicus(Selenka)
序号No.位点locus引物序列(5'-3') primer sequence退火温度/℃annealing temperature产物长度/bp product size GenBank索引号GenBank accession No. 1 HC312 F:CATTTCTCTCGTTAGTGCGGGT R:CCTGCCATTGTTGATTTATCGTG 60 274~368 HM 191716 2 IS45 F:CAGTGATGACATTATATTGGGC R:TAGGAAGCGTCCTGTAGTTGC 56 143~207 JF 692821.1 3 EAJ03 F:GATGTTGATGAAAAGCACAATGG R:GTGATTGGGAATTAGTTGTCTTG 60 246~332 GH 549789 4 EAJ04 F:TGACTCCTCATAAGAACACACC R:CACCGAGACTGTCACAAACTG 60 108~143 GH 549826 5 SP033 F:TTGGGTGTGTAGAGTAGGCG R:GTGTTTAGCATCAAGGCGTC 60 102~124 EF 032092.1 6 SJ04 F:ACTAAAAAGTCATGGACACCC R:AATCATAGCCCATTTTTCTGT 56 176~230 AB 106631.1 7 SJ09 F:GCTAGTCGTGTGGACTTCTC R:TAGGTATTGATGCATTTAGGG 56 178~222 AB 106636.1 8 SJ18 F:TTCCTGCCCCTTACAAAATAG R:GCAGCAGAATGATGAGTGTG 60 115~152 AB 106645.1 9 SJ19 F:TGTTGCTGGTCGGAGCTACTG R:AAGAGTTGGGGCAGGTGATGT 55 151~190 AB 106646.1 10 SJ20 F:CGTATTTACAAGCCCCCAACA R:GGCGTAGAAAGCAAGGGAAAG 58 150~181 AB 106647.1 11 SC57 F:TGTATGTGATGTTCCCTGTA R:AATAGCCTTTGACCCTGA 46 229~312 文献[12] 12 SCZ05 F:CCCTCATTATGGATCTGCCATG R:TTCTCTCCCTACCTCAACTACCC 55 148~203 AB 106639.1 13 DH04 F:GACATTCCATAGTAAGAAGAACC R:CGTACCCTAACCTTAACAATACC 45 332~422 AC 153625.2 14 EAJ07 F:CAAACTTGACATAGTCTGCC R:TCCACCCTTCCTCATCTTAC 58 102~139 GH 549971 15 EAJ11 F:AACTGATCTTTGGATAGACG R:AACACTCTCAGTGTGAGTCG 58 160~180 GH 550117 16 SP072 F:TCTATCACTCTTTCATTTCCAC R:TATCATTCTACCCTTAGCAGC 60 102~132 EF 032132.1 17 SP086 F:TTTGTTTACTGTTTCCCCT R:ACTTTGACTGATTTTTGCTTA 50 201~231 EF 043574.1
1.3 数据处理
1.3.1 刺参群体遗传参数的计算 利用PopGene 1.32软件计算各刺参群体的等位基因数 (Na)、有效等位基因数 (Ne)、观测杂合度 (Ho)、期望杂合度 (He);单个群体水平近交系数 (Fis)、总群体水平近交系数 (Fit)、群体间分化系数 (Fst)和基因流 (Nm);群体间遗传相似系数、遗传距离。同时利用该软件检测7个刺参群体在各个位点的Hardy-Weinberg平衡,并计算7个群体在各个微卫星位点上的多态性信息含量 (PIC)。
1.3.2 微卫星分子标记与经济性状间的相关分析
用SPSS 13.0软件进行刺参经济性状与17个微卫星位点的最小二乘法分析,确定二者的相关性。
2 结果
2.1 刺参群体微卫星PCR扩增
利用表1中1~12号微卫星引物对7个刺参群体进行PCR扩增,结果表明,12个微卫星引物均能在7个群体中稳定、重复地扩增出相应条带,部分扩增结果见图1。12个微卫星位点共获得83个等位基因,每个位点的等位基因数为3~12。
2.2 刺参群体位点的遗传多样性分析
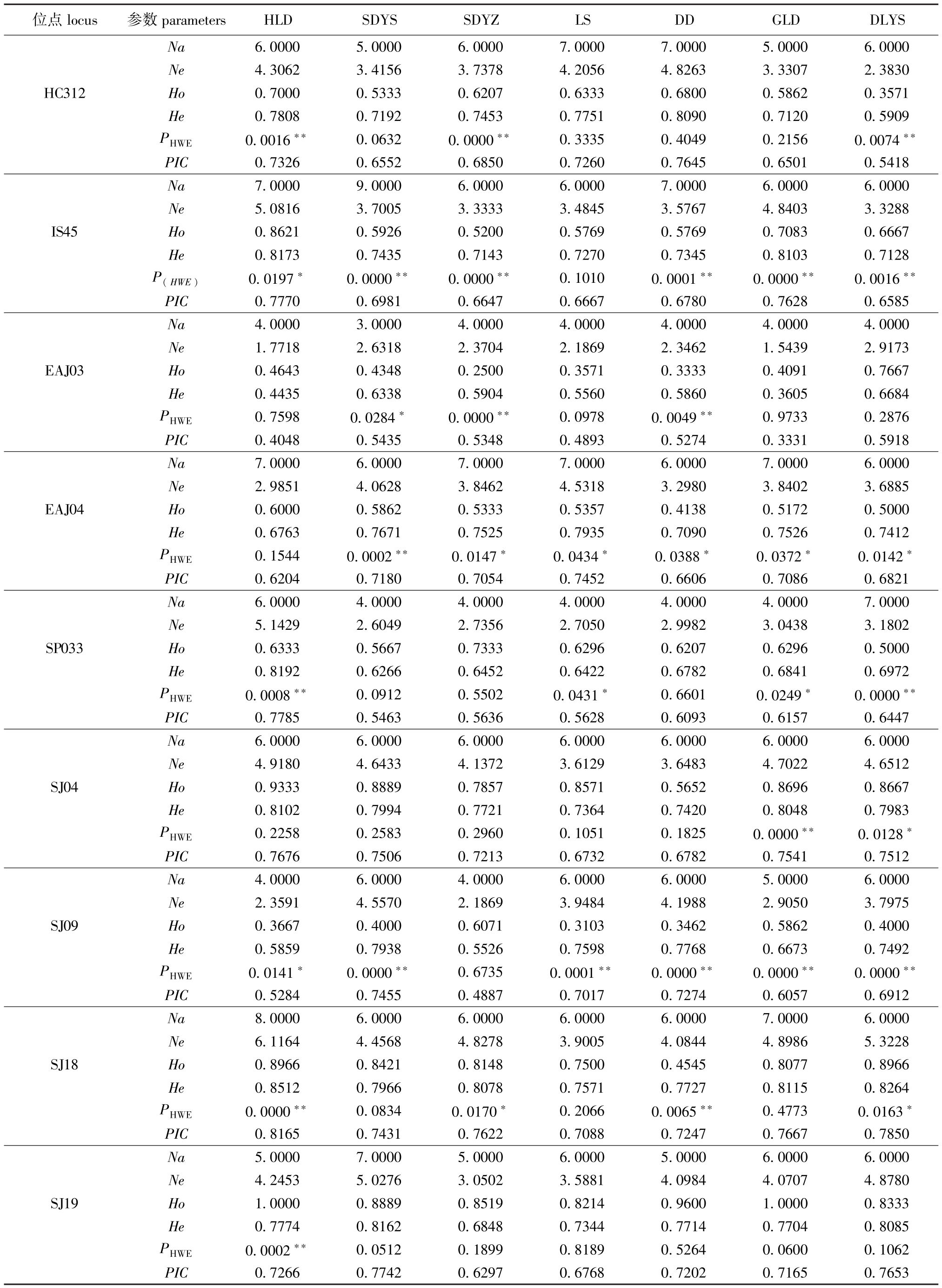
从表2可见:7个刺参群体的Na为5.083 3~5.416 7,平均值为 5.261 9;Ne为 3.205 0~3.701 1,平均值为 3.446 8;Ho为 0.547 5~0.686 9,平均值为 0.614 9;He为 0.649 1~0.697 1,平均值为 0.674 1;PIC为 0.598 9~0.638 6,平均值为0.620 0。对各群体的Hardy-Weinber平衡检测显示,7个刺参群体均存在不同程度的平衡偏离。
2.3 刺参群体的遗传分化与基因流
从表3可见:7个刺参群体间Fst为0.013 7~0.180 5,平均值为 0.078 2;Nm为 1.141 3~18.055 8,平均值为5.301 9。
2.4 刺参群体的变异分析
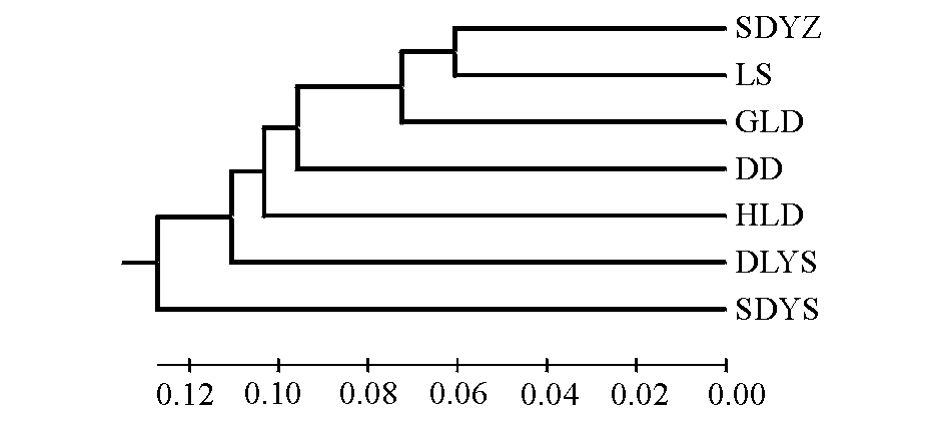
从表4可见:7个刺参群体中SDYZ与LS群体的遗传相似度最高,相似系数为0.886 0,遗传距离为0.121 1;HLD与SDYS群体的遗传相似度最低,相似系数为0.731 8,遗传距离为0.312 2。7个刺参群体UPMGA聚类图见图2,SDYS群体单独为一类,SDYZ群体先与LS群体聚为一类,再与GLD、DD、HLD和DLYS群体聚在一起。
2.5 刺参经济性状与微卫星位点的相关性分析
测定的180头刺参主要经济性状均表现出连续变异的特点,且符合正态分布。采用最小二乘法进行17个微卫星位点 (表1)与刺参体长、体宽、体积、体质量和总棘数5个性状的相关性分析,结果表明:HC312、EAJ07位点与刺参体长、体积、体质量显著相关 (P<0.05);IS45、EAJ03、EAJ04位点与刺参体长、体积、体宽、体质量显著相关(P<0.05);SJ04位点仅与刺参总棘数显著相关(P<0.05);SJ09位点与刺参体积、体宽、体质量显著相关 (P<0.05);SJ18位点仅与刺参体宽显著相关 (P<0.05);SJ19位点与刺参体积、体宽、体质量、总棘数显著相关 (P<0.05);SP072位点与刺参体积、体质量显著相关 (P<0.05)。具体计算结果限于篇幅未一一列出。此外,本研究中所获得的这10个与刺参经济性状相关的分子标记中,存在一因多效或多因一效现象。
表2 7个刺参群体在12个位点的遗传多样性分析
Tab.2 The diversity analysis of 12 loci in 7 sea cucumber populations
位点locus 参数parameters HLD SDYS SDYZ LS DD GLD DLYSNa6.0000 5.0000 6.0000 7.0000 7.0000 5.0000 6.0000Ne4.3062 3.4156 3.7378 4.2056 4.8263 3.3307 2.3830 HC312Ho0.7000 0.5333 0.6207 0.6333 0.6800 0.5862 0.3571He0.7808 0.7192 0.7453 0.7751 0.8090 0.7120 0.5909PHWE0.0016**0.0632 0.0000**0.3335 0.4049 0.2156 0.0074**PIC0.7326 0.6552 0.6850 0.7260 0.7645 0.6501 0.5418Na7.0000 9.0000 6.0000 6.0000 7.0000 6.0000 6.0000Ne5.0816 3.7005 3.3333 3.4845 3.5767 4.8403 3.3288 IS45Ho0.8621 0.5926 0.5200 0.5769 0.5769 0.7083 0.6667He0.8173 0.7435 0.7143 0.7270 0.7345 0.8103 0.7128P(HWE)0.0197*0.0000**0.0000**0.1010 0.0001**0.0000**0.0016**PIC0.7770 0.6981 0.6647 0.6667 0.6780 0.7628 0.6585Na4.0000 3.0000 4.0000 4.0000 4.0000 4.0000 4.0000Ne1.7718 2.6318 2.3704 2.1869 2.3462 1.5439 2.9173 EAJ03Ho0.4643 0.4348 0.2500 0.3571 0.3333 0.4091 0.7667He0.4435 0.6338 0.5904 0.5560 0.5860 0.3605 0.6684PHWE0.7598 0.0284*0.0000**0.0978 0.0049**0.9733 0.2876PIC0.4048 0.5435 0.5348 0.4893 0.5274 0.3331 0.5918Na7.0000 6.0000 7.0000 7.0000 6.0000 7.0000 6.0000Ne2.9851 4.0628 3.8462 4.5318 3.2980 3.8402 3.6885 EAJ04Ho0.6000 0.5862 0.5333 0.5357 0.4138 0.5172 0.5000He0.6763 0.7671 0.7525 0.7935 0.7090 0.7526 0.7412PHWE0.1544 0.0002**0.0147*0.0434*0.0388*0.0372*0.0142*PIC0.6204 0.7180 0.7054 0.7452 0.6606 0.7086 0.6821Na6.0000 4.0000 4.0000 4.0000 4.0000 4.0000 7.0000Ne5.1429 2.6049 2.7356 2.7050 2.9982 3.0438 3.1802 SP033Ho0.6333 0.5667 0.7333 0.6296 0.6207 0.6296 0.5000He0.8192 0.6266 0.6452 0.6422 0.6782 0.6841 0.6972PHWE0.0008**0.0912 0.5502 0.0431*0.6601 0.0249*0.0000**PIC0.7785 0.5463 0.5636 0.5628 0.6093 0.6157 0.6447Na6.0000 6.0000 6.0000 6.0000 6.0000 6.0000 6.0000Ne4.9180 4.6433 4.1372 3.6129 3.6483 4.7022 4.6512 SJ04Ho0.9333 0.8889 0.7857 0.8571 0.5652 0.8696 0.8667He0.8102 0.7994 0.7721 0.7364 0.7420 0.8048 0.7983PHWE0.2258 0.2583 0.2960 0.1051 0.1825 0.0000**0.0128*PIC0.7676 0.7506 0.7213 0.6732 0.6782 0.7541 0.7512Na4.0000 6.0000 4.0000 6.0000 6.0000 5.0000 6.0000Ne2.3591 4.5570 2.1869 3.9484 4.1988 2.9050 3.7975 SJ09Ho0.3667 0.4000 0.6071 0.3103 0.3462 0.5862 0.4000He0.5859 0.7938 0.5526 0.7598 0.7768 0.6673 0.7492PHWE0.0141*0.0000**0.6735 0.0001**0.0000**0.0000**0.0000**PIC0.5284 0.7455 0.4887 0.7017 0.7274 0.6057 0.6912Na8.0000 6.0000 6.0000 6.0000 6.0000 7.0000 6.0000Ne6.1164 4.4568 4.8278 3.9005 4.0844 4.8986 5.3228 SJ18Ho0.8966 0.8421 0.8148 0.7500 0.4545 0.8077 0.8966He0.8512 0.7966 0.8078 0.7571 0.7727 0.8115 0.8264PHWE0.0000**0.0834 0.0170*0.2066 0.0065**0.4773 0.0163*PIC0.8165 0.7431 0.7622 0.7088 0.7247 0.7667 0.7850Na5.0000 7.0000 5.0000 6.0000 5.0000 6.0000 6.0000Ne4.2453 5.0276 3.0502 3.5881 4.0984 4.0707 4.8780 SJ19Ho1.0000 0.8889 0.8519 0.8214 0.9600 1.0000 0.8333He0.7774 0.8162 0.6848 0.7344 0.7714 0.7704 0.8085PHWE0.0002**0.0512 0.1899 0.8189 0.5264 0.0600 0.1062PIC0.7266 0.7742 0.6297 0.6768 0.7202 0.7165 0.7653
续表2 7个刺参群体在12个位点的遗传多样性分析
Cont.tab.2 The diversity analysis of 12 loci in 7 sea cucumber populations
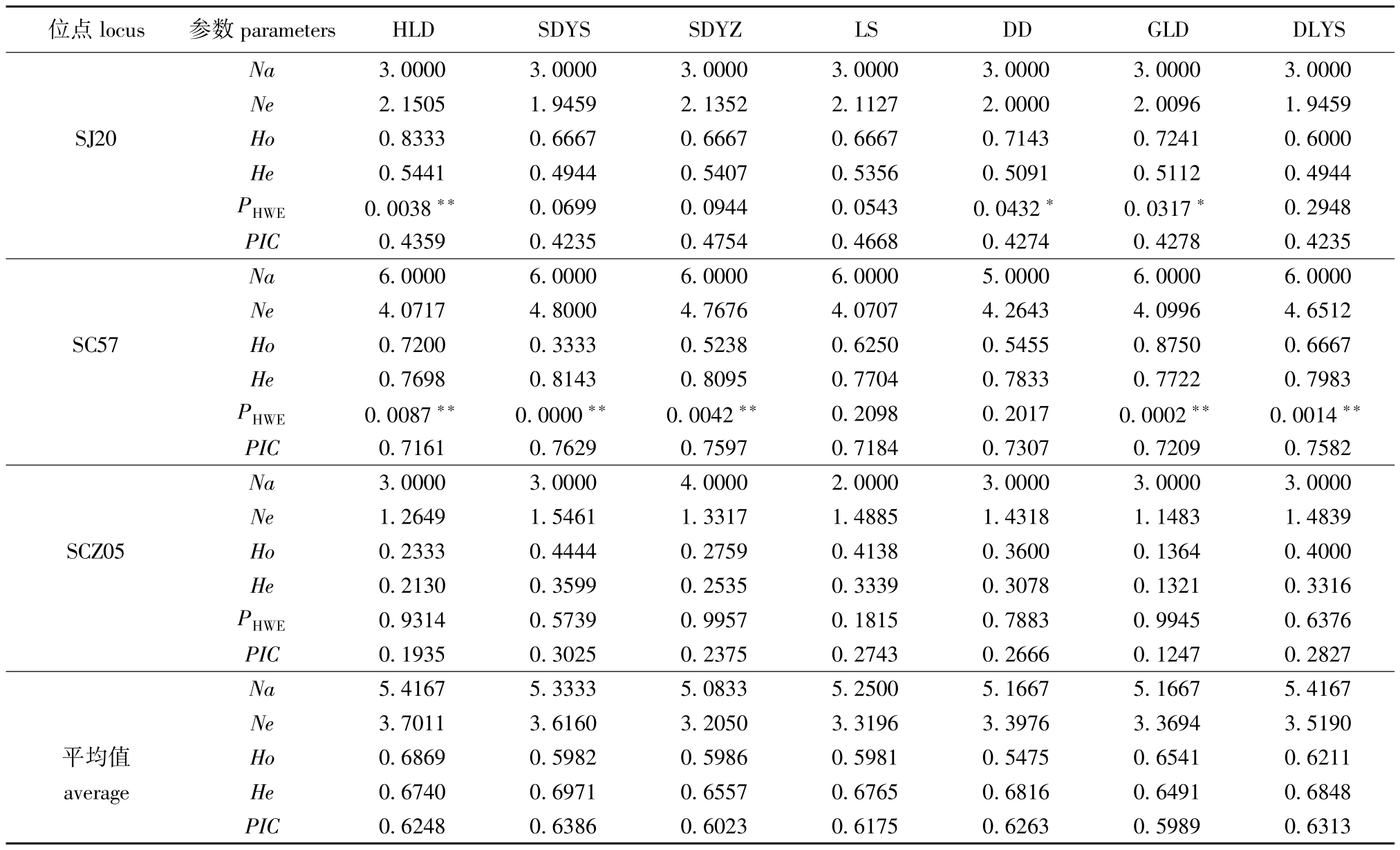
注:*表示显著偏离Hardy-Weinberg平衡(P<0.05);**表示极显著偏离Hardy-Weinberg平衡(P<0.01);PHWE为Hardy-Weinberg平衡检验P值。
Note:*indicates significant deviation from genetic equilibrium(P<0.05);**indicates very significant deviation from genetic equilibrium(P<0.01);PHWEisPvalues of Hardy-Weinberg balancing test.
位点locus 参数parameters HLD SDYS SDYZ LS DD GLD DLYSNa3.0000 3.0000 3.0000 3.0000 3.0000 3.0000 3.0000Ne2.1505 1.9459 2.1352 2.1127 2.0000 2.0096 1.9459 SJ20Ho0.8333 0.6667 0.6667 0.6667 0.7143 0.7241 0.6000He0.5441 0.4944 0.5407 0.5356 0.5091 0.5112 0.4944PHWE0.0038**0.0699 0.0944 0.0543 0.0432*0.0317*0.2948PIC0.4359 0.4235 0.4754 0.4668 0.4274 0.4278 0.4235Na6.0000 6.0000 6.0000 6.0000 5.0000 6.0000 6.0000Ne4.0717 4.8000 4.7676 4.0707 4.2643 4.0996 4.6512 SC57Ho0.7200 0.3333 0.5238 0.6250 0.5455 0.8750 0.6667He0.7698 0.8143 0.8095 0.7704 0.7833 0.7722 0.7983PHWE0.0087**0.0000**0.0042**0.2098 0.2017 0.0002**0.0014**PIC0.7161 0.7629 0.7597 0.7184 0.7307 0.7209 0.7582Na3.0000 3.0000 4.0000 2.0000 3.0000 3.0000 3.0000Ne1.2649 1.5461 1.3317 1.4885 1.4318 1.1483 1.4839 SCZ05Ho0.2333 0.4444 0.2759 0.4138 0.3600 0.1364 0.4000He0.2130 0.3599 0.2535 0.3339 0.3078 0.1321 0.3316PHWE0.9314 0.5739 0.9957 0.1815 0.7883 0.9945 0.6376PIC0.1935 0.3025 0.2375 0.2743 0.2666 0.1247 0.2827Na5.4167 5.3333 5.0833 5.2500 5.1667 5.1667 5.4167Ne3.7011 3.6160 3.2050 3.3196 3.3976 3.3694 3.5190平均值Ho0.6869 0.5982 0.5986 0.5981 0.5475 0.6541 0.6211 averageHe0.6740 0.6971 0.6557 0.6765 0.6816 0.6491 0.6848PIC0.6248 0.6386 0.6023 0.6175 0.6263 0.5989 0.6313
表3 7个刺参群体的遗传分化与基因流
Tab.3 Genetic differentiation and gene flow in 7 sea cucumber popu lations
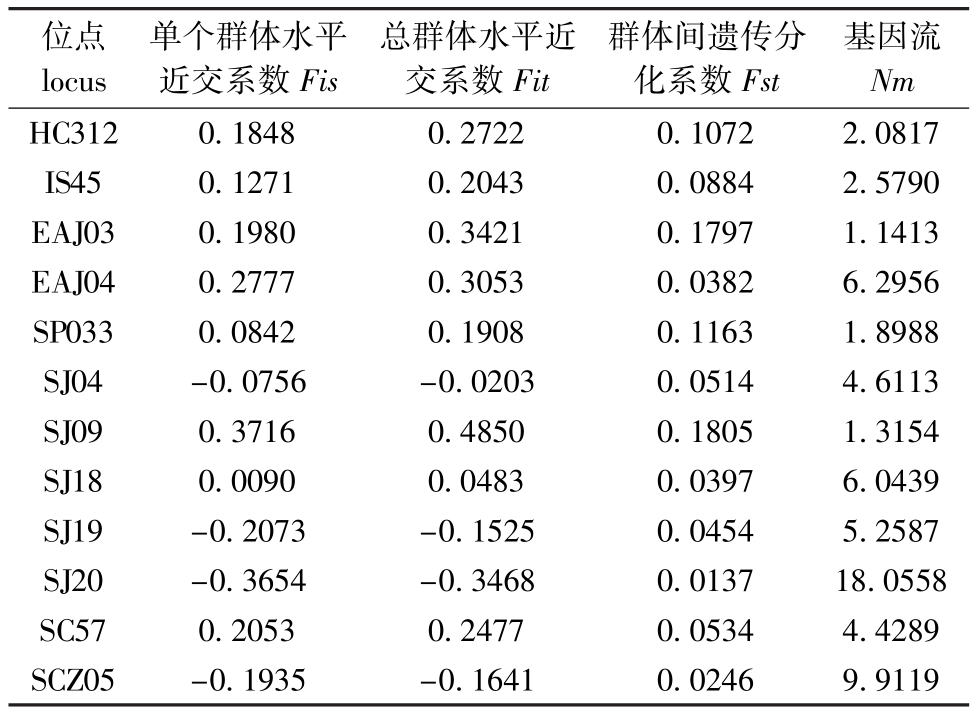
位点locus单个群体水平近交系数Fis总群体水平近交系数Fit群体间遗传分化系数Fst基因流NmHC312 0.1848 0.2722 0.1072 2.0817 IS45 0.1271 0.2043 0.0884 2.5790 EAJ03 0.1980 0.3421 0.1797 1.1413 EAJ04 0.2777 0.3053 0.0382 6.2956 SP033 0.0842 0.1908 0.1163 1.8988 SJ04 -0.0756 -0.0203 0.0514 4.6113 SJ09 0.3716 0.4850 0.1805 1.3154 SJ18 0.0090 0.0483 0.0397 6.0439 SJ19 -0.2073 -0.1525 0.0454 5.2587 SJ20 -0.3654 -0.3468 0.0137 18.0558 SC57 0.2053 0.2477 0.0534 4.4289 SCZ05 -0.1935 -0.1641 0.0246 9.9119
3 讨论
3.1 刺参群体的遗传多样性和遗传变异分析
本研究中发现:7个刺参群体的Ho和He平均值分别为0.614 9、0.674 1,与Kim等[11]报道的韩国5个刺参群体的Ho和He接近,Ho高于Chang等[12]报道的5个刺参群体的Ho;12个微卫星位点在7个刺参群体中的等位基因数为3~12个,野生刺参群体多态性信息含量高于底播群体和养殖群体的结论与谭杰等[14]用微卫星分析山东野生与养殖刺参群体所得的结论一致。
表4 7个刺参群体的遗传相似系数(对角线以上)和Nei氏遗传距离(对角线以下)
Tab.4 Genetic identity(above diagonal)and Nei's genetic distance(below diagonal)in 7 sea cucumber populations
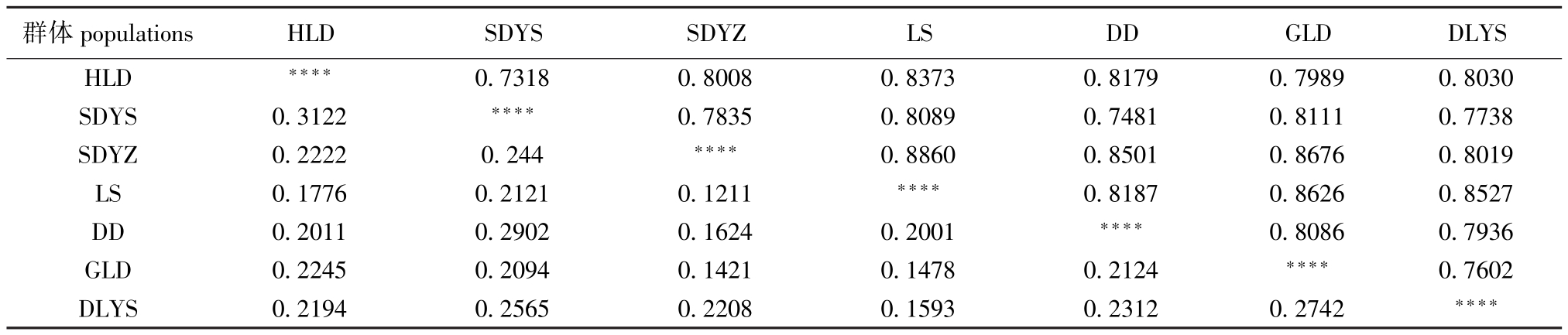
群体populations HLD SDYS SDYZ LS DD GLD DLYS HLD****0.7318 0.8008 0.8373 0.8179 0.7989 0.8030 SDYS 0.3122****0.7835 0.8089 0.7481 0.8111 0.7738 SDYZ 0.2222 0.244****0.8860 0.8501 0.8676 0.8019 LS 0.1776 0.2121 0.1211****0.8187 0.8626 0.8527 DD 0.2011 0.2902 0.1624 0.2001****0.8086 0.7936 GLD 0.2245 0.2094 0.1421 0.1478 0.2124****0.7602 DLYS 0.2194 0.2565 0.2208 0.1593 0.2312 0.2742****
UPGMA聚类图反映出山东野生群体单独为一类,与其他6个群体的遗传距离相对较远,其他6个群体聚类顺序与其地理分布有一定的相关性。7个刺参群体的遗传分化系数平均值为0.078 2,表明有 7.82%的变异是由群体分化导致的,而92.18%的变异来源于群体内,群体间的遗传分化水平较低,作为影响群体间遗传分化的主要因素[15],Wright[16]认为,基因流Nm大于1时,能够发挥其均质化作用,从而抑制由遗传漂变引起的群体间分化。本研究结果显示,12个位点在7个刺参群体中的Nm值均大于 1,平均值达到5.301 9,群体间存在一定程度的基因交流,通过对研究结果与实际情况分析,推测不同地域刺参的交流是影响其群体间遗传分化的主要因素。
3.2 刺参经济性状与微卫星位点的相关性分析
水产动物的体长、体宽等经济性状表现为连续变异,受多基因控制,易受环境影响,借助与数量性状连锁的分子标记可实现水产育种过程中的早期选择,利用与经济性状紧密相关的遗传标记还可提高选择的准确性、缩短育种周期。目前,已有微卫星分子标记与扇贝、大菱鲆、文蛤、牡蛎等海洋生物经济性状连锁分析的报道[17-21]。在刺参微卫星标记与经济性状相关性研究方面,Wang等[22]利用10个微卫星位点对大连2个刺参群体进行了微卫星标记与刺参体质量的相关性分析。孙国华等[23]进行了10个微卫星标记与刺参体长、体质量的相关性分析,获得了4个与刺参体长、体质量显著相关的微卫星标记。本研究中共获得10个与刺参主要经济性状相关的标记,经济性状与标记间存在一因多效或多因一效现象,这些与经济性状相关的微卫星标记还可进一步定位到刺参遗传连锁图谱上,用于刺参经济性状的QTL定位研究。
参考文献:
[1] 李明,马悦欣,刘志明,等.刺参机体酵母菌组成及其拮抗活性的研究[J].大连海洋大学学报,2012,27(5):436-440.
[2] 王忠华,董西征,钱国英.DNA分子标记技术在水生动物遗传多样性研究中的应用[J].科技通报,2008,24(5):624-630.
[3] Wang Xiuli,Song Bin,Qiu Xuemei,et al.Development of ESTSSRS in scallopPatinopecten yessoensisfrom sequence database [J].Conservation Genetics,2009,10(4):1129-1131.
[4] Wang Xiuli,Feng Teng,Yang Lei,et al.dbEST-derived SSR markers in sea urchinHemicentrotuspulcherrimus[J].Conservation Genetics,2009,10:729-731.
[5] LiQi,Wang Qingzhi,QiMingjun,et al.Development,characterization and inheritance of 113 novel EST-SSR markers in the Pacific oysterCrassostrea gigas[J].Genes﹠Genomics,2011,33:313-316.
[6] 李春艳,丁君,常亚青,等.虾夷扇贝微卫星标记的分离及其养殖群体的遗传结构分析[J].中国水产科学,2009,16(1):39-46.
[7] Chen Limei,LiQi.Identification and characterization ofmicrosatellitemarkers derived from expressed sequence tags of the sea cucumberStichopus japonicus[J].Molecular Ecology Notes,2007,7: 1057-1059.
[8] Zhan Aibin,Bao Zhenmin,Lu Wei,et al.Developmentand characterization of 45 novel microsatellite markers for sea cucumber (Apostichopus japonicus)[J].Molecular Ecology Notes,2007,7 (6):1345-1348.
[9] PengWei,Bao Zhenmin,Du Huixia,et al.Development and characterization of38 novel EST-SSRs for the sea cucumberApostichopus japonicus[J].Conservation Genetics Resources,2009(1): 447-450.
[10] Kang JH,Kim Y K,Kim M J,etal.Genetic differentiation among populations and color variants of sea cucumber(Stichopus japonicus)from Korea and China[J].International Journal of Biological Sciences,2011,7(3):323-332.
[11] Kim M J,Choi T J,An H S.Population genetic structure of sea cucunber,Stichopus japonicusin Korea usingmicrosatellitemarkers[J].Aquaculture Research,2008,39(10):1038-1045.
[12] Chang Yaqing,Feng Zhigang,Yu Jiaping,etal.Genetic variability analysis in five populations of the sea cucumberStichopus(Apostichopus)japonicusfrom China,Russia,South Korea and Japan as revealed bymicrosatellitemarkers[J].Marine Ecology,2009,30 (4):455-461.
[13] 冯志纲,于佳平,丁君,等.仿刺参基因组DNA提取方法改进[J].生物技术通报,2010(增刊):352-355.
[14] 谭杰,孙惠玲,刘萍,等.仿刺参自然群体和养殖群体间遗传变异的微卫星标记研究[J].海洋水产研究,2007,28(3):38-43.
[15] Slatkin M.Gene flow and the geographic structure of natural populationsn[J].Science,1987,236:787-792.
[16] W right S.Evolution in Mendelian population[J].Genetics,1931, 16:91-159.
[17] 陈蒙,常亚青,孙谦,等.虾夷扇贝群体的遗传结构及微卫星标记与体尺、体质量的相关性分析[J].大连水产学院学报, 2009,24(4):311-316.
[18] Zhan Aibin,Hu Jingjie,Hu Xiaoli,etal.Construction ofmicrosatellite-based linkagemapsand identification of size-related quantitative trait loci for Zhikong scallop(Chlamys farreri)[J].Animal Genetics,2009,40(6):821-831.
[19] Ruan Xiaohong,Wang Weijie,Kong Jie,et al.Genetic linkage mapping of turbotScophthalmusmaximusL.using microsatellite markers and its application in QTL analysis[J].Aquaculture, 2010,308:89-100.
[20] Lu Xia,Wang Hongxia,Liu Baozhong,et al.Three EST-SSR markers associated with QTL for the growth of the clamMeretrix meretrixrevealed by selective genotyping[J].Marine Biotechnology,2013,15(1):16-25
[21] Guo Xiang,LiQi,Wang Qingzhi,et al.Geneticmapping and QTL analysis of growth-related traits in the Pacific oyster[J].Marine Biotechnology,2012,14(2):218-226.
[22] Wang Xiuli,Shan Xue,Qiu Xuemei,et al.Microsatellite DNA polymorphisms and the relation with body weight in sea cucumberApostichopus japonicus[J].Chinese Journal of Oceanology and Limnology,2009,27(2):331-336.
[23] 孙国华,杨建敏,孙效德,等.刺参微卫星标记与生长性状体质量、体长的相关分析[J].水产学报,2011,35(4):501-508.
Population genetic structure and relationship between microsatellite markers and economic traits in sea cucumber
LI Yun-xia,LI Jiao,DING Jun,CHANG Ya-qing
(Key Laboratory of Mariculture & Stock Enhancement in North China's Sea,Ministry of Agriculture,Dalian Ocean University,Dalian 116023,China)
Abstract:Microsatellite marker technique was used to estimate extent of genetic diversity among 7 sea cucumber Apostichopus japonicus(Selenka) populations including farmed Huludao(HLD)in Laoning Province,wild Yantai (SDYS) in Shandong Province,farmed Liazhou (SDYZ) in Shandong Province,ranched Lüshun (LS) in Laoning Province,farmed Dandong (DD) in Laoning Province,ranched Guanglu (GLD) in Laoning Province,and wild Heishijiao(DLYS) in Laoning Province,and the relationship between economic characters of sea cucumbers and microsatellite markerswere analyze in 180 individuals sampled from 7 populations.The results showed that 83 allelic genes were amplified with 12 microsatellite markers and the number of alleles ranged from 3 to 12 per locus containing 102-368 bp.The locus diversity analysis showed that the alleles were found to be ranged from 5.083 3 to 5.416 7 in number in each sea cucumber population with an average of5.261 9,effective number of alleles ranged from 3.205 0 to 3.701 1 with an average of3.446 8,observed heterozygosity ranged from 0.547 5 to 0.686 9 with an average of 0.614 9,expected heterozygosity ranged from 0.649 1 to 0.697 1 with an average of 0.674 1,and polymorphic information content ranged from 0.598 9 to 0.638 6 with an average of0.620 0.Hardy-Weinber test indicated that there was a certain degree deviation from equilibrium in 7 sea cucumber populations.There was a certain degree of genetic exchange in sea cucumber populations,with genetic differentiation of0.078 2,largely derived from the variation within populations,and average gene flow of 5.301 9.Sea cucumber populations showed genetic similarity from 0.731 8 to 0.886 0.UPGMA showed that the SDYS population was clustered into one group alone.The relationship between economic characters of the sea cucumber and microsatellite markers revealed that HC312 and EAJ07 were significantly correlated with body length,body volume and body weight(P<0.05),while IS45,EAJ03 and EAJ04 were significantly related with body length,body volume,bodywidth and body weight(P<0.05);SJ04 had a significant relationship with total tubercle number(P<0.05)only,while SJ09 was significantly correlated with body volume,body width and body weight(P<0.05);SJ18 was significantly correlated only with body width(P<0.05),while SJ19 was significantly related with body volume,body width,body weight and total tubercle number(P<0.05);SP072 was significantly involved in body volume and body weight(P<0.05). The findings provide some guidance for sea cucumber cultivation and breeding.
Keywords:Apostichopus japonicus(Selenka);microsatellite;genetic diversity;economic trait
中图分类号:S917.4
文献标志码:A
文章编号:2095-1388(2013)05-0438-07
收稿日期:2013-01-13
基金项目:国家 “863”计划项目 (2012AA10A412,2010AA10A401);辽宁省农业攻关计划重大项目 (2011203003)
作者简介:李云霞 (1989-),女,硕士研究生。E-mail:ernan1989@163.com
通信作者:丁君 (1973-),女,研究员。E-mail:dingjun1119@dlou.edu.cn