
图1 3种蛤仔品系个体
Fig.1 Individuals in the three clam strains
摘要:首次建立并优化了菲律宾蛤仔Ruditapes philippinarum SRAP分子标记技术体系,并利用17对引物组合对经过连续选育的白蛤、橙蛤和墨蛤3个菲律宾蛤仔品系进行了分子标记分析。结果表明:3个品系蛤仔共扩增出510个位点,白蛤品系的Nei指数和Shannon指数均显著高于墨蛤和橙蛤品系 (P<0.05);遗传分化指数 (Gst)为0.000 1~0.644 8,平均为0.046 7,表明有4.67%的变异存在于群体间,95.33%的变异存在于群体内;基因流 (Nm)为0.275 4~2 000,平均每个位点的Nm为10.202 7;白蛤和墨蛤品系间的遗传距离最近 (0.028 2),白蛤和橙蛤品系间的遗传距离最远(0.039 6)。研究结果表明,白蛤、橙蛤和墨蛤3个蛤仔品系间有很强的基因交流,遗传分化程度较弱,提示蛤仔品种的人工选育工作需持续进行。
关键词:菲律宾蛤仔;SRAP;品系;遗传学
蛤仔是中国四大养殖贝类之一,目前年产量约300万t,占世界贝类总产量的90%,占中国海水养殖总产量的20%,占滩涂贝类总产量的73%[1]。国内外对菲律宾蛤仔的研究有较多报道,主要涉及养殖和人工繁育[1-2]、生理生态学[3-5]、凝集素的分离[6]等方面。随着养殖规模的不断扩大,蛤仔传统育种学的工作已经大规模展开[7-8],但涉及分子标记及辅助育种方面内容较少,目前仅见利用RAPD[9]、AFLP[10]和微卫星[11-14]标记进行群体遗传学的研究报道。育种工作相对滞后,已成为蛤仔养殖业发展的瓶颈问题。
SRAP由美国加州大学蔬菜作物系Li等[15]于2001年提出,又称基于序列扩增多态性 (Sequence-related amplified polymorphism,SRAP)。该标记利用其独特的引物设计原则对开放读码框(ORFs)进行扩增,其具有简便、稳定、产率高、成本低、使用广泛和便于克隆目标片段的优点,已广泛应用于多种作物的遗传多样性研究和图谱构建[16-17]。SRAP标记用于水产动物研究的报道仅见少数甲壳动物[18-19]、 部分鱼类[20-21]、 扇贝[22]、 皱纹盘鲍[23]等。本研究中,以连续多年选育所得的3个菲律宾蛤仔品系为研究对象,首次建立并优化了蛤仔SRAP分子标记体系,并在此基础上开展了3个蛤仔品系SRAP分子标记的研究,期待对不同蛤仔品系的遗传多样性及遗传分化状况加以评价,并通过筛选各品系间差异显著的SRAP标记位点,揭示各蛤仔品系优良性状的遗传机理,为蛤仔分子遗传学和分子标记辅助育种工作奠定基础。
1.1 材料
试验蛤仔取自庄河海洋贝类育苗场,经连续多代选育出的白、墨、橙3种不同壳色的蛤仔品系(选育代数分别为6、6、5代)(图1),各取30个个体,壳长分别为(2.03±0.54)、(1.89±0.46)、(2.15±0.67)cm,在取样地点将新鲜样品运回实验室,取其闭壳肌、外套膜等组织分别用体积分数为75%的乙醇固定,于冰箱 (4℃)中保存。
1.2 方法
1.2.1 基因组DNA的提取及检测 采用常规酚-氯仿法提取蛤仔基因组DNA,用10 g/L琼脂糖凝胶电泳 (含1.5 μL EB)检测模板质量。

图1 3种蛤仔品系个体
Fig.1 Individuals in the three clam strains
1.2.2 引物序列的确定 按照已经公布的SRAP通用引物序列,筛选Me和Em引物各6条,引物序列如表1,上下游交叉共得到36对引物组合。
表1 SRAP引物序列
Tab.1 Sequences of the SRAP primers

正向引物forward primer序列(5'-3') sequences反向引物reverse primer序列(5'-3') sequences M1 TgAgTCCAAACCggAAA E1 gACTgCgTACgAATTAAT M2 TgAgTCCAAACCggACT E2 gACTgCgTACgAATTATg M3 TgAgTCCAAACCgggTA E3 gACTgCgTACgAATTTAg M4 TgAgTCCAAACCggACA E4 gACTgCgTACgAATTTCA M5 TgAgTCCAAACCggCAg E5 gACTgCgTACgAATTCAT M6 TgAgTCCAAACCggTAg E6 gACTgCgTACgAATTCTC
1.2.3 SRAP-PCR扩增体系的优化及产物的检测
PCR扩增采用25 μL反应体系,分别对模板(1.0、2.0 μL),10 μmol/L引物 (1.0、1.5 μL), 2.5 mmol/L dNTP(2.0、3.0 μL),25 mmol/L Mg2+(1.0、1.5、2.0 μL),5 U/μL Taq酶 (0.2、0.35、0.5 μL)设置不同用量并进行体系优化。
PCR扩增程序:94℃下预变性5 min;94℃下变性1 min,35℃下退火1 min,72℃下延伸1 min,共进行5个循环;之后在94℃下变性1 min, 50℃下退火1 min,72℃下延伸1 min,共进行30个循环;最后在72℃下延伸10 min,并于4℃下保存。产物经8%非变性聚丙烯酰胺凝胶电泳检测,确定带型清晰、稳定、杂带少的25 μL PCR反应体系为:DNA模板2.0 μL,10 μmol/L引物1.5 μL,2.5 mmol/L dNTP 2.0 μL,25 mmol/L Mg2+1.0 μL,5 U/μL Taq酶0.2 μL,ddH2O 13.5 μL。
利用上述PCR反应体系和反应程序,对36对引物组合进行筛选,选择带型清晰、稳定、杂带少的17对引物组合 E1/M1、E1/M3、E1/M4、E1/ M5、E1/M6、E2/M1、E3/M3、E3/M4、E3/M5、E3/M6、E4/M1、E4/M2、E4/M4、E5/M4、E5/ M6、E6/M3、E6/M5,用于3个不同壳色蛤仔品系的遗传学分析。
1.2.4 SRAP谱带统计分析及遗传学指标的计算
根据分子量标准,对照反应产物在凝胶上的相应位置,估计扩增产物的分子量大小,以 “引物名 + f+分子量大小”方式对各个位点命名。
按照显性标记进行带型统计,有带视为显性基因型 (包括显性纯合和显性杂合),无带视为隐性纯合基因型。有带且清晰的带型记为 “1”,无带的带型记为 “0”,不清晰的带型记为 “.”,统计形成0/1矩阵。多态位点比例、Nei指数和Shannon指数等遗传多样性指标以及遗传相似度、遗传距离等遗传分化指标均由PopGene 32软件计算得出。应用Mega 3.1软件对所有个体进行遗传关系聚类图的构建。使用SPSS 11.5软件对试验数据进行单因素方差分析和检验。
2.1 SRAP扩增带型
17对引物组合在3个蛤仔品系中进行PCR扩增,扩增片段长度为50~1 310 bp。其中E1/M6和E1/M5引物在部分蛤仔个体中的扩增结果见图2。所有引物共产生了510个清晰的标记 (多态位点比例为100%),每对引物产生的标记为20~38个,平均每对引物产生30个标记。其中13对引物产生的位点在 3个品系中的多态性比例均为100%。
17对引物在白蛤、墨蛤和橙蛤品系中分别扩增出508、505和504个位点,多态性比例分别为100%、99.41%和99.80%。3个品系的多态性位点比例无显著性差异(P>0.05)。
2.2 3个蛤仔品系的遗传多样性及遗传分化

图2 引物E1/M6和E1/M5在3个品系部分蛤仔中的扩增结果
Fig.2 SRAP fingerprinting of some individuals in three strains of clams by primers E1/M6 and E1/M5
17对引物的扩增片段统计成0/1矩阵,经计算得到平均Shannon指数、Nei指数见表2。方差分析结果表明,这两个指标在3个品系之间存在显著差异 (P<0.05)。多重比较结果表明,白蛤品系的两个遗传多样性指标均显著高于墨蛤和橙蛤品系(P<0.05),而墨蛤和橙蛤品系之间无显著性差异(P>0.05)。化指数 (Gst)为0.000 1(E3/M4f399) ~0.644 8 (E1/M5f84),平均为0.046 7,说明有4.67%的变异存在于群体间,95.33%的变异存在于群体内,不同位点的Nm为0.275 4(E1/M5f84) ~2 000 (E3/M4f399),平均每个位点的Nm为10.202 7。
白蛤和墨蛤品系间的遗传距离最近(0.028 2),遗传相似度为0.972 2;白蛤和橙蛤品系间的遗传距离最远 (0.039 6),遗传相似度为0.961 1。白蛤、墨蛤和橙蛤群体内的平均遗传距离分别为0.591 2、0.527 5和0.513 1,群体内的遗传相似度分别为0.556 3、0.591 3和0.599 8。3个品系共90个个体群体内的遗传距离为0.553 0,群体内的遗传相似度为0.576 6。从3个品系90个个体的遗传聚类图 (图3)可以看出,绝大多数同种颜色的蛤仔聚在一起。
表2 3种壳色蛤仔品系遗传多样性分析
Tab.2 The genetic diversity of three clam strains with 3 shell colors

注:同列中标有不同小写字母者表示组间有显著性差异(P<0.05),标有相同小写字母者表示组间无显著性差异(P>0.05)。
Note:The means with the different letters within the same column are significant differences at the 0.05 probability level,and the means with the same letters within the same column are not significant differences.
品系strain Shannon指数Shannon's index Nei指数Nei's index多态性位点polymorphic loci数量number比率/% percent白蛤W 0.5041±0.1543a0.3323±0.2660a508 100.00墨蛤B 0.4781±0.1691b0.3120±0.1357b505 99.41橙蛤O 0.4762±0.1776b0.3117±0.1414b504 99.80 P 0.012 0.020 0.998
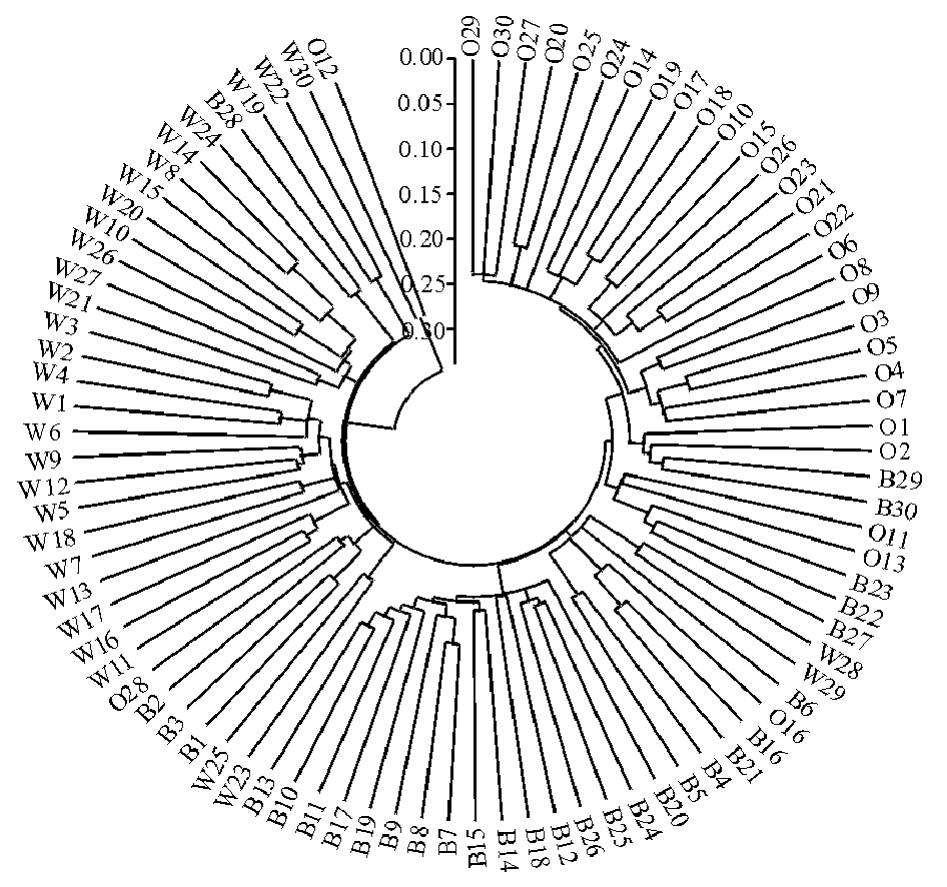
图3 3个品系90个蛤仔的遗传聚类图 (NJ法)
Fig.3 Clustal analysis among individuals of clams in three strains(NJ method)
注:O1~O30为橙蛤;W1~W30为白蛤;B1~B30为墨蛤。
Note:O1-O30,orange;W1-W30,white;B1-B30,black.
3个品系总的Nei指数为0.334 4±0.120 1,总的Shannon指数为0.508 1±0.143 5。群体总的遗传多样度 (Ht)为0.334 3±0.014 4,群体内的遗传多样度 (Hs)为0.318 7±0.012 9,群体间的遗传多样度 (Ht-Hs)为0.015 6。不同位点的遗传分
2.3 3个蛤仔品系间出现频率差异的SRAP位点分析
用SPSS 11.5软件的R×C检验法进行检验,在17对SRAP标记获得的510个位点中,3个品系间呈现显著频率差异的位点为147个 (P<0.05),占所有位点的28.82%;呈现极显著频率差异的位点为82个 (P<0.01),占16.08%。引物E1/M5和E6/M5多态位点检出率较高 (>40%)(表3)。
表3 不同蛤仔品系间出现频率差异的SRAP位点数及比例
Tab.3 The number and percent of SRAP loci showing significant differences in frequency among the three clam strains

注:1)出现频率差异显著的位点数 (P<0.05);2)出现频率差异极显著的位点数 (P<0.01)。
Note:1)The number of loci showing significant differences in frequency (P<0.05);2)The number of loci showing very significant differences in frequency(P<0.01).
引物组合primer combination位点总数total number of loci位点数A1)(占比例/%) locus number (percent)位点数B2)(占比例/%) locus number (percent) E1/M1 37 12(32.43) 5(13.51) E1/M3 35 11(31.43) 10(28.57) E1/M4 36 11(30.56) 5(13.89) E1/M5 24 16(66.67) 12(50.00) E1/M6 25 5(20.00) 4(16.00) E2/M1 30 13(43.33) 6(20.00) E3/M3 26 7(26.92) 3(11.54) E3/M4 35 10(28.57) 4(11.43) E3/M5 20 6(30.00) 4(20.00) E3/M6 24 3(12.50) 3(12.50) E4/M1 26 8(30.77) 4(15.38) E4/M2 34 9(26.47) 3(8.82) E4/M4 38 4(10.53) 1(2.63) E5/M4 36 4(11.11) 2(5.56) E5/M6 30 11(36.67) 5(16.67) E6/M3 28 3(10.71) 0(0.00) E6/M5 26 14(53.85) 11(42.30)合计total 510 147(28.82) 82(16.08)
其中白蛤中出现频率显著高于墨蛤和橙蛤(后二者差异不显著)的位点为30个,显著低于墨蛤和橙蛤 (后二者差异不显著)的位点9个;橙蛤中出现频率显著高于白蛤和墨蛤 (后二者差异不显著)的位点24个,显著低于白蛤和墨蛤(后二者差异不显著)的位点16个;墨蛤中出现频率显著高于和低于白蛤和橙蛤 (后二者差异不显著)的位点均为18个;在3个品系两两间均存在显著差异的位点8个,而3个品系中仅在两个品系间存在显著差异的位点24个。
3.1 蛤仔SRAP标记与群体遗传学
利用分子标记对菲律宾蛤仔进行分子遗传学的研究报道主要集中在RAPD、AFLP和SSR等标记手段方面,如裴赢等[9]用RAPD方法,对大连渤海沿海6个菲律宾蛤仔群体进行研究,结果表明,多态位点比例为58.65% ~71.15%,Nei指数为0.159 9~0.197 3。刘相全等[10]对青岛菲律宾蛤仔群体 (18个个体)进行了AFLP标记研究,发现4对AFLP引物在蛤仔群体中共得到216个位点,多态性比例为87.9%,群体内遗传相似度为0.605 1, Shannon指数为0.252 6。闫喜武等[11]利用13个微卫星座位在大连、莆田和青岛的蛤仔群体中共检测到154个等位基因,平均观测杂合度为0.238 3~0.438 7,3个蛤仔群体内的遗传距离分别为0.517 8、0.659 2和0.548 0,遗传相似度分别为0.693 0、0.542 6和0.598 6,3个群体间的遗传距离为0.110 8~0.432 3,遗传相似度为0.649 0~0.895 2。虞志飞等[12]利用12对微卫星引物对大连菲律宾蛤仔群体连续4个选育世代进行了遗传多样性分析,4个世代的平均观测杂合度分别为0.378 6、0.339 1、0.386 0和0.357 4。
本研究中17对SRAP引物在3个连续选育形成的蛤仔品系中共产生了510个清晰的标记,多态性比例为98.43% ~99.41%,标记效率较RAPD、SSR标记要高,且多态性位点检出率也显著高于AFLP、RAPD和SSR标记。这是因为虽然外显序列在不同的物种间是保守的,多态性水平较低,但启动子、内含子及其间隔序列在物种间甚至个体间都有很大的差异,有很高的多态性[24]。因此认为,采用SRAP标记法研究种内与种间的多样性时非常有效,尤其适用于品种间的分析[25]。另外,3个蛤仔品系群体Shannon指数为0.476 2~0.504 1, Nei指数为0.311 7~0.332 3,这两个遗传学指标较裴赢等[9]、刘相全等[10]所报道的结果要高,这可能是因研究对象群体地理位置差异、检测手段的差异造成的。而本研究结果中的遗传多样性指标、遗传距离及遗传相似度指标均与闫喜武等[11]、虞志飞等[12]利用共显性标记 (微卫星)研究所得的结果相近。众所周知,微卫星标记作为共显性标记,对遗传多样性及遗传分化指标的计算较稳定准确,而本研究中采用SRAP标记所得结果与共显性标记所得结果相近,说明SRAP标记可以作为蛤仔群体遗传学研究的分子标记手段。
另外,对基因组开放阅读框扩增,提高了扩增结果与表现型的相关性,能更多地反映材料的表型差异[16]。目前,利用SRAP标记进行基因定位并筛选标记、基因的研究较多[17,26],但在水产动物中的应用较少,仅见吕耀平等[21]对瓯江彩鲤体色相关的SRAP标记进行了筛选,并进行了SCAR标记分析。基于笔者对蛤仔的前期研究结果,发现蛤仔具有差异明显的贝壳颜色,并在生长、抗逆性方面均存在差异,因此,根据不同的育种目标,定向培育出了白蛤、橙蛤和墨蛤3个蛤仔品系。通过SRAP标记研究发现,3个品系蛤仔群体间呈现显著频率差异的位点147个,呈现极显著频率差异的位点82个。这些标记的获得,加之SRAP标记扩增位点的特殊性,将为揭示蛤仔壳色、生长和抗逆性的遗传机理奠定基础。
3.2 3个蛤仔品系的遗传分化
群体间遗传相似度与遗传距离能反映群体间的亲缘关系。Thrope[27]认为,同科属间遗传距离(D)为0.5~0.9,同属种间D为0.2~0.8,同种种群间D为0.03~0.2。本研究中发现,3个蛤仔品系间的遗传距离为0.028 2~0.039 6,遗传相似度为0.972 2~0.961 1,由此可见,3个蛤仔品系属于同种不同种群的位置。
Wright等[28]对遗传分化指数作了界定:Gst为(0~0.05]时,群体遗传分化较弱;Gst为(0.05~0.15]时,群体遗传分化中等;Gst为(0.15~0.25]时,群体遗传分化较大;Gst>0.25时,表示分化极大。本研究中计算得出不同位点的Gst为0.000 1~0.644 8,平均为0.046 7,说明有4.67%的变异存在于群体间,95.33%的变异存在于群体内。另外,本研究中不同位点的 Nm为0.275 4~2 000,平均每个位点的Nm为10.202 7。基因流主要是指基因在群体之内和群体之间的流动,当Nm>1时,说明种群间有很强的基因交流,足以抵制由于选择等行为导致的遗传漂变的作用[29]。所有结果均表明,3个蛤仔品系没有明显的遗传分化,这与其遗传相似性指数的结果较高(0.961 1~0.972 2)相符合。有报道表明,海洋贝类往往杂合度较高[30],这也预示着对贝类的育种及品种培育工作历程势必较长,因此,对菲律宾蛤仔各品系的人工选育还需要持续下去。
参考文献:
[1] 闫喜武.菲律宾蛤仔养殖生物学、养殖技术与品种选育[D].青岛:中国科学院研究生院(海洋研究所),2005.
[2] 梁俊,闫喜武,李霞,等.菲律宾蛤仔性腺发育生物学零度的研究[J].海洋科学,2007,31(9):67-72.
[3] 蒋红,崔毅,陈碧娟,等.乳山湾菲律宾蛤仔可溶性氮、磷排泄及其与温度的关系[J].中国水产科学,2006,13(2):237-242.
[4] 董波,薛钦昭,李军.环境因子对菲律宾蛤仔摄食生理生态的影响[J].海洋与湖沼,2000,31(6):636-641.
[5] 丁鉴锋,杨霏,闫喜武,等.不同壳色菲律宾蛤仔免疫机能的比较研究[J].大连海洋大学学报,2012,27(5):411-416.
[6] 李伟,陈文,佟长青,等.菲律宾蛤仔凝集素的分离纯化及抑菌活性研究[J].大连海洋大学学报,2012,27(4):333-337.
[7] 闫喜武,张跃环,霍忠明,等.不同壳色菲律宾蛤仔品系间的双列杂交[J].水产学报,2008,32(6):864-875.
[8] 闫喜武,张跃环,孙焕强,等.菲律宾蛤仔两道红与白斑马品系的三元杂交[J].水产学报,2010,34(8):1190-1197.
[9] 裴赢,王晓红,张恒庆.大连渤海沿海菲律宾蛤仔种群的RAPD分析[J].水产科学,2006,25(5):250-252.
[10] 刘相全,包振民,胡景杰,等.两种蛤仔群体遗传多样性的形态参数及AFLP分析[J].海洋与湖沼,2010,41(3):359-364.
[11] 闫喜武,虞志飞,秦艳杰,等.菲律宾蛤仔EST-SSRs标记开发及不同地理群体遗传多样性[J].生态学报,2011,31(15): 4190-4198.
[12] 虞志飞,闫喜武,杨霏,等.菲律宾蛤仔大连群体不同世代的遗传多样性[J].生态学报,2011,31(15):4199-4206.
[13] Mura L,Cossu P,Lai T,et al.Survey of the genetic variability of populations of Ruditapes philippinarum from the Gulf of Olbia(NE Sardinia)by mocrosatellites[J].Biol Mar Mediterr,2011,18 (1):266-267.
[14] An H S,Park K J,Cho K C,et al.Genetic structure of Korean population of clam Ruditapes philippinarum inferred from microsatellite marker analysis[J].Biochemical Systematics and Ecology, 2012,44:186-195.
[15] Li G,Quiros C F.Sequenced related amplified polymorphism (SRAP),a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica[J].Theoreticale and Applied Genetics,2001,103(3):455-461.
[16] 赵雪,谢华,马荣才.植物功能基因组研究中出现的新型分子标记[J].中国生物工程杂志,2007,27(8):104-110.
[17] 邓思立,潘俊松,何欢乐,等.黄瓜M基因连锁图谱的SRAP分子标记[J].上海交通大学学报:农业科学版,2006,24(3):240-244.
[18] 周劲松,曹哲明,杨国梁,等.罗氏沼虾缅甸引进种和浙江本地种及其杂交种的生长性能与SRAP分析[J].中国水产科学,2006,13(4):667-673.
[19] 陈华增,李健,王清印,等.“黄海1号”中国明对虾抗逆性状SRAP标记[J].中国水产科学,2011,18(6):1243-1249.
[20] 张志伟,韩曜平,仲霞铭,等.草鱼野生群体和人工繁殖群体遗传结构的比较研究[J].中国水产科学,2007,14(5):720-725.
[21] 吕耀平,胡则辉,王成辉,等.与“全红”瓯江彩鲤体色相关的SRAP及SCAR分子标记[J].水生生物学报,2011,35(1):45-50.
[22] 程宁宁,杨爱国,刘志鸿,等.栉孔扇贝(♀)×虾夷扇贝(♂)子一代杂种优势的SRAP分析[J].海洋科学,2009,33(10): 107-111.
[23] 刘明泰,李杨,刘小林,等.皱纹盘鲍SRAP反应体系的正交优化[J].大连海洋大学学报,2013,28(1):12-16.
[24] Ding W D,Cao Z M,Cao L P.Molecular analysis of grass carp Ctenopharyngodon idella by SRAP and SCAR molecular markers [J].Aquaculture International,2010,18:575-587.
[25] 谭祖猛,李云昌,胡琼,等.通过分子标记估算遗传距离预测甘蓝型油菜的杂种优势[J].中国油料作物学报,2007,29(2): 126-132.
[26] Liu Y Q,Wu W R.Identification of salt-responsive genes in English cordgrass Spartina anglica roots using SRAP technique[J]. Journal of Zhejiang University(Agriculture and Life Sciences), 2006,32(5):511-514.
[27] Thrope J P.The molecular clock hypothesis:biochemical evolution,genetic differentiation and systematics[J].Ann Rev Evol Syst,1982,13(2):139-168.
[28] Wright J M,Bentzen P.Microsatellites:genetic markers for the future[J].Reviews in Fish Biology and Fisheries,1994,4(3): 384-388.
[29] 张婧,常亚青,丁君,等.虾夷扇贝不同壳色间的遗传结构及微卫星标记与生长性状相关分析[J].中国农学通报,2011,27 (26):83-91.
[30] Saavedra C,Bachère E.Bivalve genenomic[J].Aquaculture, 2006,256:1-14.
Establishment of SRAP reaction conditions and the genetic analysis of three strains with different shell colors in Manila clam Ruditapes philippinarum
Abstract:A SRAP-PCR system was established and the genetic diversity were analyzed in Manila clam Ruditapes philippinarum with white(W),black(B)and orange(O)shell colors through selective breeding by 17 pairs of SRAP primers screened from 36 primer combinations.A total of 510 loci were detected with 17 primer pairs,and Shannon's information index and Nei's gene diversity were found significant higher in W strain than those in B and O strain(P<0.05),without significant differences between the last two strains.F-statistics(Gst)were varied from 0.000 1 to 0.644 8,with the mean value of 0.046 7,indicating that 4.67%of variations were observed between B and O strains,and 95.33%of variations within the populations.The gene flow were found between 0.275 4 and 2 000,with mean 10.202 7,and genetic distances between 0.028 2(W and B)and 0.039 6(W and O).The findings showed that there were weak genetic differentiations and strong gene flow among the three clam strains,and that the selective breeding for the clam was continued in order to establish new breeding.The 147 SRAP loci showing significant differences in frequency among the three strains(P<0.05),and the 82 SRAP loci showing very significant differences in frequency among the three strains(P<0.01),providing the foundation for explanation of the genetic basis of the good traits such as good growth,strong resistance and special shell colors in the three strains.
Key words:Ruditapes philippinarum;SRAP;strain;genetics
中图分类号:Q959.215
文献标志码::A
文章编号:2095-1388(2013)04-0361-06
收稿日期:2012-10-29
基金项目:国家 “863”计划项目 (2012AA10A410-2);现代农业产业技术体系建设专项 (CARS-48)