皱纹盘鲍SRAP反应体系的正交优化
刘明泰1,李杨2,刘小林2,许淑芬1,常亚青3
(1.大连海宝渔业有限公司,辽宁大连116045;2.西北农林科技大学动物科技学院,陕西杨凌712100;3.大连海洋大学农业部北方海水增养殖重点实验室,辽宁大连116023)
摘要:采用L16(45)正交试验设计方法,对影响皱纹盘鲍Haliotis discus hannai SRAP反应体系的Mg2+浓度、dNTPs浓度、引物浓度、模板DNA以及Taq酶活性进行了优化,建立了适用于皱纹盘鲍的SRAP反应体系。优化后的反应体系(10 μL)包括Mg2+1.5 mmol/L,dNTPs 0.15 mmol/L,引物0.2 μmol/L,Taq酶活性0.25 U,模板DNA 20 ng。采用不同模板和引物对体系进行验证,表明优化后的反应体系能够高效地扩增出可识别条带,为进一步应用SRAP标记对皱纹盘鲍进行遗传分析奠定了基础。
关键词:皱纹盘鲍;SRAP;正交试验设计;分子标记;反应体系
皱纹盘鲍Haliotis discus hannai是中国北方地区重要的经济贝类[1],随着工厂化养殖的发展,迫切需要对其开展分子育种的相关研究工作。目前常用的分子标记主要有随机扩增片段多态性(RAPD)、微卫星、单核苷酸多态性(SNP)、扩增性片段多态性(AFLP)等。其中微卫星标记应用最为广泛,但其特异性引物开发成本较高,SNP分析需要进行大量测序,RAPD技术重复性较差,而AFLP技术则操作繁琐[2]。上述各种标记各有优劣,而一种理想的标记要求不仅成本低廉、易于操作,而且重复性好,相关序列扩增多态性(Sequence-Related Amplified Polymorphism,SRAP)便是这样一种标记。SRAP是基于聚合酶链式反应(Polymerase Chain Reaction,PCR)技术的一种分子标记,它通过独特的引物设计优先扩增基因组内的开放阅读框(ORFs),因为开放阅读框是可以编码完整的多肽链结构基因,SRAP标记的正、反引物分别与外显子和内含子(或启动子)区域配对,在不同个体间因外显子和内含子间隔长度不同而产生多态[3]。SRAP技术已经大量应用于植物的遗传多样性分析和遗传图谱的构建[4-8],在水产动物中,此技术主要应用于日本沼虾[9]、草鱼[10]、三疣梭子蟹[11]、黄颡鱼[12]和团头鲂[13]的遗传学研究中,在贝类中目前尚未见相关报道。有研究认为, PCR反应条件是影响SRAP标记扩增效率的重要因素[2]。本研究中,作者利用正交试验设计方法探讨了SRAP标记在皱纹盘鲍群体中的最佳反应条件,以期为SRAP标记应用于皱纹盘鲍的种质资源评估和遗传育种奠定基础。
1 材料与方法
1.1 材料
试验用皱纹盘鲍194头(体质量为69.3±8.3 g),于2011年4月取自大连太平洋海珍品有限公司第一育苗场,外套膜组织样本保存于体积分数为95%的乙醇中以备提取基因组DNA。
试验用主要试剂Taq DNA聚合酶(简称为Taq酶)、dNTPs和100 bp DNA Ladder等购自Tiangen生化科技(北京)有限公司,核酸染料Gelred购自上海开放生物科技有限公司。SRAP引物由南京金斯瑞公司合成,SRAP引物序列设计如表1所示。
1.2 方法
1.2.1 基因组 DNA的提取 皱纹盘鲍基因组DNA的提取参照Strauss[14]的方法进行,采用10g/L琼脂糖凝胶电泳检测DNA质量。
表1 SRAP引物序列
Tab.1 The sequences of SRAP primers
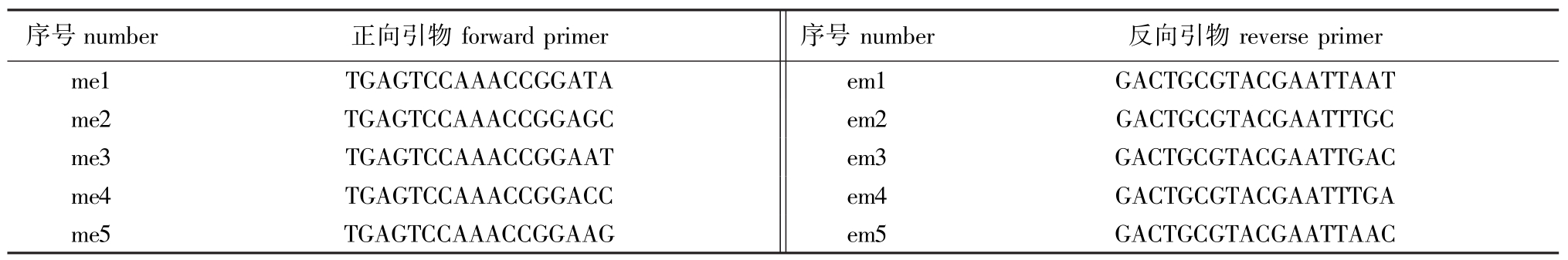
序号number 正向引物forward primer 序号number 反向引物reverse primer me1 TGAGTCCAAACCGGATA em1 GACTGCGTACGAATTA AT me2 TGAGTCCAAACCGGAGC em2 GACTGCGTACGAATTT GC me3 TGAGTCCAAACCGGAAT em3 GACTGCGTACGAATTG AC me4 TGAGTCCAAACCGGACC em4 GACTGCGTACGAATTT GA me5 TGAGTCCAAACCGGAAG em5 GACTGCGTACGAATTAAC
1.2.2 SRAP-PCR反应程序 随机选用引物me1/ em3进行优化,采用10 μL反应体系,按照 Li等[3]的方法,以皱纹盘鲍基因组 DNA为模板的SRAP-PCR扩增程序为:94℃下预变性5 min;94℃下变性1 min,35℃下退火1 min,72℃下延伸1 min,共进行5个循环;94℃下变性1 min,50℃下退火1 min,72℃下延伸1 min,共进行35个循环;最后在72℃下再延伸10 min。PCR产物首先用10 g/L琼脂糖凝胶电泳检测,然后使用8 g/L的非变性聚丙烯酰胺凝胶电泳分型。
1.2.3 反应体系因素水平的确定及正交设计 影响PCR扩增效果的因素主要有Mg2+浓度、dNTPs浓度、引物浓度、模板DNA以及Taq酶活性。根据徐宗大等[15]的设计方案,本研究中采用 L16(45)正交试验设计对以上5个因素进行4水平筛选,共16个处理,每个处理设3个重复,各因素水平见表2。根据电泳条带的多少、清晰度及背景颜色进行打分。条带最丰富、清晰度最高、背景颜色最浅的记为16分,反之则扣分,最差得1分,计算每个因素在不同水平下的得分总和及其均值k,以及各因素的极差R。
表2 SRAP-PCR体系的因素水平
Tab.2 Factors and levels of SRAP-PCR reaction system
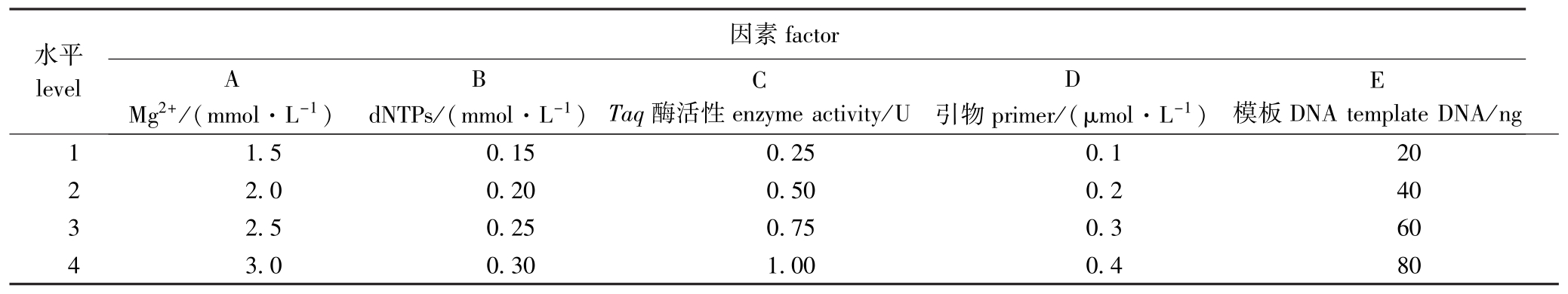
水平level因素factor A Mg2+/(mmol·L-1) B C D E dNTPs/(mmol·L-1)Taq酶活性enzyme activity/U 引物primer/(μmol·L-1)模板DNA template DNA/ng 1 1.5 0.15 0.25 0.1 20 2 2.0 0.20 0.50 0.2 40 3 2.5 0.25 0.75 0.3 60 4 3.0 0.30 1.00 0.4 80
1.2.4 体系稳定性检测 随机选用5对引物组合(me1/em2,me2/em1,me3/em5,me4/em4,me5/ em3)对反应体系的稳定性进行检测,每对引物设置2次重复。
2 结果与分析
2.1 电泳结果评分和极差分析
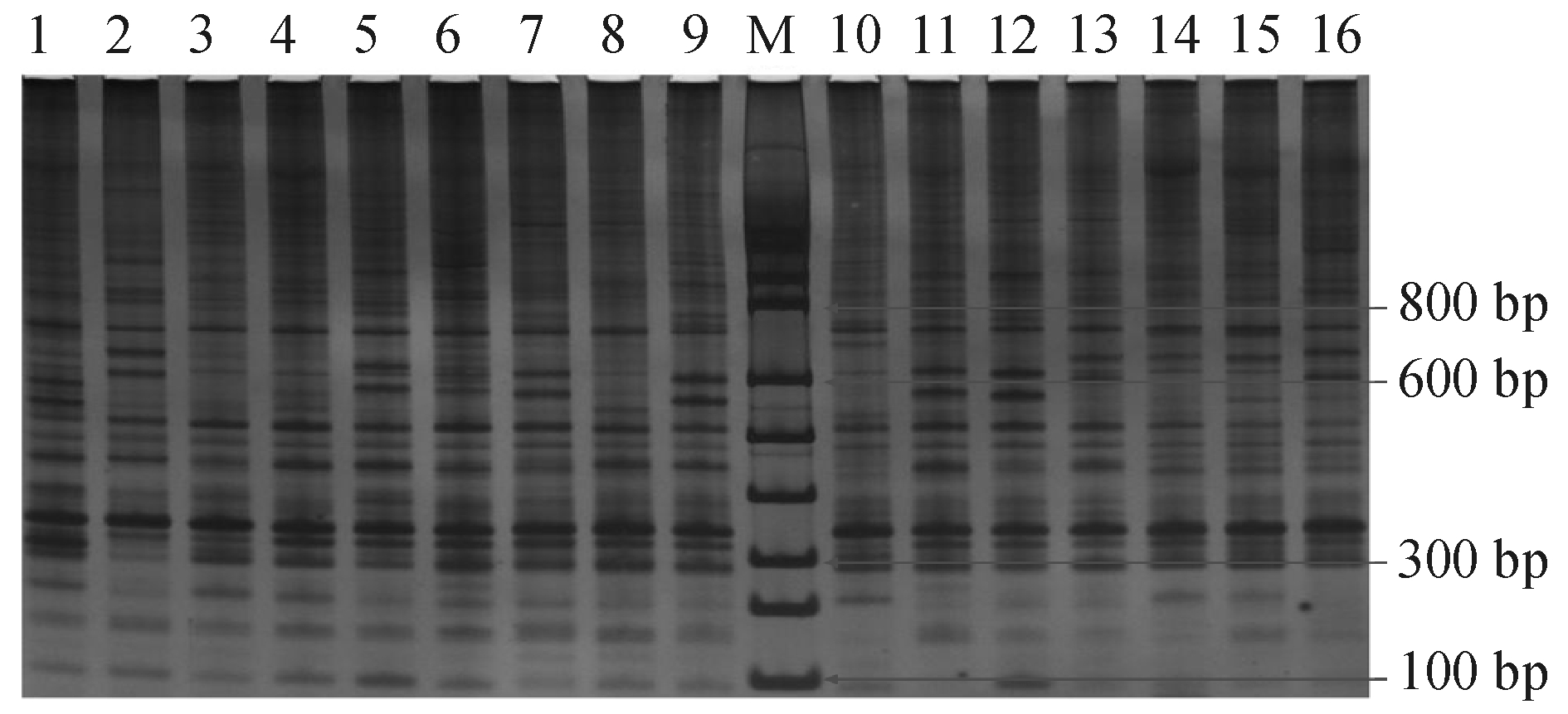
根据评分标准,对3次重复试验所得的电泳图谱(图1,仅显示1次电泳结果)分别统计,得分见表3。从3次电泳的得分来看,各重复差异不大,试验的一致性较好。其中以1号和10号试验组效果最好,评分分别达到15.67和14.67分,以12号试验组效果最差,评分仅为2.33分,这与图1显示的结果一致。各因素的极差值大小反映了其对反应结果整体的影响程度,由表3的极差分析可见,模板量对反应结果影响最大,Taq酶活性影响最小,各因素对PCR扩增结果的影响从大到小依次为模板DNA>Mg2+浓度>dNTPs浓度>引物浓度>Taq酶活性。
2.2 各因素水平对PCR反应结果的影响
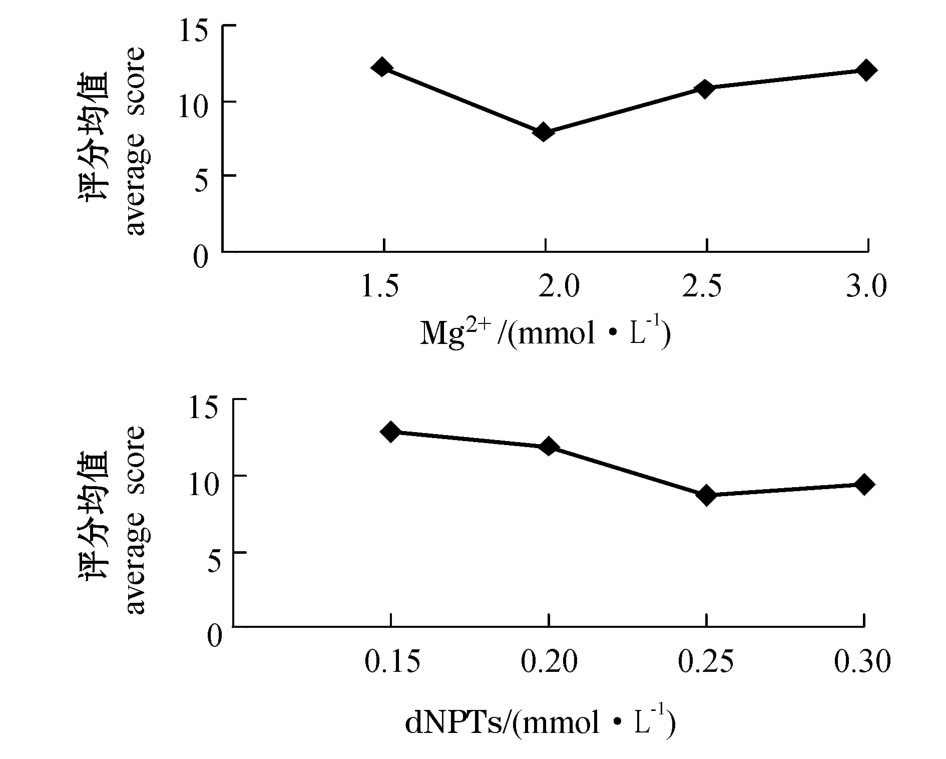
Mg2+是影响PCR反应结果最重要的因素之一。较高的 Mg2+浓度能够提高 Taq酶的活性,提高PCR的产率,但是Mg2+浓度过高又容易导致非特异性扩增,反映在电泳结果上就是影子带增多,背景变深,降低了结果的可读性和可重复性。从图2可见:Mg2+浓度为1.5 mmol/L时,实现最佳扩增; Mg2+浓度为2.0 mmol/L时,扩增效果反而下降,可读性变差;Mg2+浓度为2.5~3.0 mmol/L时,随着离子浓度的增加扩增效果再次开始好转,但最终没有超过1.5 mmol/L浓度时的扩增效果。故最佳的Mg2+反应浓度为1.5 mmol/L。
表3 SRAP-PCR反应因素水平的正交试验设计及试验结果
Tab.3 Design and results of orthogonal test for SRAP-PCR reaction
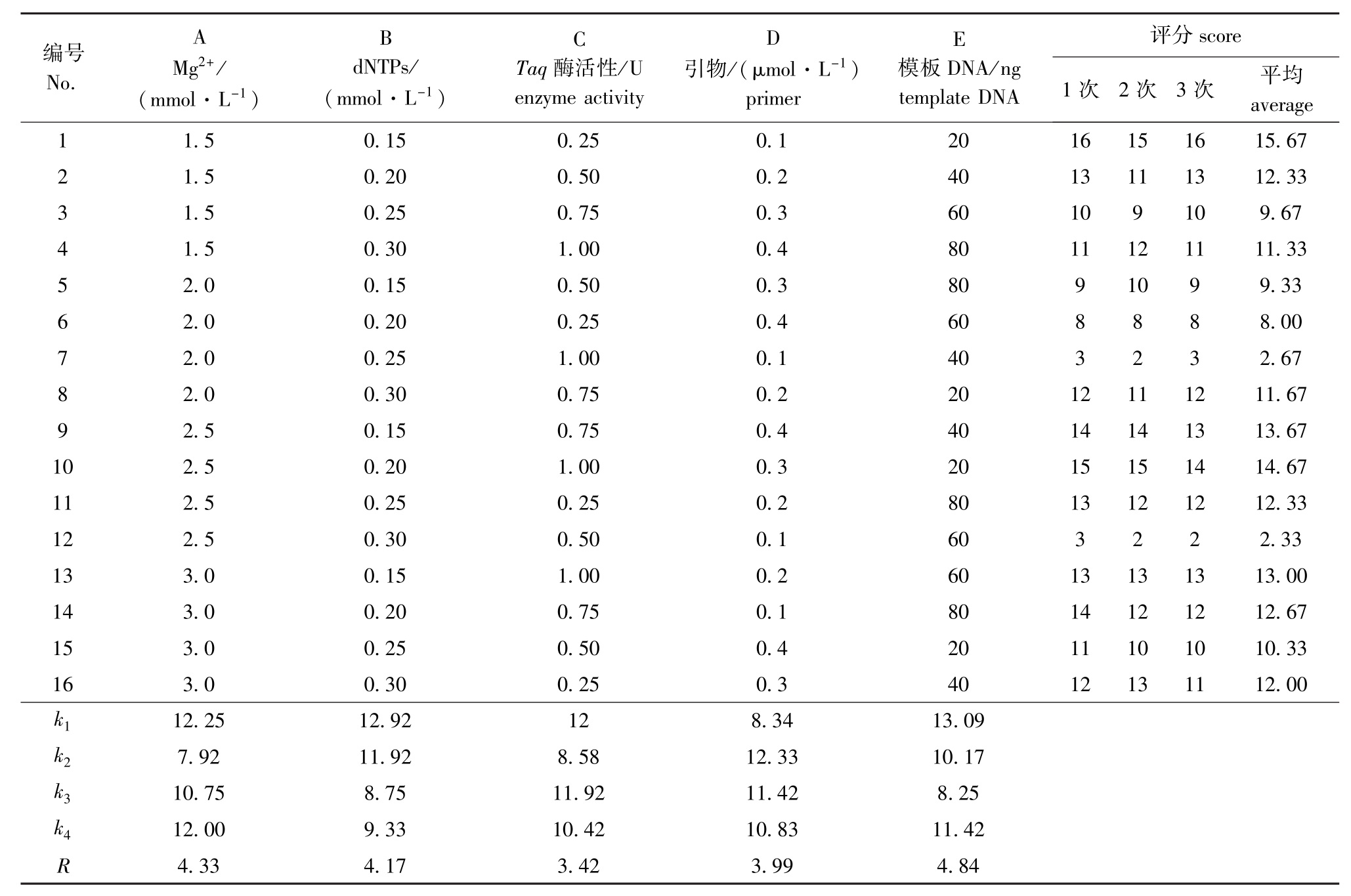
编号No. A B C D E 评分Mg2+/(mmol·L-1) dNTPs/(mmol·L-1) Taq酶活性/U enzyme activity引物/(μmol·L-1) primer模板DNA/ng template DNA score 1次 2次 3次 平均average 1.5 0.15 0.25 0.1 20 16 15 16 15.67 2 1.5 0.20 0.50 0.2 40 13 11 13 12.33 3 1.5 0.25 0.75 0.3 60 10 9 10 9.67 4 1.5 0.30 1.00 0.4 80 11 12 11 11.33 5 2.0 0.15 0.50 0.3 80 9 10 9 9.33 6 2.0 0.20 0.25 0.4 60 8 8 8 8.00 7 2.0 0.25 1.00 0.1 40 3 2 3 2.67 8 2.0 0.30 0.75 0.2 20 12 11 12 11.67 9 2.5 0.15 0.75 0.4 40 14 14 13 13.67 10 2.5 0.20 1.00 0.3 20 15 15 14 14.67 11 2.5 0.25 0.25 0.2 80 13 12 12 12.33 12 2.5 0.30 0.50 0.1 60 3 2 2 2.33 13 3.0 0.15 1.00 0.2 60 13 13 13 13.00 14 3.0 0.20 0.75 0.1 80 14 12 12 12.67 15 3.0 0.25 0.50 0.4 20 11 10 10 10.33 16 3.0 0.30 0.25 0.3 40 12 13 11 12.00 1 k112.25 12.92 12 8.34 13.09 k27.92 11.92 8.58 12.33 10.17 k310.75 8.75 11.92 11.42 8.25 k412.00 9.33 10.42 10.83 11.42 R 4.33 4.17 3.42 3.99 4.84
dNTPs是PCR反应的底物。底物浓度过低,直接影响PCR反应的合成效率,浓度过高则会导致聚合酶将错误碱基渗入。从图2可见:当dNTPs浓度为0.15~0.25 mmol/L时,随着dNTPs浓度的升高,评分值反而降低,这主要是由于非特异性扩增增加,导致背景变深,可读性变差;当dNTPs浓度为0.3 mmol/L时,评分值略有上升,但仍低于0.15 mmol/L浓度时的评分值。故最优的dNTPs反应浓度为0.15 mmol/L。
Taq酶是PCR反应的核心,酶量过多会导致非特异性扩增,过少则可能导致条带不清晰甚至完全没有条带。从图3可见:酶活性为0.25~0.50 U时,评分值有所下降;酶活性为0.50~0.75 U时,评分值开始回升;酶活性超过0.75 U时,评分值又开始下降。故最佳Taq酶活性水平为0.25 U。
引物浓度过高,可能会产生错配或二聚体,体现在电泳结果上就是小片段增多,重复性变差;浓度过低则可能造成PCR产量不足,条带不清晰。从图3可见:引物浓度为0.1~0.2 μmol/L时,评分值提高,这主要是因为条带变清晰;引物浓度为0.2~0.4 μmol/L时,评分值呈降低趋势,主要是因为影子带和二聚体增多。故最佳引物反应浓度为0.2 μmol/L。
模板DNA水平过低,会导致PCR扩增结果不稳定;过高则可能造成条带冗余,给识别带来困难。从图3可见:模板DNA为20 ng时反应体系获得了最高分,此时条带最清晰;模板 DNA为20~60 ng时,评分值逐渐降低,背景增强;模板DNA为60~80 ng时,评分值又有所上升,因为模板DNA的增加造成PCR产物增加,扩增条带再次变清晰。故最佳模板DNA反应水平为20 ng。
2.3 最优因子组合的筛选和验证
从表3的极差分析表明,最优试验组合为A1B1C1D2E1,即 10 μL反应体系中,Mg2+1.5 mmol/L、dNTPs 0.15 mmol/L、引物0.2 μmol/L、Taq酶活性0.25 U以及模板DNA 20 ng时,PCR扩增结果最佳。在此条件下,对最佳试验组合进行验证,电泳结果如图4所示。从图4可以看出,相对于优化前的反应体系(图1),新体系扩增效率更稳定,可读条带更多,可以认为优化反应体系起到了预期的效果。
3 讨论
良好的可读性和可重复性,是分子标记应用于遗传学研究的重要前提。一般来说,对于二倍体生物,一次只扫描一个位点的微卫星标记和SNP标记更易被准确识别。而一次扩增产生大量片段的RAPD、AFLP等标记均存在着影子带多、识别困难、可重复性较差等问题。这就要求在应用这些分子标记之前花费一定的精力去优化其反应条件。作为一种高效和新颖的分子标记,SRAP从诞生伊始就受到了遗传学家的喜爱,廉价、高效和简单的操作特点,迅速在植物育种中普及开来。相对于AFLP,SRAP扩增的条带数较少,识别相对简单;相对于RAPD,SRAP的扩增稳定性大大提高,假阳性率相对较低;相对于微卫星和SNP,SRAP不需要预先知道基因组序列,成本低廉[2]。这些都说明SRAP标记在种群动态、种质资源评估和育种中都将有更为广泛地应用。在水产动物上SRAP技术的应用刚刚起步,在贝类中则尚未见报道。从目前的研究看来,对于不同物种SRAP的反应体系往往有较大的差异[16-17],在应用到新物种上时需要进行反应体系的优化。从本研究结果来看,优化反应体系确实提高了扩增条带的识别率,降低了假阳性和误判发生的概率。
作为一种基于 PCR技术的分子标记,优化SRAP反应体系实际上就是优化PCR反应体系,其影响因素自然也就是影响PCR扩增效率的几大因素——Mg2+浓度、dNTPs浓度、引物浓度、模板DNA以及Taq酶活性。优化方法一般有3种:单因素试验、均匀试验和正交试验。相对其他两种试验,正交试验不仅试验次数较少而且能够顾及各因素间的交互作用,是相对较佳的一种方法。目前,大多数物种的SRAP反应体系优化都是采用正交试验[16,18-19],少数采用单因素试验[8], 本研究中也采用正交试验设计法优化了SRAP反应体系,用较少的试验取得了良好的结果。
在皱纹盘鲍SRAP反应体系优化涉及到的5个因素中,模板DNA和Mg2+浓度对反应结果影响最大,Taq酶活性的影响最小,该结果与对三疣梭子蟹的研究结果相类似[11],与对大多数植物的研究结果不同[5,15-17],这可能是由于海产动物的细胞组织含盐量高以及对动植物DNA提取方法不同所致。该SRAP-PCR反应体系的建立和优化为今后利用这种标记在皱纹盘鲍种质鉴定、遗传多样性分析和标记辅助选择等方面提供了较先进的技术方法,也必将加快分子标记辅助选择在皱纹盘鲍育种中的应用。
参考文献:
[1] 王志松,张明,李洪臣,等.我国工厂化养鲍技术现状与展望[J].水产科学,2005,24(3):42-43.
[2] 柳李旺,龚义勤,黄浩,等.新型分子标记——SRAP与TRAP及其应用[J].遗传,2004,26(5):777-781.
[3] Li G,Quiros C.Sequence-related amplified polymorphism(SRAP),a new marker system based on a simple PCR reaction:its application to mapping and gene tagging in Brassica[J].Theor Appl Genet,2001,103:455-461.
[4] 潘俊松,王刚,李效尊,等.黄瓜SRAP遗传连锁图的构建及始花节位的基因定位[J].自然科学进展,2005,15(2):167-172.
[5] 任羽,王得元,张银东,等.辣椒SRAP-PCR反应体系的建立与优化[J].分子植物育种,2004,2(5):689-693.
[6] 林忠旭,张献龙,聂以春.新型标记SRAP在棉花F2分离群体及遗传多样性评价中的适用性分析[J].遗传学报,2004,31(6):622-626.
[7] 高丽霞,胡秀,刘念,等.中国姜花属基于SRAP分子标记的聚类分析[J].植物分类学报,2008,46(6):899-905.
[8] 李莉,彭建营,白瑞霞.中国枣属植物亲缘关系的SRAP分析[J].中国农业科学,2009,42(5):1713-1719.
[9] 姚建华,傅洪拓,龚永生,等.日本沼虾SRAP反应体系正交设计及优化[J].华北农学报,2008,23(S2):122-125.
[10] 丁炜东,曹丽萍,曹哲明.草鱼种质相关SRAP及SCAR的分子标记[J].动物学报,2008,54(3):475-481.
[11] 胡则辉,徐君卓,王跃斌,等.基于正交试验设计优化三疣梭子蟹SRAP-PCR反应体系[J].南方水产,2010,6(5):68-73.
[12] 辛文婷,孙中武,尹洪滨,等.黄颡鱼雌雄差异的SRAP标记[J].东北林业大学学报,2009,37(5):112-113.
[13] 冉玮,张桂蓉,王卫民,等.利用SRAP标记分析3个团头鲂群体的遗传多样性[J].华中农业大学学报,2010,29(5):601-606.
[14] Strauss W.Preparation of genomic DNA from mammalian tissues [M]//Current Protocol in Molecular Biology.New York:John Wiley and Softs,1989:221-222.
[15] 徐宗大,赵兰勇,杨志莹.利用正交试验优化玫瑰SRAP-PCR反应体系[J].中国农学通报,2010,26(11):215-217.
[16] 刘冲,葛才林,任云英,等.SRAP、ISSR技术的优化及在甘蓝类植物种子鉴别中的应用[J].生物工程学报,2006,22(4): 657-661.
[17] 孙佳琦,梁建国,石少川,等.SRAP标记在观赏植物遗传育种中的应用[J].分子植物育种,2010,8(3):577-588.
[18] 陈华增,李健,何玉英,等.中国对虾SRAP分子标记体系正交优化及在遗传多样性分析中的应用[J].渔业科学进展,2010, 31(6):43-53.
[19] 杨兵,王天志,罗禹,等.青牛胆SRAP标记反应体系的建立与优化[J].中草药,2007,38(2):272-275.
Optimization of SRAP-PCR System for disk abalone Haliotis discus hannai based on orthogonal design
LIU Ming-tai1,LI Yang2,LIU Xiao-lin2,XU Shu-fen1,CHANG Ya-qing3
(1.Dalian Haibao Fisheries Company Limited,Dalian 116045,China;2.College of Animal Science and Technology,Northwest Agricultural&Forestry University,Yangling 712100,China;3.Key Laboratory of Mariculture&Stock Enhancement in North China's Sea,Ministry of Agriculture, Dalian Ocean University,Dalian 116023,China)
Abstract:A L16(45)orthogonal test was employed in the present study to evaluate the influences of five important factors such as concentration of Mg2+,dNTPs,primers,templates and activity of Taq DNA polymerase influencing amplification of SRAP-PCR,and optimization of SRAP-PCR System for disk abalone Haliotis discus hannai.The results showed that the optimum concentrations were found to be 1.5 mmol/L in Mg2+,0.15 mmol/L in dNTPs, 0.2 μmol/L in primer 0.25 U in activity of Taq enzyme,DNA polymerase and 20 ng in template DNA in a total volume of 10 μL reaction solution.The system was proved by other primers and template test to amplify reliable bands and to be used in the genetic analysis of disk abalone.
Key words:Haliotis discus hannai;SRAP;orthogonal design;molecular marker;reaction system
中图分类号:Q953.1
文献标志码:A
收稿日期:2012-05-26
基金项目:国家 “863”计划项目(2006AA10A411)
作者简介:刘明泰(1970-),男,硕士,高级工程师。E-mail:wl1970@163.com
通信作者:刘小林(1961-),男,教授。E-mail:liuxiaolin@nwsuaf.edu.cn;常亚青(1967-),男,教授。E-mail:yqchang@dlou.edu.cn
文章编号:2095-1388(2013)01-0012-05