历山中国大鲵线粒体片段序列的测定及其遗传差异研究
黄立群1、3,仪慧兰1,崔松林2,郭军1
(1.山西大学生命科学学院,山西太原030006;2.山西省水产科学研究所,山西太原030006;3.中国辐射防护研究院,山西太原030006)
摘要:对山西历山2尾野生大鲵Andrias davidianus mtDNA中Cyt b和ATPase 6基因的部分序列进行了检测,并与GenBank中收集的14尾外地样本进行比较,用Mega 5.0软件对305 bp Cyt b片段和371 bp ATPase 6片段进行分析。结果表明:Cyt b片段有10个不同的单倍型,共检测出14个变异位点,占核苷酸总数的4.58%,其中有12个简约信息位点;ATPase 6片段有13个不同的单倍型,检测出30个变异位点,占核苷酸总数的8.09%,其中有18个简约信息位点,Cyt b比ATPase 6更为保守,但两序列中碱基组成均表现为鸟嘌呤缺乏 (<18.9%),且多数变异发生在密码子第3位。合并序列构建的分子系统树表明,历山大鲵与陕西、四川及部分湖南个体聚为一支,历山大鲵与广西样本的遗传距离最远,与四川、陕西样本的遗传距离较近。
关键词:中国大鲵;历山地区;Cyt b;ATPase 6;mtDNA
中国大鲵Andrias davidianus俗称 “娃娃鱼”,属于两栖纲、有尾目、隐鳃鲵科,是现存个体最大的两栖动物。隐鳃鲵科有隐鳃鲵属Cryptobranchus和大鲵属Andrias 2个属,共计3个种,其中隐鳃鲵属的北美隐鳃鲵Cryptobranchus alleganiensis分布于美国东部,大鲵属的日本大鲵Andrias japonicus和中国大鲵分别分布于日本和中国[1]。中国大鲵是中国特有的珍稀有尾两栖动物,属于国家二级保护动物,从水系看主要分布于长江中上游、珠江上游及黄河流域的部分地区[2]。杨丽萍等[3]对中国大鲵5个野生种群的遗传多样性和遗传分化水平进行了研究,并结合化石证据,推测中国大鲵由北向南进行扩散。山西省翼城、垣曲、阳城、沁水四县交界处的历山是现今野生大鲵分布的最北端,历山国家自然保护区的野生大鲵资源是中国大鲵重要的组成部分,可能在种群演化历史中起到了重要作用。近年来,由于大鲵栖息地环境恶化以及人为乱捕滥杀,造成野生大鲵资源量锐减,部分地区濒临灭绝[4]。为此,开展大鲵群体遗传学研究,确定中国大鲵不同种群之间的遗传关系,对大鲵的人工繁育和种质资源保护具有重要意义。
线粒体DNA(mtDNA)由于呈母系遗传,且碱基替换速率快,无组织特异性,是两栖类进化研究中重要的分子标记,被广泛应用于种群遗传多样性、种群遗传结构及进化史等方面的研究中[5-6]。Cyt b由于进化速度适中,被认为是解决系统发育问题最可信的分子标记之一[7-9],但单个线粒体基因在反映脊椎动物系统发生关系时可能效果不佳[10]。为此,本研究中作者以mtDNA中Cyt b和ATPase 6序列为分子标记,比较了历山野生大鲵与其他地域中国大鲵的序列差异,从分子水平上探讨它们之间的相互关系,以期为大鲵的保护、繁殖和育种提供参考依据。
1 材料与方法
1.1 材料
试验用野生大鲵2尾,于2010年取自历山国家自然保护区,1号样本取其肌肉组织,2号样本采用大鲵自然生理过程中脱掉的皮,经生理盐水清洗后用体积分数为75%的乙醇浸泡,两种样品均在-20℃下保存备用。
1.2 方法
1.2.1 基因组DNA的提取 大鲵组织用生理盐水浸洗后,剪碎,称取约50 mg置于1.5 mL离心管中,采用常规酚-氯仿抽提法提取基因组DNA,经TE溶解后用琼脂糖凝胶电泳检测。
1.2.2 PCR扩增及测序 PCR扩增时Cyt b引物参考Murphy等[11]的引物序列,ATPase 6引物为自行设计引物 (表1),PCR反应程序:94℃下预变性5 min;94℃下变性30 s,退火30 s,72℃下延伸40 s,共进行30个循环;最后在72℃下延伸10 min。退火温度分别为51℃和57℃。用10 g/L琼脂糖凝胶电泳检测PCR产物,回收目的条带送华大基因公司测序。
表1 PCR扩增引物
Tab.1 Primers used in PCR amplification

基因gene 引物序列primer sequence产物长度product length/bp Cyt b L 5′CCATCCAACATCTCAGCATGATGAAA 3′358 H 5′GCCCCTCAGAATGATATTTGTCCTCA 3′ATPase 6 L 5′CTGACCATGAATTTAAGCTTTTTTGACC AATT 3′ 487 H 5′ATTAGTAGGTGACCTGCTGTTAGATTTGCTGTT 3′
1.3 数据处理
为了比较大鲵种群线粒体序列变异,从NCBI上收集到已知来源的中国大鲵样本14尾 (表2)。中国大鲵样本按地域来源及水系进行编号,其中AH代表安徽样本,GX代表广西样本,HN代表湖南样本,SC代表四川样本,ShX代表陕西样本, SX代表山西 (垣曲)样本;Y代表黄河水系,YZ代表长江水系,P为珠江水系;SX1-Y和SX2-Y为所测历山样本。
使用软件 Geneious Pro 5.1.7辅助人工校对DNA序列,相关序列的收集、比对由Geneious软件完成[12]。用Mega 5.0软件统计序列之间的碱基差别,计算核苷酸使用频率,采用ML(Maximum likelihood)法和MP(Maximum parsimony)法分别构建分子系统树,ML采用Kimura双参数模型, Bootstrap 1000循环检验结果为各分支的置信度。
表2 大鲵样本来源
Tab.2 The samples of Chinese giant salamander collected from different areas
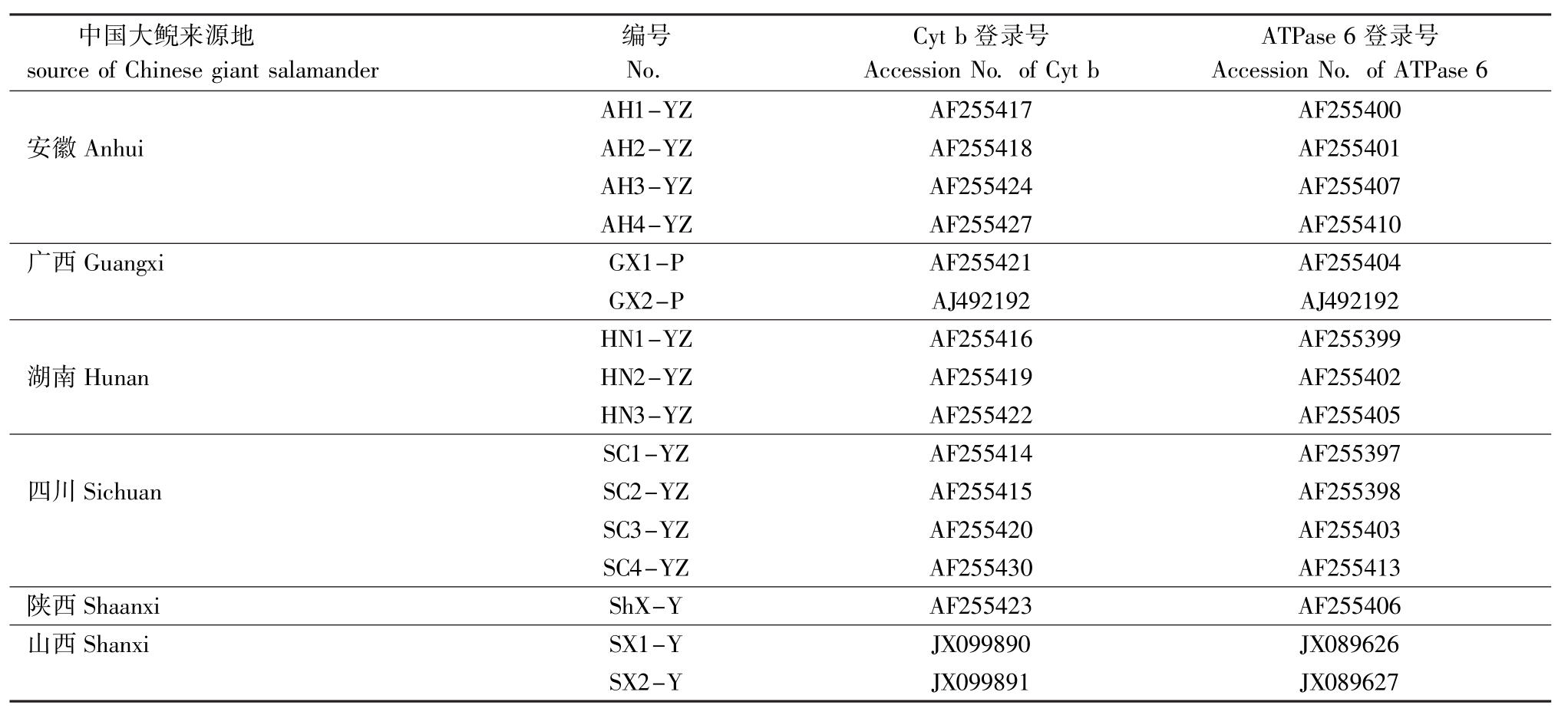
中国大鲵来源地source of Chinese giant salamander编号No. Cyt b登录号Accession No.of Cyt b ATPase 6登录号Accession No.of ATPase 6 AH1-YZ AF255417 AF255400安徽Anhui AH2-YZ AF255418 AF255401 AH3-YZ AF255424 AF255407 AH4-YZ AF255427 AF255410广西Guangxi GX1-P AF255421 AF255404 GX2-P AJ492192 AJ492192 HN1-YZ AF255416 AF255399湖南Hunan HN2-YZ AF255419 AF255402 HN3-YZ AF255422 AF255405 SC1-YZ AF255414 AF255397四川Sichuan SC2-YZ AF255415 AF255398 SC3-YZ AF255420 AF255403 SC4-YZ AF255430 AF255413陕西Shaanxi ShX-Y AF255423 AF255406山西Shanxi SX1-Y JX099890 JX089626 SX2-Y JX099891 JX089627
2 结果与分析
2.1 DNA提取
目前,国内学者普遍采用大鲵肌肉或肝脏组织提取DNA,但Matsui等[13-14]曾从大鲵脚趾尖、尾尖及表皮中提取DNA。大鲵在饲养过程中会经常脱皮 (10~30 d脱皮一次)[15],本试验中尝试采用常规酚-氯仿抽提法从2号大鲵样本所脱皮中提取DNA。用琼脂糖凝胶电泳检测,所提DNA样品产生单一条带,纯度高,无降解,与肌肉来源的DNA相似。这说明,对于大鲵这样的珍稀保护动物,完全可以采用非损伤取样进行分子遗传学研究。
2.2 Cyt b区域DNA序列的组成与变异
测定和收集了中国大鲵6个不同地理区域16个样本的Cyt b基因序列,有效片段长为305 bp,共发现10个不同的单倍型。对核苷酸序列进行分析,共检测出14个变异位点 (图1-A),占核苷酸总数的4.58%,其中有12个简约信息位点,多数变异发生在密码子的第3位点上 (占64.3%),第1、第2位点变异数分别占总变异数的28.6%和7.1%。没有任何碱基的插入或缺失,其中A、T、G和 C碱基平均含量分别为 23.9%、35.1%、18.9%和22.1%,碱基组成的百分比显示了G的相对缺乏,A+T平均含量为59%,G+C含量为41%。核苷酸的替换以转换为主,转换远多于颠换(13/1),表现出高的转换偏倚[16],其中A←→G转换 (8)比T←→C(5)转换略多。

图1 历山大鲵Cyt b和ATPase 6序列多态性位点分布
Fig.1 Polymorphic sites in the Cyt b and ATPase 6 sequences of Chinese giant salamander in Lishan region
注:A区为Cyt b序列;B区为ATPase 6序列;纵向数字表示变异的核苷酸位点。
Note:The part A is Cyt b sequence;The part B is ATPase 6 sequence;Vertical numbers mean varied nucleotide sites.
2.3 ATPase 6区域DNA序列的组成与变异
检测ATPase 6基因序列有效片段长为371 bp,有13个不同的单倍型,发现30个变异位点,占核苷酸总数的8.09%(图1-B),其中有18个简约信息位点,多数变异发生在密码子的第3位点上(占50%),第1、第2位点变异数分别占总变异数的43.3%和6.7%。没有碱基的插入或缺失,其中A、T、G和 C碱基平均含量分别为 29.1%、35.2%、12.3%和23.4%,碱基组成百分比同样显示了G的相对缺乏,A+T平均含量为64.3%,而G+C含量只有35.7%。核苷酸的替换以转换为主,转换远多于颠换 (22/8),同样表现出高的转换偏倚,其中A←→G转换 (14)也多于T←→C转换 (8)。与Cyt b序列相比,ATPase 6序列具有更高的变异度,但两序列碱基组成同样表现出了G的缺乏,多数变异发生在的密码子的第三位,且变异以转换为主。
2.4 分子系统树
大鲵种群地理关系比较复杂,因此采用ML和MP两种方法构建6个不同地理区域中国大鲵Cyt b和ATPase 6合并序列的分子系统树。合并序列全长为676 bp,以新疆北鲵Ranodon sibiricus作为外群,Bootstrap 1000次循环检验结果作为各分支的置信度 (图2)。两种方法构建的分子系统树具有基本相同的拓扑结构,拓扑结构分为3个分支,其中安徽个体聚在一起为一单独分支,广西个体和湖南一个体聚为一支,而山西、陕西、四川及湖南两个体聚为另一分支。历山大鲵样本与广西样本的遗传距离最远,为0.032,与四川、陕西样本的遗传距离较近。长江流域种群与黄河流域种群共享有单倍型HN1-YZ(SX1-Y),长江流域种群与珠江流域种群共享有单倍型GX2-P(HN2-YZ),而黄河流域与珠江流域的地理种群没有共享的单倍型。从流域来看,黄河流域大鲵聚在第1分支,珠江流域大鲵聚在第2分支,长江流域大鲵则分布于第3分支。由此可见,大鲵的遗传分化呈现出地理区域上的连续性:长江流域介于黄河流域和珠江流域之间,其种群与其它种群之间遗传距离较近且存在交叉,而黄河流域与珠江流域种群没有交叉,这与陶峰勇等[17]的研究结果一致。
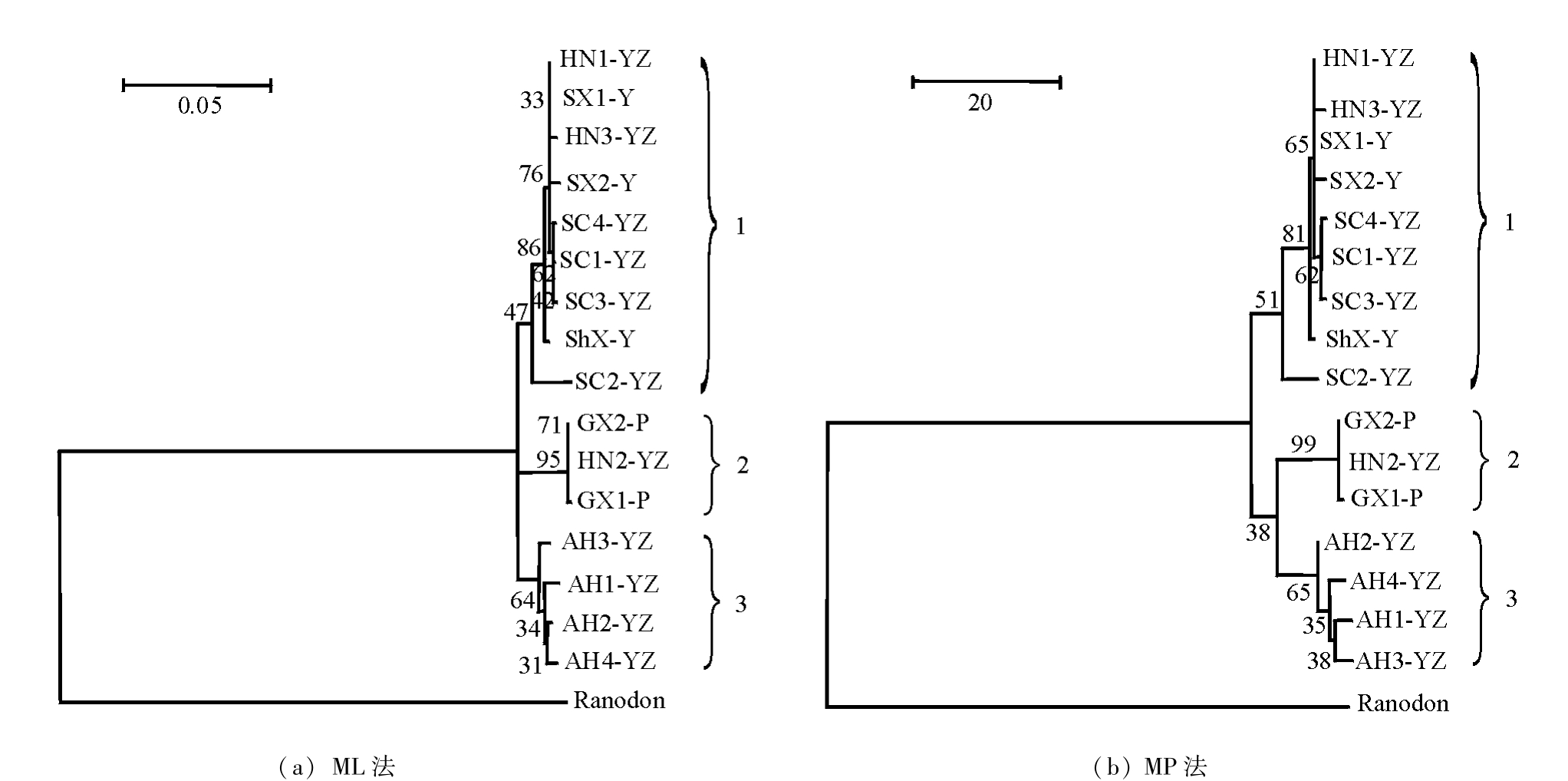
图2 以Cyt b和ATPase 6合并序列构建的分子系统树
Fig.2 Molecular phylogenetic trees based on the combined sequences of Cyt b and ATPase 6 genes
3 讨论
中国大鲵迁徙能力差,对水环境的依赖性很大,且不同水系种群间的基因交流不太可能,部分地区种群可能形成独特的种群遗传特征。中国大鲵主要分布于23.5~35°N的区域,本研究中大鲵样本采集于历山地区35°12′N处。历山作为现今中国大鲵分布的最北端,研究其种群结构及遗传特性对于大鲵种质资源的保护具有重要意义。本研究结果表明:2尾历山大鲵基因序列在个别位点有地域特异性,历山大鲵与四川、陕西样本的遗传距离较近,与广西样本的遗传距离最远。分析不同来源16个样品核苷酸序列的差异,所测ATPase 6序列变异率比Cyt b序列高,但两基因序列碱基组成同样表现出了鸟嘌呤的缺乏,多数变异发生在密码子第3位,且变异以转换为主。本研究中虽然收集的大鲵样品数量有限,只检测了2段常用DNA分子标记序列,但还是能够说明中国大鲵不同地理位置种群间存在遗传差异,历山野生大鲵种群具有一定的遗传特征。Murphy等[11]在对中国大鲵mtDNA序列分析的基础上,认为安徽黄山大鲵为一单独分支,具有独特的遗传特征,但没有确定出三流域之间地理种群的亲缘关系。本研究中,作者认为长江流域种群可能充当了黄河流域种群与珠江流域种群的桥梁作用。
历山地区大鲵分布比较集中,主要位于李家河上游部分溪流中。山西省曾于1984年对历山地区大鲵资源进行了调查,发现大鲵主要分布于同善公社东北角和铜矿峪一带。2010年作者对大鲵栖息地调查时发现,铜矿峪地区由于铜矿的开发,河道污染严重,大鲵已经绝迹,另一主分布区西阳河上游西哄哄段大鲵数量也急剧减少。因此,保护历山野生大鲵资源刻不容缓。
目前,大鲵人工繁殖及规模化繁育技术已取得成功[18],种群数量已有所恢复,但其种群多样性短期内仍难以有效恢复[19]。Lin等[20]通过 RAPD分析了大鲵亲本及子二代的多样性,发现子二代的多样性远远低于亲本。因此,保护野生大鲵种群对于保护大鲵资源并有效利用其资源具有重要意义。历山地区是野生大鲵分布区域中纬度最高的地域,其大鲵种群在中国大鲵资源中占有不可替代的重要位置。在有关部门的倡导与资助下,垣曲县境内成立了山西省首个大鲵保护繁育站,但由于资金、设施及配套管理措施不到位,尤其是专业技术人员的缺乏,工作难度较大,亟待多方的大力支持。
致谢:本研究得到了山西省水产科学研究所、垣曲县水利局和历山大鲵保护繁育站的大力支持,在此表示感谢!
参考文献:
[1] 侯进慧,朱必才,童玉玮,等.中国大鲵研究进展[J].四川动物,2004,23(3):262-276.
[2] 章克家,王小明,吴巍,等.大鲵保护生物学及其研究进展[J].生物多样性,2002,10(3):291-297.
[3] 杨丽萍,蒙子宁,刘晓春.中国大鲵5个野生种群的AFLP分析[J].中山大学学报,2011,50(2):99-104.
[4] 郑合勋.河南省卢氏县大鲵种群生态学及生态适应特征研究[D].上海:华东师范大学,2006.
[5] Feller A E,Hedges S B.Molecular evidence for the early history of living amphibians[J].Molecular Phylogenetics and Evolution, 1998,9:509-516.
[6] Saccone C,Giorgi C D,Gissi C,et al.Evolutionary genomics in Metazoa:the mitochondrial DNA as a model system[J].Gene, 1999,238:195-209.
[7] 陶峰勇.中国大鲵Andrias davidianus不同地理种群遗传分化的初步研究[D].上海:华东师范大学,2005.
[8] 王义权,许群山,彭宣宪,等.通过Cyt b基因同源序列比较评估厦门文昌鱼的分类学地位[J].动物学报,2004,50(2):202-208.
[9] Zhang P,Chen Y Q,Liu Y F,et al.The complete mitochondrial genome of the Chinese giant salamander,Andrias davidianus(Amphibia:Caudata)[J].Gene,2003,311:93-98.
[10] Zardoya R,Meyer A.Phylogenetic performance of mitochondrial protein-coding genes in resolving relationships among vertebrates [J].Molecular Biology and Evolution,1996,13:933-942
[11] Murphy R W,Fu J Z,Upton D E,et al.Genetic variability among endangered Chinese giant salamander,Andrias davidianus[J].Molecular Ecology,2000,9:1539-1547.
[12] Drummond A J,Ashton B,Buxton S,et al.Geneious Pro 5.1.7 [EB/OL].[2011-10-01].http://www.geneious.com.
[13] Matsui M,Tominaga A,Liu W Z,et al.Reduced genetic variation in the Japanese giant salamander,Andrias japonicus(Amphibia: Caudata)[J].Molecular Phylogenetics and Evolution,2008,49: 318-326.
[14] Matsui M,Tominaga A,Hayashi T,et al.Phylogenetic relationships and phylogeography of Hynobius tokyoensis(Amphibia:Caudata)using complete sequences of cytochrome b and control region genes of mitochondrial DNA[J].Molecular Phylogenetics and Evolution,2007,44:204-216.
[15] 陈云祥.大鲵高效养殖技术一本通[M].北京:化学工业出版社,2008.
[16] 陶峰勇,王小明,郑合勋.中国大鲵五地理种群Cyt b基因全序列及其遗传关系分析[J].水生生物学报,2006,30(5):625-628.
[17] 陶峰勇,王小明,郑合勋,等.中国大鲵四种群的遗传结构和地理分化[J].动物学研究,2005,26(2):162-167.
[18] Chen L J,Tao F Y,Lu Q B.Advances in research of Chinese giant salamander[J].Journal of Shanghai Fisheries University, 2003,12:110-114.
[19] Thorbjarnarson J,Wang X M.The conservation status of the Chinese alligator[J].Oryx,1999,33(2):152-159.
[20] Lin M,Huang J,Li Z,et al.RAPD analysis on the wild parents and second filial generation of artificial breeding of Andrias davidianus[J].Journal of Shanghai Fisheries University,2003,12:20-23.
Nucleotide sequence differences in some mitochondrial DNA regions in wild Chinese giant salamander Andrias davidianus in Lishan region
HUANG Li-qun1,3,YI Hui-lan1,CUI Song-lin2,GUO Jun1
(1.School of Life Science,Shanxi University,Taiyuan 030006,China;2.Fisheries Research Institute of Shanxi Province,Taiyuan 030006, China;3.China Institute for Radiation Protection,Taiyuan 030006,China)
Abstract:Partial sequences of Cyt b and ATPase 6 genes in mtDNA were sequenced in two wild individuals of Chinese giant salamander Andrias davidianus from Lishan,Shanxi and compared with 14 samples collected from other areas in China(from GenBank).The analysis of Mega 5.0 showed that there were 14 variable loci in the 305 bp Cyt b fragment,while there were 30 variable loci in the 371 bp ATPase 6 fragment.Nucleotide variation in the two sequences indicates that the Cyt b was found to be more conserved than ATPase 6.However,both sequences were shown to be lack guanine(<18.9%)in base composition and that the variations were mostly observed at the third site of the codon.The phylogenetic trees based on the combined sequences of Cyt b and ATPase 6 revealed that Lishan population was clustered into a group with Shaanxi and Sichuan populations.Lishan giant salamander population is found to be special in the corresponding fragments of mtDNA,which is genetically different from Guangxi population and closer to Sichuan and Shaanxi populations.
Key words:Andrias davidianus;Lishan region;Cyt b;ATPase 6;mtDNA
中图分类号:Q346
文献标志码:A
文章编号:2095-1388(2012)06-0513-05
收稿日期:2012-04-02
基金项目:山西省高校高新技术产业化项目 (20080002)
作者简介:黄立群 (1986-),男,硕士研究生。E-mail:huangliqun369@163.com
通信作者:仪慧兰 (1963-),女,博士生导师,教授。E-mail:yihuilan@yahoo.com.cn



