双齿围沙蚕Hsp70 cDNA基因的克隆及序列分析
李颖,周一兵,万良,赵欢,杨大佐
(大连海洋大学辽宁省海洋生物资源恢复与生境修复重点实验室,辽宁大连116023)
摘要:根据已知巨型管状虫Riftia pachyptila热休克蛋白70(Hsp70)基因序列设计引物,应用同源克隆策略和RACE技术首次从双齿围沙蚕Perinereis aibuhitensis体壁肌肉中克隆获得Hsp70 cDNA序列。结果表明:双齿围沙蚕Hsp70 cDNA序列全长为2 336 bp,包括5′端非翻译区88 bp、3′端非翻译区286 bp和开放阅读框1 962 bp;整个开放阅读框编码653个氨基酸,包含Hsp70家族3个特征区域;通过生物信息学分析表明,该蛋白是一种无信号肽跨膜蛋白,理论等电点为5.14,相对分子质量为71 396,半胱氨酸 (Cys)含量为0.8%;同源性分析表明,双齿围沙蚕Hsp70氨基酸序列与其他真核生物的Hsp70序列具有很高的相似性。该研究结果可为进一步开发沙蚕Hsp70作为监测海洋污染的一种生物标记物提供重要的参考依据。
关键词:双齿围沙蚕;热休克蛋白70;基因分析
热休克蛋白70(Hsp70)由于其具有多方面的生物学功能成为被广泛研究的一组蛋白[1-2]。Hsp70作为蛋白成熟过程中的分子伴侣,能与变性蛋白质结合,修复错误折叠蛋白或加速其降解,参与应激条件下细胞必需蛋白质空间构象的维持,在对抗包括高温、低氧、缺血、紫外线、重金属离子、DNA损伤、环境毒物等环境胁迫引起的损伤中发挥重要作用[3-4]。同时,热休克蛋白还具有抗细胞凋亡、促进细胞增殖、抗氧化、使生物获得耐热性、稳定细胞膜结构、免疫等生物学作用。正常条件下机体Hsp70表达量较少,外界环境胁迫下机体Hsp70表达量会明显升高。鉴于Hsp70与环境胁迫引起的机体损伤之间的关系,Hsp70作为生物标记物用于环境监测受到广泛关注。沈骅等[5-7]探讨了鲫Carassius auratus肝脏组织Hsp70作为重金属污染条件下生物标记物的潜力。对Hsp70的初步研究结果显示,Hsp70作为生物标记物在水环境检测方面具有一定潜力。Radlowska等[8]研究表明,高浓度重金属污染可导致紫贻贝Mytilus edulis体内Hsp70表达水平升高,且其表达量在铜、镉联合作用下变化更加显著。另有研究表明,海胆、贝类和多种鱼类体内Hsp70表达量会随着温度变化发生显著变化[9-12]。目前,已对多种海洋生物Hsp70基因序列和蛋白功能进行了研究,然而对双齿围沙蚕Perinereis aibuhitensis Hsp70基因的研究迄今尚未见报道。
双齿围沙蚕是广泛分布于中国沿海潮间带及河口潮滩的优势底栖动物,隶属于环节动物门、多毛纲,是重要的经济多毛类。由于其底栖生活习性,双齿围沙蚕受环境污染物影响的可能性增大,其对污染物的耐受性亦随之增强。研究发现,沙蚕对多种环境污染物如重金属、多环芳烃等有较强的富集能力和污染耐受性[13]。深入开展沙蚕热休克蛋白研究,可从分子生物学水平探索其在沙蚕生理过程中的作用,为进一步分析重金属胁迫条件下该基因对细胞和机体的保护作用,以及建立以沙蚕为模式生物的海洋污染沉积质生物修复技术提供重要的参考依据。
本研究中,作者采用同源克隆及RACE技术首次从双齿围沙蚕体壁肌肉中克隆得到Hsp70基因,并进行了基因结构分析,同时构建系统发生树,将双齿围沙蚕Hsp70基因序列和其他已知Hsp70基因序列进行了聚类分析。
1 材料与方法
1.1 材料
试验用双齿围沙蚕采自辽宁省盘锦市双台子河口,选取体质量为1.5~2.0 g的个体运回到大连海洋大学辽宁省海洋生物资源恢复与生境修复重点实验室,于18℃水中暂养一周,每24 h更换海水1次。试验用海水取自大连市黑石礁海区,经沉淀、沙滤后贮存备用。试验海水的Cu(Ⅱ)、Hg(Ⅱ)、Zn(Ⅱ)、Pb(Ⅱ)和Cd(Ⅱ)的背景浓度分别为0.38、0.01、75.00、0.27、0.06 μg/L,盐度为31~32,pH为 8.25±0.10,温度为 (18± 0.5)℃。
RNAisoTMPlus、RNA PCR Kit(AMV)Ver.3.0、LA Taq®with GC Buffer、5′-Full RACE Kit、Agarose Gel DNA Purification Kit Ver.2.0、DNA Ligaton Kit Ver.2.0(Code No.D6022)均购自TAKARA公司;EZ Spin Column DNA Gel Extraction Kit购自上海生工生物工程技术服务有限公司;其它试剂均为国产分析纯。
1.2 方法
1.2.1 总RNA的提取研钵使用液氮预冷,取0.1 g双齿围沙蚕体壁肌肉,迅速放入预冷研钵中,加入适量液氮将组织研磨成粉末状,再按照RNAisoTMPlus试剂盒说明书提取总 RNA,最终溶于DEPC处理水中。经10 g/L琼脂糖凝胶电泳检验总RNA的完整性,并使用紫外分光光度计测定其浓度[14-16]。将提取的总RNA于-80℃中保存备用。
1.2.2 引物设计 根据近源物种巨型管状虫Riftia pachyptila部分基因热休克蛋白 Hsp70 CDS (GenBank登录号为FN860145)寻找保守区域设计简并引物 (表1)。以双齿围沙蚕总RNA为模板,反转录扩增出特异性片断,并对其进行序列测定和比对。根据得到的Hsp70基因片段设计3′RACE和5′RACE引物。
1.2.3 第一链合成及特异性片段扩增 取总RNA,按照RNA PCR Kit(AMV)Ver.3.0试剂盒说明书合成cDNA及PCR反应。cDNA合成反应条件:30℃下反应10 min,42℃下反应60 min,70℃下反应15 min。同源克隆反应条件:94℃下预变性3 min;94℃下变性30 s,55℃下退火30 s, 72℃下延伸2 min,共进行30个循环;最后在72℃下延伸10 min。
表1 双齿围沙蚕Hsp70基因全长扩增引物设计
Tab.1 Relative primers of Hsp70 gene cloning in Perinereis aibuhitensis

引物primer序列sequence CTD553-F 5′CAAGCGCAAGWWCAAGAARGAC 3′CTD553-R 5′CGGCTGRTTGTCYGAGTAKGT 3′CTD553 R173 5′CAAAACGAGCCCTGGTGATGG-3′CTD553 R86 5′GGGTCCTCTTCGCACGCTCACA 3′CTD553 R400 5′CAATGCTTTTTGTCTGACTGAA 3′CTD553 F437 5′TTGCTTTTGCTTGATGTGACCC 3′CTD553 F534 5′AGCAGACGCAGACCTTCACAACATAC 3′CTD553 F174 5′CAAACGACCAGGGTAACAGGA 3′
取5 μL PCR产物,用10 g/L琼脂糖凝胶电泳检验,再用EZ Spin Column DNA Gel Extraction Kit切胶回收试剂盒回收。回收后的产物用DNA Ligaton Kit Ver.2.0(Code No.D6022)载体连接并进行克隆,然后热转化至 E.coli Competent Cells JM109中,涂布于平板上,于37℃下过夜培养,挑选阳性菌斑加至LB液体培养基 (含Amp)中培养16 h,送宝生物工程 (大连)有限公司进行测序。
1.2.4 RACE扩增 使用引物CTD553 F/R进行5′RACE和3′RACE扩增,引物信息详见表1。
1)5′RACE PCR扩增
按照LA Taq®with GC Buffer和5′-Full RACE Kit试剂盒说明书对总RNA进行去磷酸化处理和“去帽子”反应,并与5′RACE Adaptor连接后,反转录合成5′RACE的cDNA。采用巢式PCR方法扩增沙蚕Hsp70 5′端序列,设立M-MLV(-)阴性对照。Outer PCR反应使用引物CTD553 R173和5′RACE Outer Primer。Outer PCR反应条件:94℃下预变性3 min;94℃下变性30 s,65℃下退火30 s,72℃下延伸2 min,共进行30个循环;最后在72℃下延伸 10 min。Inner PCR反应使用引物CTD553 R86 Primer和5′RACE Inner Primer。Inner PCR反应条件同Outer PCR。使用Agarose Gel DNA Purification Kit Ver.2.0试剂盒切胶回收,回收后的产物使用 DNA Ligation Kit Ver.2.0中的连接酶,与pMD20-T Vector连接,热转化至E.coli Competent Cells JM109中,涂布于平板上,于37℃下过夜培养,挑选阳性菌落植菌并测序。
2)3′RACE PCR扩增
使用LA Taq®进行巢式PCR扩增,Outer PCR反应使用引物CTD553 F437 Primer和3′RACE Out-er Primer。Outer PCR反应条件:94℃下预变性3 min;94℃下变性30 s,60℃下退火30 s,72℃下延伸2 min,共进行30个循环;最后在72℃下延伸10 min。Inner PCR反应使用引物CTD553 F534 Primer和3′RACE Inner Primer。Inner PCR反应条件同 Outor PCR。切胶回收及克隆测序方法同5′RACE。序列测定由 TAKARA公司完成,应用DNA Star软件将得到的序列拼接成完整的cDNA序列。
1.2.5 序列分析 用DNA Star软件翻译序列,获得编码序列的氨基酸序列,用Blast对推导的氨基酸序列进行同源性比对;用Clustal W和Mega 4.0进行系统进化分析;用ProtParam和DNA Star预测蛋白的理化性质、等电点和分子质量;用Tmhmm进行跨膜区分析,用SignalP进行信号肽预测,用ExPasy-ProtScale进行疏水性分析。
2 结果与分析
2.1 特异性片段扩增及RACE扩增
经过DNase I纯化处理后的总RNA用10 g/L琼脂糖凝胶电泳进行检测,可清晰地看到5S、18S和28S核糖体 RNA条带 (图 1)。结果显示, OD260nm/OD280nm为2.0左右,RNA有较高的完整性和均一性,无降解现象,符合基因克隆要求。
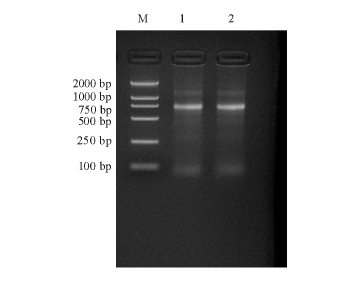
图1 双齿围沙蚕RNA的提取结果
Fig.1 The total RNA from P.aibuhitensis
注:M为DL2000 Marker;1、2为双齿围沙蚕体壁肌肉RNA。
Note:M shows DL2000 Marker;Lane 1 and Lane 2 shows total RNA of P.aibnhitensis.
根据巨型管状虫Hsp70 CDS寻找保守区域设计引物CTD553 F/R,经PCR产物切胶后克隆测序得到574 bp的核苷酸序列 (图2)。通过优化PCR反应条件,3′RACE PCR扩增得到931 bp核苷酸序列 (图3-A),5′RACE PCR克隆获得819 bp核苷酸序列 (图3-B)。经DNA Star软件进行序列拼接,得到全长为 2 336 bp的双齿围沙蚕 Hsp70 cDNA序列,并提交到 GenBank,登录号为HQ449186。
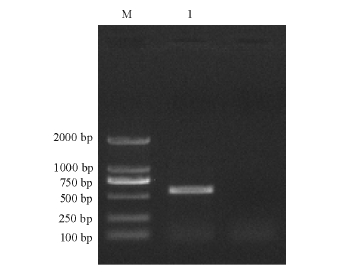
图2 Hsp70特异性片段扩增
Fig.2 PCR amplification product of Hsp70 fragment
注:M为DL2000 Marker;1为Hsp70特异性片段扩增。
Note:M shows DL2000 Marker;Lane 1 shows PCR product of Hsp70 fragment.
2.2 基因序列分析
序列分析显示,双齿围沙蚕Hsp70基因cDNA包括5′非翻译区88 bp、3′非翻译区286 bp和开放阅读框为1 962 bp,整个开放阅读框编码653个氨基酸。预测双齿围沙蚕Hsp70的相对分子质量为71 396,理论等电点为5.14,原子总数为10 036,分子式为C8645H13530N2378O2691S53。对Hsp70氨基酸序列的跨膜区进行分析,结果显示,Hsp70无明显跨膜结构,推测其可能是可溶性蛋白。根据ProtScale预测结果,Hsp70疏水性最大值为1.944,最小值为-3.133,整个多肽链中大多数氨基酸的分值较低,亲水性氨基酸多于疏水性氨基酸,故推测该蛋白可能为亲水性蛋白。氨基酸序列分析表明, Hsp70存在 IDLGTTYS、IFDLGGGTFDVSIL和 IVLVGGSTRIPKIQK 3个特征基序,并具有简并重复四肽GGMP。Hsp70氨基酸具有ATPase活性区,N端此活性区的保守性比C端更高。C末端具有以EEVD为特征的基序。
将双齿围沙蚕Hsp70序列提交到NCBI中,使用在线Blast软件进行同源性比较。结果表明,双齿围沙蚕 Hsp70与褐牙鲆 Paralichthys olivaceus Hsp71同源性为78%,与斑鳟属Salmo salar Hsp70同源性为78%,与斑马鱼Danio rerio Hsp8同源性为78%,与脊尾白虾 Exopalaemon carinicaud同源性为77%,与文昌鱼Branchiostoma Hsp同源性为76%,与小鼠Mus musculus Hsp8同源性为76%,与长牡蛎Crassostrea giga Hsc71同源性为77%,与铃夜蛾属Helicoverpa Hsp70同源性为76%,与中华绒鳌蟹Eriocheir sinensis Hsp70同源性为77%。双齿围沙蚕Hsp70氨基酸序列通过GenBank数据库比较,经Clustal W多重序列比对分析,用Mega 4.1构建不同物种Hsp70基因的系统进化树 (图4)。从图4可见:双齿围沙蚕和庞贝蠕虫Alvienlla pompejana、可口革囊星虫Phascolosoma esculenta的Hsp70亲缘关系最近;稻飞虱Nilaparvata lugens和红火蚁Solenopsis invicta等昆虫聚为一簇;人Homo sapiens、野猪Sus scrofa、非洲象Loxodonta africana、火鸡Meleagris gallopavo、密西西比鳄Alligator mississippiensis、非洲爪蟾Xenopus laevis等脊椎动物为一簇。
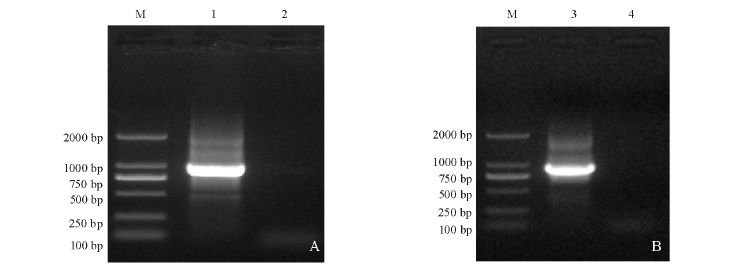
图3 双齿围沙蚕Hsp70 PCR扩增产物电泳图
Fig.3 Electrophoretogram of PCR amplified product of Hsp70 from P.aibuhitensis
注:M为DL2000 Marker;1为3′RACE PCR产物;3为5′RACE PCR产物;2、4为M-MLV(-)对照。
Note:M shows DL2000 Marker;Lane 1 represents PCR product of 3′RACE;Lane 3 shows PCR product of 5′RACE;Lane 2 and Lane 4 M-MLV (-)denote blank control.
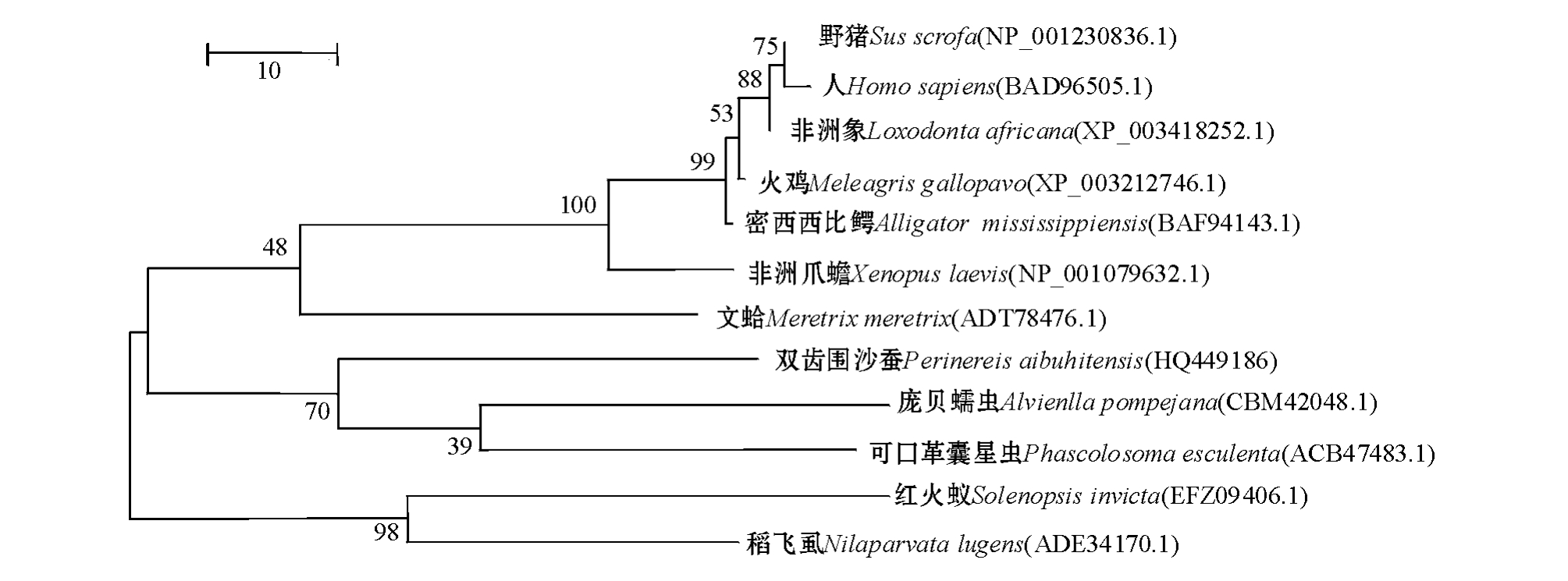
图4 不同物种Hsp70基因的系统进化树
Fig.4 The phylogenetic tree of Hsp70 gene in different species
3 讨论
Hsp70基因在不同生物体内均有分布,具有较高的保守性。Flynn等[17]研究表明,Hsp70氨基酸的一级结构近N端具有相对分子质量为45 000的高度保守结构域,该结构域具有 ATPase活性; Hsp70氨基酸一级结构近C端具有相对分子质量为25 000的底物结合结构域,包括相对分子质量为15 000的p2夹层结构和相对分子质量为10 000的螺旋结构亚区。Hsp70家族具有 IDLGTTYS、IFDLGGGTFDVSIL和IVLVGGSTRIPKIQK 3个特征基序[18-21]以及C末端具有EEVD特征基序。本试验中,克隆获得的双齿围沙蚕Hsp70序列也具有这些特征基序。
Hsp70 C末端的GGMP重复功能目前尚不清楚,相关资料表明,可能是分子伴侣因子的结合位点[22],Piano等[23]认为只有Hsc70才有GGMP四肽重复。但Boutet等[18]发现,食用牡蛎Crassostrea gigas的Hsp70和Hsc70氨基酸序列C末端均含有GGMP重复。目前报道的Hsp70有的含有GGMP四肽重复[18,24],有的则没有[23,25]。 甚至同一物种会出现有或没有两种情况[22,26-27]。对本研究中克隆获得的双齿围沙蚕Hsp70序列结构进行分析发现,在其C末端具有GGMP四肽重复,对此末端重复功能还需要进一步研究。
根据不同物种Hsp70氨基酸序列的比对分析结果,进行系统进化分析。结果显示,稻飞虱和红火蚁等昆虫聚为一簇,人、野猪、非洲象、火鸡、密西西比鳄、非洲爪蟾等脊椎动物为一簇,双齿围沙蚕和庞贝蠕虫、可口革囊星虫的Hsp70亲缘关系最近。这与双齿围沙蚕生物进化情况相一致,可为今后环节动物门的系统学和进化研究提供重要的参考依据。
利用Hsp70作为分子生物标记,是目前海洋生物的一个热点研究领域[28-29]。Kilemade等[30]研究发现,虹鳟鱼体内Hsp70的表达水平与水生环境中2,4-二氯苯胺的含量有关,认为水生生物体内Hsp70的表达水平可以作为水生环境毒性的指标。Veldhuizen-Tsoerkan等[31]将地中海贻贝Mytilus galloprovincialis放在污染水中,结果发现,经水体污染物诱导的贻贝Hsp高度表达。Snyder等[32]观察到贻贝和鲍鱼Haliotis rufescens暴露在高热和化学药品超标的水体中,其体内的Hsp70、Hsp90和Hsp60含量均发生显著变化。由此看出,Hsp70在防御应急中发挥一定的作用。笔者将进一步对其功能进行分析,以期对多毛类耐受环境污染的分子调控机制进行深入研究,为建立海洋沉积质污染早期生态风险测评和评价方法提供重要的参考依据。
参考文献:
[1] Moiimoto R L,Tiieles A.Stress Proteins in Biology and Medicine [M].Cold Soring Harboe:CSHL Press,1990:1-36.
[2] Bond U,Schlesinger M J.Heat shock proteins and development [J].Adv Genet,1987,24(1):1-19.
[3] Lindquist S,Craig E A.The heat shock protein[J].Annu Rev Genet,1988,22:613-677.
[4] 段燕英,杨瑾,牛丕业,等.Hsp70对苯并芘所致DNA损伤修复的影响[J].工业卫生与职业病,2009,35(8):257-260.
[5] 沈骅,王晓蓉,张景飞,等.低浓度Zn对幼龄鲫鱼肝脏组织应激蛋白Hsp70诱导的影响[J].农业环境科学学报,2004,23 (3):441-443.
[6] 沈骅,王晓蓉,张景飞.应用应激蛋白Hsp70作为生物标志物研究锌、铜及其联合毒性对鲫鱼肝脏的影响[J].环境科学学报,2004,24(9):895-899.
[7] 沈骅,王晓蓉,张景飞,等.低浓度Pb2+、Cd2+对鲫鱼肝脏组织中Hsp70诱导的影响[J].环境污染与防治,2004,26(4):244-247.
[8] Radlowska M,Pempkowiak J.Stress-70 as indicator of heavy metals accumulation in blue mussel Mytilus edulis[J].Environ Int, 2002,27(8):605-608.
[9] Hartl F U.Molecular chaperones in protein folding[J].Nature, 1996,381:571-580.
[10] Basu N,Todgham A E,Ackerman P A,et al.Heat shock protein genes and their functional significance in fish[J].Gene,2002,295 (2):173-183.
[11] Snyder M J,Ross S.Stress protein(Hsp70 family)expression in intertidal benthic organisms:the example of Anthopleura elegantissima(Cnidaria:Anthozoa)[J].Sci Mar,2004,68(suppl.1):155-162.
[12] Matranga V,Toia G,Bonaventura R,et al.Cellular and biochemical responses to environmental and experimentally induced stress in sea urchin coelomocytes[J].Cell Stress&Chaperones,2000,5 (2):113-120.
[13] Saiz-Salina J I,France's-Zubillaga G.Enhanced growth in juvenile Nereis diversicolor after its exposure to anaerobic polluted sediments[J].Marine Pollution Bulletin,1997,34:437-442.
[14] Scaps P.A review of the biology,ecology and potential use of the commom ragworm Hediste diversicolor(Annelida:Polychaeta)[J]. Hydrobiologia,2002,470:203-218.
[15] 周一兵,陈雪,杨大佐,等.双齿围沙蚕CYP4基因的克隆及序列分析[J].大连海洋大学学报,2011,26(6):507-513
[16] 郑珂,陈秋实,李霞.仿刺参体壁总RNA提取方法的建立[J].生物技术通讯,2007,18(1):84-87.
[17] Flynn G C,Chappell T G,Rothman J E.Peptide bingding and release by proteins implicated as catalysts of protein assembly[J]. Science,1989,245:385-390.
[18] Boutet I,Taaguy A,Moraga D.Organization and nucleotide sequence of the Europe flat oyster Ostrea edulis heat shock cognate 70(Hsc70)and heat shock protein 70(Hsp70)genes[J].Aquat Toxicol,2003,65:221-225.
[19] Gupta R S,Singh B.Phylogenetic analysis of 70 kD heat shock protein sequences suggests a chimeric origin for the eukaryotic cell nucleus[J].Curr Biol,1994,4:1104-1114.
[20] Boorstein W R,Ziegelhoffer T,Craig E A.Molecular evolution of the Hsp70 multigene family[J].J Mol Evol,1994,38:1-17.
[21] 田照辉,徐绍刚,王巍,等.西伯利亚鲟热休克蛋白HSP70 cDNA的克隆、序列分析和组织分布[J].大连海洋大学学报,2012,27(2):150-157.
[22] Demand J,Luders J,Hohfeld J.The carboxy-terminal domain of Hsc70 provides binding sites for a distinct set of chaperone cofactors[J].Mol Cell Biol,1998,18(4):2023-2028
[23] Piano A,Franzellitti S,Tiniti F,et al.Sequencing and expression pattern of inducible heat shock gene products in the European flat oyster,Ostrea edulis[J].Gene,2005,361:119-126.
[24] Boutet I,Tanguy A,Rousseau S,et al.Molecular identification and expression of Heat Shock Cognate 70(Hsc70)and Heat Shock Protein 70(Hsp70)genes in the Pacific oyster Crassostrea gigas [J].Cell Stress Chap,2003,8:76-85.
[25] RathinamA V,Vhent T,Grossfeld R M,et al.Cloning and sequence analysis of a cDNA for an inducible 70 kDa heat shock protein(Hsp70)of the American oyster(Crassostrea virginica)[J]. DNA Seq,2000,11:261-264.
[26] Kimura M.A simple method for estimating evolutionary rate of base substitution through comparative studies of nucleotide sequence[J].Mol Evol,1980,16:111-120.
[27] Yu D H,Chu K H.Species identity and phylogenetic relationship of the pearl oysters in Pinctada Röding,1798 based on ITS sequence analysis[J].Biochem Syst Ecol,2006,34(3):240-250.
[28] Lyons C,Dowling V,Tedengren M,et al.Variability of heat shock proteins and glutathione S-transferase in gill and digestive gland of blue mussel,Mytilus edulis[J].Mar Environ Res,2003,56(5): 585-597.
[29] Sanders B M,Marrin L S.Stress proteins as biomarkers of contaminant exposure in archived environmental samples[J].Scitotal Environ,1993,193:459-470.
[30] Kilemade M,Mothersill C.Heat shock protein 70 levels in rainbow trout primary epidermal cultures in response to 2,4-dichloroanoline exposure:a novel in vitro aquatic toxicity marker[J].Environ Toxicol,2001,16(3):253-259.
[31] Veldhuizen-Tsoerkan M B,Holwerda D A,Van der Mast C A,et al.Synthesis of stress proteins under normal and heat shock conditions in gill tissue of sea mussels(Mytilus edulis)after chronic exposure to cadmium[J].Comp Biochem Physiol C,1991,100(3): 699-706.
[32] Snyder M J,Girvetz E,Mulder E P.Induction of marine mollusc stress proteins by chemical or physical stress[J].Arch Environ Contam Toxicol,2001,41(1):22-29.
Cloning and analysis of Hsp70 cDNA from Perinereis aibuhitensis (Annelida:Polychaeta)
LI Ying,ZHOU Yi-bing,WAN Liang,ZHAO Huan,YANG Da-zuo
(Key Laboratory of Marine Bio-resources Restoration and Habitat Reparation in Liaoning Province,Dalian Ocean University,Dalian 116023,China)
Abstract:In present study,the partial cDNA of Hsp70 was amplified from the total RNA of epidermis muscle in polychaete,Perinereis aibuhitensis by homology cloning strategy and RACE technology,using specific primers based on the reported Riftia pachyptila Hsp70 gene sequence.Sequence analysis revealed that there was a 2 336 bp cDNA containing a 88 bp 5′-untranslated region,a 286 bp 3′-untranslated region and 1 962 bp open reading frame encoding 653 amino acids.Three conserved motifs of hsp70 family were found in this sequence.The bioinformatic analysis indicated that the deduced amino acid is a non-transmembrane protein with no signal peptide,with isoelectric point of 5.14 and molecular weight of 71 396 and 0.8%of cysteine(Cys)content.The sequence similarity analysis revealed that the deduced amino acid had high homology to other organisms.The findings will provide insight for identifying hsp70 as biomarker of environmental pollution.
Key words:Perinereis aibuhitensis;Hsp70;gene analysis
中图分类号:Q78
文献标志码:A
文章编号:2095-1388(2012)06-0502-06
收稿日期:2012-04-10
基金项目:国家 “863”计划项目 (2006AA10Z410);国家海洋公益性行业科研专项 (200805069)
作者简介:李颖(1986-),女,硕士研究生。E-mail:lyyfx326@163.com
通信作者:周一兵 (1957-),男,教授。E-mail:ybzhou@dlou.edu.cn




