虾夷马粪海胆硬脂酰辅酶A去饱和酶和蛋白酪氨酸磷酸酶基因的克隆与表达
丁君,孙巍,常亚青
(大连海洋大学农业部北方海水增养殖重点实验室,辽宁大连116023)
摘要:采用同源克隆方法,结合cDNA末端快速扩增 (RACE)技术,成功克隆了虾夷马粪海胆Strongylocentrotus intermedius硬脂酰辅酶A去饱和酶 (Stearoyl-coenzyme A desaturase,SCD)基因和蛋白酪氨酸磷酸酶 (Protein tyrosine phosphatase 1B,PTP1B)基因,并对其结构进行了分析,同时采用实时定量PCR方法对这两个基因在海胆胚胎不同发育时期的表达进行了研究。结果表明:SCD基因的cDNA序列全长为1 934 bp(Genbank登录号为HM208174),开放阅读框819 bp,编码273个氨基酸残基,存在跨膜结构和信号肽,为分泌性蛋白;实时定量PCR检测显示,SCD基因在虾夷马粪海胆八腕期表达量最高,在2细胞时期表达量最低。PTP1B基因的cDNA序列全长为1 249 bp(Genbank登录号为HM208173),开放阅读框657 bp,编码219个氨基酸残基,存在跨膜结构和信号肽,为分泌性蛋白;PTP1B基因在海胆2细胞时期表达量最高,在八腕期表达量最低。本研究为在分子水平上探讨海胆脂代谢过程中的基因功能提供了科学依据。
关键词:虾夷马粪海胆;硬脂酰辅酶A去饱和酶;蛋白酪氨酸磷酸酶;实时定量PCR
海胆隶属于棘皮动物门Echinodermata、海胆纲Echinoidea,是一种营养、药用和科研价值很高的海洋动物。海胆是世界范围内优良的海水养殖品种之一,在亚洲,日本、中国、韩国是海胆的主产区。1989年,大连海洋大学首次将虾夷马粪海胆Strongylocentrotus intermedius引入中国,现已成为中国北方重要海水养殖品种之一。海胆的营养价值主要体现在其性腺 (俗称海胆黄)中脂肪酸的食用、药用功效方面,海胆性腺中脂肪酸的组成、含量与其繁育生理密切相关,不同性别、不同发育时期的海胆,其性腺中脂肪酸的组成、含量明显不同[1]。海胆性腺不但是性腺营养储备场所和能量来源,同时还是其繁殖器官,海胆脂肪酸积累是否充足也直接影响其配子的质量。因此,以虾夷马粪海胆作为研究对象,进行海胆性腺脂肪酸代谢相关基因的研究,对提高海胆品质,增加其营养、药用功效,培育出繁育性状优良的海胆具有理论意义和实际意义。
硬脂酰辅酶A去饱和酶 (Stearoyl-coenzyme A desaturase,SCD)基因是调控饱和脂肪酸向单一不饱和脂肪酸转化的关键酶,在脂肪酸生物合成中起着中心调控作用[2],可通过信号传导调控动物的采食量、能量代谢和机体内分泌等生理活动。SCD作为脂肪细胞分化的标记基因,其活性的高低会影响动物脂肪细胞的增殖和增长[3]。目前,有研究者 已 对 人[4-5]、 鸡[6]、 猪[7-8]、 绵 羊[9]、 山羊[10-11]、 牛[12]、 小鼠[13-14]和大鼠[15]等的 SCD 基因进行了克隆和测序,但对无脊椎动物SCD基因的研究较少。
蛋白酪氨酸磷酸酶 (Protein tyrosine phosphatase 1B,PTP1B)基因是一种广泛表达于细胞质并锚定在内质网上的磷酸酶,它是蛋白酪氨酸磷酸酶(PTP)胞内酶的代表[16]。PTP1B基因没有特异性受体,通过调节细胞内不同蛋白底物的酪氨酸磷酸化水平而参与各种细胞功能的调节。现已证实, PTP1B基因可与胰岛素受体、表皮生长因子等某些受体型酪氨酸激酶及多种生长因子、底物互相作用,以调控细胞的生长分化[17]。
本研究中,作者查阅了KEGG数据库中海胆脂肪酸代谢通径,选取并克隆了SCD和PTP1B基因,检测了SCD和PTP1B基因在海胆胚胎不同发育时期的表达情况,旨在为从分子水平上探讨海胆脂代谢过程中的基因功能提供科学的依据。
1 材料与方法
1.1 材料
试验用虾夷马粪海胆为农业部北方海水增养殖重点实验室培育的2龄海胆。
Trizol Reagent、RNA PCR Kit(AMV)3.0、载体pMD19-T Vector均为宝生物工程 (大连)有限公司产品;异丙醇、分析乙醇、RnaseA、DEPC、MOPS、氨苄青霉素钠盐均为上海生工生物工程技术服务有限公司产品。SMARTTMRACE cDNA Amplification Kit为美国 Clontech公司产品;Gel Extraction Kit和 Plasmid Miniprep Kit购自 Omega Biotek公司;限制性内切酶购自 Promega公司; Tryptone、YeastExtract购自Oxoid公司;其它试剂均为国产分析纯试剂。Taq DNA聚合酶聚核苷酸引物由宝生物工程 (大连)有限公司合成;大肠杆菌表达宿主为农业部北方海水增养殖重点实验室保存。
1.2 方法
1.2.1 设计与筛选 通过与加州紫海胆和马粪海胆SCD、PTP1B基因序列的比对,找出保守序列,设计扩增片段引物SCD-F1、SCD-R1、PTP1BF1、PTP1B-R1(表1)。
1.2.2 胚胎不同发育时期虾夷马粪海胆总RNA的提取、cDNA的合成及目的基因的PCR扩增 分别取发育至2细胞期、囊胚期、原肠期和八腕期的虾夷马粪海胆胚胎、幼虫样品各 100 μL,提取总RNA,经琼脂糖电泳检测总RNA质量。
使用TAKARA公司的cDNA Library Construction Kit试剂盒,利用反转录酶 M-MLV、Oligo (dT)18 Anchor Primer和5-methyldCTP合成第一链cDNA(含有NotⅠ酶切位点)。
以cDNA为模板,根据所设计的上、下游引物进行PCR扩增,反应条件为:94℃下预变性5 min;94℃下变性1 min,57℃下退火1 min,72℃下延伸1 min,共进行30个循环;最后在72℃下保温10 min。PCR产物使用10 g/L的琼脂糖电泳检测,分离纯化的目的基因片段连接到pMD19-T载体上,转化至大肠肝菌DH5感受态细胞中,挑选阳性克隆进行测序。
1.2.3 采用RACE PCR技术获得全长序列 采用cDNA末端快速扩增技术(Rapid Amplification of cDNA Ends,RACE),根据已经获得的目的基因部分片段设计基因特异性引物进行5′cDNA和3′cDNA末端克隆。5′cDNA和3′cDNA末端克隆按照SMARTTMRACE cDNA Amplification Kit说明书进行操作, PCR产物使用10 g/L琼脂糖电泳检测,分离纯化的目的基因片段连接到pMD19-T载体上,转化至大肠肝菌DH5感受态细胞中,挑选阳性克隆进行测序,将测序后的序列与以获得的目的片段进行拼接,获得cDNA全长。所用的特异性引物见表1。
表1 克隆SCD、PTP1B基因的主要引物
Tab.1 The cloning primers of SCD and PTP1B genes

引物primer 序列sequence(5′-3′) SCD(F1)GAATACACCATACCCGGCAG SCD(R1) TGGATCTTCCATAAGGTCGG PTP1B(F2) AAAATTCAACCAAACCAGCC PTP1B(R2) CGCCTCCTCCAATCAATTT 5′SCD(R2) GGAATGACCAGAACCTTAGTGACG 3′SCD(F2) AAGCAGTGGTATCAACGCAGAGT 5′PTP1B(R2) CGCCTCCTCCAATCAATTT 3′PTP1B(F2) GTAAGATACAGAAACGGGACGAG O-SCD(F3) CCGATCCACACAATGCACTAC Q-SCD(R3) TTCTTTCCACTTTCACAGACATCC Q-PTP1B(F3) GTCTCTATGATGCCATTGAACTGCC Q-PTP1B(R3) CGCCGTCAGATACAGAATAATGCTC β-actin(F) AGAGGCGTAGAGGGAAAGCAC β-actin(R)ACAGGGAAAAGATGGCACAGA
1.2.4 目的基因的生物信息学分析 测序结果用VecScreen(http://www.ncbi. nlm.nih.gov/Vec-Screen/VecScreen.html)去除载体序列,用bl2seq (http://www.ncbi.nlm.nih.gov/blast/bl2seq/wblast2.cgi)进行比对拼接,得到各基因的全长cDNA序列;用 ExPasy软件包中的 Compute pI/Mw tool (http://cn.expasy.org/tools/pi_tool.html.) 预测等电点和分子质量,用 the program SignalP(http://www.cbs.dtu.dk/services/SignalP/) 寻找信号肽序列;用Mega 4.0软件中的邻接法构建系统进化树。
1.2.5 用实时定量PCR法检测目的基因的表达采用ΔΔCT法检测胚胎不同发育时期海胆SCD、PTP1B基因的表达,实时荧光定量PCR反应体系以cDNA第一链为模板,以β-actin作为内参基因, β-actin引物和目的基因定量引物经PCR扩增后电泳检测无引物二聚体,且每对定量引物扩增出单一的含有已知序列的片段,再经预试验对引物和模板浓度进行优化,反应条件如下:94℃下预变性30 s;94℃下变性5 s,60℃下退火32 s,共进行30个循环;最后在95℃下变性15 s,60℃下退火1 min,95℃下延伸15 s。
1.3 数据处理
实时荧光定量PCR结果由ABI step one Software系统进行分析,以β-肌动蛋白表达量校正目的基因的相对表达量。采用SPSS 16.0软件进行单因素方差分析、标本重复性及组间差异的比较。
2 结果
2.1 总RNA的提取
经10 g/L琼脂糖凝胶电泳检测海胆胚胎的总RNA,可见清晰的28S、18S和5S 3条RNA条带。表明总RNA没有降解,提取的RNA质量较高。
2.2 虾夷马粪海胆SCD和PTP1B基因片段的PCR扩增
以总RNA逆转录得到的cDNA为模板,用设计的引物进行PCR扩增,获得与预期大小一致的片段。将目的片段切胶回收、连接质粒后测序,得到440、630 bp两条序列片段。
2.3 RACE-PCR扩增
以总RNA逆转录得到的5′RACE 1 st cDNA为模板,用设计的引物进行5′RACE扩增,获得了与预期大小一致的424 bp的5′SCD片段和433 bp的 5′PTP1B片段。
以总RNA逆转录得到的3′RACE 1 st cDNA为模板,用设计的引物进行3′RACE扩增,获得了与预期大小一致的962 bp的3′SCD片段和488 bp的3′PTP1B片段。
2.4 虾夷马粪海胆SCD基因的序列分析
将用5′RACE、3′RACE引物获得的SCD基因片段和用引物调取的SCD中间片段进行拼接,得到全长为1 934 bp的SCD基因cDNA序列 (Genbank登录号为HM208174),该序列包括153 bp的5′非编码区 (Untranslated region,UTR)、819 bp的开放阅读框 (Open reading frame,ORF)和962 bp的3′非编码区。推测的SCD基因cDNA多聚腺苷酸加尾信号 (Polyadenylation signal)为AATAA,开放阅读框编码273个氨基酸残基,该多肽的理论相对分子质量为31 600,等电点为8.43。
使用SingaIP软件分析虾夷马粪海胆SCD基因的氨基酸序列,表明SCD基因存在跨膜结构和信号肽,为分泌性蛋白。用Predict Protein软件分析推测的SCD基因氨基酸序列含有以下位点:2个N-糖基化位点,1个蛋白酶C磷酸化位点,3个酪蛋白激酶Ⅱ磷酸化位点,3个N-肉逗蔻酰化位点。多序列比对发现,不同物种的SCD基因中间部分序列保守性较高,序列两端的差异性较大。
2.5 虾夷马粪海胆SCD基因的进化分析
采用Neibor-Joining方法构建不同物种SCD基因的系统进化树。从图1可见:虾夷马粪海胆与紫球海胆首先聚到一起,进化关系最近;鱼类和哺乳动物也各自聚为一类。海胆在进化地位上位于脊椎动物和昆虫之间。

图1 不同物种SCD基因的系统进化树
Fig.1 The phylogenetic tree of SCD gene in different species
2.6 虾夷马粪海胆SCD基因在胚胎不同发育时期的表达
利用实时定量 PCR方法分析虾夷马粪海胆SCD基因在胚胎不同发育时期的表达。从图2可见:随着海胆胚胎的发育,SCD基因表达量呈逐渐升高趋势。统计学分析结果显示:SCD基因在八腕期的表达量显著高于其他3个时期 (P<0.01),在2细胞期与囊胚期的表达量无统计学差异 (P>0.05)。
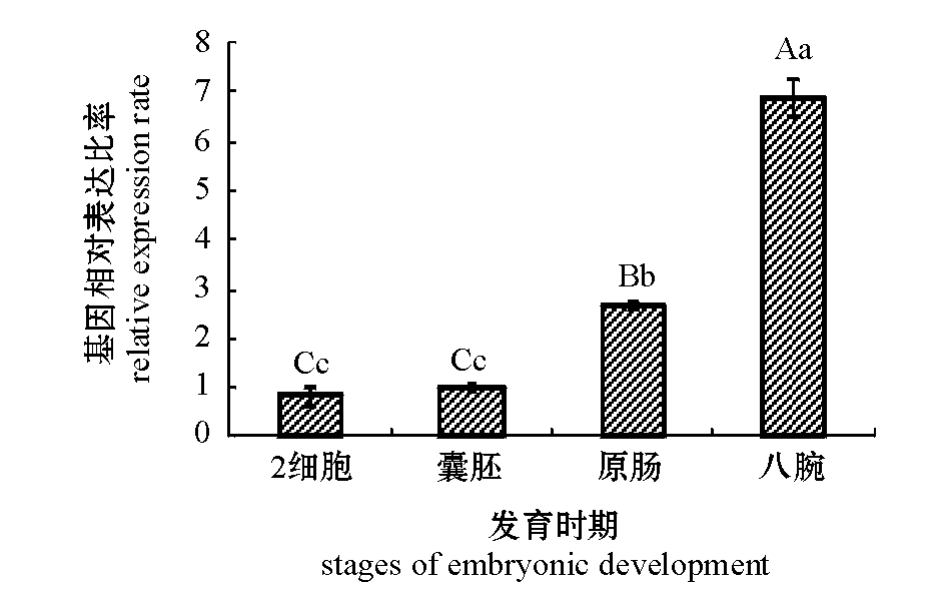
图2 SCD基因在胚胎不同发育时期的表达情况
Fig.2 The expression of SCD gene at different stages
of embryonic development
注:标有不同大写字母者表示组间有极显著性差异 (P<0.01),标有不同小写字母者表示组间有显著性差异 (P<0.05),标有相同小写字母者表示组间无显著性差异 (P>0.05),下同。
Note:The means with different capital letters are very significantly different at the 0.01 probability level,with different letters being significantly different at the 0.05 probability level,and the means with the same letters are not significant differences,et sequentia.
2.7 虾夷马粪海胆PTP1B基因的序列分析
将用5′RACE、3′RACE引物获得的PTP1B基因片段和用引物调取的PTP1B的中间片段进行拼接,得到全长为1 249 bp的PTP1B基因cDNA序列 (Genbank登录号为HM208173),该序列包括104 bp的5′非编码区、657 bp的开放阅读框和488 bp的3′非编码区。推测的PTP1B基因cDNA多聚腺苷酸加尾信号为AATAA,开放阅读框编码219个氨基酸残基,该多肽的理论相对分子质量为25 200,等电点为8.37。
用SingaIP软件分析虾夷马粪海胆PTP1B基因的氨基酸序列,表明PTP1B存在跨膜结构和信号肽,为分泌性蛋白。用Predict Protein软件分析推测的PTP1B基因氨基酸序列含有以下位点:1个N -糖基化位点,1个蛋白酶C磷酸化位点,3个酪蛋白激酶Ⅱ磷酸化位点,3个N-肉逗蔻酰化位点, 1个酪氨酸蛋白磷酸酶的活性位点。推测的PTP1B氨基酸序列含有两个半胱氨酸 (Cys4和Cys58),可形成二硫键。多序列比对发现,不同物种的PTP1B基因保守性较高。
2.8 虾夷马粪海胆PTP1B基因的进化分析
采用Neibor-Joining方法构建不同物种PTP1B基因的系统进化树。从图3可见,虾夷马粪海胆与紫球海胆先聚为一类,进化关系最近;哺乳动物聚为一类;海胆虽为后口动物,但进化地位仍低于鱼类和两栖类。
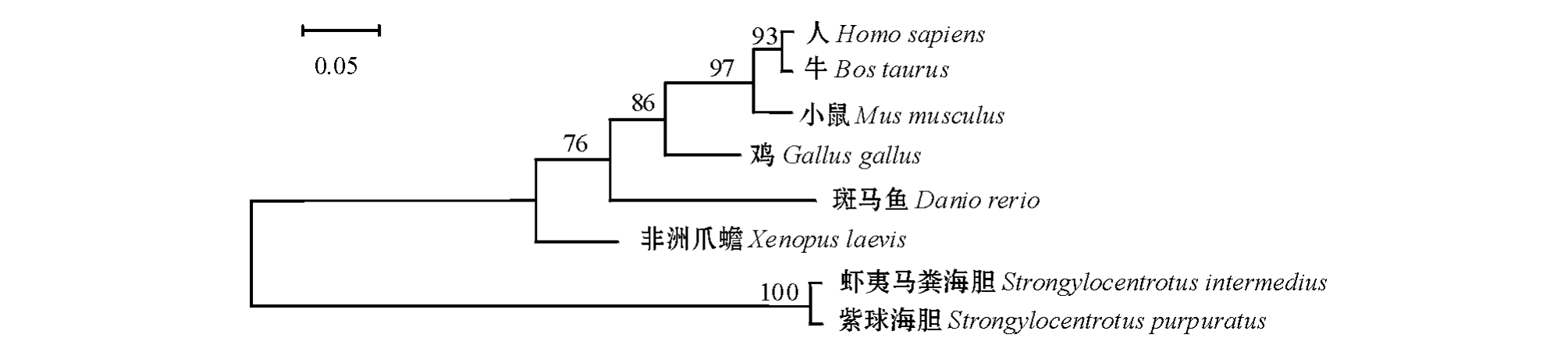
图3 不同物种PTP1B基因的系统进化树
Fig.3 The phylogenetic tree of PTP1B gene in different species
2.9 虾夷马粪海胆PTP1B基因在胚胎不同发育时期的表达
利用实时定量 PCR方法分析虾夷马粪海胆PTP1B基因在胚胎不同发育时期的表达。从图4可见,PTP1B基因表达量随着海胆胚胎发育呈逐渐下降趋势。统计学分析结果显示:PTP1B基因在2细胞期表达量显著高于其他3个时期 (P<0.01),在原肠期与八腕期的表达量无统计学差异 (P>0.05)。
3 讨论
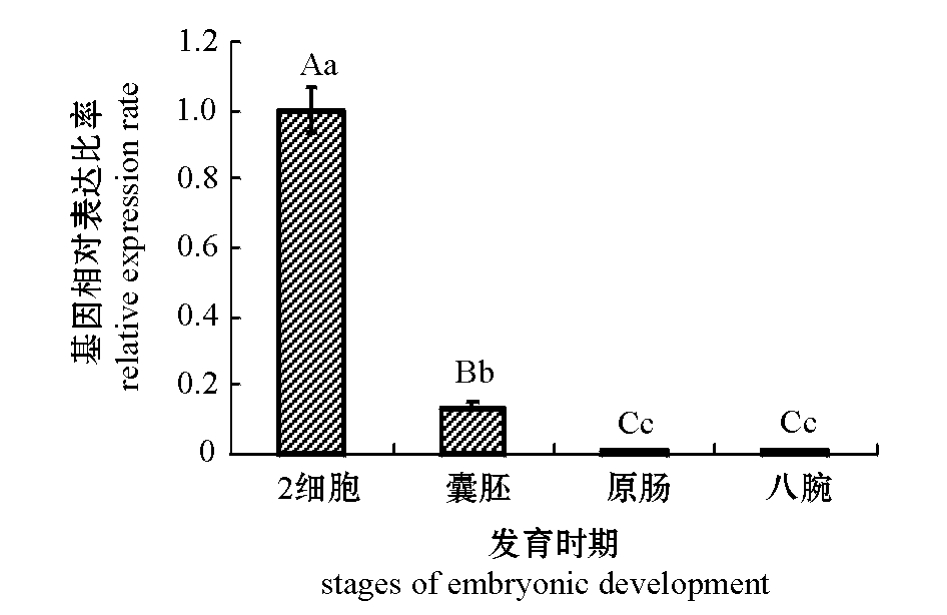
图4 PTP1B基因在胚胎不同发育时期的表达情况
Fig.4 The expression of PTP1B gene at different stages of embryonic development
有关SCD基因克隆和测序多用于对哺乳动物和植物的研究,而对无脊椎动物的研究较少。本研究中成功克隆了虾夷马粪海胆SCD基因,推测SCD全长有273个氨基酸,存在信号肽,推测的SCD基因氨基酸序列有2个N-糖基化位点,1个蛋白激酶C磷酸化位点,3个酪蛋白激酶Ⅱ磷酸化位点,3个N-肉豆蔻酰化位点。通常基因中的功能位点与蛋白质翻译后的修饰、加工有关,其中N-糖基化位点能使蛋白质抵抗消化酶的作用,并且某些蛋白只有在糖基化之后才能正确折叠,具有传导信号的功能[18];蛋白激酶C磷酸化位点与介导胞外分泌有关[19];酪蛋白激酶Ⅱ磷酸化位点与调节细胞凋亡和细胞周期有关[20],N-肉豆蔻酰化位点可能定位于内质网或线粒体外膜[21],表明其为分泌性蛋白,参与介导胞外分泌。研究发现,虾夷马粪海胆SCD基因含有3个跨膜区域,这与现有报道的SCD基因均存在4个跨膜区域不同,其原因和效应还需进一步探讨。SCD基因在不同物种中的序列保守性较高,本研究中通过比对虾夷马粪海胆与其他物种的SCD基因序列,发现SOD基因中部序列相对保守,C端和N端则缺乏同源性,这一结论与Man等[22]的研究结果一致。
分析不同物种SCD基因的系统进化树可知,虾夷马粪海胆和紫球海胆进化关系最近,海胆在进化地位上位于脊椎动物和昆虫中间。因此,根据不同物种SCD基因序列构建的系统进化树能较真实地体现虾夷马粪海胆SCD基因的分类地位。
推测的虾夷马粪海胆 PTP1B全长有219个氨基酸,存在1个N-糖基化位点,1个蛋白酶C磷酸化位点,3个酪蛋白激酶Ⅱ磷酸化位点,3个N-肉豆蔻酰化位点,1个酪氨酸蛋白磷酸酶活性位点,酪氨酸激酶位点与调控细胞增殖和分化、细胞周期以及从细胞外环境传递各种信号有关。实时定量PCR结果显示,PTP1B基因表达量随着海胆胚胎的发育呈逐渐下降趋势。推测其与生长因子及底物互相作用,在胚胎发育的较早阶段调控细胞的生长分化。
虾夷马粪海胆SCD、PTP1B基因的成功克隆及其在胚胎不同发育期的表达研究,对于深入探讨棘皮动物脂肪酸代谢相关基因在胚胎不同发育时期的作用机制及调控机理具有重要意义,进而在分子水平上为虾夷马粪海胆的良种培育提供科学依据。
参考文献:
[1] Russell M P.Resource allocation plasticity in sea urchins:rapid, diet induced,phenotypic changes in the green sea urchin,Strongylocentrotus droebachiensis[J].Journal of Experimental Marine Biology and Ecology,1998,200:12-14.
[2] Sampath H,Miyazaki M,Dobrzyn A,et al.Stearoyl-CoA desaturase -1 mediates the pro-lipogenic effects of dietary saturated fat[J]. Journal of Biological Chemistry,2007,282(4):2483-2493.
[3] 尚秀国,朱晓萍,李藏兰.日粮脂肪酸组成对硬脂酰辅酶A去饱和酶的调控[J].中国畜牧杂志,2009,45(1):54-56.
[4] Wang J,Yu L,Schmidt R E,et al.Characterization of HSCD5,a novel human stearoyl-CoA desaturase unique to primates[J].Biochemical and Biophysical Research Communications,2005,332 (3):735-742.
[5] Campbell E M,Gallagher D S,Davis S K,et al.Rapid communication:mapping of the bovine stearoyl-CoA desaturase(SCD)gene to BTA26[J].Journal of Animal Science,2001,79:1945-1955.
[6] Fillon V,Langlois P,Douaire M,et al.Assignment of stearoyl Coenzyme A Desaturase gene(SCD1)to chicken chromosome R-band 6q14 by in situ hybridization[J].Cytogenet Cell Genet, 1997,78:229-230.
[7] Ren J,Knorr C,Habermann F A,et al.Assignment of the porcine stearoyl-CoA desaturase(SCD)gene to SSC14q27 by fluorescence in situ hybridization and by hybrid panel mapping[J].Anim Genet,2003,34:471-473.
[8] Ren J,Christoph K,Huangl S,et al.Isolation and molecular characterization of the porcine stearoyl-CoA desaturase gene[J]. Gene,2004,340:19-30.
[9] Vaziri H,Dessain S K,Ngcaton E,et al.hSIR2(SIRT1)functions as an NAD-dependent p53 deacetylase[J].Cell,2001,107:149-159.
[10] Bernard L,Leroux C,Hayes H,et al.Characterization of the caprine stearoyl-CoA desaturase gene and its mRNA showing an unusually long 3′-UTR sequence arising from a single exon[J]. Gene,2001,281:53-61.
[11] Yahyaoui M H,Sanchez A,Folch J M.Partial nucleotide sequence of the goat stearoyl coenzyme A desaturase cDNA and gene structure[J].Anim Sci,2002,80:866-867.
[12] Jiang Z,Michal J J,Tobey D J,et al.Significant associations of stearoyl-CoA desaturase(SCD1)gene with fat deposition andcomposition in skeletal muscle[J].International Journal of Biological Sciences,2008,4(6):345-351.
[13] Ntambi J M,Buhrow S A,Kaestner K H,et al.Differentiation-induced gene expression in 3T3-L1 preadipocytes:characterization of differentially expressed gene encoding stearoyl-CoA desaturase [J].Biol Chem,1988,263:17291-17300.
[14] Kaestner K H,Ntambi J M,Kelly T J,et al.Differentiation-induced gene expression in 3T3-L1 preadipocytes:a second differentially expressed gene encoding stearoyl-CoA desaturase[J].Biol Chem,1989,264:14755-14761.
[15] Mihara K.Structure and regulation of rat liver microsomal stearoyl -CoA desaturase gene[J].Biochem,1990,108:1022-1029.
[16] 马宁,刘素筠,冯艳凌.蛋白酪氨酸磷酸酶1B与细胞信号转导[J].国际内分泌代谢杂志,2008,28:10-13.
[17] Winter C L,Lange J S,Davis M C,et al.A nonspecific phosphotyrosine phosphatase inhibitor,bis(maltolato)oxovanadium(Ⅳ), improves glucose tolerance and prevents diabetes in Zucker diabetic fatty rat[J].Exp Biol Med(Maywood),2005,230:207-216.
[18] Christensen L L,Jensen U B,Bross P,et al.The C-terminal N-glycosylation sites of the human 1,3/4-fucosyltransferaseⅢ,-Ⅴ, and-Ⅵ are necessary for the expression of full enzyme activity [J].Glycobiology,2000,10:931-939.
[19] Morgan A,Burgoyne R D,Barclay J W,et al.Regulation of exocytosis by protein kinase C[J].Biochem Soc Trans,2005,33:1341-1344.
[20] Yamane K,Kinsella T J.Casein kinase regulates both apoptosis and the cell cycle following DNA damage induced by 6-thioguanine[J].Clin Cancer Res,2005,11:2355-2363.
[21] Colombo S,Longhi R,Alcaro S,et al.N-myristoylation determines dual targeting of mammalian NADH-cytochrome b reductase to ER and mitochondrial outer membranes by a mechanism of kinetic partitioning[J].J Cell Biol,2005,168:735-745.
[22] Man W C,Miyazaki M,Chu K,et al.Membrance topology of mouse stearoyl-CoA desaturase 1[J].J Biol Chem,2006,281 (2):1251-1260.
Cloning and expression of stearoyl-coenzyme A desaturase and protein tyrosine phosphatase 1B genes from sea urchin Strongylocentrotus intermedius
DING Jun,SUN Wei,CHANG Ya-qing
(Key Laboratory of Mariculture&Stock Enhancement in North China's Sea,Ministry of Agriculture,Dalian Ocean University,Dalian 116023,China)
Abstract:Stearoyl-coenzyme A desaturase(SCD)and Protein tyrosine phosphatase 1B(PTP1B)genes were cloned in sea urchin Strongylocentrotus intermedius by homology-base technique and RACE technique,and the structures of these genes were studied.A quantitative reverse transcriptase real-time PCR assay was developed to assess mRNA expression of these genes in different periods of embryonic development.SCD(Genbank No. HM208174)was found to be secreted protein whose full-length cDNA was 1 934 bp with an ORF of 819 bp encoding 273 amino acids with transmembrane domain,signal peptide.Higher-level mRNA expression of SCD was detected in the periods of 8-arm stage.PTP1B(Genbank No.HM208173)as secreted protein had full-length cDNA of 1 249 bp with an ORF of 657 bp encoding 219 amino acids with transmembrane domain,signal peptide.The RT -PCR detection revealed that the maximal expression level of PTP1B was observed in 2-cell stage,while the minimal level expression was found in 8-arm stage.The findings provided the foundation for understanding of the function of genes in the sea urchin metabolic processes at the molecular level.
Key words:Strongylocentrotus intermedius;SCD;PTP1B;real-time PCR
中图分类号:S917
文献标志码:A
文章编号:2095-1388(2012)06-0489-06
收稿日期:2012-04-17
基金项目:国家自然科学基金资助项目 (30972269);国家 “863”计划项目 (2012AA10A412);辽宁省百千万人才工程入选项目(2008921059);辽宁省科技计划项目 (2007203004)
作者简介:丁君 (1973-),女,博士,副研究员。E-mail:dingjun1119@dlou.edu.cn
通信作者:常亚青 (1967-),男,博士,博士生导师,教授。E-mail:yqchang@dlou.edu.cn




