鲤KCTD15基因的克隆和表达
刘伟1、2,孙婷1、3,张研1,孙效文1
(1.中国水产科学研究院生物技术研究中心,北京100141;2.大连海洋大学水产与生命学院,辽宁大连116023; 3.上海海洋大学水产与生命学院,上海201306)
摘要:通过克隆得到鲤Cyprinus carpio含钾离子通道四聚化结构域15(KCTD15)两个基因KCTD15a和KCTD15b,其开放阅读框 (Open read frame,ORF)均为774 bp,编码含257个氨基酸的多肽,编码的蛋白质相对分子量均为29 000,PI分别为7.0和6.5。经氨基酸比对和进化树分析表明:鲤KCTD15基因与斑马鱼Danio rerio、人Homo sapien、食蟹猴Macaca fascicularis、牛Bos taurus、小鼠Mus musculus、家鼠Rattus norvegicus、爪蟾Xenopus laevis等的 KCTD15基因具有高度同源性,均含有一个 BTB(Broad-Complex, Tramtrack and Bric a brac)模块,鱼类比哺乳动物少26个氨基酸。采用半定量RT-PCR法分析了KCTD15基因在鲤组织中的表达,结果表明:KCTD15a基因只在头肾中表达且较低,在其他组织中均不表达; KCTD15b基因在卵巢中表达最高,在鳃和皮肤中次之,在脑、肝胰脏、头肾、肠等组织中表达较低。采用实时荧光定量PCR法检测KCTD15在鲤胚胎发育时期的表达,结果表明:KCTD15a和KCTD15b基因都在受精后0 h表达量最高 (P<0.05),在受精后6 h和12 h均迅速下降 (P<0.05);KCTD15a基因在受精后36 h有所上升,但未达到6 h的水平,在受精后72 h及破膜后3 d、10 d时表达量逐渐降低;而KCTD15b基因在受精后36 h降至最低,在受精后72 h及破膜后3 d时逐渐升高,但幅度不大,破膜后10 d时再次下降;KCTD15b基因的表达量总体高于KCTD15a基因的表达量。
关键词:鲤;KCTD15基因;克隆;表达
水产品具有低脂肪、高蛋白的特点,是合理膳食结构中不可缺少的重要部分,是人们摄取动物蛋白的主要方式之一[1]。鱼类的肉质主要取决于肌肉中水分、脂肪、氨基酸含量特别是鲜味氨基酸的含量等因素[2]。脂肪对鱼类本身的营养以及鱼类脂肪酸对人类的健康都具有重要作用[3-4]。通过基因组关联研究 (GWAS)发现,KCTD15(Potassium channel tetramerization domain containing 15)在多个人群中被确定为与脂肪过多相关联的基因之一[5-7]。Dutta等[8]研究表明,KCTD15基因异常表达抑制神经嵴的分化,而敲除KCTD15基因则导致神经嵴的扩张,即KCTD15基因可抑制神经嵴的扩张使其保持正常范围,而神经系统的发育与生长发育密切相关。目前,关于KCTD15基因的研究报道较少,其功能也尚未确定[7]。鲤Cyprinus carpio是中国最重要的淡水养殖品种之一,KCTD15基因在鲤中的表达规律和功能目前尚未见报道。本研究中,作者克隆了鲤KCTD15基因,并利用半定量RT-PCR和实时荧光定量PCR方法检测其在鲤不同组织和胚胎发育时期的表达差异,为进一步研究KCTD15基因的功能,从而改善鲤的脂肪含量和促进其生长 (胚胎)发育提供基础资料。
1 材料与方法
1.1 材料
试验用鲤采自中国水产科学研究院黑龙江水产研究所,暂养于中国水产科学研究院生物技术研究中心。取新鲜鲤组织(血液、脑、肝胰脏、脾、头肾、体肾、肠、鳃、心脏、皮肤、肌肉、卵巢和精巢)分别保存在RNAsafeguard里;取受精后0、6、12、24、36、48、72 h的受精卵及破膜后3 d和10 d的全鱼,均保存在冰箱(-80℃)中备用。
1.2 方法
1.2.1 RNA的提取及cDNA的合成 用TRIZOL法 (Invitrogen,USA)分别提取血液、脑、肝胰脏、 脾、 头肾、 体肾、 肠、 鳃、 心脏、 皮肤、 肌肉、卵巢和精巢以及受精后0、6、12、24、36、48、72 h、破膜后3 d和10 d的全鱼总RNA,并用无核甘酸水处理。反转录为 cDNA之前,用NanoVue Plus Spectrophotometer(GE Healthcare Life Sciences,Piscataway,NJ,USA)检测RNA的含量,并用15 g/L琼脂糖胶检测其完整性。每种组织取3 μg RNA,先用DNaseI(Amplification grade,Invitrogen)进行前处理以去掉其中的DNA,再用反转录试剂盒 (引物为Oligo(dT))合成cDNA第一条链,最后用RNase去掉剩余RNA。cDNA样品均用无核甘酸水稀释10倍备用。
1.2.2 KCTD15基因在鲤中的克隆、测序与进化树的构建 利用斑马鱼KCTD15a与KCTD15b基因序列与鲤EST以及基因组序列进行Blastn比对,得到鲤KCTD15a与KCTD15b基因序列。采用3 μg全鱼 RNA(包含所有组织)反转录得到全鱼cDNA,用引物设计软件 Primer 5.0设计扩增KCTD15a与KCTD15b基因的引物为KCTD15a-3F、3R、4F、4R及KCTD15b-5F、5R、6F、6R(表1),设计的引物要跨越至少一个内含子,以防基因组污染而产生假阳性现象。所有的引物均由上海生工生物工程技术服务有限公司合成,使用前将引物稀释至10 mm/L。扩增得到的片段用PMD-18T载体克隆并测序。通过Blastn比对分析,调取与KCTD15基因开放阅读框序列相似度较高的其他物种KCTD15基因开放阅读框序列,使用Mega 5.0软件构建系统进化树,计算方法为邻位相连法(Neighbor-joining),采用自展内部分枝法 (Bootstrapping)评定进化树的可靠性,重复次数为1 000,并利用BioEdit 7.0.1软件推测氨基酸序列。
1.2.3 KCTD15基因在鲤组织中的表达 采用半定量RT-PCR法检测KCTD15基因在鲤组织中的表达,用Primer 5.0设计引物为KCTD15a-3F、3R和KCTD15b-5F、5R以及β-actin-F、β-actin-R (表1)。确定适当的循环数,使扩增产物处在平台期前的线性增长范围内,并用15 g/L琼脂糖凝胶电泳检测。基因β-actin与KCTD15的PCR反应体系共20 μL,包括鲤cDNA模板1.0 μL,10×buffer 2.0 μL,0.2 mmol/L dNTPs 2.0 μL,Taq酶 (1.0 U)0.1 μL,2.0 mmol/L MgCl21.2 μL,引物 1 μL,最后用ddH2O补至20 μL。反应程序为:95℃下预变性5 min;95℃下变性30 s,60℃下退火30 s,72℃下延伸30 s,共进行13~36个循环;最后在72℃下延伸10 min,4℃下保存。用15 g/L琼脂糖凝胶电泳检测PCR扩增产物,用溴化乙锭染色后在紫外灯下观察确定循环数。根据每个目的基因所确定的循环数对目的基因进行13个鲤组织cDNA的PCR扩增,用15 g/L琼脂糖凝胶电泳检测扩增产物。采用SPSS 15.0软件进行单因素方差分析。
1.2.4 KCTD15基因在鲤胚胎发育时期的表达选取鲤受精后0、12、24、36、48、72 h和破膜后3 d、10 d的cDNA,采用实时定量 (荧光定量)法检测鲤胚胎发育时期的表达。用Primer 5.0设计引物为KCTD15a-5F、5R和KCTD15b-7F、7R以及β-actin-F、β-actin-R(表1)。目的基因的标准曲线是以全鱼 cDNA 10倍系列稀释为模板(1.0×10-1,1.0×10-2,1.0×10-3,1.0×10-4, 1.0×10-5copies/mL),PCR反应体系共15 μL,包括鲤cDNA(100 ng/μL)1.0 μL,上、下游引物混合液 (10 μmol/L)0.3 μL,SYBR Green reagent (东洋纺)7.5 μL,最后用ddH2O补至15 μL。反应程序为:95℃下预变性15 min;95℃下变性15 s,60℃下退火30 s,72℃下延伸30 s,共进行40个循环。平行试验设3个重复,所有样品检测均设置阴性对照 (无模板)。反应在7500 PCR仪 (Applied Biosystems)上进行。每个目的基因的相对表达以内参 β-actin为校准,采用2-ΔΔCT法[9]来计算,所得数据由7500 PCR仪自带的软件分析。采用SPSS 15.0软件进行单因素方差分析。
表1 试验设计的引物序列
Tab.1 The primers used in the experiment

引物primers 序列sequences(5′-3′) KCTD15a-3F ACCAAATACCCAGACTCCAG KCTD15a-3R AATCCTCTCGCCCAGGTCAG KCTD15a-4F TGACCTGGGCGAGAGGATTG KCTD15a-4R ATGGGTGTGTGGCTGGTCTG KCTD15b-5F CTCACCAAATACCCAGACTCAC KCTD15b-5R TGGTTCCAGCCAGCGTTGAC KCTD15b-6F AACTCGGTCAACGCTGGCTG KCTD15b-6R TTCCACTGAATGGCTACTGC KCTD15b-7F TTAACGGAACCGAGCCCATC KCTD15b-7R TAACGCGCCTCCTCGTACAG β-actin-F TGCAAAGCCGGATTCGCTGG β-actin-R AGTTGGTGACAATACCGTGC
2 结果与讨论
2.1 鲤KCTD15基因全长cDNA序列及分析
利用斑马鱼KCTD15基因序列以鲤全鱼cDNA为模板进行同源克隆,确定了2个鲤KCTD15基因,分别与斑马鱼KCTD15a、KCTD15b基因相对应。从图1可见,鲤KCTD15a和KCTD15b基因全长cDNA均为774 bp,包含4个外显子,编码257个氨基酸残基的多肽。通过 Blastn比对,鲤KCTD15a和 KCTD15b基因与斑马鱼 KCTD15a、KCTD15b基因的相似性分别为90%和93%,用ExPASY在线软件预测其编码的蛋白质相对分子量均为29 000,PI分别为7.0和6.5。

图1 鲤KCTD15基因ORF序列及其编码氨基酸
Fig.1 The ORF nucleotide and deduced amino acid sequences of the gene KCTD15 in common carp
用Smart在线软件分析鲤KCTD15a、KCTD15b基因的氨基酸序列 (图1),结果表明二者在30~132个氨基酸均含有相同的BTB模块 (BTB模块被发现位于某些C2H2型的锌指结构转录因子N末端,是一种进化保守的蛋白质——蛋白质相互作用模块[10])。
从图2可见:鲤KCTD15a和KCTD15b与斑马鱼[8]、 人[5-7]、 小鼠[11]具有高度同源性, 且在鱼类中同源性较高,但鱼类比哺乳动物少26个氨基酸,推测可能与物种进化有关,且第150和155位点分别为D和E,与其他种类也不同,这可能是导致同物种不同基因间或不同物种相同基因间功能不同的原因之一。刘春伟等[12]指出,可能由于进化程度的不同,导致OLR1基因在不同物种间同源性差异较大。
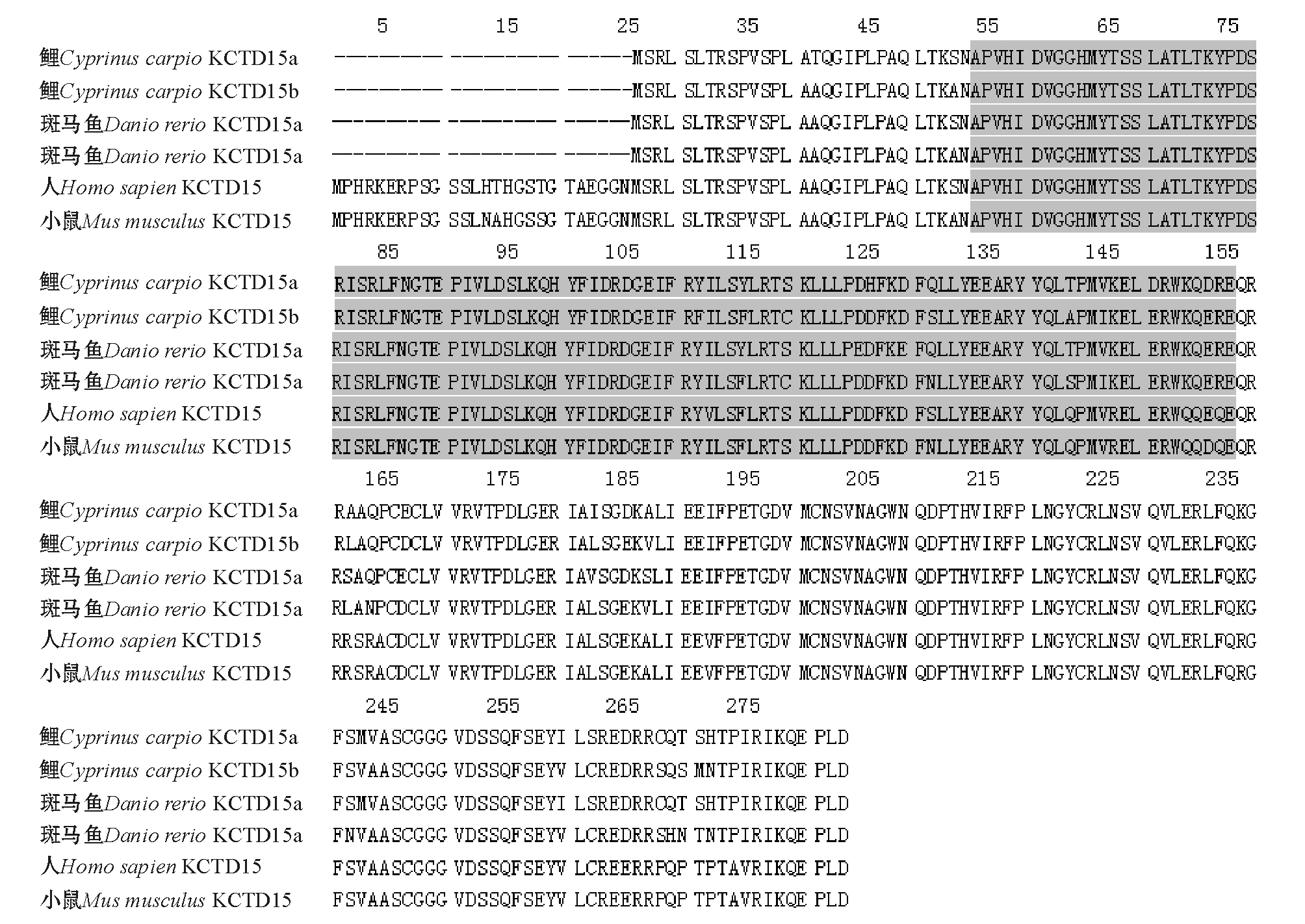
图2 KCTD15基因氨基酸序列Blastp比对结果
Fig.2 Blastp results of the amino acid sequences of the gene KCTD15
注:灰色部分表示BTB模块。Note:The gray color shows BTB domain.
进化树 (图3)分析表明,哺乳动物、鱼类和爪蟾的KCTD15基因分别聚类,这与物种的分类地位相符。鲤KCTD15a基因与斑马鱼KCTD15a基因聚类,鲤KCTD15b基因与斑马鱼KCTD15b基因聚类,推测KCTD15a与KCTD15b基因的分化早于斑马鱼和鲤的分化,但需要进一步的研究方能确定。Clark等[13]指出,果蝇的基因和基因组的进化可能与它们的生物学特点和适应性相关联。
2.2 KCTD15基因在鲤组织中的表达
采用RT-PCR方法检测KCTD15基因在鲤组织中的表达。将β-actin与KCTD15基因在同一管中扩增进行竞争性试验,只有β-actin基因有表达,目的基因没有表达,且两者到达扩增平台期的循环数不同,因此,需进行单独扩增。分别用6个不同循环数 (21、24、27、30、33、36个循环)对目的基因进行扩增,分析扩增产物的15 g/L琼脂糖凝胶电泳结果,以条带亮度观察是否进入平台期作为选择循环数的依据。从图4可见:KCTD15a基因在36个循环时达到最亮,在33个循环时亮度基本保持不变,说明在33个循环时已进入平台期,最后选取32个循环为 PCR扩增循环数。同理, KCTD15b基因在32个循环时进入平台期,β-actin基因在23个循环时进入平台期。

图3 KCTD15基因进化树
Fig.3 The evolutionary tree of the gene KCTD15
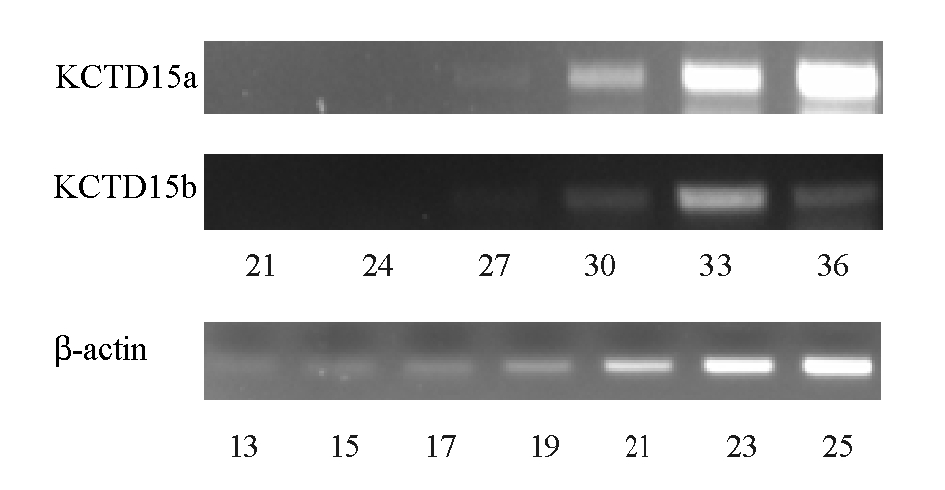
图4 确定循环数的凝胶电泳图
Fig.4 The amplification numbers confirmed
注:鲤KCTD15基因与β-actin循环数的确定,数字代表循环数。
Note:The amplification numbers of the gene KCTD15 and β-actin in common carp and the figure means the amplification numbers.
RT-PCR结果表明:KCTD15a基因只在头肾中表达且较低,在其他组织中均不表达;KCTD15b基因在卵巢中表达最高,在鳃和皮肤中次之,在脑、肝胰脏、头肾、肠等组织中表达最低(图5、图6)。
鲤KCTD15a和KCTD15b虽结构相似,但功能可能存在较大差异。KCTD15是与人身体质量指数(BMI)和脂肪量有关的基因之一[5-7],鲤卵巢含有丰富的脂肪酸[14-15],KCTD15b在卵巢中表达最高,KCTD15b可能与鲤卵巢脂肪酸含量较多有关,推测与鲤的产卵、繁殖有关。KCTD15a和KCTD15b的组织表达差异性,间接地证实了之前推测的KCTD15b与哺乳动物同源基因的亲缘性比KCTD15a近是正确的。
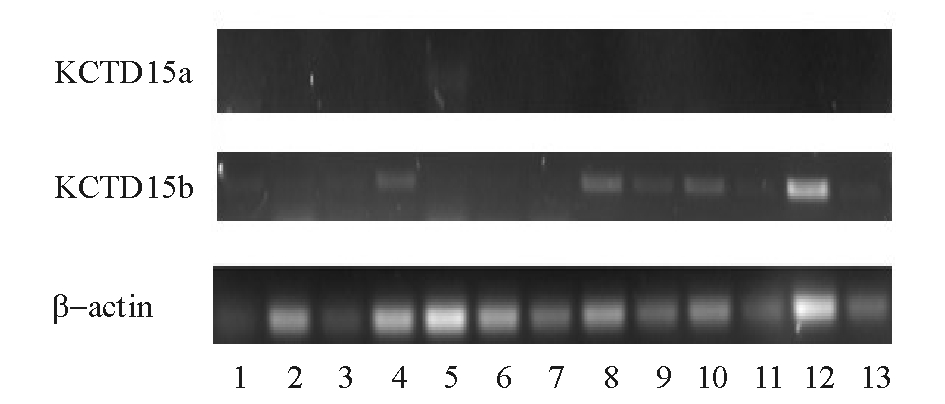
图5 鲤KCTD15基因组织表达凝胶电泳图
Fig.5 The expression of the gene KCTD15 in tissues of common carp
注:1~13分别表示血液、脑、肝胰脏、脾、头肾、体肾、肠、腮、心脏、皮肤、肌肉、卵巢、精巢。
Note:1-13 represents the blood cells,brain,liver,spleen,head kidney,body kidney,intestine,gill,heart,skin,muscle,ovary and testicle,respecticely.
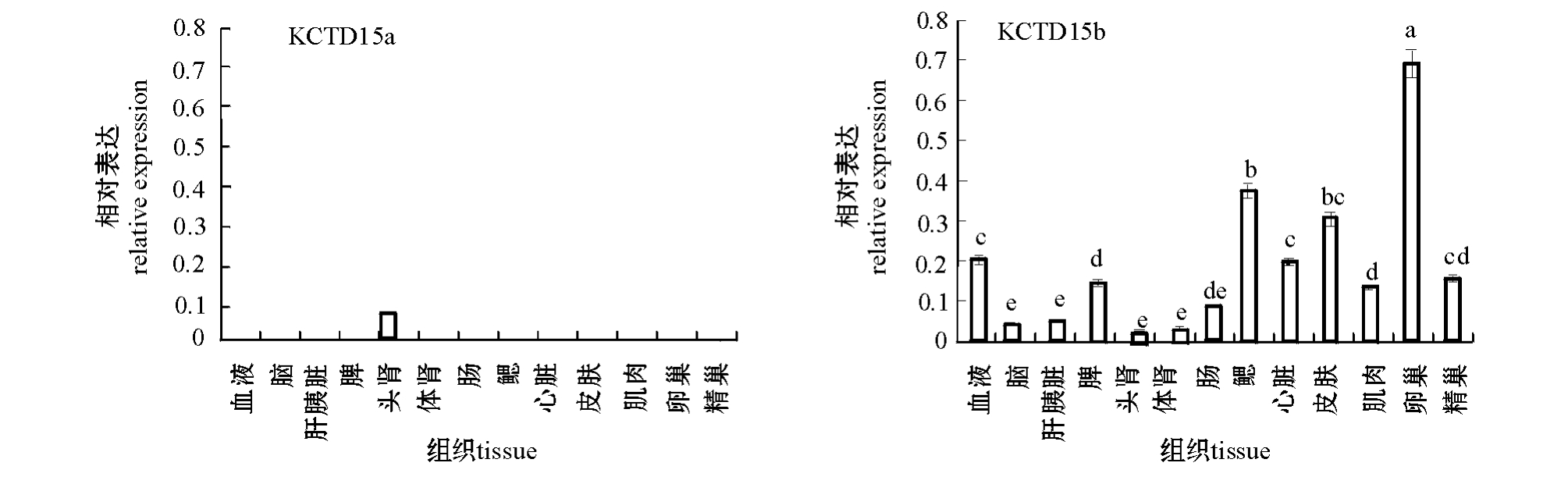
图6 鲤KCTD15基因在各组织中的表达量
Fig.6 The relative expression of the gene KCTD15 in tissues of common carp
注:标有不同小写字母者表示组间差异显著 (P<0.05),标有相同小写字母者表示组间差异不显著 (P>0.05),下同。
Note:The means with the different letters are significant differences at the 0.05 probability level,and the means with the same letters are not significant differences,et sequentia.
2.3 实时荧光定量PCR
采用实时荧光定量相对定量中的2-ΔΔCT法检测鲤 KCTD15基因的时间表达。从图 7可见: KCTD15a基因在受精后 0 h表达量最高 (P<0.05),在受精后6 h和12 h均迅速下降 (P<0.05),在受精后36 h有所上升,但未达到6 h的水平,在受精后72 h及破膜后3 d、10 d时表达量逐渐降低;KCTD15b基因同样在受精后0 h表达量最高 (P<0.05),在受精后6 h和12 h均迅速呈梯度下降 (P<0.05),在受精后36 h降至最低,在受精后72 h及破膜后3 d时逐渐升高,但幅度不大,在受精后10 d时再次下降。KCTD15b基因的表达量总体上高于KCTD15a基因的表达量。推测鲤胚胎前期 (12 h前),KCTD15a和KCTD15b基因的功能具有一定的相关性。
Dutta等[8]指出,在斑马鱼和爪蟾发育初期KCTD15基因异常表达抑制神经嵴的分化,而敲除KCTD15基因则导致神经嵴的扩张。本研究中KCTD15基因在鲤发育初期表达量较高,后期表达较低,可能与神经嵴的分化发育有关,而且KCTD15a与KCTD15b基因在脑中的表达量都较低,推测随着鲤的生长,KCTD15在神经系统的表达减少或被抑制,说明KCTD15在鱼类的胚胎及生长发育中具有重要的作用。

图7 鲤KCTD15基因在胚胎发育时期的表达量
Fig.7 The relative expression levels of the gene KCTD15 in common carp during embryonic and larval periods
3 结语
本研究中利用斑马鱼KCTD15基因与鲤基因组进行Blastn比对,经过克隆测序得到与斑马鱼相对应的鲤KCTD15a和KCTD15b两个基因。系统进化树和氨基酸序列比对分析显示,鲤KCTD15基因与斑马鱼、人、食蟹猴、牛、小鼠、家鼠、爪蟾等的KCTD15基因存在高度同源性,KCTD15b基因与哺乳动物同源基因的亲缘性比KCTD15a基因近。采用半定量RT-PCR和实时荧光定量PCR检测了KCTD15a和KCTD15b基因的组织表达和胚胎发育时期的表达,结果显示鲤KCTD15a和KCTD15b基因在组织中的表达存在较大差异,推测KCTD15b基因可能与鲤卵巢的脂肪含量有关;KCTD15a和KCTD15b基因在发育初期表达趋势相似,即受精后达到最大表达量,6 h后迅速下降,发育后期表达量均较少,推测KCTD15基因在胚胎发育初期有重要作用。KCTD15基因对鱼类脂肪含量有何影响及其具体的生物学功能还没有被确定,需进一步通过荧光原位杂交、基因突变与敲除等技术研究该基因在鱼类生长发育过程中的规律与作用。
致谢:对赵紫霞博士、李炯棠博士、冀培丰、万玉美等人在采集试验样本、试验操作和数据分析过程中提供的帮助表示衷心地感谢!
参考文献:
[1] 张慜,肖功年.国内外水产品保鲜盒保活技术研究进展[J].食品与生物技术,2002,21(1):104-107.
[2] 中国预防医学科学院,营养与食品卫生研究所.食物成分表(全国代表值)[M].北京:人民卫生出版社,1991.
[3] Watanabe T.Lipid nutrition in fish[J].Comp Biochem Physiol, 1982,73B:3-15.
[4] Steffens W.Effects of variation in essential fatty acids in fish feeds on nutritive value of freshwater fish for humans[J].Aquaculture, 1997,151:97-119.
[5] Willer C J,Speliotes E K,Loos R J,et al.Six new loci associated with body mass index highlight a neuronal influence on body weight regulation[J].Nat Genet,2009,41(1):25-34.
[6] Thorleifsson G,Walters G B,Gudbjartsson D F,et al.Genomewide association yields new sequence variants at seven loci that associate with measures of obesity[J].Nat Genet,2009,41:18-24.
[7] Hotta K,Nakamura M,Nakamura T,et al.Association between o-besity and polymorphisms in SEC16B,TMEM18,GNPDA2,BDNF,FAIM2 and MC4R in a Japanese population[J].Hum Genet, 2009,54:727-731.
[8] Dutta S,Dawid I B.KCTD15 inhibits neural crest formation by attenuating Wnt/β-catenin signaling output[J].Development, 2010,137:3013-3018.
[9] Livak K J,Schmittgen T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta DeltaC (T))method[J].Methods,2001,25:402-408.
[10] Ahmad K F,Engel C K,Prive G G.Crystal structure of the BTB domain from PLZF[J].Biochemistry,1998,95:12123-12128.
[11] Bailey P J,Klos J M,Andersson E,et al.A global genomic transcriptional code associated with CNS-expressed genes[J].Exp Cell Res,2006,312(16):3108-3119.
[12] 刘春伟,孙超.猪OLR1基因克隆及生物信息学分析[J].西北农业学报,2008,17(5):51-55.
[13] Clark A G,Eisen M B,Smith D R,et al.Evolution of genes and genomes on theDrosophilaphylogeny[J].Nature,2007:203-218.
[14] Kaitaranta J K,Linko R R.Fatty acids in the roe lipids of common food fishes[J].Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology,1984,79(3):331-334.
[15] 陆丽丽,陈舜胜.鲤、鲫卵的脂质和脂肪酸特性[J].食品科学,2007,28:100-104.
Molecular cloning and expression of KCTD15 in common carp
LIU Wei1,2,SUN Ting1,3,ZHANG Yan1,SUN Xiao-wen1
(1.The Center for Applied Aquatic Genomics,Chinese Academy of Fishery Sciences,Beijing 100141,China;2.College of Fisheries and Life Science,Dalian Ocean University,Dalian 116023,China;3.College of Fisheries and Life Science,Shanghai Ocean University,Shanghai 201306, China)
Abstract:Potassium channel tetramerization domain containing 15(KCTD15)was one of genes controlling the fat and mass by affecting human nerve center.We used Blastn program at the NCBI to identify near matches in the common carp(Cyprinus carpio)genome database to zebrafish KCTD15.KCTD15a and KCTD15b were identified respectively.ORF of KCTD15a and KCTD15b were cloned and sequenced in common carp.KCTD15a and KCTD15b were both 774 bp encoding polypeptides of 257 amino acids with a calculated Mw of 29 000 and PI of 7.0 and 6.5.They all have four exons and three introns.KCTD15a and KCTD15b all contained one conservative BTB domain.Phylogenetic relationship analysis indicated that KCTD15a and KCTD15b were clustered with KCTD15 of zebrafish to the corresponding subgroup respectively.RT-PCR analysis revealed that KCTD15a only expressed at a low level in head kidney.KCTD15b was predominantly expressed in ovary and lower expressed in gill and skin and lowest expressed in brain,hepatopancreas,head kidney and intestine.These results suggest that KCTD15a and KCTD15b may have the similar structures but different functions in common carp.Quantitive real time PCR was used to detect the expression of KCTD15 after fertilization.KCTD15a was strongest expressed at 0 hour after fertilization(hpf),it declined very much at 6 hpf and 12 hpf(P<0.05),it increased at 36 hpf lower than the level at 6 hpf and then decreased again at 72 hpf,3 dpf,and 10 dpf.KCTD15b was similar with KCTD15a at 0 hpf to 12 hpf,it increased from 36 hpf to 3 dpf and then decreased at 10 dpf.The expression level of KCTD15a was lower than that of KCTD15b which was corresponding to their expression in tissues.
Key words:common carp;KCTD15;clone;gene expression
中图分类号:Q752;S917.4
文献标志码:A
文章编号:2095-1388(2012)05-0464-07
收稿日期:2011-12-17
基金项目:国家 “863”计划项目 (2011AA100401);公益性行业 (农业)科研专项项目 (200903045);中国水产科学院研究基金资助项目 (2010C016)
作者简介:刘伟 (1987-),男,硕士研究生。E-mail:liuweihaoyang@163.com
通信作者:孙效文 (1955-),男,研究员,博士生导师。E-mail:sunxw2002@163.com







