马志,张峰,张慧晓,杨威
(大连海洋大学农业部北方海水增养殖重点实验室,辽宁大连116023)
摘要:应用分子动力学方法研究了人类miRNA 3′末端3碱基与PAZ功能域的结合模式,并采用线性相互作用能方法计算miRNA 3′末端片段与PAZ之间的结合自由能。结果表明:miRNA 3′末端碱基组成决定了其和PAZ功能域的结合模式以及结合自由能,UGU与PAZ的结合最为稳定,结合自由能为-37.248 kJ/mol,而CGA与PAZ的结合情况相对不太稳定,结合自由能为-5.418 kJ/mol;miRNA 3′端与PAZ的结合自由能与其在人类miRNA基因中的分布频率存在一定的相关性,UGU在人类基因中占6.768%,而CGA仅占0.414%。本研究结果可为进一步研究miRNA与Argonaute蛋白的相互作用以及对人类疾病防治和生物进化探索提供重要信息。
关键词:miRNA;PAZ功能域;分子动力学模拟;线性自由能
MicroRNAs(miRNAs)是由21~23个核苷酸(nt)组成的一类RNA分子。近年来越来越多的miRNA被发现[1-2],miRNA通过在转录后水平上调控基因表达来参与生命过程中的一系列重要进程,包括早期发育、细胞增殖、细胞分化以及细胞凋亡[3-4]。miRNAs广泛存在于真核生物中,是真核生物中最大的基因家族之一,约占人类基因组的1%,且在植物领域中很多都高度保守[5]。对于大多数动植物,miRNA能与若干个蛋白结合组成RNA诱导沉默复合体 (RNA-induced silencing complex,RISC)。RISC中的miRNA通过碱基匹配原则识别靶基因3′非翻译区的靶位点。其中, miRNA 5′端的1-7nt在与靶mRNA的结合中比3′端序列更为重要,因此,第1-7nt的碱基序列所组成的区域被称为种子区 (seed region)。
Argonaute(Ago)蛋白是 RISC的核心元件,具有剪切功能,能够降解与miRNA互补配对的目标mRNA[6],从而抑制编码蛋白靶基因的翻译或(和)降解靶基因[7]。该蛋白由PAZ和PIWI功能域组成[8],其中PAZ功能域决定了对miRNA 3′末端的识别与结合。PAZ功能域中央是一个 β桶,一侧是两个N端螺旋,另一侧是一个保守的组件:由一个 β发夹和一个α螺旋组成[9]。β桶与αβ组件之间能形成一个口袋状的结构,可以插入miRNA或siRNA 3′末端突出的碱基,从而形成有效的结合(图1)。图1(a)为用Ribbon模型表示的PAZ功能域和miRNA 3′端,图1(b)为miRNA 3′末端3碱基与 PAZ功能域的结合模式,其中miRNA用Stick模型表示,PAZ功能域用Surface模型表示。研究发现,miRNA 3′末端的2碱基会与PAZ功能域发生作用,其中由3个保守氨基酸 (Tyr354、Pro363和Tyr386)形成了一个酚羟基区域,从而和3′末端碱基形成很强的氢键作用[10]。但是PAZ对miRNA存在怎样的识别机制和选择性结合,目前仍然未知。
本研究中,作者统计了所有已知的人类miRNA,取其3′末端3碱基的所有组成作为研究对象,通过分子对接和分子动力学模拟方法获得了3碱基与Ago蛋白稳定的复合结构,并采用线性相互作用能 (linear interaction energy,LIE)方法计算出miRNA 3′末端片段与Ago蛋白之间的结合自由能,旨在为miRNA的进一步研究提供重要的信息。
1.1 模型构建
模拟所用的结构是以人类 Ago eIF2c为参考(PDB1SI2)[11],对缺失的残基用Swiss-PDBviewer 4.0[12]进行修复补齐。然后采用Gromacs软件包[13]在300 K下进行100 ps的MD模拟,取其平均结构作为对接时的大分子结构。配体小分子为含有3个RNA碱基的片段,miRNA片段由人类的miRNA 3′末端的3个碱基组成,通过统计共有64个碱基片段,本研究中取出其中的12个具有典型结构的片段来进行研究。配体小分子采用ISI-draw构建。
1.2 分子对接
首先采用 Auto Dock 4.0(http://autodock. scripps.edu)程序包对一系列miRNA片段与 Ago PAZ功能域进行分子对接[14]。对接时受体大分子的格点盒子大小为6.7 nm×7.6 nm×6.8 nm,间距为0.0375 nm,用Lamarckian遗传算法[15]将局部能量搜索与遗传算法相结合,以半经验势函数作为能量打分函数,对分子构象和位置进行全局搜索,并对每个配体进行100次独立的对接试验。以miRNA与PAZ结合模式的晶体结构为参考,对有配体的100次对接结果进行分析。最后,以最低对接能和miRNA 3′末端与PAZ的结合情况来选取合理的miRNA片段与PAZ结合模式作为初步的复合物结构。
1.3 分子动力学模拟
本研究中采用Gromacs 4.0软件进行分子动力学模拟,力场为Gromos96[13]。小分子配体的原子信息通过Prodrg软件[16]获得。对12个复合物分别在水溶液中进行分子动力学模拟。复合体均取自分子对接所得到的结果。每个复合体的质心置于盒子中心,选择溶质原子到盒子壁的距离为0.75 nm,然后加入水分子,水分子模型采用 SPC模型[17]。用最陡下降法和共轭梯度法进行能量优化使体系于100 kJ/(mol·nm)处正常收敛,并用热浴耦合使系统温度保持在300 K,然后在 300 K下进行10 000 ps的MD模拟。本研究中模拟采用NPT系综,原子的初始速度根据Maxwell分布在300 K、1 bar下产生,模拟步长取2 fs,每5 ps记录一次轨迹。在模拟中,采用PME算法计算静电作用的大小,范德华作用的截断半径为1.4 nm,分别对蛋白质环境和溶剂环境中miRNA 3′末端3个碱基与环境之间的相互作用能进行系综平均,进而计算结合自由能。
1.4 主成分分析
主成分分析 (PCA)是用于确定蛋白质和RNAs主要运动模式的一种分析方法,这与Wlodek等[18]的描述基本相同。从每个运动轨迹中平衡部分,计算出协方差矩阵C。对于一个由N个原子组成的系统,对该系统取M个快照,这个系统的协方差矩阵C是一个3N×3N的矩阵:
协方差矩阵C的特征向量V以及与其相关联的特征值λ,都可以通过协方差矩阵C的对角化来获得:
特征向量代表了结构变化的主要模式,特征值则表示了相应的特征向量在整个运动轨迹中所占的比例,可以通过![]() 计算得出。
计算得出。
1.5 结合自由能的计算
线性相互作用能方法是一种基于分子动力学采样的自由能计算方法,这种方法把结合自由能分解为极性部分和非极性部分[19]。极性部分通过线性响应理论处理静电项得到,非极性部分通过由一系列试验数据拟合的经验公式得到。因此,受体和配体的结合自由能可表示为
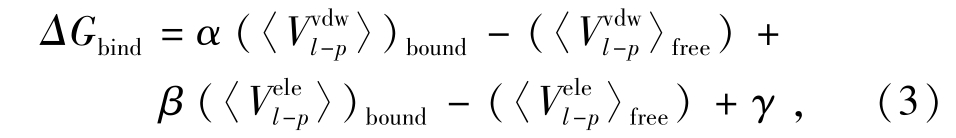
式中:ΔGbind为结合自由能;α、β分别为非极性和极性相互作用对结合自由能的响应系数;![]() 分别表示配体与环境之间的范德华相互作用能以及静电相互作用能的系综平均,下标bound和free分别表示配体在与受体结合状态下以及配体在溶剂中的相互作用能,下标l-p表示配体分别在孤立状态和与环境相互作用状态时势能的差值。
分别表示配体与环境之间的范德华相互作用能以及静电相互作用能的系综平均,下标bound和free分别表示配体在与受体结合状态下以及配体在溶剂中的相互作用能,下标l-p表示配体分别在孤立状态和与环境相互作用状态时势能的差值。
考虑到自由状态和结合状态下配体和环境之间的相互作用能,蛋白质和溶剂以及溶剂和溶剂之间的相互作用,配体受体结合过程中平动熵和转动熵的变化,以及去溶剂化过程中水分子的重排所引起的熵变等,Ben-Naim等[20]加入了常数项γ来弥补这些因素在配体-受体相互作用中的影响。在具体模拟时,可分别对配体化合物在蛋白环境和溶剂环境下进行分子动力学模拟,式 (3)中的4项系综平均值可从收集得到的分子动力学轨迹进行系综平均得到。Åqvist等[19]选取了几个蛋白质-抑制剂体系,对其结合自由能进行计算,当β=0.5,α= 0.16时预测值与试验值相吻合。
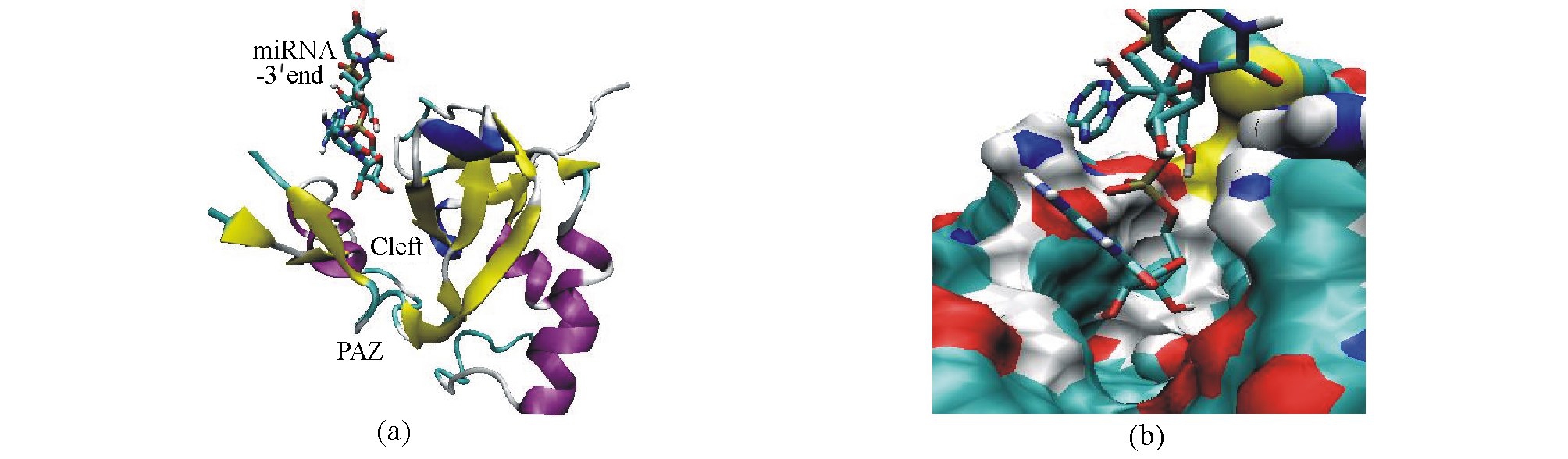
图1 PAZ-miRNA复合体的结构
Fig.1 The structure of PAZ-miRNA complex
本次模拟中,结合自由能采用下式计算:
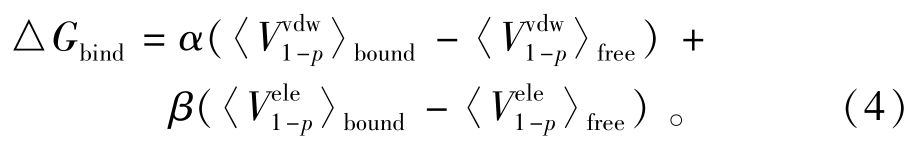
由MD轨迹可以分析得到配体与受体结合状态下以及配体在溶剂中与环境之间的静电相互作用能和范德华相互作用能,由式 (4)可用LIE方法计算miRNA片段与PAZ的结合自由能。本研究中,也取β=0.5,α=0.16进行计算。
2.1 动力学分析
均方根偏差 (root-mean-square deviation, RMSD)是用来判断一个体系是否稳定的重要依据。各个体系的RMSD是根据它们初始结构的位置来进行计算的。从UUC-PAZ、CGA-PAZ和PAZ的RMSD曲线图 (图2)可以看出,经过2 ns之后选取的3个体系的RMSD波动幅度保持在0.25 nm左右,说明整个模拟系统已经达到了平衡状态。在整个模拟过程中PAZ并没有其他构象形式出现,表明在整个模拟过程中PAZ一直处于稳定状态,同时也证明了本研究中模拟过程的准确性。
主成分分析方法可为解决大规模非线性数据的特征提取提供一条可行的途径。与正常震荡型分析类似,这种分析方法旨在利用降维的思想,把多指标转化为少数几个综合指标。本研究中,每个系统的模拟轨道的分析应用PAZ全原子波动的PCA分析[21]。对UUC-PAZ、CGA-PAZ、PAZ 3个模拟体系的PCA分析结果 (表1、表2)表明,各个特征向量对总变化的贡献值大致相同,表明在模拟过程中PAZ处于非常稳定的状态,结构并没有发生大的变化,也没有其他构象出现。选取各个体系PAZ的前3个特征向量来分析整个模拟过程中PAZ的结构变化,从各个构象的均匀分布图 (图3)可以看出,PAZ在整个模拟过程中保持稳定的构象,此结果与RMSD结果一致。从PCA分析结果中看出,该系统处于稳定的结构,没有出现显著的波动,也没有出现其他构象,这在一定程度上显示了PAZ和miRNA结合的稳定性。
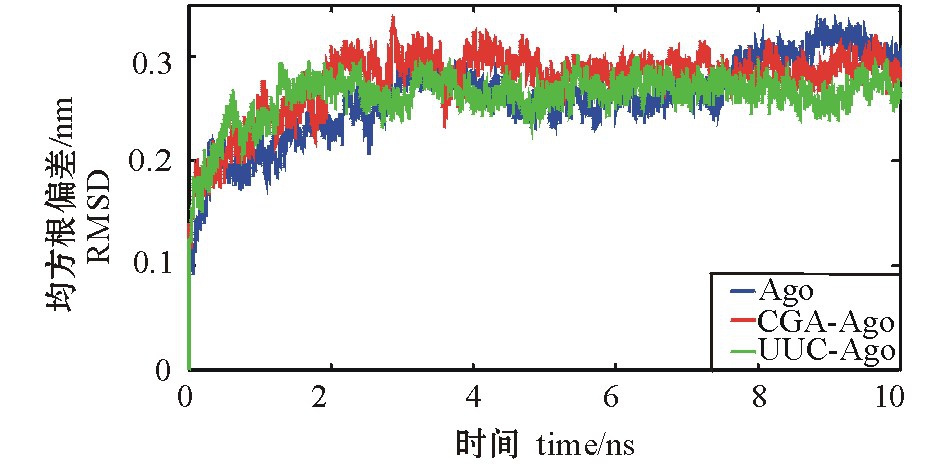
图2 PAZ、UUC-PAZ和CGA-PAZ体系的RMSD曲线图
Fig.2 RMS deviations with respect to the starting structure in the simulation of the PAZ,UUCPAZ and CGA-PAZ complex
2.2 miRNA的统计结果
从Mirbase数据库中获得人类成熟的miRNA共724条,对724条miRNA进行统计,取各条miRNA 3′末端3碱基进行计数,得出64个不同的miRNA 3′片段在整个miRNA中的分布情况,其中分布最多的是片段UGU(6.768%),分布较少的是片段CGA(0.414%)和CAA(0.414%)。
鉴于计算时间等原因,本研究中根据miRNA片段的分布情况选取12个具代表性的miRNA片段作为研究对象,它们分别为UGU、GUU、UGA、AAU、AUC、CAA、CAC、CUG、UGC、UUA、UUC和CGA(表3)。这样的选择保证了miRNA片段在总体分布上的平均性,能够更好的解释模拟结果。
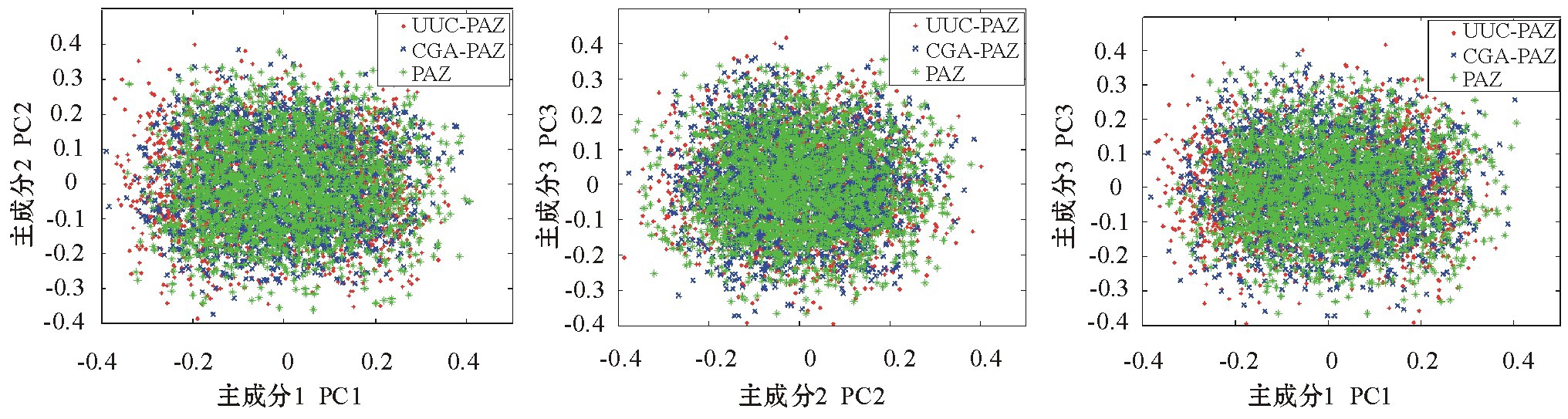
图3 PAZ、UUC-PAZ和CGA-PAZ的主成分散点图
Fig.3 Charts of the first three principal components(PC1,PC2,and PC3)of PAZ,UUC-PAZ and CGA-PAZ
2.3 结合自由能的计算
经MD模拟后,在蛋白质和水环境下,miRNA片段与受体结合状态的静电相互作用能和范德华相互作用能的计算结果见表3。这些能量是由MD轨迹得到的配体与环境之间相互作用能的系综平均值。从表3可以看出:在蛋白质环境中,范德华相互作用能大于静电相互作用能,说明miRNA片段与蛋白质的作用中范德华作用起到了主要作用,有利于miRNA与PAZ的结合;在水环境中,范德华相互作用能依然大于静电相互作用能,进一步说明了miRNA有与PAZ功能域结合的趋势。
试验中,结合自由能一般采用下式计算:
其中:R为理想气体常数;T为温度 (K);Kd为能量平衡常数。根据式 (5)计算试验所得的结合自由能应在-9 kJ/mol和-22 kJ/mol之间[22-23]。
本次模拟采用式 (4),选取 α=0.16,β= 0.5,进行结合自由能的计算,所得到的结果符合试验结果。
2.4 结合模式分析
不同的miRNA片段与PAZ功能域的结合模式不同,所得到的结合自由能也不同。现以 UUC (ΔGbind=-29.754 kJ/mol) 和 CGA(ΔGbind= -5.418 kJ/mol)为例来说明它们的结合模式。
miRNA 3′末端悬挂于PAZ形成的口袋区域内,由图4(a)可以看出,miRNA片段U1U2C3的RC3碱基上的O2与Y107 N上的H形成氢键,它们之间的距离为0.201 nm;RC3两个羟基的氢与Y107上的O形成氢键,距离分别为0.325 nm和0.358 nm;RC3磷酸上的O1P与H46上的HE2形成氢键,距离为0.213 nm;RU2上的O3′与Y80的HH以及Y85的 HH形成氢键,距离分别为 0.328 nm、0.388 nm;RU2上的HO′2与Y85的HH形成氢键,距离为0.196 nm;RU1羟基上的氢与Y80的HH形成氢键,距离为0.21 nm。UUC与PAZ之间形成的这8对氢键使得它们之间的结合自由能为-29.754 kJ/mol。图4(b)为片段C1G2A3与PAZ之间的结合情况,RA3磷酸上的O2P与PAZ的口袋区域的Y54上的HH形成氢键,距离为0.262 nm。它们之间仅形成一对氢键,使得CGA与PAZ之间的结合自由能为-5.418 kJ/mol。

图4 UUC-PAZ和CGA-PAZ的结合模式图
Fig.4 The binding mode of UUC-PAZ and CGA-PAZ
表1 UUC-PAZ、CGA-PAZ、PAZ的主成分分析结果
Tab.1 Principal component analysis of the simulation trajectories of UUC-PAZ complex,CGA-PAZ complex, and single PAZ
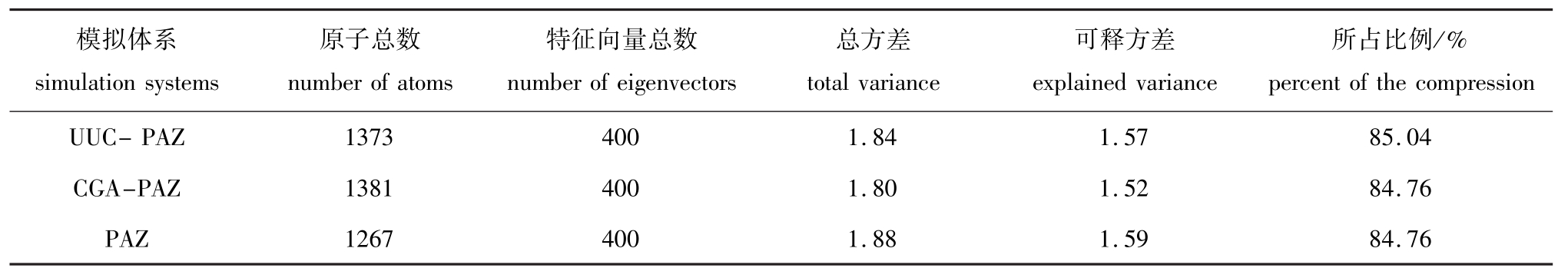
模拟体系simulation systems原子总数number of atoms特征向量总数number of eigenvectors总方差total variance可释方差explained variance所占比例/% percent of the compression UUC-PAZ 1373 400 1.84 1.57 85.04 CGA-PAZ 1381 400 1.80 1.52 84.76 PAZ 1267 400 1.88 1.59 84.76
表2 UUC-PAZ、CGA-PAZ和PAZ各主成分向量的分析结果
Tab.2 Principal component vector analysis of the simulation trajectories of UUC-PAZ complex,CGA-PAZ complexn and single PAZ
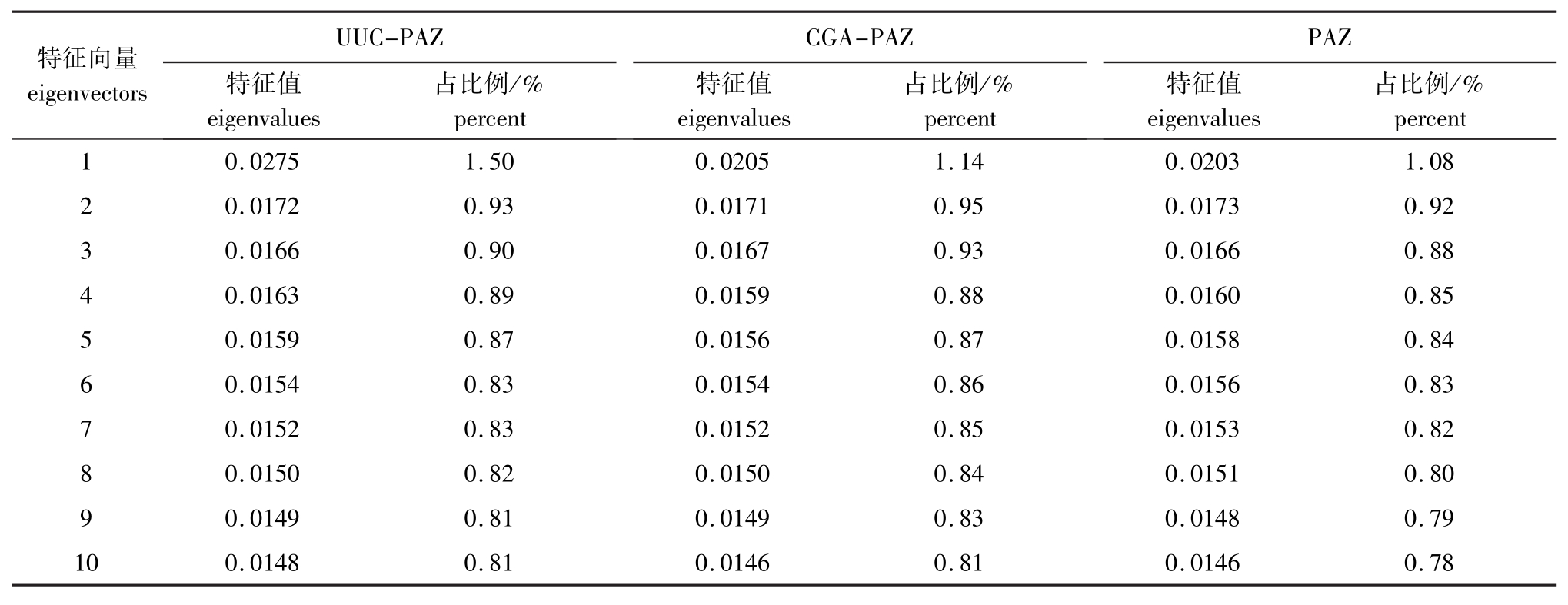
特征向量eigenvectors UUC-PAZ特征值eigenvalues占比例/%占比例/%占比例/% percent CGA-PAZ特征值eigenvalues percent PAZ特征值eigenvalues percent 1 0.0275 1.50 0.0205 1.14 0.0203 1.08 2 0.0172 0.93 0.0171 0.95 0.0173 0.92 3 0.0166 0.90 0.0167 0.93 0.0166 0.88 4 0.0163 0.89 0.0159 0.88 0.0160 0.85 5 0.0159 0.87 0.0156 0.87 0.0158 0.84 6 0.0154 0.83 0.0154 0.86 0.0156 0.83 7 0.0152 0.83 0.0152 0.85 0.0153 0.82 8 0.0150 0.82 0.0150 0.84 0.0151 0.80 9 0.0149 0.81 0.0149 0.83 0.0148 0.79 10 0.0148 0.81 0.0146 0.81 0.0146 0.78
表3 miRNA 3′末端片段与Ago蛋白的结合自由能分析
Tab.3 The analysis of the binding free energy between miRNA 3′end fragment and Ago kJ/mol
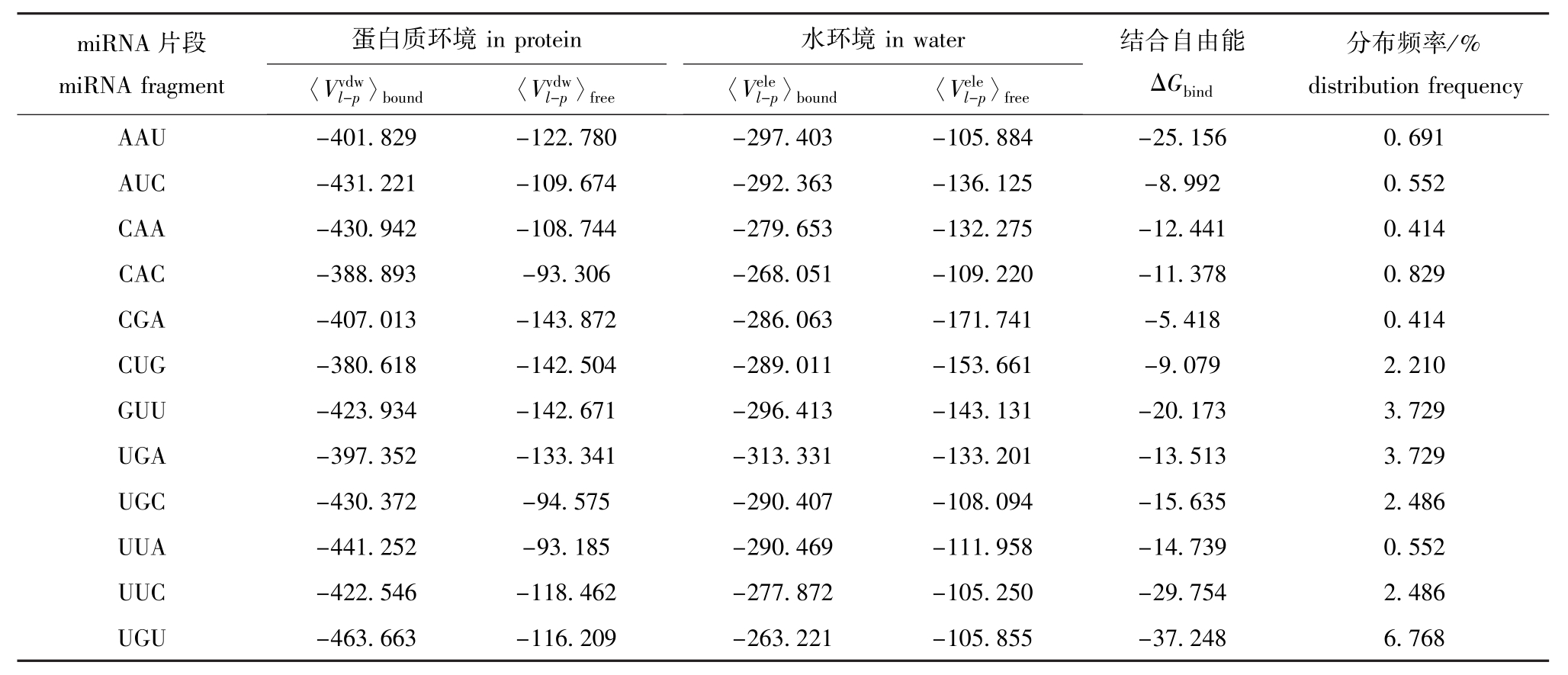
miRNA片段miRNA fragment蛋白质环境分布频率/% in protein〈Vvdw水环境distribution frequency AAU -401.829 -122.780 -297.403 -105.884 -25.l-p〉bound 〈Vvdwl-p〉free in water〈Velel-p〉bound 〈Velel-p〉free结合自由能ΔGbind 156 0.691 AUC -431.221 -109.674 -292.363 -136.125 -8.992 0.552 CAA -430.942 -108.744 -279.653 -132.275 -12.441 0.414 CAC -388.893 -93.306 -268.051 -109.220 -11.378 0.829 CGA -407.013 -143.872 -286.063 -171.741 -5.418 0.414 CUG -380.618 -142.504 -289.011 -153.661 -9.079 2.210 GUU -423.934 -142.671 -296.413 -143.131 -20.173 3.729 UGA -397.352 -133.341 -313.331 -133.201 -13.513 3.729 UGC -430.372 -94.575 -290.407 -108.094 -15.635 2.486 UUA -441.252 -93.185 -290.469 -111.958 -14.739 0.552 UUC -422.546 -118.462 -277.872 -105.250 -29.754 2.486 UGU -463.663 -116.209 -263.221 -105.855 -37.248 6.768
2.5 相关性分析
通过拟合miRNA 3′末端3碱基的分布频率与miRNA 3′末端和Ago PAZ功能域的结合自由能(图5)可以得到,回归系数R=0.68,说明两者之间存在着一定的相关性,即miRNA 3′末端3碱基的分布越多,与PAZ作用的结合能越低。低能量说明miRNA与PAZ结合稳定,表明miRNA越易于和PAZ功能域结合。以UGU为例来看,其在人类总miRNA 3′末端3个碱基中的分布比例最大,高达6.768%,有趣的是其与PAZ功能域的结合自由能最低。根据这些结果可以推断,在人类中miRNA 3′末端3碱基分布得越多、越广泛,其就更易与PAZ结合。由此可以推测,PAZ对miRNA的结合和该3′末端3碱基的分布频率相关,这也从一定程度证明了生物体系是一个高度高效的节能系统。
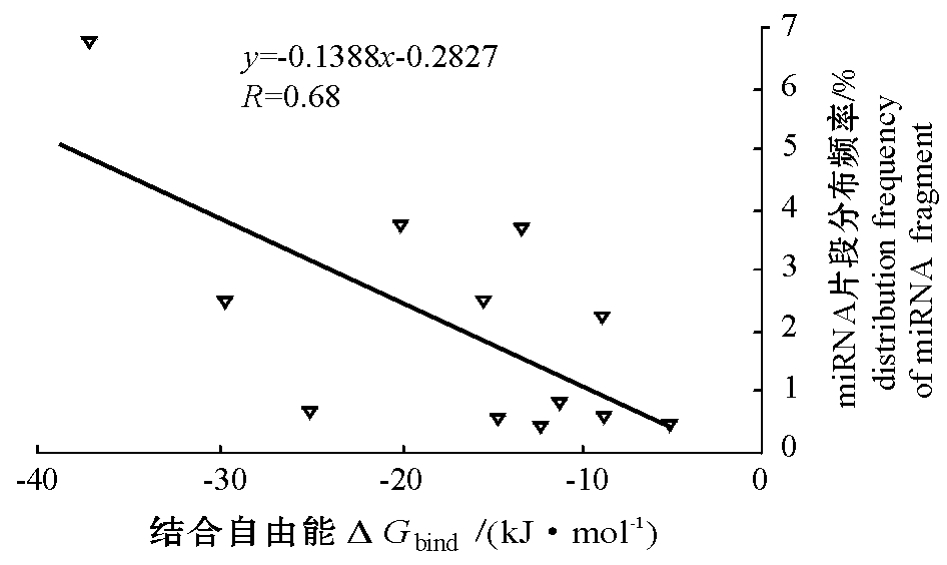
图5 miRNA 3′片段分布频率与结合自由能的拟合
Fig.5 The distribution frequency of miRNA fragment to the binding free energy
本研究中采用一系列分子模拟的方法对Ago蛋白PAZ功能域的晶体结构进行了研究分析。首先,采用分子动力学模拟的方法分别对PAZ与miRNA 3′端3碱基的结合复合体进行模拟,找到蛋白上与相应碱基发生作用的氨基酸,并分析其结合模式,然后通过线性自由能的方法对这些结构的结合自由能进行了计算。结果发现,miRNA 3′末端碱基组成决定了其和PAZ的结合模式,同时结合自由能与其在人类miRNA基因中的分布频率存在一定的相关性。
miRNA对细胞的增殖、分化和凋亡有重要的调节作用,其在正常组织和肿瘤组织中的表达显著不同,miRNA参与了肿瘤的发生、发展。研究表明, miRNA在肿瘤的形成中起到很大的作用。在肺癌组织中,miR-21在肺癌组织中表达显著上调,抑制凋亡基因产物表达,与肺癌形成、预后和总存活数有关[24]。在肝癌组织中,miR-122可以抑制癌细胞的某些特性,例如克隆存活、不依赖支持物生长、转移、侵袭、上皮-间质转化、裸鼠成瘤。这些结果表明,在肝脏中miR-122作为肿瘤抑制因子行使其功能。在乳腺癌研究中,Iorio等[25]用miRNA芯片比较了76个乳腺肿瘤和34个正常乳腺标本中miRNA分子的表达,结果表明有29个miRNA的水平在乳腺癌中发生了明显的变化,说明miRNA在肿瘤的发生过程中起到了重要的作用。miRNA行使拟制肿瘤发生和发展的功能均发生在RNA干扰的过程中。
RNA干扰在基因沉默方面具有高效性和简单性,是基因功能研究的重要工具,在疾病治疗方面也发挥着重要的作用。miRNA和Argonaute蛋白在RNA干扰作用中起到了关键的作用,研究表明, Ago蛋白的PAZ功能域能够与miRNA 3′端进行结合,进而与miRNA组成RISC,完成RNA干扰的过程。
本研究中通过分子模拟方法研究了Argonaute蛋白PAZ功能域与miRNA 3′末端3碱基的结合模式,发现miRNA 3′末端碱基组成对其与PAZ的结合模式以及结合的稳定程度起到了决定性作用。同时研究发现,miRNA 3′末端碱基与PAZ的结合自由能和其在人类miRNA基因中的分布频率存在相关性,这为研究miRNA治疗肿瘤疾病方面提供了重要的信息。
参考文献:
[1] Bahadori M.New advances in RNAs[J].Arch Iranian Med,2008, 11:435.
[2] Gregory R I,Shiekhattar R.MicroRNA biogenesis and cancer[J]. Cancer Res,2005,65:3509-3512.
[3] Bartel D P.MicroRNAs:genomics,biogenesis,mechanism,and function[J].Cell,2004,116:281-297.
[4] Eulalio A,Huntzinger E,Izaurralde E.Getting to the root of miRNA-mediated gene silencing[J].Cell,2008,132:9-14.
[5] Kloosterman W P,Plasterk R H A.The diverse functions of microRNAs in animal development and disease[J].Dev Cell,2006, 11:441-450.
[6] Song J J,Smith S K,Hannon G J,et al.Crystal structure of Argonaute and its implications for RISC slicer activity[J].Science, 2004,305:1434-1437.
[7] Carrington J C,Ambros V.Role of microRNAs in plant and animal development[J].Science,2003,301:336-338.
[8] Carmell M A,Xuan Z,Zhang M Q,et al.The Argonaute family: tentacles that reach into RNAi,developmental control,stem cell maintenance,and tumorigenesis[J].Genes Dev,2002,16:2733-2742.
[9] Lingel A,Simon B,Izaurralde E,et al.Structure and nucleic acid binding of the Drosophila Argonaute 2 PAZ domain[J].Nature, 2003,426:4652-4691.
[10] Yan K S,Yan S,Farooq A,et al.Structure and conserved RNA binding of the PAZ domain[J].Nature,2003,426:468-474.
[11] Ma J B,Ye K,Patel D J.Structural basis for overhang-specific small interfering RNA recognition by the PAZ domain[J].Nature, 2004,429:318-322.
[12] Guex N,Peitsch M C.SWISS-MODEL and the Swiss-PDBViewer:an environment for comparative protein modeling[J].Electrophoresis,1997,18:2714-2723.
[13] Van der Spoel D,Lindahl E,Hess B,et al.Gromacs User Manual,Version 3.2[M].Groningen.The Netherlands:University of Groningen,2004.
[14] Morris G M,Goodsell D S,Halliday R S,et al.Automated docking using a Lamarckian genetic algorithm and an empirical binding free energy function[J].Comput Chem,1998,19:1639-1662.
[15] Van der Spoel D,Van Buuren A R,Tieleman D P,et al.Molecular dynamics simulations of peptides from BPTI:A closer look at amide—aromatic interactions[J].Biomol NMR,1996,8:229-238.
[16] Aalten D M F,Bywater R,Findlay J B C,et al.PRODRG,a program for generating molecular topologies and unique molecular descriptors from coordinates of small molecules[J].Comput-Aided Mol Des,1996,10:255-262.
[17] Berendsen H J C,Postma J P M,Van Gunsteren W F,et al.Interaction models for water in relation to protein hydration[M]//Pullman B.Intermolecular Forces.Dordrecht:Reidel Publishing Company,1981:331-342.
[18] Wlodek S T,Clark T W,Scott L R,et al.Molecular dynamics of acetylcholinesterase dimer complexed with tacrine[J].Am Chem Soc,1997,119:9513-9522.
[19] Åqvist J,Medina C,Samuelsson J K.A new method for predicting binding affinity in computer-aided drug design[J].Protein Eng 1994,7:385.
[20] Ben-Naim A,Marcus Y.Solvation thermodynamics of nonionic solutes[J].Chem Phys,1984,81:2016.
[21] Amadei A,Linssen A B,Berendsen H J.Essential dynamics of proteins[J].Proteins,1993,17:412-425.
[22] Vargason J M,Szittya G,Burgyan J,et al.Size selective recognition of siRNA by an RNA silencing suppressor[J].Cell,2003,15: 799-811.
[23] Parker J S,Parizotto E A,Wang W H,et al.Enhancement of the seed-target recognition step in RNA silencing by a PIWI/MID domain protein[J].Cell,2009,33:204-214.
[24] Zhu S,Si M L,Wu H,et al.MicroRNA-21 targets the tumor suppress or gene tropomyosin(TPM1)[J].Biol Chem,2007,282: 14328-14336.
[25] Iorio M V,Ferracin M,Liu C G,et al.MicroRNA gene expression deregulation in human breast cancer[J].Cancer Res,2005,65: 7065-7070.
The interaction simulation of miRNA 3′end with Argonaute PAZ domain by molecular dynamics
MA Zhi,ZHANG Feng,ZHANG Hui-xiao,YANG Wei
(Key Laboratory of Mariculture&Stock Enhancement in North China's Sea,Ministry of Agriculture,Dalian Ocean University,Dalian 116023,China)
Abstract:The binding model of the human miRNA with the PAZ domain was studied by molecular dynamics,and binding free energies of the miRNA-PAZ complexes were calculated via linear interaction energy(LIE).The analyses revealed that the binding formation of the miRNA with the PAZ domain and the binding free energies of the complexes were depended on the base composition of the 3′end fragment,with the stable binding of UGU to PAZ at the binding free energy of-37.248 kJ/mol,while the binding free energy between CGA and PAZ was found to be -5.418 kJ/mol.Simultaneously,the binding free energies were also found associated with distribution frequency of the miRNA gene,UGU accounting for 6.768%,and CGA accounting for 0.414%.The findings provide information with understanding of the interactions between miRNAs and Ago,the human diseases,and the biological evolution.
Key words:miRNA;PAZ domain;molecular dynamics simulation;linear interaction energy
中图分类号:O643
文献标志码:A
文章编号:2095-1388(2012)05-0441-07
收稿日期:2011-12-25
基金项目:国家自然科学基金资助项目 (30471323)
作者简介:马志 (1986-),男,硕士研究生。E-mail:mshappy1986@126.com
通信作者:张峰 (1957-),男,教授,博士。E-mail:Fengz57@gmail.com