厌氧氨氧化反应器载体生物膜细菌多样性的研究
王银华1,马悦欣1,刘长发2,朱学惠2,朱莹1
(1.大连海洋大学农业部北方海水增养殖重点实验室,辽宁大连116023;2.大连海洋大学海洋科技与环境学院,辽宁大连116023)
摘要:采用细菌16S rRNA通用引物1055F/1392R-GC获得的PCR产物进行变性梯度凝胶电泳(DGGE),分析了两个厌氧氨氧化反应器在不同运行时间其载体生物膜上的细菌多样性。结果表明,两个反应器在不同运行时间其细菌种群多样性有一定差异。DGGE优势条带序列系统发育分析结果表明,反应器载体生物膜上的细菌类群主要是陶厄氏菌属Thauera、鞘氨醇单胞菌属Sphingomonas,外硫红螺旋菌科Ectothiorhodospiraceae、酸杆菌门Chloroflexi、绿弯菌门Acidobacteria及不可培养细菌。当反应器运行208 d时,水体中氨氮和亚硝态氮的去除率维持较高水平,两者去除率之比为1.1,表明反应器内发生了厌氧氨氧化反应。针对厌氧氨氧化细菌16S rRNA基因引物Pla46F/Amx368R-GC获得的PCR产物,采用DGGE技术对载体生物膜上的厌氧氨氧化细菌进行了检测。DGGE优势条带序列分析结果表明,在反应器中富集得到的厌氧氨氧化细菌分别与Planctomycete KSU-1、Candidatus Jettenia asiatica的相似性均为96%,可以认为它们是反应器内起厌氧氨氧化作用的主要细菌。
关键词:厌氧氨氧化反应器;生物膜;PCR-DGGE;细菌多样性;厌氧氨氧化细菌
厌氧氨氧化是在厌氧条件下,微生物以NO 为电子受体,NH
为电子受体,NH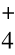 为电子供体,将NH
为电子供体,将NH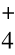 、NO
、NO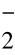 转变为N2的生化过程[1]。厌氧氨氧化工艺是一种有效的、低成本的生物脱氮新技术[2]。厌氧氨氧化采用的反应器主要有上流式厌氧污泥床(UASB)反应器[3]、流化床反应器(FBR)[4]、序批式反应器(SBR)[5]、气提反应器[5]、膜生物反应器[6]和生物转盘[7]等。厌氧氨氧化细菌属于分枝很深的浮霉菌,已鉴定出的有5属9种[8-14]。杨洋等[3]采用DGGE技术研究了厌氧氨氧化反应器污泥中细菌的多样性。Innerebner等[15]针对浮霉状菌门16S rRNA基因引物获得的PCR产物,采用DGGE技术分析了脱氨反应器内粒状和絮状污泥中细菌的多样性,发现两者的DGGE带谱不同。国内外学者使用克隆文库[3,14,16-18]技术研究了不同厌氧氨氧化反应器污泥中厌氧氨氧化菌的多样性。本研究中,作者采用PCR-DGGE技术研究了厌氧氨氧化反应器运行过程中载体生物膜上细菌及厌氧氨氧化细菌的多样性,旨在为厌氧氨氧化工艺在废水处理中的应用提供参考资料。
转变为N2的生化过程[1]。厌氧氨氧化工艺是一种有效的、低成本的生物脱氮新技术[2]。厌氧氨氧化采用的反应器主要有上流式厌氧污泥床(UASB)反应器[3]、流化床反应器(FBR)[4]、序批式反应器(SBR)[5]、气提反应器[5]、膜生物反应器[6]和生物转盘[7]等。厌氧氨氧化细菌属于分枝很深的浮霉菌,已鉴定出的有5属9种[8-14]。杨洋等[3]采用DGGE技术研究了厌氧氨氧化反应器污泥中细菌的多样性。Innerebner等[15]针对浮霉状菌门16S rRNA基因引物获得的PCR产物,采用DGGE技术分析了脱氨反应器内粒状和絮状污泥中细菌的多样性,发现两者的DGGE带谱不同。国内外学者使用克隆文库[3,14,16-18]技术研究了不同厌氧氨氧化反应器污泥中厌氧氨氧化菌的多样性。本研究中,作者采用PCR-DGGE技术研究了厌氧氨氧化反应器运行过程中载体生物膜上细菌及厌氧氨氧化细菌的多样性,旨在为厌氧氨氧化工艺在废水处理中的应用提供参考资料。
1 材料与方法
1.1 试验装置及运行参数
试验采用两个上流式生物滤池(UBF)反应器,编号为1号、2号,其反应装置见图1。反应器为圆柱形,有效体积为6 L。1号反应器以聚乙烯塑料小球为载体,2号反应器以无纺布为载体。将大连凌水污水处理厂生化池活性污泥接种于反应器中,进水为人工模拟废水,采用NaOH调节pH为7.5~8.5。循环水浴温度为(33±0.5)℃,水力停留时间(HRT)为1 d,试验过程中充氮气除氧。1号、2号反应器于2009年3月5日启动,1号反应器中直接培养厌氧氨氧化菌,2号反应器中先采用反硝化生物膜培养而后富集培养厌氧氨氧化菌。
模拟废水有以下两种:1)反硝化培养的模拟废水包括KH2PO40.03 g/L,KHCO30.5 g/L, MgSO4·7H2O 0.2 g/L,FeCl30.1 g/L,CaCl20.03 g/L,以NaNO3和CH3COONa分别作为电子受体和电子供体;2)厌氧氨氧化培养的模拟废水包括
KHCO30.5 g/L,KH2PO40.027 g/L,MgSO4·7H2O 0.3 g/L,CaCl20.18 g/L,微量元素Ⅰ、Ⅱ各1.25 mL/L,以NaNO2和NH4Cl分别提供电子受体和电子供体[4]。其中微量元素Ⅰ包括EDTA 5 g/L, FeSO45 g/L;微量元素Ⅱ包括EDTA 15 g/L, ZnSO4·7H2O 0.43 g/L,CoCl2·6H2O 0.24 g/L, MnCl2·4H2O 0.99 g/L,CuSO4·5H2O 0.25 g/L, Na2MoO4·2H2O 0.22 g/L,NiCl2·6H2O 0.19 g/L,Na2SeO4·10H2O 0.21 g/L,H3BO40.014 g/L[19]。
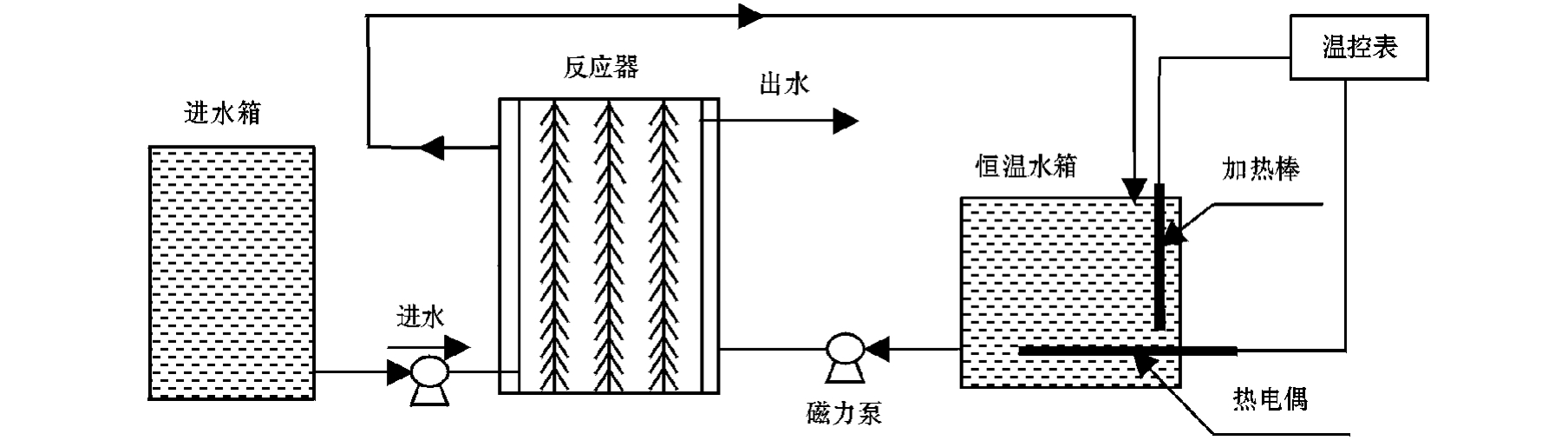
图1 UBF反应器的结构
Fig.1 Structure of the UBF reactor
1.2 方法
1.2.1 化学指标的测定 氨氮、亚硝态氮采用德国SEAL公司生产的流动注射分析仪(Bran Luebbe Auto Analyzer 3)测定。氨氮或亚硝态氮去除率= [(进水氨氮或亚硝态氮浓度-出水氨氮或亚硝态氮浓度)/进水氨氮或亚硝态氮浓度]×100%。
1.2.2 生物膜总DNA的提取和PCR扩增 于反应器运行第88天和第208天,从生物膜表层5~10 cm处取约4 g的样品加入10 mL无菌水中,用力震荡20 min。取振荡液加入1.5 mL微量离心管中,离心,弃上清液,使用SDS裂解法抽提沉积物中的DNA。用Ferris等[20]描述的细菌16S rRNA基因通用引物1055F/1392R-GC对1号、2号反应器运行第88天和第208天的样品总DNA进行PCR扩增;采用厌氧氨氧化菌特异引物Pla46F/ Amx368R-GC对1号、2号反应器运行第208天的样品总DNA进行PCR扩增[13,21],两者的反应体系和反应程序相同。50 μL反应体系包括:5 μL 10× PCR Buffer,4 μL dNTPs,各2 μL(20 mmol/L)引物,2.5 U Taq酶,2 μL DNA模板,34 μL ddH2O。反应程序为:94℃下预变性2 min;94℃下变性30 s,57℃下退火1 min,72℃下延伸1 min,共进行28个循环;最后在72℃下延伸10 min。用10 g/L的琼脂糖凝胶电泳检测PCR产物的大小。
1.2.3 DGGE分析 采用Bio-Rad公司D-code通用突变检测系统对PCR产物进行DGGE分析。将PCR反应产物(质量分数为8%)直接上样于含体积分数为35%~60%(第88天样品)或35%~50%(第208天样品)梯度变性剂聚丙烯酰胺凝胶中,在TAE中电泳5 h(温度为60℃,电压为200 V),然后采用银染方法染色,并用凝胶成像仪进行观察。
1.2.4 克隆、测序及系统发育分析 切下DGGE优势条带浸泡于50 μL无菌水中,4℃下过夜。取2 μL作为模板,细菌、厌氧氨氧化菌分别用引物1055F/1392R、Pla46F/Amx368R进行PCR扩增,反应体系和反应程序同“1.2.2”。PCR产物用15 g/L的琼脂糖凝胶电泳进行检测,并切胶纯化。纯化后的PCR产物与pMD18-T载体连接,并转化到E.coli DH5α感受态细胞中,在含有X-gal、IPTG和Amp的LB平板上培养16 h(37℃);将蓝白斑筛选阳性转化子接种于LB培养基中,37℃下振荡过夜,以培养的菌液为模板,用引物RV-M/MB -47进行PCR反应检测阳性克隆,将阳性克隆产物送北京天根生化科技有限公司测序。将所测DNA序列在GenBank数据库中进行检索,从Blast比对结果中取相似性最近的序列。将DGGE条带序列与从GenBank数据库中获得的相似序列,用系统发生推断软件包Mega 4.0进行统计和聚类分析[22]。采用邻接法(neighbor-joining method)获得分支系统树,并通过自举分析(boostrap)进行置信度检测,自举数据集为1 000次。
2 结果
2.1 总DNA的提取和PCR扩增
从图2可见,反应器中样品总DNA条带较亮,
无拖带现象。
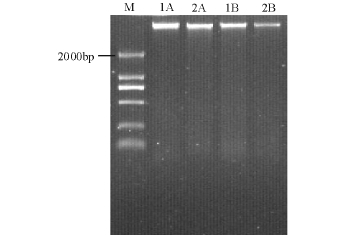
图2 1号、2号反应器运行88 d(1A、2A)和208 d (1B、2B)的样品总DNA的琼脂糖凝胶电泳
Fig.2 Agarose gel electrophoresis of DNA extraction from samples collected on 88th(1A,2A)and 208th(1B,2B)from reactor 1 and reactor 2
使用通用引物1055F/1392R-GC和厌氧氨氧化菌特异性引物Pla46F/Amx368R-GC分别对总DNA进行PCR扩增,将扩增产物用10 g/L的琼脂糖凝胶电泳进行检测,结果见图3和图4,通过与Marker对比,可知目标片段长度分别约为350 bp和370 bp,其条带较亮,无拖带现象,说明PCR扩增效果良好。
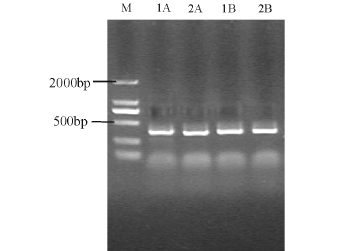
图3 1号、2号反应器运行88 d(1A、2A)和208 d (1B、2B)的样品总DNA用引物1055F/1392RGC获得的PCR扩增产物的琼脂糖凝胶电泳
Fig.3 Agarose gel electrophoresis of PCR products amplified with primers 1055F/1392R-GC from samples collected on 88th(1A,2A)and 208th(1B,2B)from reactor 1 and reactor 2
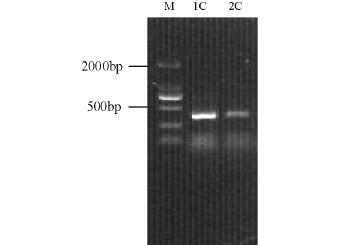
图4 1号、2号反应器运行208 d的样品用Pla46F/ Amx368R-GC获得的PCR产物琼脂糖凝胶电泳(1C,2C)
Fig.4 Agarose gel electrophoresis of PCR products amplified with primers Pla46F/Amx368R-GC from samples collected on 208th(1C,2C) from reactor 1 and reactor 2
2.2 DGGE分析
两个反应器载体生物膜样品的DGGE结果见图5、图6。可以看出,反应器运行88 d,生物膜中的细菌多样性(DGGE条带数)较高(图5-a),但两个反应器中的细菌种群组成有一定差异,此时出水氨氮和亚硝态氮虽然能同时被去除(1号反应器氨氮和亚硝态氮的去除率分别为35%、27%,2号的为25%、7%),但去除量少且两者去除量之比没有规律。反应器运行208 d(图5-b),生物膜中的细菌多样性有所减少,同一反应器不同时间段的细菌区系也有所不同,这时1号反应器氨氮和亚硝态氮的去除率分别为86%、99%,2号的为82%、90%,两者去除率之比约为1.1。通过厌氧氨氧化菌的检测(图6),结果发现,反应器内有3种起厌氧氨氧化作用的细菌。
对DGGE优势条带序列的Blast分析结果见表1。将所测序列与已知的相似序列用Mega 4.0软件进行聚类分析,采用邻接法构建了生物膜中优势细菌的系统发育树(图7)。DGGE优势条带序列系统发育分析结果表明,反应器载体生物膜上的细菌类群主要是陶厄氏菌属Thauera、鞘氨醇单胞菌属Sphingomonas,外硫红螺旋菌科、酸杆菌门、绿弯菌门及不可培养的细菌。在反应器中富集得到的厌氧氨氧化细菌分别与Planctomycete KSU-1和Candidatus Jettenia asiatica的相似性均为96%。
3 讨论
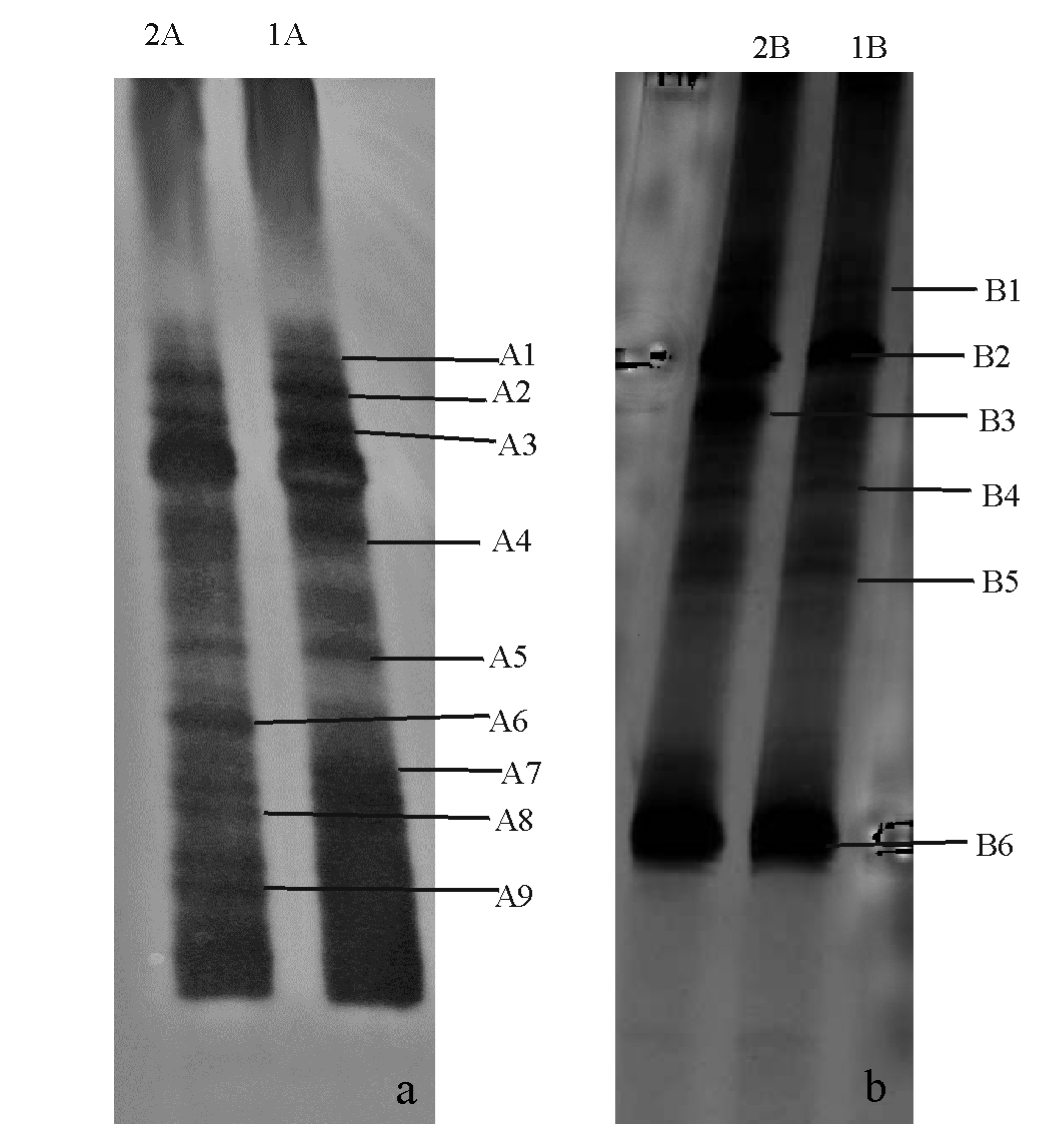
图5 1号、2号反应器运行88 d(a)和208 d(b)的生物膜样品总DNA用引物1055F/1392R-GC获得的PCR扩增产物的DCGE图谱
Fig.5 DGGE profile of PCR amplified 16S rRNA gene fragments of biofilm samples with primers 1055F/ 1392R-GC collected on 88th(a),and 208th(b) from reactor 1 and reactor 2
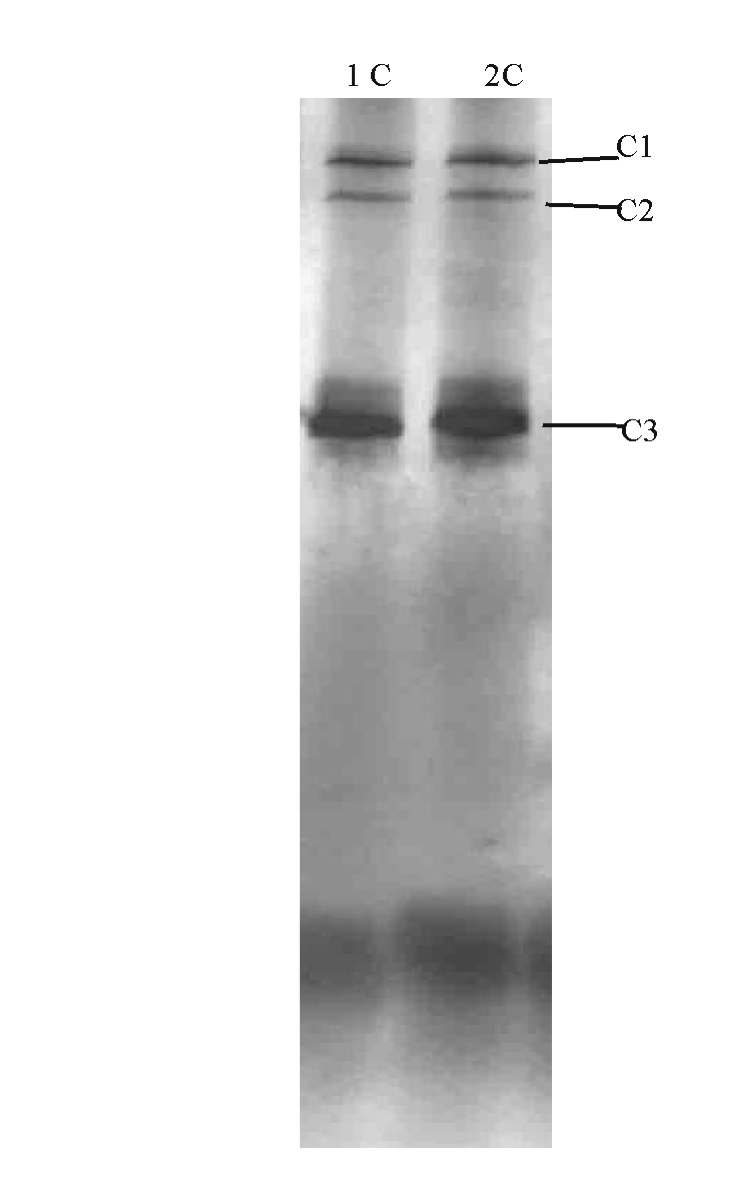
图6 1号、2号反应器运行208 d的生物膜样品总DNA用引物Pla46F/Amx368R-GC获得的PCR扩增产物的DCGE图谱
Fig.6 DGGE profile of PCR amplified 16S rRNA gene fragments of biofilm samples with primers Pla46F/ Amx368R-GC collected on 208thfrom reactor 1 and reactor 2
表1 DGGE条带序列的Blast结果
Tab.1 The blast analysis of the sequences of DGGE bands
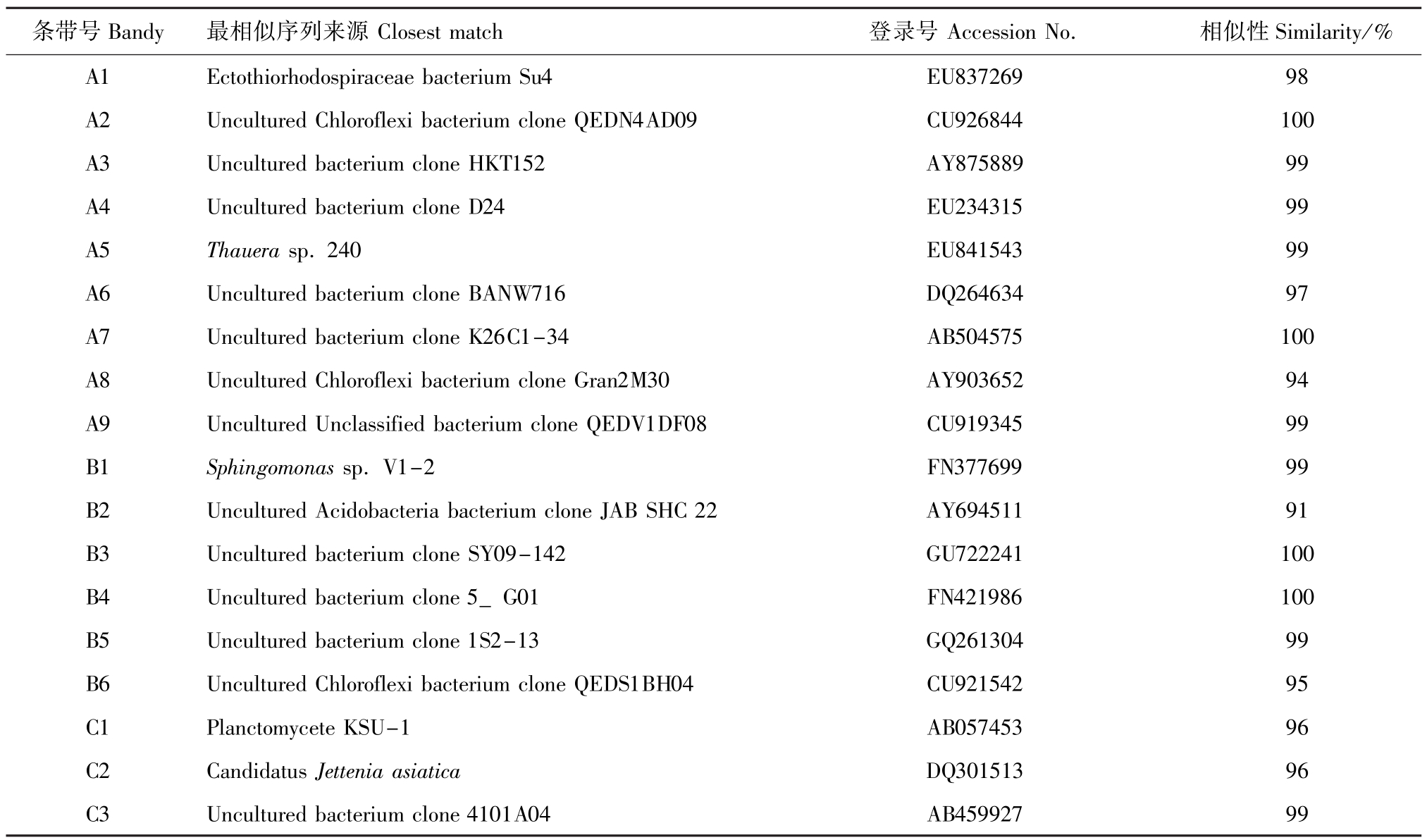
条带号Bandy最相似序列来源Closest match登录号Accession No.相似性Similarity/% A1Ectothiorhodospiraceae bacterium Su4EU83726998 A2Uncultured Chloroflexi bacterium clone QEDN4AD09CU926844100 A3Uncultured bacterium clone HKT152AY87588999 A4Uncultured bacterium clone D24EU23431599 A5Thauera sp.240EU84154399 A6Uncultured bacterium clone BANW716DQ26463497 A7Uncultured bacterium clone K26C1-34AB504575100 A8Uncultured Chloroflexi bacterium clone Gran2M30AY90365294 A9Uncultured Unclassified bacterium clone QEDV1DF08CU91934599 B1Sphingomonas sp.V1-2FN37769999 B2Uncultured Acidobacteria bacterium clone JAB SHC 22AY69451191 B3Uncultured bacterium clone SY09-142GU722241100 B4Uncultured bacterium clone 5_G01FN421986100 B5Uncultured bacterium clone 1S2-13GQ26130499 B6Uncultured Chloroflexi bacterium clone QEDS1BH04CU92154295 C1Planctomycete KSU-1AB05745396 C2Candidatus Jettenia asiaticaDQ30151396 C3Uncultured bacterium clone 4101A04AB45992799
本研究中采用污水处理厂的污泥作为接种物,通过直接培养厌氧氨氧化菌,以及先培养反硝化生物膜后再富集培养厌氧氨氧化菌,启动上流式生物
滤池反应器。反应器运行88 d时,载体生物膜中的细菌多样性较高,但两个反应器中的细菌种群有一定差异,可能与启动方法不同有关;反应器运行208 d时,生物膜中的细菌多样性有所减少,同一反应器不同时间段的细菌区系也有所不同,可能是随着反应器运行时间的推移,某些种类的细菌不适应反应器的运行条件而死亡。这时从反应器的外观看,反应器内污泥的颜色由最初的黑色变为红褐
色,充分表明厌氧氨氧化逐渐成为反应器的主导反应。通过检测发现,反应器内确实存在厌氧氨氧化细菌。
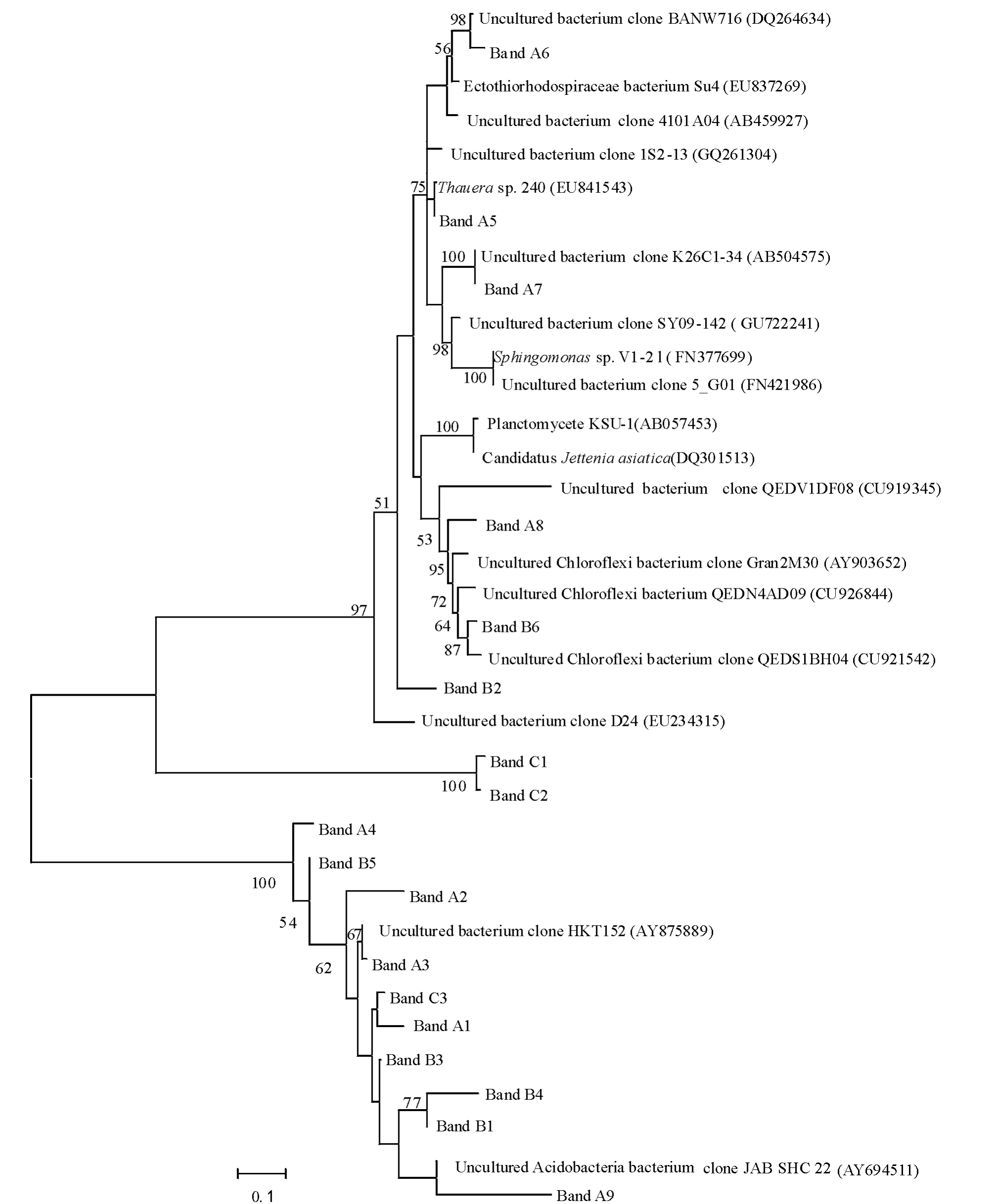
图7 根据所测序列和Blast结果构建的反应器载体生物膜细菌系统发育树
Fig.7 Phylogenetic tree of the bacteria in boifilms associated with media of two anammox bioreactors based on the results of Blast of DGGE bands and the sequences
DGGE图谱和优势条带序列分析结果表明:条带A1为1号反应器特有,与外硫红螺旋菌科细菌Su4的相似性为98%;条带A2、A8、B6与不可培养的绿弯菌门细菌克隆的相似性为94%~100%。研究表明:绿弯菌门是厌氧污泥消化器中的主要微生物[23],普遍存在于生物废水处理厂的活性污泥中[24],也出现于圆柱形上流式厌氧反应器中[16];在上流式颗粒床厌氧氨氧化反应器中也发现类似绿弯菌门的丝状细菌[25];秦玉洁等[18]用传统方法从试验的两套厌氧氨氧化反应器中分离到一种绿弯菌。本研究中,条带A5的序列与陶厄氏菌Thauera sp.240 bp序列的相似性为99%,该菌属于反硝化细菌。DGGE和16S rRNA基因文库分析结果表明,在去除喹啉的反硝化反应器中,陶厄氏菌属的细菌起着重要作用[26]。Mao等[27]从焦化废水处理厂的反硝化生物反应器中也可分离到若干陶厄氏菌。条带B1与鞘氨醇单胞菌Sphingomonas sp.V1-2的相似性为99%。Lu等[28]用培养方法从一厌氧/好氧SBR反应器中分离到鞘氨醇单胞菌属的细菌。Egli等[29]结合16S rDNA克隆文库、荧光原位杂交(FISH)和斑点杂交技术研究分析了无有机碳富氨废水的生物转盘生物膜的微生物组成,发现鞘氨醇单胞菌、氨氧化细菌Nitrosomonas和厌氧氨氧化细菌Candidatus Kuenenia stuttgartiensis等的存在。条带B2与不可培养的酸杆菌门细菌克隆JAB SHC 22的相似性为91%。Li等[16]用构建浮霉状菌门的特定16S rRNA基因文库分析了厌氧氨氧化反应器中的细菌多样性,发现反应器中除了有厌氧氨氧化细菌外,还存在酸杆菌门等非浮霉状菌门的细菌。
条带C1与Planctomycete KSU-1的相似性为96%,KSU-1菌株被鉴定为以无纺布为载体的生物膜厌氧氨氧化反应器中的优势菌种[30]。条带C2与已知厌氧氨氧化细菌Candidatus Jettenia asiatica的相似性为96%。Quan等[14]通过建立浮霉状菌目Planctomycetales的特定16S rRNA基因文库分析了粒状(0.6~1.0 mm)污泥厌氧氨氧化反应器中厌氧氨氧化细菌的多样性,发现了一种在反应器中占50%的新的厌氧氨氧化细菌,并命名为Candidatus Jettenia asiatica。其余条带均与数据库中主要来源于环境样品的不可培养细菌克隆序列有较高的相似性(97%~100%),这意味着厌氧氨氧化反应器中可能存在大量新的种类(phylotypes)。
本研究结果表明,反应器运行稳定时,厌氧氨氧化细菌和其它异养细菌共同存在于生物膜中,这些共同存在的异养细菌的具体生理作用和功能目前还不清楚,有待进一步研究。
参考文献:
[1] Jetten M S,Wagner M,Fuerst J,et al.Microbiology and application of the anaerobic ammonium oxidation(anammox)process[J]. Current Opinion in Biotechnology,2001,12(3):283-288.
[2] Grommen R,Verstraete W.Environmental biotechnology:the ongoing quest[J].Journal of Biotechnology,2002,98(1):113-123.
[3] 杨洋,左剑恶,全哲学,等.UASB反应器中厌氧氨氧化污泥的种群分析[J].中国环境科学,2006,26(1):52-56.
[4] Van de Graaf A A,de Bruijn P,Robertson L A,et al.Autotrophic growth of anaerobic ammonium-oxidizing microorganisms in a fluidized bed reactor[J].Microbiology,1996,142(8):2187-2196.
[5] Dapena-Mora A,Campos J L,Mosquera-Corral A,et al.Stability of the annammox process in a gas-lift reactor and a SBR[J].Journal of Biotechnology,2004,110(2):159-170.
[6] Wang T,Zhang H M,Yang F L,et al.Start-up of the Anammox process from the conventional activated sludge in a membrane bioreactor[J].Bioresource Technology,2009,100(9):2501-2506.
[7] Egli K,Fanger U,Alvarez P J,et al.Enrichment and characterization of an anammox bacterium from a rotating biological contactor treating ammonium-rich leachate[J].Archives of Microbiology, 2001,175(3):198-207.
[8] Kuenen J G,Jetten M S M.Extraordinary anaerobic ammonium-oxidizing bacteria[J].American Society for Microbiology News, 2001,67:456-463.
[9] Schmid M,Twachtmann U,Klein M,et al.Molecular evidence for genus level diversity of bacteria capable of catalyzing anaerobic ammonium oxidation[J].Systematic and Applied Microbiology,2000, 23(1):93-106.
[10] Kartal B J,Rattray L A.Candidatus“Anammoxoglobus propionicu”a new propionate oxidizing species of anaerobic ammonium oxidizing bacteria[J].Systematic and Applied Microbiology,2007 (30):39-49.
[11] Gong Z,Yang F,Liu S,et al.Feasibility of a membrane aerated biofilm reactor to achieve single-stage autotrophic nitrogen removal based on anammox[J].Chemosphere,2007,69:776-784.
[12] Kuypers M M M,Slikers A O,Lavik G,et al.Anaerobic ammonium oxidation by anammox bacteria in the Black Sea[J].Nature, 2003,422:608-611.
[13] Schmid T,Walsh M K.Candidatus“Scalindua brodae”sp.nov., Candidatus“Scalindua wagneri”sp.nov.,two new species of anaerobic ammonium oxidizing bacteria[J].Systematic and Applied Microbiology,2003,26(4):529-538.
[14] Quan Z X,Rhee S K,Zuo J E,et al.Diversity of ammonium-oxidizing bacteria in a granular sludge anaerobic ammonium-oxidizing (anammox)reactor[J].Environmental Microbiology,2008,10 (11):3130-3139.
[15] Innerebner G,Insam H,WhittleI H,et al.Identification of anammox bacteria in a ful1-scale deammonification plant making use of anaerobic ammonia oxidation[J].Systematic and Applied Microbiology,2007,30(5):408-412.
[16] Li X R,Du B,Fu H X,et al.The bacterial diversity in an anaerobic ammonium-oxidizing(anammox)reactor community[J].Systematic and Applied Microbiology,2009,32(4):278-289.
[17] Date Y,Isaka K,Ikuta H,et al.Microbial diversity of anammox bacteria enriched from different types of seed sludge in an anaerobic continuous-feeding cultivation reactor[J].Journal of Bioscience and Bioengineering,2009,107(3):281-286.
[18] 秦玉洁,周少奇,朱明石.厌氧氨氧化反应器微生态的研究[J].环境科学,2008,29(6):1638-1643.
[19] Strous M,Fuerst J A,Kramer E H M,et al.Missing lithotroph identified as new planctomycete[J].Nature,1999,400:446-449.
[20] Ferris M J,Muyzer G,Ward D M.Denaturing gradient gel electrophoresis profiles of 16S rRNA-defined populations inhabiting a hot spring microbial mat community[J].Applied and Environmental Microbiology,1996,62(2):340-346.
[21] Neef A R,Schlesner A H,Schleifer K H.Monitoring a widespread bacterial group:in situ detection of planctomycetes with 16S rRNA -targeted probes[J].Microbiology,1998,144:3257-3266.
[22] Tamura K,Dudley J,Nei M,et al.Mega4:Molecular evolutionary genetics analysis(MEGA)software version 4.0[J].Molecular Biology and Evolution,2007,24(8):1596-1599.
[23] Rivière D,Desvignes V,Pelletier E,et al.Towards the definition of a core of microorganisms involved in anaerobic digestion of sludge[J].ISME Journal,2009,3(6):700-714.
[24] Lovisa B,Philip H,Gene W T.Filamentous Chloroflexi(green non-sulfur bacteria)are abundant in wastewater treatment processes with biological nutrient removal[J].Microbiology,2002,148: 2309-2318.
[25] Cho S,Takahashi Y,Fujii N,et al.Nitrogen removal performance and microbial community analysis of an anaerobic up-flow granular bed anammox reactor[J].Chemosphere,2010,78:1129-1135.
[26] Liu B,Zhang F,Feng X,et al.Thauera and Azoarcusas functionally important genera in a denitrifying quinoline-removal bioreactor as revealed by microbial community structure comparison[J]. FEMS Microbiology Ecology,2006,55(2):274-286.
[27] Mao Y,Zhang X,Zhang B,et al.Specific-PCR and denaturing gradient gel electrophoresis assistant isolation of Thauera spp.from a coking wastewater treatment plant[J].Acta Microbiologica Sinica,2008,48(12):1634-1641.
[28] Lu S P,Park,M J,Ro H S,et al.Analysis of microbial communities using culture-dependent and culture-independent approaches in an anaerobic/aerobic SBR reactor[J].The Journal of Microbiology,2006,44(2):155-161.
[29] Egli K,Bosshard F,Werlen C,et al.Microbial composition and structure of a rotating biological contactor biofilm treating ammonium-rich wastewater without organic carbon[J].Microbial Ecology,2003,45:419-432.
[30] Fujii T,Sugino H,Rouse J,et al.Characterization of the microbial community in an anaerobic ammonium-oxidizing biofilm cultured on a nonwoven biomass carrier[J].Journal of Bioscience and Bioengineering,2002,94:412-418.
The bacterial diversity in boifilms associated with media of anammox bioreactors
WANG Yin-hua1,MA Yue-xin1,LIU Chang-fa2,ZHU Xue-hui2,ZHU Ying1
(1.Key Laboratory of North Mariculture,Ministry of Agriculture,Dalian Ocean University,Dalian 116023,China;2.College of Marine Science and Environment,Dalian Ocean University,Dalian 116023,China)
Abstract:The bacterial diversity was studied in boifilms associated with media of two anammox bioreactors running at different time by PCR-DGGE using bacterial 16S rRNA universal primers 1055F/1392R-GC.Results showed that the bacterial diversity was different in two reactors at different sampling time.Phylogenetic analysis of sequences from prevalent DGGE bands revealed that the bacteria were found to be Thauera,Sphingomonas,Ectothiorhodospiraceae,Chloroflexi,Acidobacteria and uncultured bacteria.When the bioreactors were operated for 208 days,the removal rates of ammonia and nitrite nitrogens were kept high with the ratio of ammonia nitrogen level to nitrite nitrogen level=1.1,indicating that the anammox reaction took place in the bioreactors.Anammox bacteria in the biofilms associated with media of bioreactors were detected by PCR-DGGE using anammox bacteria specific primers Pla46F/Amx368R-GC.Analysis of sequences from prevalent DGGE bands showed anammox bacteria had 96%similarity to Planctomycete KSU-1 and Candidatus Jettenia asiatica,respectively.It is suggested that these bacteria remove nitrogen by anaerobic ammonium-oxidation in reactors.
Key words:anaerobic ammonium oxidation reactor;biofilm;PCR-DGGE;anaerobic ammonium oxidation bacterium;anammox bacterium
中图分类号:X172
文献标志码:A
文章编号:2095-1388(2011)06-0500-07
收稿日期:2011-01-20
基金项目:国家“863”计划项目(2007AA10Z410)
作者简介:王银华(1985-),女,硕士研究生。E-mail:414795838@qq.com
通信作者:马悦欣(1963-),女,博士,教授。E-mail:mayuexin@dlou.edu.cn
 为电子受体,NH
为电子受体,NH 为电子供体,将NH
为电子供体,将NH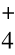 、NO
、NO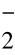 转变为N2的生化过程[1]。厌氧氨氧化工艺是一种有效的、低成本的生物脱氮新技术[2]。厌氧氨氧化采用的反应器主要有上流式厌氧污泥床(UASB)反应器[3]、流化床反应器(FBR)[4]、序批式反应器(SBR)[5]、气提反应器[5]、膜生物反应器[6]和生物转盘[7]等。厌氧氨氧化细菌属于分枝很深的浮霉菌,已鉴定出的有5属9种[8-14]。杨洋等[3]采用DGGE技术研究了厌氧氨氧化反应器污泥中细菌的多样性。Innerebner等[15]针对浮霉状菌门16S rRNA基因引物获得的PCR产物,采用DGGE技术分析了脱氨反应器内粒状和絮状污泥中细菌的多样性,发现两者的DGGE带谱不同。国内外学者使用克隆文库[3,14,16-18]技术研究了不同厌氧氨氧化反应器污泥中厌氧氨氧化菌的多样性。本研究中,作者采用PCR-DGGE技术研究了厌氧氨氧化反应器运行过程中载体生物膜上细菌及厌氧氨氧化细菌的多样性,旨在为厌氧氨氧化工艺在废水处理中的应用提供参考资料。
转变为N2的生化过程[1]。厌氧氨氧化工艺是一种有效的、低成本的生物脱氮新技术[2]。厌氧氨氧化采用的反应器主要有上流式厌氧污泥床(UASB)反应器[3]、流化床反应器(FBR)[4]、序批式反应器(SBR)[5]、气提反应器[5]、膜生物反应器[6]和生物转盘[7]等。厌氧氨氧化细菌属于分枝很深的浮霉菌,已鉴定出的有5属9种[8-14]。杨洋等[3]采用DGGE技术研究了厌氧氨氧化反应器污泥中细菌的多样性。Innerebner等[15]针对浮霉状菌门16S rRNA基因引物获得的PCR产物,采用DGGE技术分析了脱氨反应器内粒状和絮状污泥中细菌的多样性,发现两者的DGGE带谱不同。国内外学者使用克隆文库[3,14,16-18]技术研究了不同厌氧氨氧化反应器污泥中厌氧氨氧化菌的多样性。本研究中,作者采用PCR-DGGE技术研究了厌氧氨氧化反应器运行过程中载体生物膜上细菌及厌氧氨氧化细菌的多样性,旨在为厌氧氨氧化工艺在废水处理中的应用提供参考资料。






