基于两个核基因序列研究龟鳖类的系统进化特征
郑将臣1、3,万全2,程起群1,赵金良3
(1.中国水产科学研究院东海水产研究所,农业部海洋与河口渔业资源及生态重点开放实验室,上海200090;2.安徽农业大学动物科技学院,安徽合肥230036;3.上海海洋大学农业部水产种质资源与利用重点开放实验室,上海201306)
摘要:为从核基因的角度探讨龟鳖类的系统发育特征,采用PCR扩增和测序的方法,获得两种龟的R35内含子部分序列以及6种龟的RAG2基因部分序列,结合NCBI其它龟鳖的同源序列一并进行分析。结果表明:将R35序列比对后得到941 bp的一致序列,将RAG2比对后得到620 bp的一致序列,二者合并后,比对排序后得到1561 bp的一致序列,其中共有505个可变核苷酸位点,总变异率为32.35%;简约信息位点为239个,插入/缺失为139个,转换/颠换比率为1.64。碱基A、T、G、C的平均含量分别为29.5%、28.5%、22.8%、19.2%。海龟科和鳄龟科之间Kimura双参数(K-2-P)遗传距离最小(0.025),鳖科和南美侧颈龟科之间的遗传距离最大(0.182)。分子系统树结果显示:1)陆龟科与潮龟科先聚在一起,再与龟科聚在一起,陆龟科与潮龟科构成一个单系类群;2)支持陆龟总科与鳄龟科+海龟总科构成姐妹群; 3)鳄龟科和海龟总科是姐妹群的关系。
关键词:龟鳖类;R35基因;RAG2基因;遗传趋异;分子分类;系统进化
全世界现有龟鳖类动物约14科、324种[1]。目前,国外关于龟鳖目分子系统学的研究较多[2-10],国内对这方面的研究相对较少,且大多数是基于龟鳖类线粒体的研究[11-15],而用核基因来分析龟鳖类系统进化的研究鲜见报道。RAG2是重组激活基因(Recombinationactivatinggenes, RAGs)的一种。RAGs在脊椎动物中普遍存在,在进化过程中非常保守,不同物种之间其碱基组成变化很少,近年来被较多地运用于物种进化和亲缘关系的分析[16-20]。RNA指纹蛋白35(RNA fingerprint protein 35,R35)也是一种核内基因,目前关于这个蛋白的功能尚不明确,但是RNA指纹蛋白35基因被认为是单拷贝基因[21],因此,国外已有若干学者运用RNA指纹蛋白35基因作为标记来研究龟鳖类动物的系统进化[7,22]。
本研究中,作者测定了两种龟的R35内含子序列和6种龟的RAG2部分序列,并结合NCBI上其它龟鳖类的相关序列,以此来初步探讨龟鳖类的系统进化特征。
1 材料与方法
1.1 材料
在安徽省长江水生动物保护中心采集6种龟的样品,每种龟各取1只个体的肌肉组织保存于冰箱(-20℃)中备用。龟鳖类分类地位均依据国际龟类动物分类工作组2009年修订的最新分类系统[1]。
1.2 方法
1.2.1 基因组DNA提取、PCR扩增和测序 基因组DNA采用标准的酚/氯仿法提取[23],略作修改。R35基因扩增采用引物R35EX1和R35EX2[22],序列分别为
5'-ACGATTCTCGCTGATTCTTGC-3',
5'-GCAGAAAACTGAATGTCTCAAGG-3'。
RAG2基因扩增采用引物F2(RAG2)和R2-1 (RAG2)[24],序列分别为
5'-CAGGATGGACTTTCTTTCCATGT-3',
5'-CAGTTGAATAGAAAGGAACCCAAGT-3'。
PCR反应在50 μL体系中进行,包括10×PCR Buffer(Mg2+Plus)5 μL,dNTP Mixture(各2.5 mmol/L)2 μL,引物(10 μmol/L)各2 μL,Taq酶2.5 U,模板DNA 40 ng。反应条件为:94℃下预变性5 min;94℃下变性45 s,退火(R35和RAG2分别为58℃和51℃)45 s,72℃下延伸60 s,共进行40个循环;最后于72℃下再延伸7 min。PCR产物经割胶纯化后在ABI3730型DNA测序仪上双向测序。
1.2.2 数据分析 对所测得的序列进行拼接,再用Clustal X 1.83[25]、DnaSP[26]、MEGA 4.1[27]等软件对所有序列进行比对,计算其多态位点数、简约信息位点数、碱基组成和转换/颠换比率等遗传信息指数,并基于Kimura双参数模型(Kimura-2 -parameter,K-2-P)[28]计算种、属、科间的遗传距离。为了进一步全面研究龟鳖类的系统发生关系,从NCBI下载龟鳖类的R35和RAG2序列(表1)一并分析。
分别对R35和RAG2序列进行系统进化分析,再将二者合并进行分析。采用PAUP4.0bl0软件中的PHT(partition homogeneity test)方法对R35和RAG2序列的数据进行同质性检验[29]。
为了判断这些序列是否适合系统进化分析,用两种方法对序列的饱和度进行检测。一种是基于转换和颠换与K-2-P的作图分析[28];另一种是运用指数Iss检测[30]。两种检测均在DAMBE软件[31]上进行。
采用邻接法(Neighbor-joining,NJ)、最大简约法(Maximum-parsimony,MP)和最大似然法(Maximum likelihood,ML)构建分子系统树。NJ树用MEGA软件构建,遗传距离模型选择Kimura双参数模型,将序列中的转换和颠换位点均视为信息位点并对所有位点一致性加权,对于序列中的插入/缺失位点则采用成对删除。MP树在PAUP4.0bl0软件[32]上进行,选择简约标准(Parsimony criterion)、启发式搜寻(Heuristic search)、树二分再连接(Tree bisection reconnection,TBR)、逐步加入法随机加入序列(10 000 random stepwise addition sequence replicates),所有数据均未加权。当几个相同的简约树出现时,最后结果将由严格一致树来表示。在ML树构建之前,采用Modeltest软件[33]确定序列最适合的进化模式,ML树也用PAUP4.0bl0软件构建,参数与MP法相同。采用Bootstrap检验分子系统树各节点的置信值(其中NJ和MP树重复1 000次,ML树重复100次)。NJ树、MP树和ML树拓扑结构的一致性用Templeton检验[34]评估。
2 结果
2.1 PCR扩增及序列分析
本研究中成功扩增并测序了两种龟的R35内含子部分序列(长度为1 080 bp)以及6种龟的RAG2基因部分序列(长682 bp),所有序列均已提交GenBank数据库(登录号见表1,用*示出)。其中,RAG2基因682 bp的核苷酸序列共编码227个氨基酸,其中有59个发生变异。
将从NCBI上下载的其他龟鳖类R35序列和RAG2序列一并分析(表1),经比对后,R35的一致序列为941 bp,RAG2一致序列为620 bp,二者合并后得到1 561 bp的一致序列,其中共有505个可变核苷酸位点,总变异率为32.35%;简约信息位点有239个,单变异多态位点有266个,插入/缺失为139个。碱基A、T、G、C的平均含量分别为29.5%、28.5%、22.8%、19.2%。A+T含量为58%,高于G+C含量42%。在505个核苷酸位点中,转换数为54,颠换数为33,转换/颠换比率(R)为1.64。龟鳖目9个科间的K-2-P遗传距离见表2,其中海龟科和鳄龟科之间的遗传距离最小,为0.025;鳖科和南美侧颈龟科之间的遗传距离最大,为0.182。PHT检验显示,P=0.02>0.01,因此R35和RAG2序列可以合并。Modeltest检测结果显示,TVM+G为最佳模型,该模型的相关参数为:-ln L=6 274.6504,K=8,AIC= 12 565.3008,伽马分布的形状参数(Gamma distribution shape parameter)=1.2752。
基于转换和颠换与遗传距离的线性关系分析碱基替代饱和的结果,显示碱基转换和颠换在K-2-P遗传距离小于0.195的范围内均未达到替代饱和(图1)。Iss的指数检测结果显示,在两尾t检验下,P=0.0000,Iss值为0.1328,极端对称树临界值Iss.c为0.7620,极端非对称树临界值Iss.c为0.5599,则Iss<Iss.c,表明碱基替代未饱和。两种检验方法都显示这些序列碱基转换和颠换均未达到替代饱和,因此可用于构建进化树。
2.2 系统发生分析
Templeton检验显示,基于R35/RAG2序列的NJ树、MP树和ML树拓扑结构基本一致(图2)。分子系统树显示,根据传统形态学划分的各科成员
均以较高的支持率聚在一起,说明基于R35/RAG2序列的系统树能将各科清晰明确的区分开。其中潮龟科和陆龟科汇聚成一支后,再与龟科构成姐妹群
(支持率大于95%)。棱皮龟科与海龟科聚在一起,尔后再与鳄龟科汇聚成一支,说明这三个科之间可能有较近的亲缘关系。鳖科则位于树的基部,推测鳖科可能是曲颈龟亚目中分化较早的科之一。
表1 24种龟及其R35和Rag2序列登陆号
Tab.1 The R35&Rag2 accession numbers of 24 turtle species
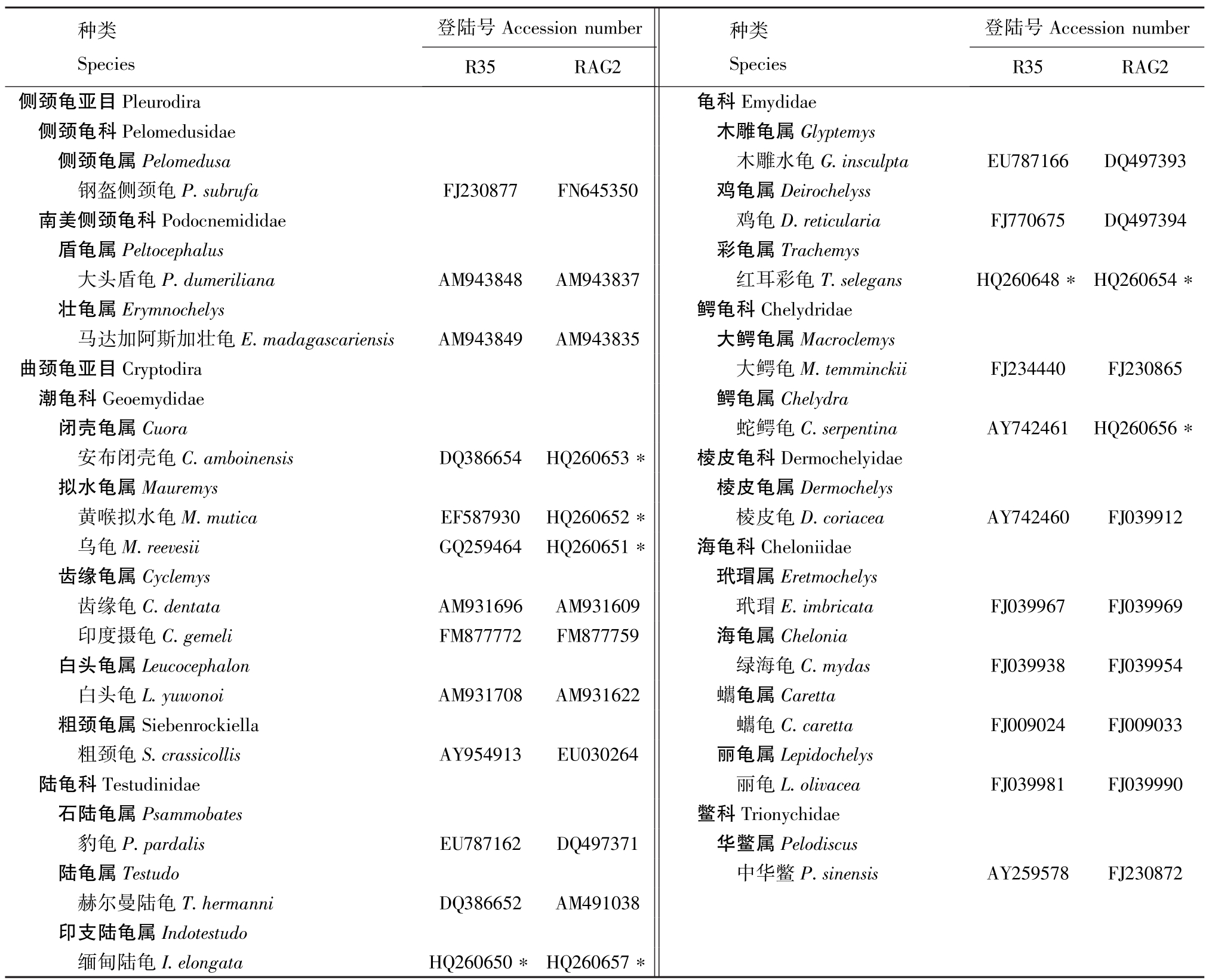
注:*为本研究中所获得的序列。
Notes:*means the sequences obtained in this study.
登陆号登陆号Accession number Accession number R35RAG2侧颈龟亚目Pleurodira龟科种类Species R35RAG2种类Species Emydidae侧颈龟科Pelomedusidae 木雕龟属Glyptemys侧颈龟属Pelomedusa 木雕水龟G.insculpta Deirochelyss南美侧颈龟科Podocnemididae 鸡龟D.reticularia EU787166DQ497393钢盔侧颈龟P.subrufaFJ230877FN645350 鸡龟属FJ770675DQ497394盾龟属Peltocephalus 彩龟属Trachemys大头盾龟P.dumerilianaAM943848AM943837 红耳彩龟T.selegansHQ260648*HQ260654*壮龟属Erymnochelys鳄龟科Macroclemys曲颈龟亚目Cryptodira 大鳄龟M.temminckii Chelydridae马达加阿斯加壮龟E.madagascariensisAM943849AM943835 大鳄龟属FJ234440FJ230865潮龟科Geoemydidae 鳄龟属Chelydra闭壳龟属Cuora 蛇鳄龟C.serpentinaAY742461HQ260656* Dermochelyidae拟水龟属Mauremys 棱皮龟属安布闭壳龟C.amboinensisDQ386654HQ260653*棱皮龟科Dermochelys黄喉拟水龟M.muticaEF587930HQ260652* 棱皮龟D.coriacea Cheloniidae齿缘龟属Cyclemys 玳瑁属AY742460FJ039912乌龟M.reevesiiGQ259464HQ260651*海龟科Eretmochelys齿缘龟C.dentataAM931696AM931609 玳瑁E.imbricata Chelonia白头龟属Leucocephalon 绿海龟C.mydas FJ039967FJ039969印度摄龟C.gemeliFM877772FM877759 海龟属Caretta粗颈龟属Siebenrockiella 蠵龟C.caretta FJ039938FJ039954白头龟L.yuwonoiAM931708AM931622 蠵龟属Lepidochelys陆龟科Testudinidae 丽龟L.olivacea FJ009024FJ009033粗颈龟S.crassicollisAY954913EU030264 丽龟属FJ039981FJ039990石陆龟属Psammobates鳖科Pelodiscus陆龟属Testudo 中华鳖P.sinensis Trionychidae豹龟P.pardalisEU787162DQ497371 华鳖属DQ386652AM491038印支陆龟属AY259578FJ230872赫尔曼陆龟T.hermanni Indotestudo缅甸陆龟I.elongataHQ260650*HQ260657*
表2 龟鳖目9个科间的Kimura-2-paramete遗传距离
Tab.2 Kimura-2-parameter distances among 9 families in Testudinata
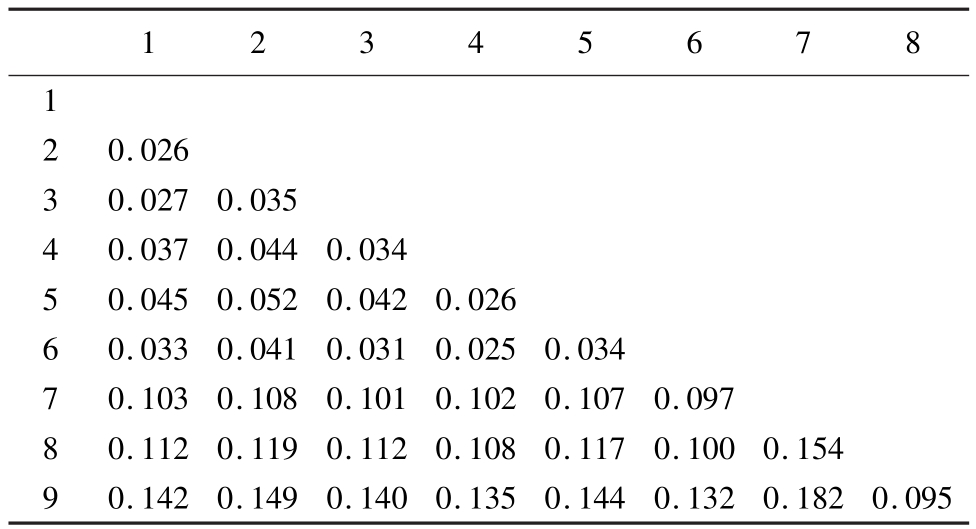
注:1——潮龟科,2——陆龟科,3——龟科,4——海龟科,5——棱皮龟科,6——鳄龟科,7——鳖科,8——侧颈龟科,9——南美侧颈龟科。
Notes:1,Geoemydidae;2,Testudinidae;3,Emydidae;4,Cheloniidae;5, Dermochelyidae;6,Chelydridae;7,Trionychidae;8,Pelomedusidae;9, Podocnemididae.
12345678 1 95 20.026 30.0270.035 40.0370.0440.034 50.0450.0520.0420.026 60.0330.0410.0310.0250.034 70.1030.1080.1010.1020.1070.097 80.1120.1190.1120.1080.1170.1000.154 90.1420.1490.1400.1350.1440.1320.1820.0
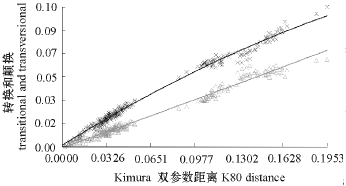
图1 基于转换和颠换对Kimura双参数遗传距离作图检测R35/RAG2序列饱和程度
Fig.1 Saturation test of R35/RAG2 by transitional and transversional variation against K-2-P distances between pairs of taxa
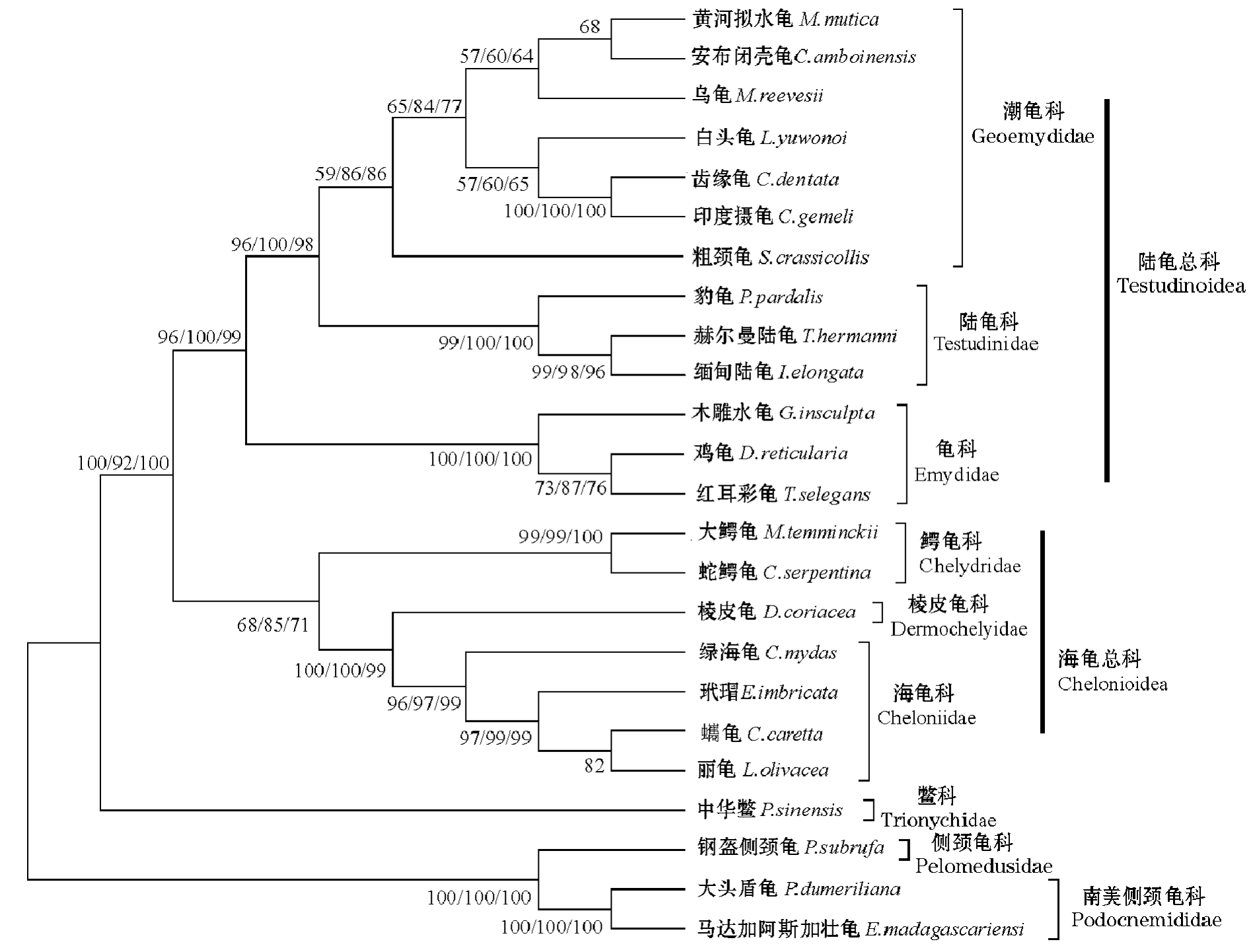
图2 基于龟鳖类R35/RAG2合并序列构建的分子系统树
Fig.2 Phylogenetic tree based on the combined R35/RAG2 date sets of turtles and tortoises
注:分支上方的数字为MP树、ML树、NJ树的节点置信值,置信值小于50%的未显示。
Note:numbers indicate bootstrap support values of nodes of MP/ML/NJ trees,and the values less than 50%were not shown.
3 讨论
3.1 龟鳖类系统进化关系的分析
随着分子生物学技术的发展,国内外有部分学者运用线粒体或核基因来研究龟鳖目的系统进化,解决了许多龟鳖类传统形态学上有争议的问题。然而,有关龟鳖类科水平或更高分类阶元的系统关系问题还有一些不确定之处,如平胸龟科、海龟类(棱皮龟科和海龟科)、鳄龟科的进化位置及其亲缘关系等[10]。
笔者构建的系统树结果显示,潮龟科与陆龟科汇聚后,再与龟科聚成姐妹群,且具有较高的支持率。从遗传距离上看,潮龟科与陆龟科的遗传距离(0.026)小于龟科和陆龟科的遗传距离(0.035),显示潮龟科与陆龟科之间的亲缘关系较近,而潮龟科与龟科的亲缘关系比与陆龟科要远。因此,本研究结果印证了陆龟科与潮龟科关系较近的观点[7,12,35-36],同时也再次验证Gaffney等[35]将Mc-Dowell[36]定义的龟科中的潮龟亚科和龟亚科提升为潮龟科和龟科的科学性[36]。这也与万全等[15]基于12SrRNA的研究结果一致。
本研究中构建的系统树结果还显示,陆龟总科,包括潮龟科、陆龟科和龟科与鳄龟科+海龟总科(包括海龟科和棱皮龟科)构成姐妹群,有较高的支持率(MP=100,ML=92,NJ=100),其中鳄龟科和海龟总科又是处于姐妹群的位置,其支持率均大于65(MP=68,ML=85,NJ=71)。这些与Krenz等[37]运用RAG-1、Cytb、12S rRNA序列重建的系统树的结果一致,与Barley等[10]用14个核基因序列重建的系统树结果也是一致的,但与Parham等[38]基于线粒体全基因组重建的系统树结果有
差异。线粒体基因和核基因的进化速率不同可能是造成这种差异的重要原因。
3.2 核基因标记在分子系统学上的适用性探讨
传统的系统进化分析多采用形态学和线粒体的数据来分析,然而,仅局限于这些数据的分析往往会产生一些误差,甚至是错误的结论。因此,结合核内更多的分子数据来分析系统进化成为一种必然。本研究中,作者采用两个核基因标记,其中关于重组激活基因(RAGs)的研究比较成熟,RAGs较为保守,因此被广泛用于动物系统进化及亲缘关系分析[16-20];采用的另外一个分子标记是RNA指纹蛋白35(R35),该蛋白属于多样性的7次跨膜蛋白超级家族的成员,R35的编码框编码一段是由723个氨基酸残基组成的多肽。然而,对于R35的具体功能还没研究清楚,目前的研究显示,R35是单拷贝基因,可能属于一个新的G蛋白偶联受体超家族。通过序列比对显示,R35或许司职着G蛋白偶联受体的作用,在小脑、骨髓、幼胚的神经节细胞中进行着发育的调节并高表达[21]。Friedel等[21]还指出R35基因有一段1 000~2 000 bp的内含子。R35基因的这一段内含子作为一种新的核内基因标记已被国外一些学者运用到龟鳖类动物的系统进化研究中[7,22]。
本研究中运用RAG2和R35这两个核基因的数据来分析24种龟鳖类的系统进化关系,构建的三种系统树均具有较高的支持率,从而更好地佐证了龟鳖类科阶元及更高分类阶元的一些进化关系。然而,研究龟鳖类的系统进化和亲缘关系是极其复杂的工作,还需要通过更多的线粒体(如线粒体全基因组)和核内的分子数据相结合来做进一步的分析。
参考文献:
[1] Rhodin A G J,Parham J F,van Dijk P P,et al.Turtles of the world:annotated checklist of taxonomy and synonymy,2009 update,with conservation status summary[M]//Conservation biology of freshwater turtles and tortoises:A Compilation Project of the IUCN/SSC tortoise and freshwater turtle specialist group,chelonian research monographs.Massachusetts:Chelonian Research Foundation, 2009:39-84.
[2] Cao Y,Sorenson M D,Kumazawa Y,et al.Phylogenetic position of turtles among amniotes:evidence from mitochondrial and nuclear genes[J].Gene,2000,259:139-148.
[3] Yasukawa Y,Hirayama R,Hikida T.Phylogenetic relationships of geoemydine turtles(Reptilia:Bataguridae)[J].Curr Herpetol, 2001,20:105-133.
[4] Honda M,Yasukawa Y,Hirayama R,et al.Phylogenetic relationships of the Asian box turtles of the genus Cuora sensu lato(Reptilia:Bataguridae)inferred from mitochondrial DNA sequences[J]. Zool Sci,2002,17:1305-1312.
[5] Honda M,Yasukawa Y,Ota H.Phylogeny of the Eurasian freshwater turtles of the genus Mauremys Gray 1869(Testudines),with special reference to a close affinity of Mauremys japonica with Chinemys reevesii[J].J Zool Syst Evol Res,2002,40:195-200.
[6] Stuart B L,Parham J F.Molecular phylogeny of the critically endangered Indochinese box turtle(Cuora galbinifrons)[J].Mol Phylogenet Evol,2004,31:164-177.
[7] Spinks P Q,Shaffer H B,Iverson J B,et al.Phylogenetic hypotheses for the turtle family Geoemydidae[J].Mol Phylogenet Evol,2004, 32:164-182.
[8] Le M,Raxworthy C J,Mccord W P,et al.A molecular phylogeny of tortoises(Testudines:Testudinidae)based on mitochondrial and nuclear genes[J].Mol Phylogenet Evol,2006,40:517-531.
[9] Eugenia N M,Le M,Fitzsimmons N N,et al.Evolutionary relationships of marine turtle:A molecular phylogeny based on nuclear and mitochondrial genes[J].Mol Phylogenet Evol,2008,49:659-662.
[10] Barley A J,Spinks P Q,Thomson R C,et al.Fourteen nuclear genes provide phylogenetic resolution for difficult nodes in the turtle tree of life[J].Mol Phylogenet Evol,2010,55:1189-1194.
[11] 吴平,周开亚,杨群.用12S rRNA基因序列研究潮龟科(BATAGURIDAE)闭壳龟类的进化[J].应用与环境生物学报,1998, 4(4):374-378.
[12] 吴平,周开亚,杨群.亚洲淡水和陆生龟鳖类12S rRNA基因片段的序列分析和系统发生研究[J].动物学报,1999,45(3): 260-267.
[13] 聂刘旺,宋娇莲,张颖,等.基于线粒体ND4基因探讨水龟组系统发生关系[J].安徽师范大学学报:自然科学版,2007,30 (3):343-348.
[14] 张颖,聂刘旺,宋娇莲.缅甸陆龟线粒体全基因组的测序及分析[J].动物学报,2007,53(1):151-158.
[15] 万全,郑将臣,程起群,等.基于12S rRNA序列研究龟鳖类的系统进化特征[J].海洋渔业,2010,32(3):264-274.
[16] Groth J G,Barrowclough G F.Basal divergences in birds and the phylogenetic utility of the nuclear RAG21 gene[J].Mol Phylogenet Evol,1999,12(2):115-123.
[17] Lovejoy N R,Collette B B.Phylogenetic relationships of new world needlefishes(Teleostei:Belonidae)and the biogeography of transitions between marine and freshwater habitats[J].Copeia,2001 (2):324-338.
[18] Venkatesh B,Erdmann M V,Brenner S.Molecular synapomorphies resolve evolutionary relationships of extant jawed vertebrates[J]. Proceedings of the National Academy of Sciences,2001,98(20): 11382-11387.
[19] Sullivan J P,Lundberg J G,Hardman M.A phylogenetic analysis of the major groups of catfishes(Teleostei:Siluriformes)using rag 1 and rag 2 nuclear gene sequences[J].Mol Phylogenet Evol,2006, 41(3):636-662.
[20] Wang X,Li J,He S.Molecular evidence for the monophyly of East Asian groups of Cyprinidae(Teleostei:Cypriniformes)derived from the nuclear recombination activating gene 2 sequences[J].Mol
Phylogenet Evol,2007,42(1):157-170.
[21] Friedel R H,Stubbusch J,Barde Y,et al.A novel 7-transmembrane receptor expressed in nerve growth factor-dependent sensory neurons[J].Mol Cell Neurosci,2001,17:31-40.
[22] Fujita M K,Engstrom T N,Starkey D E,et al.Turtle phylogeny: insights from a novel nuclear intron[J].Mol Phylogenet Evol, 2004,31:1031-1040.
[23] Sambrook J,Russel L D W.Molecular Cloning:A Laboratory Mannual[M].3rd ed.New York:Cold Spring Harbor Laboratory Press, 2001:463-471.
[24] Le M,Raxworthy C J,McCord W P,et al.A molecular phylogeny of tortoises(Testudines:Testudinidae)based on mitochondrial and nuclear genes[J].Mol Phylogenet Evol,2006,40:517-531.
[25] Thompson J D,Gibson T J,Plewniak F,et al.The CLUSTAL_X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Res,1997, 25:4876-4882.
[26] Librado P,Rozas J.Dna SP5.0:A software for comprehensive analysis of DNA polymorphism data[J].Bioinformatics,2009,25: 1451-1452.
[27] Tamura K,Dudley J,Nei M,et al.MEGA4:Molecular evolutionary genetics analysis(MEGA)software version 4.0[J].Mol Biol Evol,2007,24:1596-1599.
[28] Kimura M.A simple method for estimating evolutionary rate of base substitutions through comparative studies of nucleotide sequences[J].J Mol Evol,1980,16:111-120.
[29] Farris J S,K llersj M,Kluge A G,et al.Testing significance of incongruence[J].Cladistics,1994,10:315-320.
[30] Xia X H,Xie Z,Salemi M,et al.An index of substitution saturation and its application[J].Mol Phylogenet Evol,2003,26:1-7.
[31] Xia X,Xie Z.DAMBE:Data analysis in molecular biology and evolution[J].J Hered,2001,92:371-373.
[32] Swofford D L.PAUP*4.0610:Phylogenetic Analysis Using Parsimony(*and other methods),beta version[M].Sunderland:Sinauer Associates,2002.
[33] Posada D,Crandall K A.Modeltest:testing the model of DNA substitution[J].Bioinformatics,1998,14(9):817-818.
[34] Templeton A R.Phylogenetic inference from restriction endonuclease cleavage site maps with particular reference to the evolution of humans and apes[J].Evolution,983,37:221-244.
[35] Gaffney E S,Meylan P A.A phylogeny of turtles[M]//The Phylogeny and Classification of Tetrapods.Oxford England:Clarendon Press,1988:157-219.
[36] Mcdowell S B.Patition of the genus Clemmys and related problems in the taxonomy of the aquatic Tesudinidae[J].Proc Zool Soc London,1964,143:239-279.
[37] Krenz J G,Naylor G J P,Shaffer H B,et al.Molecular phylogenetics and evolution of turtles[J].Mol Phylogenet Evol,2005,37:178 -191.
[38] Parham J F,Feldman C R,Boore J L.The complete mitochondrial genome of the enigmatic bigheaded turtle(Platysternon):Description of unusual genomic features and the reconciliation of phylogenetic hypotheses based on mitochondrial and nuclear DNA[J]. BMC Evol Biol,2006,6:11-25.
Phylogenetic characteristics of turtles based on two nuclear gene sequences
ZHENG Jiang-chen1,3,WAN Quan2,CHENG Qi-qun1,ZHAO Jin-liang3
(1.Key Laboratory of Marine and Estuarine Fisheries Resources and Ecology,Ministry of Agriculture,East China Sea Fisheries Research Institute,Chinese Academy of Fishery Sciences,Shanghai 200090,China;2.College of Animal Sciences,Anhui Agricultural University,Hefei 230036,China; 3.Key Laboratory of Aquatic Genetic Resources and Utilization,Ministry of Agriculture,Shanghai Ocean University,Shanghai 201306,China)
Abstract:R35 intron segment sequences of 2 turtles and RAG2 gene segment sequences of 6 turtles were obtained by using PCR amplification and sequencing in order to evaluate the evolutionary character and phylogenetic relationships of turtles from the point of nuclear gene sequences.The homologous sequences of other turtles were downloaded from NCBI and the phylogenetic characters were analyzed together with the sequences the authors provided here. The results showed that 941 bp consensus sequences of R35 and 620 bp consensus sequences of RAG2 were obtained after aligned.1561bp consensus sequences of combined R35/RAG2 date set were acquired.Thereinto,505 variable sites were detected,accounting for 32.35%of total sites,with 239 parsim-informative and 139 indels. The average contents were found to be 29.5%in A,28.5%in T,22.8%in G,and 19.2%in C,with the Ts/Tv ratio of 1.64.and Kimura-2-parameter(K-2-P)distance was the minimum(0.025)between the Cheloniidae and Chelydridae.The K-2-P distance between the Trionychidae and Podocnemididae was the maximum(0.182).The phylogenetic trees indicate that 1)Geoemydidae is firstly clustered with Testudinidae,forming a monophyletic clade,and then they clustered with Emydidae,2)it strongly support that Testudinoidea is the sister taxon to Chelydridae plus Chelonioidea,and 3)Family Chelydridae is the sister taxon to Chelonioidea.
Key words:turtles;R35;RAG2;genetic divergence;molecular taxonomy;phylogeny
中图分类号:Q959.6
文献标志码:A
文章编号:2095-1388(2011)05-0452-06
收稿日期:2010-12-17
基金项目:中央级公益性科研院所基本科研业务费资助项目(2008Z02);农业部科技跨越计划资助项目(2009-跨-14);安徽教育厅科研项目(KJ2009B067)
作者简介:郑将臣(1986-),男,硕士研究生。E-mail:zjch896@163.com.
通信作者:程起群(1972-),男,研究员。E-mail:qiquncheng@gmail.com



