表1 材料来源
Tab.1 Sampling location and time
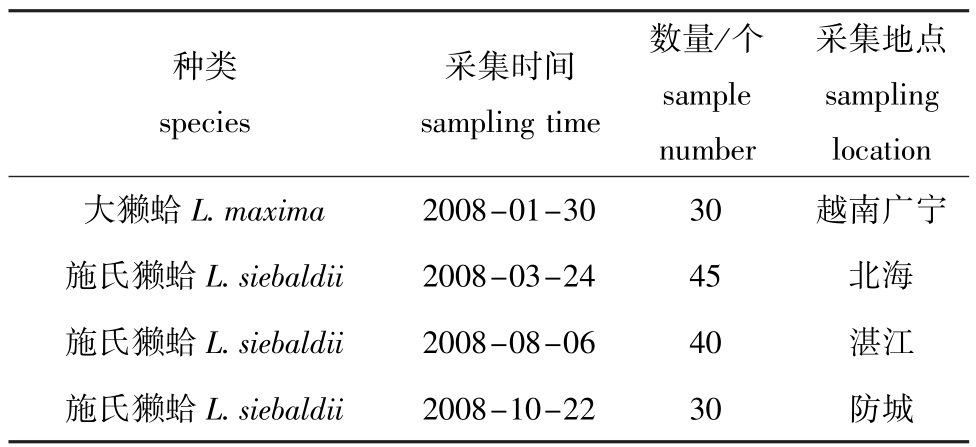
采集地点sampling越南广宁施氏獭蛤L.siebaldii2008-03-2445北海施氏獭蛤L.siebaldii2008-08-0640湛江施氏獭蛤L.siebaldii2008-10-2230 location大獭蛤L.maxima2008-01-3030种类species采集时间sampling time数量/个sample number防城
李斌,何俊锋,区小玲,苏翔驹,潘英,罗福广,郑惠芳,覃志彪
(广西大学动物科学技术学院,广西南宁530004)
摘要:采用形态特征分析和RAPD技术对采自广西北海、防城和广东湛江的施氏獭蛤Lutraria siebaldii 3个自然群体和越南大獭蛤Lutraria maxima 1个群体的遗传多样性进行分析。结果表明:用20条引物从施氏獭蛤3个群体和越南大獭蛤1个群体的145个样品中共扩增出428条带,大小为125~5 000 bp,每条引物扩增出6~17条带;多态位点比例为76.1%~91.9%,平均多态位点比例为84.5%,遗传距离为0.1321~0.2441;对施氏獭蛤3个群体和大獭蛤1个群体进行UPGMA聚类分析,施氏獭蛤北海群体和防城群体先聚为一体,再与湛江群体聚类,最后与越南的大獭蛤群体聚为一个整体;用8条引物可以区分施氏獭蛤3个自然群体和大獭蛤群体,可作为群体特征标记;形态差异分析结果显示,湛江、北海和防城施氏獭蛤3个群体的形态较接近,而越南大獭蛤群体的外部形态和遗传距离均与施氏獭蛤3个群体差异较大,已分化成为同属不同种;主成分分析表明,主成分1、主成分2和主成分3的贡献率分别为49.13%、17.18%和14.02%,累计贡献率为80.33%;施氏獭蛤3个群体判别函数的判别准确率P1为88.9%~100.0%,P2为86.7%~100.0%,综合判别率为95.9%。这说明施氏獭蛤3个群体的遗传多样性均处于较高水平,种质状况良好,且具有很好的养殖前景。
关键词:施氏獭蛤;RAPD;遗传多样性;形态差异
施氏獭蛤Lutraria siebaldii隶属软体动物门Mollusca、瓣鳃纲Lamillibranchia、异齿亚纲Heterodonta、帘蛤目Veneroida、蛤蜊科Mactridae、獭蛤亚科Lutrariinae H et A Adams、獭蛤属Lutraria,在中国主要分布于广西、广东、福建及海南沿海,以北部湾数量居多,是北部湾沿海特有的一种经济双壳类水产品[1]。施氏獭蛤呈椭圆形,其壳长为10.0~12.0 cm,广西沿海俗称象鼻螺、牛螺,广东称包螺。近年来,依靠天然苗种且通过海上圈养,施氏獭蛤已成为广西、广东沿海浅海增养殖的主要种类之一。目前,在广西施氏獭蛤生产性人工育苗已成功,滩涂养殖正逐步走向规模化。但由于滥捕,施氏獭蛤产量急剧下降,且由于不同来源的同种材料难以从外观、形态上加以区别,给施氏獭蛤的推广养殖带来一定困难。因此,有必要建立有效的分子标记鉴定方法和认证特征,以防止在生产上发生种质与种苗的“混乱”。
有关獭蛤的研究大多集中在形态发育、生态学、核型分析、营养成分等方面[2-7],而关于遗传背景的研究却很少,目前对施氏獭蛤的DNA水平的研究国内外尚未见报道。本研究中,作者以产自广西和广东两省区的3个施氏獭蛤自然群体为试验对象,结合形态特征分析与核基因DNA的RAPD标记,对其遗传多样性进行分析,旨在为系统研究施氏獭蛤遗传资源,以制定相应的资源保护措施提供科学依据。
1.1 材料
施氏獭蛤分别取自中国广西北海(BH)、防城(FC)和广东湛江(ZJ),共115个样本,采用越南广宁(VIE)的大獭蛤30个样本作外参对照,具体情况见表1。
表1 材料来源
Tab.1 Sampling location and time

采集地点sampling越南广宁施氏獭蛤L.siebaldii2008-03-2445北海施氏獭蛤L.siebaldii2008-08-0640湛江施氏獭蛤L.siebaldii2008-10-2230 location大獭蛤L.maxima2008-01-3030种类species采集时间sampling time数量/个sample number防城
1.2 方法
本试验中分别采用形态测量分析和RAPD分子标记两种方法对施氏獭蛤3个自然群体的遗传多样性进行了分析。
1.2.1 形态指标及测定 对施氏獭蛤3个群体和大獭蛤1个群体共145个样品的形态指标进行测量,分别测量壳高(SH)、壳长(SL)、壳宽(SW)、壳顶到背缘前端(DUA)、前闭壳肌高(HAR)、前闭壳肌宽(WAR)、后闭壳肌高(HPR)、后闭壳肌宽(WPR)、主齿宽(WT)、主齿到外套线边缘距离(DTL)、外套线到壳腹缘距离(DPM)、体质量(W)和软体部质量(R)13个形态指标,共测得数据2 250个。贝壳测量部位见图1,参照Kong等[8]的测量方法。
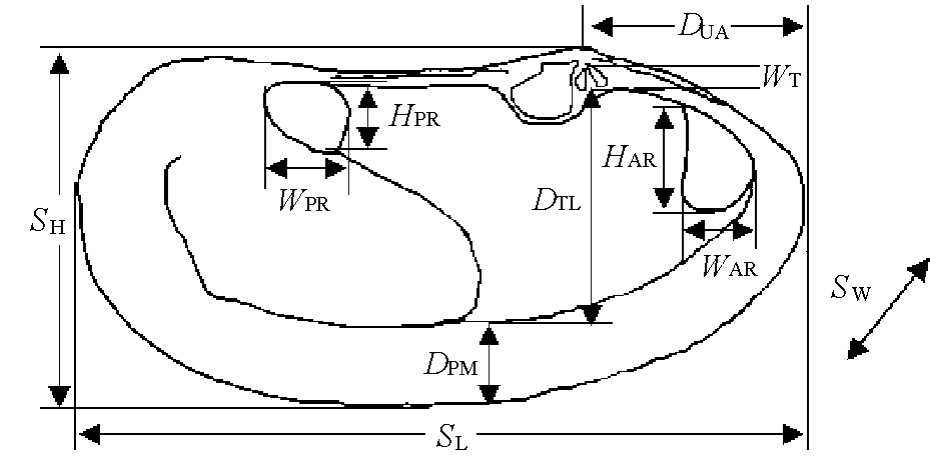
图1 施氏獭蛤的形态测量
Fig.1 Morphologic measurements of L.siebaldii
注:SH为壳高;SL为壳长;SW为壳宽;DUA为壳顶到背缘前端;HAR为前闭壳肌高;WAR为前闭壳肌宽;HPR为后闭壳肌高;WPR为后闭壳肌宽;WT为主齿宽;DTL为主齿到外套线边缘距离;DPM为外套线到壳腹缘距离。
Note:SH,shell height;SL,shell length;SW,shell width; DUA,distance between umbo and anterior end of shell;HAR, height of anterior adductor muscle scar;WAR,width of anterior adductor muscle scar;HPR,height of posterior adductor muscle scar; WPR,width of posterior adductor muscle scar;WT,width of cardinal tooth;DTL,distance between cardinal tooth and pallial line; DPM,distance between pallial line and ventral shell margin.
1.2.2 DNA的提取 参照尤仲杰等[9]的方法,根据实际需要略有改动。取50 mg肌肉组织,剪碎后放入1.5 mL灭菌EP管中,加入520 μL STE溶液和60 μL质量分数为10%的SDS,震荡混匀后再加入20 μL蛋白酶K(10 mg/mL),于56℃下消化过夜;用φ(酚)∶φ(氯仿)∶φ(异戊醇)=50∶49∶1和φ(氯仿)∶φ(异戊醇)=24∶1的混合液抽提两次,加入2倍体积的冰冻无水乙醇,颠倒摇动数次至DNA呈絮状析出,于-20℃下冰冻30 min以上;将管内液体小心倒出,加入预冷的体积分数为70%的乙醇洗涤两次,将附着有DNA的EP管置于空气中自然风干,向管中加入50 μL TE缓冲液,置于冰箱(4℃)中保存。
1.2.3 随机引物及RAPD反应 试验所用的20个随机引物是从100个引物中筛选出的,引物序列见表2。试验前进行条件优化,最终确定反应条件为:反应体系为25 μL,10×buffer 2.5 μL,dNTPs 1.0 μL,primer 1.0 μL,DNA模板2.0 μL,Taq酶0.3 μL(5 U/μL),ddH2O 18.2 μL。RAPD扩增程序为:94℃下预变性4 min;94℃下变性1 min, 36℃下退火3 min,72℃下延伸2 min,共进行44个循环;最后在72℃下再延伸10 min。PCR扩增产物用20 g/L的琼脂糖凝胶进行电泳分离,在紫外凝胶系统下观察并拍照。
表2 随机引物的编号及序列
Tab.2 The numbers and sequences of random primers
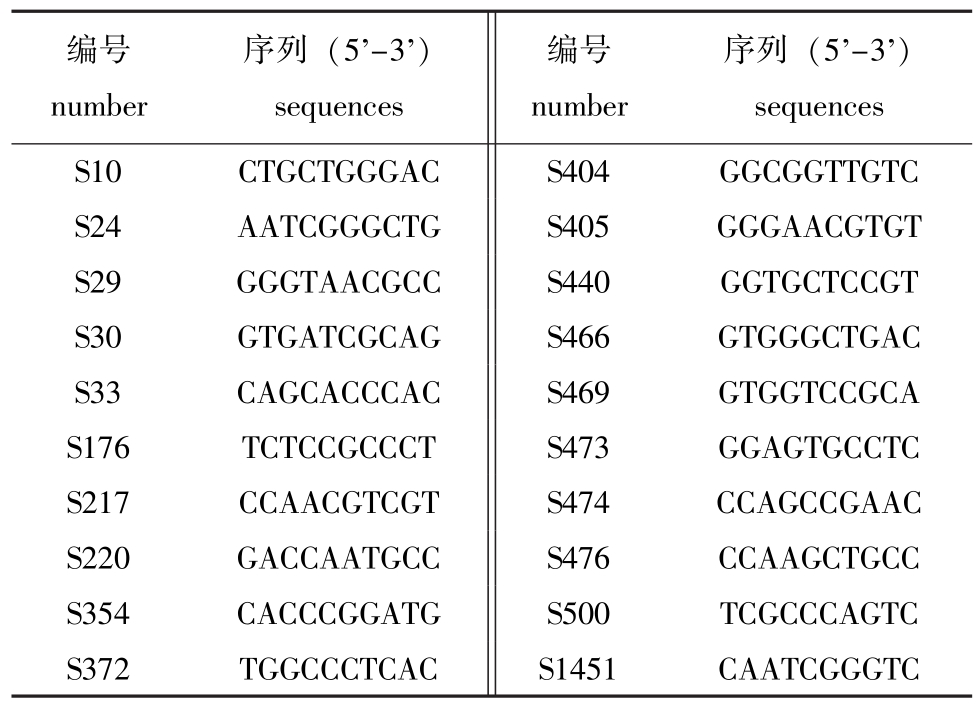
编号number序列(5'-3') sequences编号number序列(5'-3') sequences S10CTGCTGGGAC S404GGCGGTTGTC S24AATCGGGCTGS405GGGAACGTGT S29GGGTAACGCCS440GGTGCTCCGT S30GTGATCGCAGS466GTGGGCTGAC S33CAGCACCCACS469GTGGTCCGCA S176TCTCCGCCCTS473GGAGTGCCTC S217CCAACGTCGTS474CCAGCCGAAC S220GACCAATGCCS476CCAAGCTGCC S354CACCCGGATGS500TCGCCCAGTC S372TGGCCCTCACS1451CAATCGGGTC
1.3 数据处理
形态差异分析采用SPSS 13.0统计软件包的聚类分析(CA)、主成分分析(PCA)、判别分析(DA)3种多元分析方法,分别对施氏獭蛤3个群体和大獭蛤1个群体的12个形态比例性状进行单因子方差分析(ANOVA)。
根据电泳凝胶的扩增条带,以1、0表示扩增条带有无,形成0、1矩阵,将结果输入Popgen 32
软件,统计扩增结果。多态位点百分率P是反映群体内变异水平的重要指标之一,其计算公式为
其中:k为多态位点数目;n为所测定位点的总数。
种群内和种群间的遗传相似性和遗传距离根据Nei等[10]的公式计算:
其中:Nx表示样品x具有的条带数;Ny表示样品y具有的条带数;Nxy表示样品x和y的共有条带数。
用Shannon遗传多样性指数表示群体的遗传多样性,根据Luciane等[11]的方法,计算公式为
其中:H0表示全部引物所检测到的该群体中所有位点;πi表示某位点在群体中的类型频率。
2.1 形态差异分析结果
2.1.1 形态特征参数分析 施氏獭蛤3个群体和大獭蛤1个群体的形态特征描述性统计值见表3。
表3 施氏獭蛤3个群体和大獭蛤1个群体形态特征的描述性统计值
Tab.3 A descriptive statistics on morphological characters from different populations of Lutraria siebaldii and Lutraria maxima

种类species壳长SL壳高SH壳宽SW壳顶到背缘前端DUA前闭壳肌高HPR前闭壳肌宽WAR后闭壳肌高HPR 1.14±0.020.68±0.021.13±0.02湛江施氏獭蛤ZJ9.11±0.124.56±0.062.72±0.042.70±0.051.51±0.020.92±0.021.20±0.02北海施氏獭蛤BH8.09±0.063.91±0.032.39±0.022.45±0.021.36±0.020.80±0.011.11±0.02防城施氏獭蛤FC8.21±0.174.11±0.082.27±0.062.60±0.061.35±0.030.79±0.021.06±0.02种类species越南大獭蛤VIE7.29±0.063.67±0.032.34±0.022.13±0.13后闭壳肌宽WPR主齿宽WT主齿到外套线边缘距离DTL外套线到壳腹缘距离DPM体质量W软体部质量R 56.71±1.4725.82±0.58湛江施氏獭蛤ZJ1.16±0.020.92±0.022.40±0.031.04±0.0293.19±3.2651.43±1.99北海施氏獭蛤BH0.94±0.020.91±0.012.04±0.030.93±0.0170.18±1.5535.20±0.74防城施氏獭蛤FC0.95±0.020.78±0.022.35±0.050.90±0.034越南大獭蛤VIE0.86±0.020.86±0.012.30±0.031.06±0.02 2.13±2.3624.92±1.27
2.1.2 聚类分析 对施氏獭蛤3个群体和大獭蛤1个群体所有样本的12个性状形态矫正值进行种内和种间聚类分析结果见图2。施氏獭蛤3个群体中,北海与防城群体首先聚为一类,然后再与湛江群体聚为一个整体。
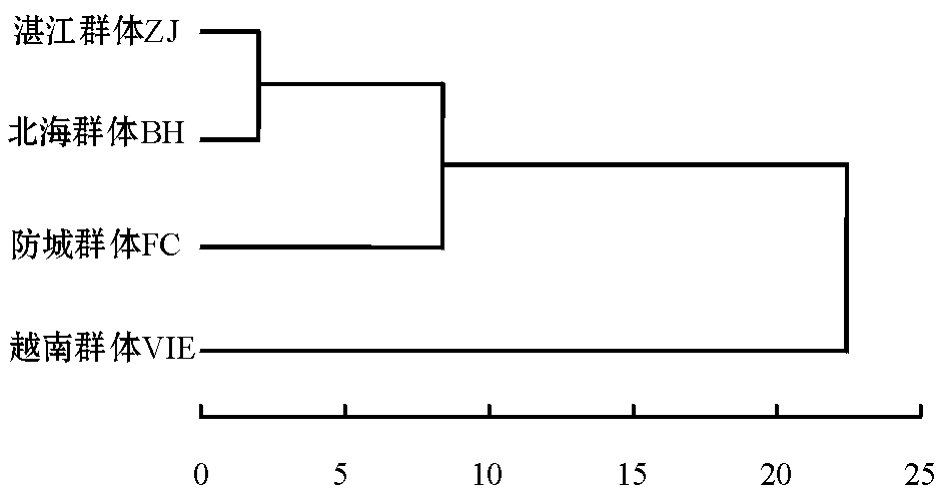
图2 施氏獭蛤3个群体和大獭蛤1个群体的聚类分析图
Fig.2 Diagram of cluster analysis from different populations of Lutraria siebaldii and Lutraria maxima
种间比较,施氏獭蛤湛江群体与北海群体距离最短,形态最为接近,最先聚为一类,然后防城群体与前者趋异程度增加再聚成一类;而越南大獭蛤群体与施氏獭蛤3个群体趋异程度最大,作为外群的越南大獭蛤群体自成一支。
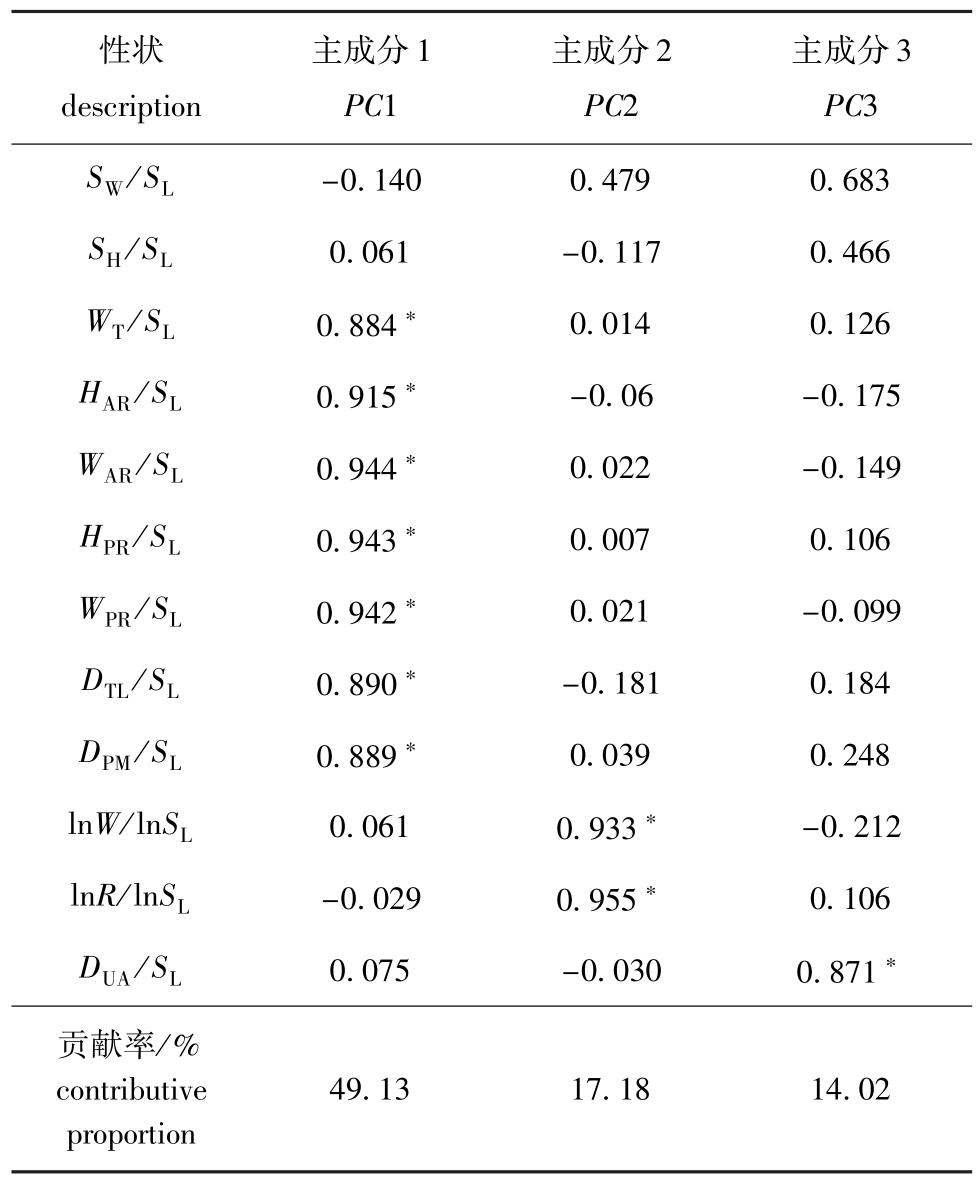
2.1.3 主成分分析 对施氏獭蛤3个群体和大獭蛤1个群体的12个性状形态矫正值进行主成分分析,获得3个主成分。3个主成分的负荷值和方差贡献率见表4。3个主成分的累积贡献率较高,说明这3个相互独立的因子适合概括不同群体之间的形态差异。
施氏獭蛤3个群体和大獭蛤1个群体的第一、第二主成分散布图见图3。从图3可以看出:施氏獭蛤3个群体重叠比较严重,说明3个群体差异较小;而越南大獭蛤群体与3个施氏獭蛤群体的形态差异较大,已分化形成两个不同的种,这与聚类分析的结果相一致。
2.1.4 判别分析 对施氏獭蛤3个群体和大獭蛤1个群体的12个性状形态矫正值用F检验进行判别分析,判别效果较好(P<0.01),说明施氏獭蛤和大獭蛤不同群体间形态差异明显。
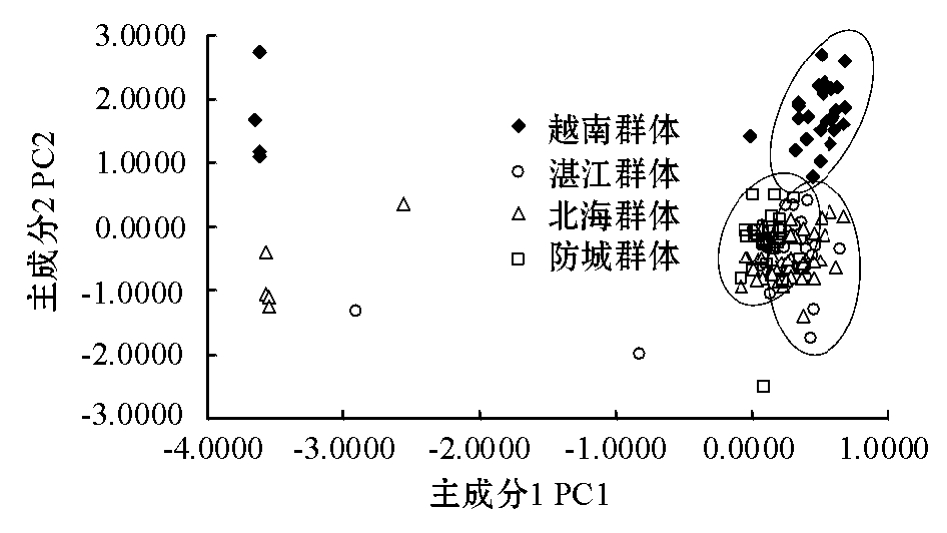
图3 施氏獭蛤3个群体和大獭蛤1个群体的主成分散布图
Fig.3 Scattering diagram of the principal components from different populations of Lutraria siebaldii and Lutraria maxima
表4 施氏獭蛤3个群体和大獭蛤1个群体12个性状的3个主成分的贡献率及负荷值
Tab.4 Contribution ratios and loading of three principal components for twelve characters from different populations of Lutraria siebaldii and Lutraria maxima
注:*表示负荷值>0.700。Note:*means a load value>0.700.
性状description主成分1 PC1主成分2 PC2主成分3 PC3 -0.1400.4790.683 SH/SL0.061-0.1170.466 WT/SL0.884*0.0140.126 HAR/SL0.915*-0.06-0.175 WAR/SL0.944*0.022-0.149 HPR/SL0.943*0.0070.106 WPR/SL0.942*0.021-0.099 DTL/SL0.890*-0.1810.184 DPM/SL0.889*0.0390.248 lnW/lnSL0.0610.933*-0.212 lnR/lnSL-0.0290.955*0.106 DUA/SL0.075-0.0300.871*贡献率/% contributive proportion SW/SL49.1317.1814.02
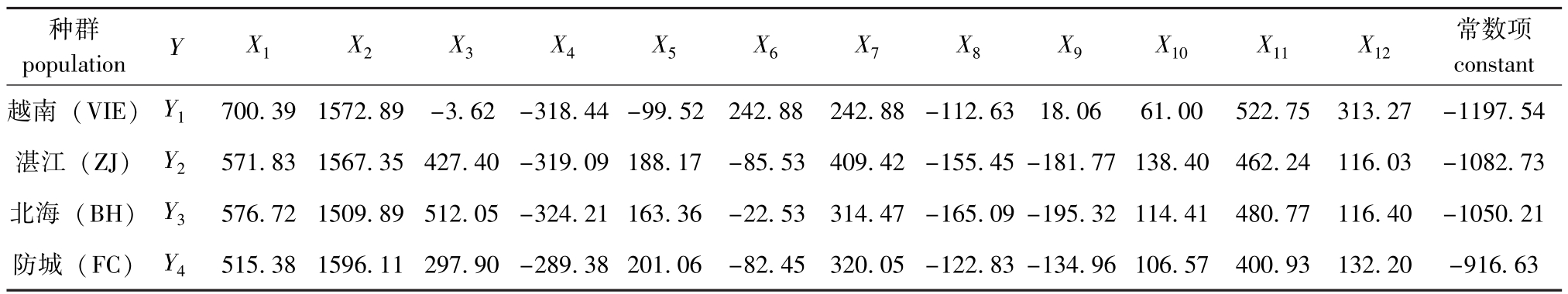
通过逐步判别法分析,建立施氏獭蛤3个群体和大獭蛤1个群体的判别函数。为了消除样本大小对形态度量指标的影响,度量指标除以壳长作为判别的特征值外,公式中的X1、X2、X3、X4、X5、X6、X7、X8、X9、X10、X11、X12分别代表SW/SL、SH/SL、WT/SL、HAR/SL、WAR/SL、HPR/SL、WPR/SL、DTL/SL、DPM/SL、lnW/lnSL、lnR/lnSL、DUA/SL12个性状形态矫正值,施氏獭蛤3个群体和大獭蛤1个群体的判别函数中的自变量系数和常数项见表5,其判别公式如下:
越南群体Y1=700.39X1+1572.89X2-3.62X3+…+313.27X12-1197.54;
湛江群体Y2=571.83X1+1567.35X2+427.40X3+…+116.03X12-1082.73;
北海群体Y3=576.72X1+1509.89X2+512.05X3+…+116.40X12-1050.21;
防城群体Y4=515.38X1+1596.11X2+297.90X3+…+132.20X12-916.63。
表5 施氏獭蛤3个群体和大獭蛤1个群体形态判别函数的各项系数及常数项
Tab.5 Coefficients and constant of discriminant functions from different populations of Lutraria siebaldii and Lutraria maxima
注:Y为函数;X为自变量系数。Note:Y is discriminant functions;X is coefficient for independent variables.
常数项constant越南(VIE)Y1700.391572.89-3.62-318.44-99.52242.88242.88-112.6318.0661.00522.75313.27-1197.种群populationYX1X2X3X4X5X6X7X8X9X10X11X1216.63 54湛江(ZJ)Y2571.831567.35427.40-319.09 188.17-85.53409.42-155.45-181.77 138.40462.24116.03-1082.73北海(BH)Y3576.721509.89512.05-324.21 163.36-22.53314.47-165.09-195.32 114.41480.77116.40-1050.21防城(FC)Y4515.381596.11297.90-289.38 201.06-82.45320.05-122.83-134.96 106.57400.93132.20-9
将12个矫正值分别代入上面的判别函数中,比较函数值的大小,被判别的个体归属于函数值最大的函数所对应的群体。为检验判别函数的应用效果,对施氏獭蛤3个群体和大獭蛤1个群体的145个样本按判别函数进行预测分类,判别分析结果见表6,表明判别函数是可靠的。
2.2 RAPD扩增结果
本试验中用筛选所得的20条引物对施氏獭蛤3个群体和大獭蛤1个群体共145个样品进行多态性分析,共获得428条清晰的条带,对单个引物扩增出6~17条带,引物扩增的片段大小为125~5 000 bp,其中引物S176-200、S30-150仅在北海施氏獭蛤群体中出现,S469-200只在防城施氏獭
蛤群体中出现,S30-300仅在湛江施氏獭蛤群体出现,而S474-3000、S1451-2000、S30-1100、S176 -850仅在越南大獭蛤群体中出现。用以上8条引物可以区分施氏獭蛤3个群体和大獭蛤1个群体,作为群体的特征标记。用引物S176对施氏獭蛤3个群体和大獭蛤1个群体的RAPD扩增结果见图4。
表6 施氏獭蛤3个群体和大獭蛤1个群体判别分析结果
Tab.6 Discriminant results from different populations of Lutraria siebaldii and Lutraria maxima
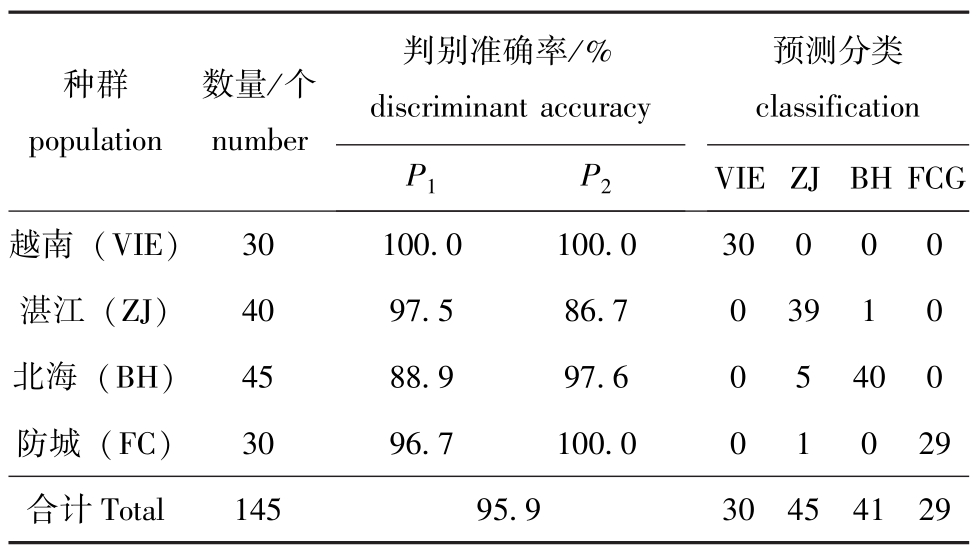
判别准确率/%预测分类种群population数量/个number classification VIE ZJBH FCG越南(VIE) discriminant accuracy P1P230100.0100.030000湛江(ZJ)4097.586.703910北海(BH)4588.997.605400防城(FC)3096.7100.001029合计Total14595.930454129
分别统计施氏獭蛤3个群体和大獭蛤1个群体RAPD扩增结果,用Popgen 32软件计算各个群体的多态位点率、遗传分化系数、香农多样性指数(表7),并根据遗传分化指数和香农指数估算出种群间的遗传多样性和分化(表8)。从表7、8可知,各组数据较一致地反映出施氏獭蛤3个群体和大獭蛤1个群体都具有较丰富的遗传多样性,其中从香农指数和遗传分化指数上来看,北海施氏獭蛤群体最为丰富。利用Nei等[12]无偏法(Unbiased measure)计算群体内和群体间的遗传距离及遗传相似性指数(表9),结果表明:施氏獭蛤3个群体之间的遗传相似性指数为0.8304~0.8378;3个群体间的遗传距离为0.1770~0.1859,其中,湛江和防城群体遗传距离最大(0.1859),而北海与防城群体间遗传距离最小(0.1770),说明施氏獭蛤群体间存在着一定的遗传差异,并且差异显著,具有明显的地理种群特征。对施氏獭蛤3个群体和大獭蛤1个群体进行UPGMA聚类分析,构建的分子系统树见图5。
遗传距离和聚类分析的结果表明,在施氏獭蛤群体内,北海与防城群体最先聚为一类,然后再与湛江群体聚为一个整体;在种间聚类分析中,施氏獭蛤群体内的北海与防城群体先聚,再与湛江群体相聚,最后才与越南大獭蛤群体聚为一个整体。
表7 施氏獭蛤3个群体和大獭蛤1个群体RAPD标记分析结果
Tab.7 RAPD results from different populations of Lutraria siebaldii and Lutraria maxima
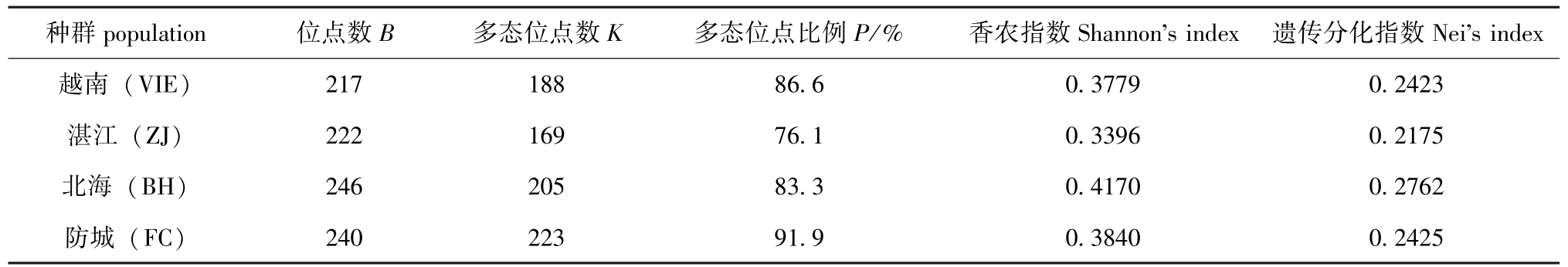
种群population位点数B多态位点数K多态位点比例P/%香农指数Shannon's index遗传分化指数Nei's index越南(VIE) 24022391.90.38400.2425 21718886.60.37790.2423湛江(ZJ)22216976.10.33960.2175北海(BH)24620583.30.41700.2762防城(FC)
表8 施氏獭蛤3个群体和大獭蛤1个群体种群内、种群间遗传多样性及分化
Tab.8 Genetic diversity and differentiation in/among different populations of Lutraria siebaldii and Lutraria maxima by Shannon's index and Nei's gene diversity

种群内遗传多样性Hp总种群遗传多样性Hs种群内遗传多样性所占比例Hp/Hs种群间遗传多样性所占比例(Hs-Hp)/Hs总种群基因多样性Ht种群内基因多样性Hs种群间遗传分化Gs种群每代迁移数Nm0.37960.45260.83870.16130.25480.14210.44220.6306
近年来,采用形态分析和分子遗传标记进行海洋贝类种质资源遗传分析的研究较为广泛[8-9],但有关施氏獭蛤群体遗传的研究尚未见报道。本研究中,作者采用形态测量分析和RAPD分子标记两种方法对广西和广东沿海施氏獭蛤3个地理群体的遗传多样性进行了分析,所得结果基本一致,均较好地反映出施氏獭蛤3个群体和大獭蛤1个群体的亲缘关系的远近程度及遗传多样性的丰富程度。但同时也表明这两种方法各有优缺点,只有将两种方法有机地结合起来,才能得出更加可信的试验结果。
3.1 应用多元分析法对数量性状的分析
在形态分析中,聚类分析可将不同种群进行初步归类,量化种群间的差异程度,分析种群间的相似程度[13],所得出的种群间的相似程度与种类亲疏和地理距离的远近有关。如本研究中,地理距离较近的北海、防城群体首先相聚,再与湛江群体相聚,最后才与越南群体相聚。主成分分析中,第1
主成分主要反映壳高数据,第2主成分的体质量和去壳质量,间接反映壳厚,表明贝壳的形状和厚度是区分施氏獭蛤不同地理群体的主要因素,这在对其它贝类的研究中也同样得到证实[14]。从判别分析结果来看,因为越南大獭蛤群体与施氏獭蛤3个群体之间属于种间判别,所以对越南群体的判别准确率较高。采用3种多元分析方法得到施氏獭蛤3个群体和大獭蛤1个群体的遗传多样性分析结果较一致,说明这3种方法在一定程度上可以区分施氏獭蛤不同地理群体。但由于自然和人为因素对贝类的外部形态影响较大,在种类鉴定、遗传分析等方面往往会存在一定误差。所以,只有将形态指标与生化、分子标记技术结合起来,才能更准确地描述施氏獭蛤各群体间的特性。
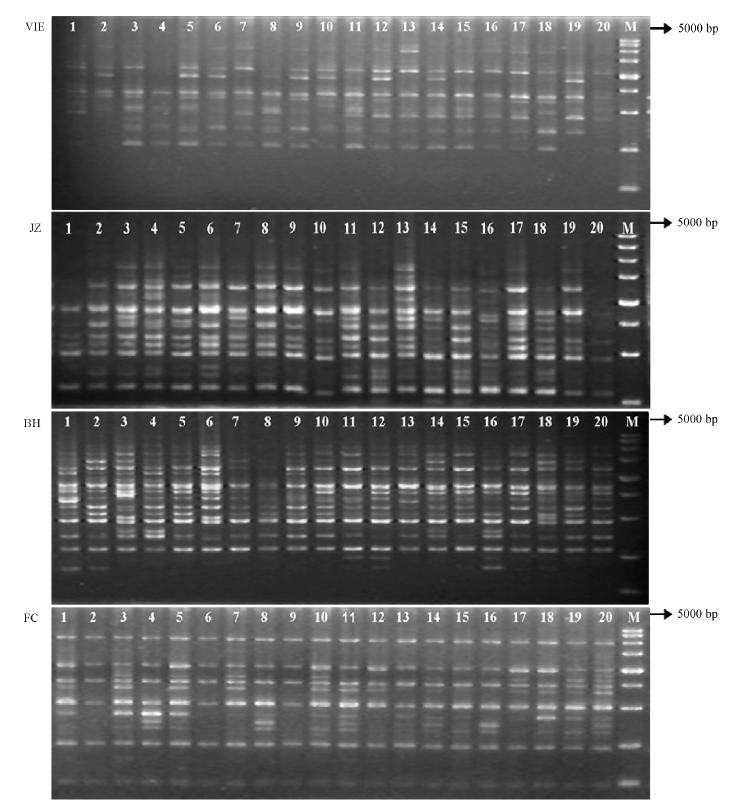
图4 引物S176对施氏獭蛤3个群体和大獭蛤1个群体RAPD扩增图谱
Fig.4 RAPD amplification profiles by primer S176 from different populations of Lutraria siebaldii and Lutraria maxima
表9 施氏獭蛤3个群体和大獭蛤1个群体的遗传相似性(上三角)和遗传距离(下三角)
Tab.9 Nei's genetic identity(above diagonal)and genetic distance(below diagonal)of different populations of Lutraria siebaldii and Lutraria maxima
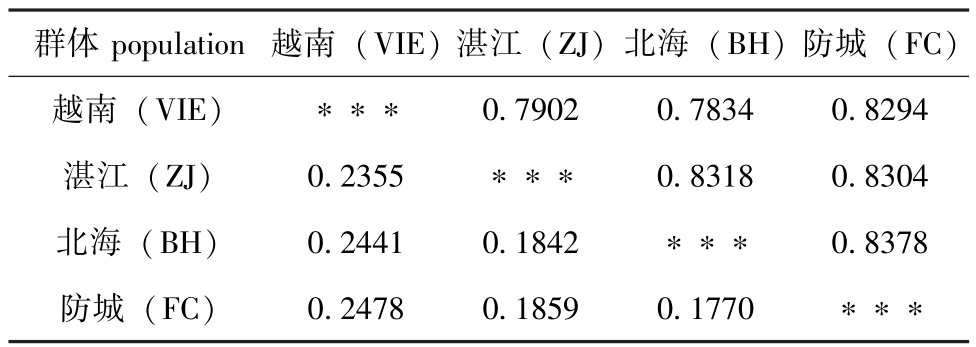
群体population越南(VIE)湛江(ZJ)北海(BH)防城(FC)越南(VIE)***0.79020.78340.8294湛江(ZJ)0.2355***0.83180.8304北海(BH)0.24410.1842***0.8378防城(FC)0.24780.18590.1770***
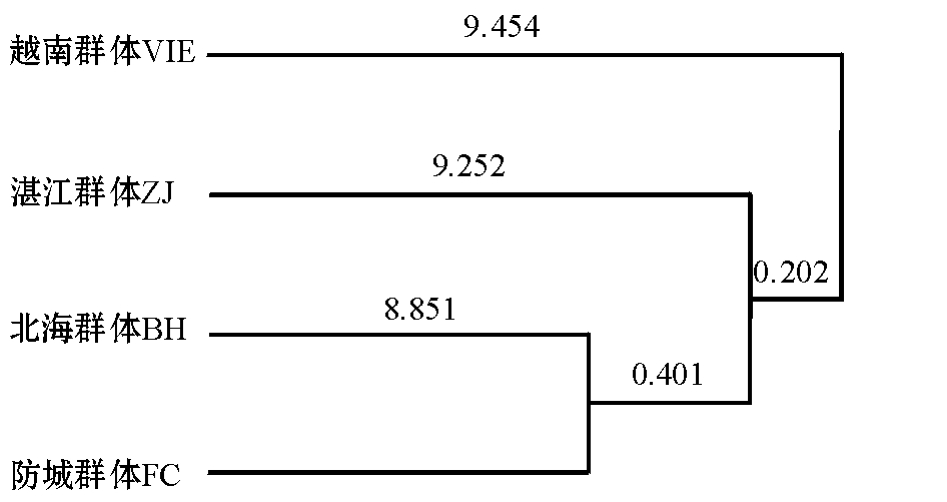
图5 施氏獭蛤3个群体和大獭蛤1个群体的UPGMA聚类图
Fig.5 UPGMA clustering of different populations of Lutraria siebaldii and Lutraria maxima
3.2 施氏獭蛤的遗传多样性
遗传多样性是生物所携带的遗传信息的总和。采用RAPD技术对施氏獭蛤3个群体和大獭蛤1个群体的样品进行研究,共获得428条带、925个位点,每条引物扩增出条带6~17条,平均每条引物扩增出11.56个位点,片段大小为125~5 000 bp。对施氏獭蛤3个群体和大獭蛤1个群体共扩增出多态位点785个,多态位点比例平均为84.9%。较高的多态位点比例表明,施氏獭蛤3个群体具有丰富的遗传多样性。目前国内施氏獭蛤的养殖还处于起步阶段,尚未形成规模化养殖,养殖群体未对野生群体造成影响,施氏獭蛤遗传多样性仍处于较高的水平,而不同地理种群间遗传多样性差异与施氏獭蛤的地理分布特点有着密切关系。
遗传多样性的丰富程度受到繁育系统和分布范围的影响,远交或分布范围较广的物种遗传多样性水平较高,反之较低。RAPD研究结果表明,施氏獭蛤3个群体和大獭蛤1个群体的Shannon指数及Nei指数与检测到的遗传多样性一致,遗传多样性由低到高依次为越南群体<湛江群体<防城群体<北海群体,表明施氏獭蛤3个群体的遗传多样性水平较高。
3.3 遗传距离和亲缘关系的比较
遗传距离是评价群体遗传变异水平的重要指标,同样能反映出群体间遗传多样性的丰富程度。Thorpe[15]认为,同科属间遗传距离(D)为0.5~0.9,同属种间D为0.2~0.8,同种种群间D为0.03~0.2。群体间遗传相似度与遗传距离指数能反映群体间的亲缘关系。本研究中,施氏獭蛤3个群体间的遗传相似度为0.8304~0.8378,遗传距离为0.1770~0.1859,防城与北海群体的亲缘关系较近,与湛江群体亲缘关系较远。施氏獭蛤群体间的遗传距离随着地理位置的距离增加而逐渐加大,遗传差异也较大。施氏獭蛤湛江、北海、防城群体间的遗传距离为0.03~0.2,说明没有达到种间遗传分化水平;而越南大獭蛤群体与施氏獭蛤3个群体的遗传距离达到了种间的分化范围,说明大獭蛤与施氏獭蛤这两个种之间因栖息地气候和海水理化因子差异较大,经过长期的自然选择,有较大的遗传差异,维持了各自种的特点,符合地理距离产生遗传分化的理论。
聚类分析结果可以明显地表现群体间亲缘关系的远近。在群体间分化较小的前提下,不同的聚类方法可能会出现不同的聚类结果[9]。本研究结果表明,采用形态差异中的主成分分析、聚类分析和RAPD的聚类分析法都一致地把施氏獭蛤各群体和大獭蛤进行了准确区分,而采用判别式分析法也得出越南大獭蛤群体较施氏獭蛤群体判别率高的结果,表明越南大獭蛤群体与施氏獭蛤3个群体之间的区别。本研究结果表明,施氏獭蛤3个群体各有自己的RAPD特征带谱,通过8个标记可以区分施氏獭蛤3个群体和大獭蛤群体,并作为施氏獭蛤群体种质鉴定的重要依据,同时香农指数等数据也表明了施氏獭蛤3个自然群体的遗传多样性较为丰富。
采用形态差异和RAPD技术对施氏獭蛤3个群体的遗传多样性进行分析,结果发现,各群体维持了较高的遗传多样性。影响遗传分化的主要原因可能是环境因素,但也要注意在地理范围较小的孤立环境中造成群体间遗传多样性降低的可能性。笔者认为,在对施氏獭蛤进行分类、种质鉴别、遗传多样性分析时,仅依靠形态分析是不够的,今后有必要结合其他分子标记(如SSR等)进行更可靠的分析。目前,尚未对施氏獭蛤进行大规模的人工育苗和养殖。施氏獭蛤3个自然群体之间的遗传距离较大,遗传多样性维持在相对高的水平,说明施氏
獭蛤仍然保持较为丰富的遗传资源,因此具有良好的育种前景。
致谢:广东海洋大学蔡英亚教授在样品鉴定方面给予了大力协助,在此谨致谢忱!
参考文献:
[1] 徐凤山,张素萍.中国海产双壳类图志[M].北京:科学出版社,2008:162-163.
[2] 李琼珍,童万平,苏琼,等.大獭蛤的胚胎、幼虫及稚贝的形态发育[J].广西科学,2003,10(4):12-14.
[3] 李琼珍,陈瑞芳,童万平,等.盐度对大獭蛤胚胎发育的影响[J].广西科学院学报,2004,20(1):33-34.
[4] 蔡英亚,劳赞,陈东.An ecological observation of Lutraria sieboldii [J].湛江海洋大学学报,2005,25(1):39-42.
[5] 杨家林,蒋艳,蔡德建.Preliminary studies on the holding of Lutmria maxima jonas at low temperature[J].广西科学院学报, 2006,22(2):75-77.
[6] 潘英,苏以鹏.大獭蛤的核型研究[J].海洋科学,2007,31(9): 87-90.
[7] 潘英,秦小明,潘红平.大獭蛤软体部营养成分的分析与评价[J].广东海洋大学学报,2007,27(3):78-81.
[8] Kong L,Li Q,Qiu Z.Genetic and morphological differentiation in the clam Coelomactra antiquata(Bivalvia:Veneroida)along the coast of China[J].Journal of Experimental Marine Biology and E-cology,2007,343(1):110-117.
[9] 尤仲杰,包永波,张爱菊.中国沿海西施舌5个自然群体形态差异和RAPD分析[J].海洋学报,2007,29(3):98-104.
[10] Nei M,Li W H.Mathematical model for studying genetic variation in terms of restriction endonucleases[J].Proc Natl Acad Sci U S A,1979,76(10):5269-5273.
[11] Luciane G,Suzana C M.Genetic variation in natural populations of mate(Liex paraguariensis A.St.Hil,Aquifoliaceae)using RAPD markers[J].Heredity,2000,84:647-656.
[12] Nei M.Estimation of average heterozygosity and genetic distance from a small number of individuals[J].Genetics,1978,89:583-590.
[13] 蔡庆华.武汉东湖浮游植物水华的多元分析[J].水生生物学报,1990,14(1):22-31.
[14] 周晓农,洪青标,孙乐平,等.中国钉螺螺壳的聚类分析[J].动物学杂志,1997(5):5-8.
[15] Thrope J P.The molecular clock hypothesis:Biochemical evolution,genetic differentiation and systematics[J].Ann Rev Evol Syst,1982,13(2):139-168.
Morphological and genetic diversity analysis among three populations of clam Lutraria siebaldii in coastal Guangxi and Guangdong provinces
LI Bin,HE Jun-feng,OU Xiao-ling,SU Xiang-ju,PAN Ying, LUO Fu-guang,ZHENG Hui-fang,QIN Zhi-biao
(College of Animal Science and Technology,Guangxi University,Nanning 530004,China)
Abstract:The random amplified polymorphic DNA(RAPD)technique and the morphological variation analysis were used to evaluate population genetics among three wild populations of clam Lutraria siebaldii in coastal Guangxi and Guangdong provinces and one population Lutraria maxima in coastal Vietnam.A total of 428 scorable markers were amplified from 20 RAPD primers in 145 samples from different populations of Lutraria siebaldii and Lutraria maxima,ranging from 125 to 5 000 bp in size,and the number of the scorable bands for each primer varied from 6 to 17,with the average polymorphic band proportion of 84.5%.The mean percent of polymorphic loci in four populations were ranged from 76.1%to 91.9%with genetic distance from 0.1321 to 0.2441 among individuals.The cluster analysis according to their genetic distances,first Zhanjiang,Beihai and Fangcheng populations,and then with Vietnam population,revealed that the 3 wild populations of Lutraria siebaldii were distinguished by 8 primers. The morphological variations showed that the samples from Zhanjiang,Beihai and Fangcheng populations were rather similar in morphology,whereas Vietnam population differed greatly from the other populations in morphology,indicating that these clams were divided into different species.In the principal component analysis,the contributory ratio of the three principal components was 49.13%,17.18%and 14.02%,respectively,and the cumulative contributory ratio was 80.33%.The discrimination functions of the four populations were set up,the discrimination accuracy being 88.9%~100.0%for P1and 86.7%~100.0%for P2,the integrative discrimination accuracy being 95.9%.The resource of Lutraria siebaldii was found in good condition with high genetic variation and Lutraria siebaldii had good culture prospects.
Key words:Lutraria siebaldii;random amplified polymorphic DNA;genetic diversity;morphological variation
中图分类号:Q959.215
文献标志码:A
文章编号:2095-1388(2011)05-0414-08
收稿日期:2010-11-09
基金项目:国家自然科学基金资助项目(31060353);广西科学研究与技术开发计划项目(0718003-3-6)
作者简介:李斌(1982-),男,硕士生。E-mail:libin064@qq.com
通信作者:潘英(1968-),女,博士,教授。E-mail:nnpying@sohu.com