应用PCR-DGGE技术研究四角蛤蜊的细菌多样性
马悦欣,王颖,杨凤,闫喜武,霍忠明
(大连海洋大学农业部海洋水产增养殖学重点开放实验室,辽宁大连116023)
摘要:应用PCR-DGGE技术对盘锦某滩涂四角蛤蜊Mactra venerformis体内的细菌多样性进行了分析。使用SDS裂解法提取样品的总DNA,采用大多数细菌通用引物F338GC/R518从总DNA中成功扩增出16S rDNA片段,然后对PCR产物进行DGGE分析,并将DGGE图谱上部分条带回收、再扩增、克隆和测序;将所测序列在GenBank核酸数据库中进行检索,用BLASTN分析DNA序列获得相似性最近的细菌。DGGE图谱显示,四角蛤蜊体内的细菌种类比较丰富,且不同时间样品细菌的优势菌群结构有一定的差异。将所测条带序列进行比对,结果表明,细菌种群中包括支原体属Mycoplasma、假单胞菌属Pseudomonas、弧菌属Vibrio、红球菌属Rhodococcus和鞘氨醇单胞菌属Sphingomonas及不可培养的细菌。
关键词:四角蛤蜊;PCR-DGGE;细菌多样性
四角蛤蜊Mactra venerformis肉质细嫩、味道鲜美,是中国颇具养殖前途的滩涂贝类。四角蛤蜊营埋栖生活,生活在潮间带的中、低潮区及浅海的泥砂滩。一般情况下,四角蛤蜊体内存在着维持微生态平衡的正常菌群,但在其抗病力低下或环境不利时,此类细菌可大量繁殖,引起感染。因此,了解四角蛤蜊体内正常细菌的菌群组成,对其健康养殖是很重要的。有关贝类体内细菌种群组成的调查已有一些报道[1-3],研究中使用的是传统微生物学方法。近年来,PCR-DGGE技术已被广泛地应用到微生物多样性研究中。Hovda等[4]、刘淮德等[5]和Amaro等[6]使用PCR-DGGE技术分别研究了大西洋鲑Salmo salar、南美白对虾Penaeus vannamei和海参Portuguese canyons肠道中细菌的多样性。本试验中,作者采用PCR-DGGE技术对四角蛤蜊体内的细菌多样性进行了研究,旨在为促进该贝类的健康养殖提供基础资料。
1 材料与方法
1.1 材料
于2009年6月2日、8月30日、11月20日和2010年3月30日、5月4日从盘锦某滩涂取四角蛤蜊,置于加冰的保温箱中运回实验室,用体积分数为70%的乙醇对四角蛤蜊进行体表消毒,用无菌刀开启,取出肌肉和内脏,置于研钵中研碎,取适量放入Eppendorf管中,于-20℃下保存。
1.2 方法
1.2.1 总DNA的提取和PCR扩增 于Eppendorf管样品中加入567 μL DNA提取液(包括100 mmol/L Tris-HCl、100 mmol/L EDTA、100 mmol/L磷酸钠、1.5 mol/L NaCl、10 g/L的CTAB、pH为8.0)和3 μL蛋白酶K,于37℃下水浴30 min;加入30 μL 10 g/L的SDS,充分混匀,于65℃下水浴2 h,期间不断混匀,取出后置于室温下离心10 min(12 000 r/min);取上清液置于另一个1.5 mL的离心管中,加入等体积的Tris-饱和酚抽提,以12 000 r/min于4℃下离心10 min;取上清液,用混合液[φ(酚)∶φ(氯仿)∶φ(异戊醇)= 25∶24∶1和φ(氯仿)∶φ(异戊醇)=24∶1]各抽提一次,于4℃下离心10 min(12 000 r/min);取上清液,加入0.6倍体积的异丙醇于室温下沉淀20 min,于4℃下以12 000 r/min离心20 min;去
上清液,用体积分数为70%的乙醇洗涤沉淀,于室温下干燥30 min后溶于30 μL无菌TE缓冲液中,加入3 μL RNA酶,于37℃下水浴30 min,取出后置于-20℃下保存。以提取的总DNA为模板,用引物F338GC(5'-CGC CCG CCG CGC GCG GCG GGC GGG GCG GGG GCA CGG GGG GAC TCC TAC GGG AGG CAG CAG-3')与R518(5'-ATT ACC GCG GCT GCT GG-3')扩增四角蛤蜊细菌DNA的16S rDNA片段。50 μL反应体系中含5 μL 10×PCR Buffer,4μL dNTPs,各2μL(20 mmol/L)引物,2.5 U Taq酶,2 μL DNA模板, 34 μL ddH2O。反应程序为:94℃下预变性5 min; 94℃下变性1 min,65℃下复性1 min(每个循环降低0.5℃),72℃下延伸1 min,共进行24个循环;94℃下变性1 min,53℃下复性1 min,72℃下延伸1 min,共进行11个循环;最后于72℃下延伸10 min。用10 g/L的琼脂糖凝胶电泳检测PCR产物的大小。
1.2.2 DGGE分析 采用Bio-Rad公司D-code通用突变检测系统对PCR产物进行DGGE分析。将PCR反应产物直接上样于含质量分数为35%~60%的梯度变性剂的80 g/L的聚丙烯酰胺凝胶中。在TAE中电泳5 h(温度为60℃,电压为200 V),采用银染方法染色核酸,用凝胶成像仪观察。
1.2.3 DGGE条带的克隆和测序 切下DGGE条带溶于50 μL无菌水中,4℃过夜,取2 μL作为模板,用引物F338/518R进行PCR扩增,反应体系和反应程序同“1.2.1”。PCR用15 g/L的琼脂糖凝胶电泳检测,并切胶纯化。纯化后的PCR产物与pMD18-T载体连接,转化到E.coli DH5a感受态细胞中,在含有X-gal、IPTG和Amp的LB培养基平板上培养16 h(37℃);将蓝白斑筛选阳性转化子接种于LB培养基中,37℃下振荡过夜,以培养的菌液为模板,用引物RV-M/MB-47进行PCR反应检测阳性克隆,将阳性克隆产物送天根公司进行测序。将所测DNA序列在GenBank (www.ncbi.nlm.nih.gov)数据库中进行检索,从BLAST比对结果中取相似性最近的序列。
2 结果与讨论
2.1 DNA的提取和PCR扩增
提取的总DNA经10 g/L的琼脂糖凝胶电泳检测,结果见图1。从图1可见,四角蛤蜊样品DNA条带较亮,无拖带现象。
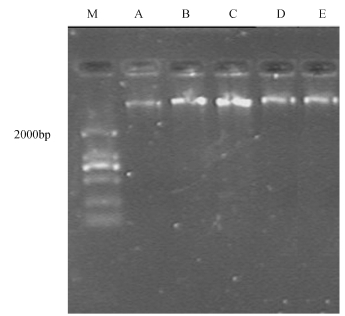
图1 对不同样品中提取的DNA的琼脂糖凝胶电泳
Fig.1 Agarose gel electrophoresis of DNA extracted from the samples collected at different time
注:A、B、C、D、E分别代表2009年6月2日、8月30日、11月20日和2010年3月30日、5月4日采集的四角蛤蜊样品,下同。
Note:Lanes A,B,C,D,and E indicate clam samples collected on 2 June, 30 August,20 November,2009,and 30 March and 4 May 2010,et sequentia.
使用通用引物F338GC/R518对总DNA进行PCR扩增,用10 g/L的琼脂糖对扩增产物进行电泳,结果见图2。通过与Marker对比,可知目标片段长度约为230 bp。该条带较亮,无拖带现象,说明PCR扩增效果良好。
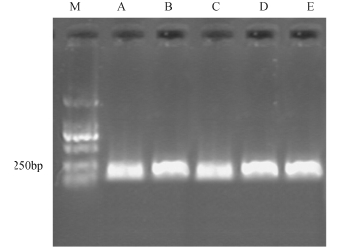
图2 对不同样品的PCR产物的琼脂糖凝胶电泳
Fig.2 Agarose gel electrophoresis of PCR products from the samples collected at different time
2.2 DGGE分析
对不同时间采集的四角蛤蜊样品进行DGGE分析,结果见图3。从图3可见,四角蛤蜊体内具有比较丰富的细菌菌群组成,不同时间采集的样品
中细菌的优势菌群组成明显不同。比较而言,2009年6月采集的样品中细菌多样性较高,有13条带; 2009年8月、2010年3月30日和5月采集的样品中各有9条带,2009年11月采集的样品中细菌多样性稍低,只有8条带。

图3 对不同月份采集的样品的DGGE凝胶电泳图
Fig.3 DGGE profile of the samples collected at different time
DGGE部分条带序列的BLAST分析结果见表1。条带A1(或E2)和条带A3分别与Mycoplasma pulmonis PG34(T)[7]和不可培养的Mycoplasma sp.clone ME97的相似性为90%和95%。条带A4与不可培养的Pseudomonas sp.clone JAB_PL01F_ E02的相似性为100%。Schulze等[8]从野生的大文蛤Panopea abrupta、成体扇贝Patinopecten yessoensis和牡蛎Crassostrea gigas体内分离出与假单胞菌属Pseudomonas spp.同源性达98%~99%的菌株,而假单胞菌属Pseudomonas是黑蛤Villorita cyprinoides var.cochinensis体内正常的细菌区系组成[3]。条带A6与Vibrio tasmaniensis UDC391(V.cyclitrophicus strain Col 15和Vibrio splendidus strain GHrC15)的相似性为100%,而弧菌属是黑蛤体内的优势种群[3]。V.cyclitrophicus和V.splendidus是Galicia养殖蛤体内的优势弧菌[9]。Schulze等[8]从野生的大文蛤、成体扇贝和幼杂色鲍Haliotis kamtschatkana体内分离出与Vibrio tasmaniensis同源性达99%的菌株。条带A7与Rhodococcus sp.的相似性为99%。红球菌属具有广泛的底物作用谱,在生物降解、生物修复、生物转化和生物表面活性剂等领域得到了越来越普遍的应用[10]。条带E1和条带E3与Sphingomonas sp.30D12和Sphingomonas sp.V1-2的相似性分别为99%和100%,后者是从Kongsfjorden的海洋沉积物中获得的[11]。Sphingomonas是一类丰富的新型微生物资源,可用于芳香化合物的生物降解,该属菌株凭借自身的高代谢能力与多功能的生理特性,在环境保护及工业生产方面具有巨大的应用潜力[12]。
表1 DGGE条带序列的BLAST结果
Tab.1 The BLAST analysis of the sequences of DGGE bands

条带号Band相似性最高的细菌Closest match登录号Accession No.相似度/% Samilarity A1 A2 A3 A4 A5 A6 A7 E1 E2 E3 Mycoplasma pulmonis strain PG34(T) Uncultured bacterium clone R76-B150 Uncultured Mycoplasma sp.clone ME97 Uncultured Pseudomonas sp.clone JAB_PL01F_E02 Uncultured bacterium isolate DGGE gel band 15 Vibrio tasmaniensis strain UDC391 Rhodococcus sp. Sphingomonas sp.30D12 Mycoplasma pulmonis strain PG34(T) Sphingomonas sp.V1-2 AF125582 AF449262 DQ917883 FJ978091 GU363940 HM031983 DQ406729 GU932978 AF125582 FN377699 90 97 95 100 100 100 99 99 89 100
3 结论
本研究中,作者采用PCR-DGGE技术分析了四角蛤蜊体内的细菌多样性。DGGE图谱显示,四角蛤蜊体内的细菌菌群较丰富,不同时间采集的样品中优势菌群的组成明显不同,6月份采集的四角
蛤蜊体内的细菌多样性较高,11月份采集的四角蛤蜊体内的细菌多样性稍低。部分DGGE条带序列比对的结果表明,四角蛤蜊体内含有支原体属Mycoplasma、假单胞菌属Pseudomonas、弧菌属Vibrio、红球菌属Rhodococcus、鞘氨醇单胞菌属Sphingomonas和不可培养的细菌。
参考文献:
[1] 刘军义.广西沿海文蛤及其生境中细菌菌群的初步调查报告[J].水产科技情报,1996,23(2):80-83.
[2] 郑国兴,周凯,于业,等.青蛤体内细菌菌群组成及致病性弧菌的初步调查[J].上海水产大学学报,1999,8(2):131-136.
[3] Lalitha K V,Surendran P K.Bacterial profile of black clam(Villorita cyprinoides var.cochinensis)and clam harvesting waters from Vembanad lake in Kerala(India)[J].Fishery Technology,2005, 42(2):183-190.
[4] Hovda M B,Lunestad B T,Fontanillas R,et al.Molecular characterisation of the intestinal microbiota of farmed Atlantic salmon (Salmo salar L.)[J].Aquaculture,2007,272(1-4):581-588.
[5] 刘淮德,王雷,王宝杰,等.应用PCR-DGGE分析南美白对虾肠道微生物多样性[J].饲料工业,2008,29(20):55-58.
[6] Amaro T,Witte H,Herndl G J,et al.Deep-sea bacterial communities in sediments and guts of deposit-feeding holothurians in Portuguese canyons(NE Atlantic)[J].Deep-Sea Research I,2009,56: 1834-1843.
[7] Pettersson B,Tully J G,Bolske G,et al.Updated phylogenetic description of the Mycoplasma hominis cluster(Weisburg et al. 1989)based on 16S rDNA sequences[J].International Journal of Systematic and Evolutionary Microbiology,2000,50:291-301.
[8] Schulze A D,Alabi A O,Tattersall-Sheldrake A R,et al.Bacterial diversity in a marine hatchery:Balance between pathogenic and potentially probiotic bacterial strains[J].Aquaculture,2006,256:50 -73.
[9] Hidalgoa R B,Cleenwerckb I,Balboaa S,et al.Diversity of Vibrios associated with reared clams in Galicia(NW Spain)[J].Systematic and Applied Microbiology,2008,31:215-222.
[10] Martínková L,Uhnáková B,Pátek M,et al.Biodegradation potential of the genus Rhodococcus[J].Environment International, 2009,35:162-177.
[11] Srinivas T N,Nageswara R S,Vishnu V R,et al.Bacterial diversity and bioprospecting for cold-active lipases,amylases and proteases,from culturable bacteria of kongsfjorden and Ny-Ålesund, Svalbard,Arctic[J].Current Microbiology,2009,59(5):537-547.
[12] 苟敏,曲媛媛,杨桦,等.鞘氨醇单胞菌:降解芳香化合物的新型微生物资源[J].应用与环境生物学报,2008,14(2):276-282.
The analysis of bacterial diversity in clam Mactra venerformis by PCR-DGGE
MA Yue-xin,WANG Ying,YANG Feng,YAN Xi-wu,HUO Zhong-ming
(Key Laboratory of Mariculture,Agriculture Ministry,PRC,Dalian Ocean University,Dalian 116023,China)
Abstract:In this study,the bacterial diversity was studied in clam Mactra venerformis collected from Panjin sea beach,Liaoning province.The genomic DNA was extracted from the calm using SDS schizolysis method.PCR amplification of the extracted bacterial 16S rDNA fragments was performed using primers F338GC and R518,and then PCR products were analyzed by DGGE.Some clear bands of 16S rDNA fragment from DGGE profiles of the samples were further recoveried,amplified,cloned and sequenced.The closest macth was observed by aligning sequences to be sequenced in the database using BLASTN program.The DGGE profiles revealed that there were abundant bacterial species in the clam samples,and various bands in the samples collected at different time.The band sequence alignment showed that bacterial populations in the clam samples included Mycoplasma,Pseudomonas,Vibrio,Rhodococcus,Sphingomonas and uncultured bacteria.
Key words:Mactra venerformis;PCR-DGGE;bacterial diversity
文章编号:2095-1388(2011)03-0243-04
中图分类号:Q938.1
文献标志码:A
收稿日期:2010-08-18
基金项目:国家“十一五”科技支撑项目(2006BAD09A09)
作者简介:马悦欣(1963-),女,教授。E-mail:mayuexin@dlou.edu.cn



