大口黑鲈遗传连锁图谱的构建
卢建峰1、2,白俊杰1,李胜杰1,樊佳佳1,徐浩1
(1.中国水产科学研究院珠江水产研究所,中国水产科学研究院热带亚热带鱼类选育与养殖重点开放实验室,广州510380; 2.上海海洋大学水产与生命学院,上海201306)
摘要:采用AFLP技术结合“拟测交”策略,以大口黑鲈Micropterus salmoides选育家系作为亲本进行单对杂交产生的F1代家系为作图群体,初步构建了大口黑鲈雌、雄性遗传连锁图谱。结果表明:用64对AFLP引物组合对父母本和106个子代个体进行遗传分析,共得到398个分离标记。母本分离标记有189个,其中172个符合1∶1孟德尔分离规律;父本分离标记有209个,其中181个符合1∶1孟德尔分离标记。雌性框架图包括75个遗传标记,分布在21个连锁群中,有6个三联体,6个连锁对,标记间平均间隔为18.22 cM,图谱总长度为983.8 cM;雄性框架图包括82个遗传标记,分布在22个连锁群中,有7个三联体,5个连锁对,标记间平均间隔为19.22 cM,图谱总长度为1153.2 cM。雌、雄性基因组估算长度分别为1 399.85和1 582.72,图谱的总覆盖率分别达到70.28%和72.86%。
关键词:大口黑鲈;AFLP;遗传连锁图谱
大口黑鲈Micropterus salmoides俗称加州鲈,原产于美国密西西比河淡水流域,是一种肉质鲜美、生长迅速、适温较广的名贵肉食性鱼类。1983年首次由台湾地区引进[1],经过20多年的养殖和推广,大口黑鲈已成为国内重要的淡水养殖种类之一,年产量逾10万t[2]。国内大口黑鲈是由野生种家养驯化而成,苗种缺少系统的人工定向选育,没有较好的遗传基础,生长速度、抗病能力还未达到良种化的程度。分子标记辅助育种技术是目前培育改良物种快速而有效的方法,进行大口黑鲈的分子标记辅助育种,首先要构架高密度的遗传连锁图谱。遗传连锁图谱作为其中一个重要的基础环节,为重要经济性状的基因(生长、抗病等)定位与克隆提供前提,使得分子标记辅助选择育种的应用成为现实。AFLP标记具有高度的特异性、快速性和高效性,且实验结果稳定可靠,易于标准化,是当今构建遗传连锁图谱最有效的方法之一,已被广泛应用于水产动物的连锁图谱构建中[3-6]。
“拟测交”策略创立初期主要用于植物作图,最早的报道见于利用RAPD标记构建桉树的遗传连锁图谱[7],其优点是仅需要两代系谱的材料,便可在较短的时间内构建生物的连锁图谱。鱼类一般具有较强的繁殖力,可以从相对较少的亲本中获得作图群体(与高等植物相类似),且普通群体往往处在相对较高的杂合状态,以此为基础的杂合群体更能满足作图的需要。近年来“拟测交”策略已成功应用于很多鱼类的图谱构建中[8]。本研究中,作者采用“拟测交”策略,利用AFLP分子标记成功构建了大口黑鲈的遗传连锁图谱,这将为定位、克隆大口黑鲈的重要数量、质量性状基因以至最终实现标记辅助选育提供重要的基础资料。
1 材料与方法
1.1 材料
试验用大口黑鲈取自广东佛山市南海区大口黑鲈选育基地。其家系构建于2008年珠江水产研究所观赏鱼培育基地,作图所用家系为大口黑鲈单对亲本繁殖所获得的F1代群体。人工繁殖培育按大口黑鲈的常规育苗方法进行,F1代群体生长2月龄后随机采集106尾个体,活体抽取血液后,加入抗凝剂,保存待用。
1.2 方法
1.2.1 基因组DNA的提取 按照上海Sangon公司DNA抽提试剂盒的方法提取血液DNA,用10 g/L琼脂糖凝胶电泳检测DNA的完整性和纯度并估计其浓度,用无菌双蒸水稀释至浓度为50 ng/μL, -20℃下保存备用。
1.2.2 AFLP反应 AFLP试验流程参照Vos等[9]的方法操作,稍加变动。
1)基因组DNA的双酶切和连接。采用酶切和连接同时进行的方法,反应体系为:10 U/μL EcoR I 0.5 μL,10 U/μL Mse I 0.5 μL,100×BSA 2 μL,20 U/μL EcoR I adapter 0.4 μL,20 U/μL Mse I adapter 0.4 μL,5 U/μL T4-DNA Ligase 1 μL,10 ×T4DNA连接酶连接缓冲液1.5 μL,50 ng/μL DNA 4 μL,加水至20 μL。37℃下温浴8 h即可用于后续扩增试验,剩余酶切连接液于-20℃下保存。
Mse I接头序列:5'-GACGATGAGTCCTGAG-3';3'-TACTCAGGACTCAT-5'。
EcoR I接头序列:5'-CTCGTAGACTGCGTACC -3';3'-CTGACGCATGGTTAA-5'。
2)PCR预扩增。取4 μL的连接产物用3'端带有1个选择性碱基的引物进行预扩增[9]。预扩增反应体系为:50 ng/μL EcoR I primer 0.6 μL,50 ng/μL Mse I primer 0.6 μL,25 mmol/L dNTPs 0.3 μL,25 mmol/L Mg2+1.3 μL,10×PCR buffer 2.0 μL,5 U/μL Taq DNA polymerase 0.12 μL,酶切连接后模板4.0 μL。
预扩增反应程序为:94℃下预变性2 min;94℃下变性30 s,56℃下退火30 s,72℃延伸1 min,共进行30个循环;72℃下再延伸5 min。预扩增产物用0.1倍的TE缓冲液稀释20倍,作为选择性反应的模板。
3)引物筛选和选择性扩增。通过预备试验,筛选出扩增产物稳定、重复性好、多态性高且分辨能力强的引物对各样品基因组DNA预扩产物进行选择性扩增。选择性扩增反应体系为:50 ng/μL EcoR I primer 0.8 μL,50 ng/μL Mse I primer 0.8 μL,25 mmol/L dNTPs 0.2 μL,25 mmol/L Mg2+1.3 μL,10×PCR buffer 2.0 μL,5 U/μL Taq DNA polymerase 0.12 μL,预扩增产物4.0 μL。
选择性扩增PCR反应程序为:94℃下预变性2 min;94℃下变性30 s,65℃下退火30 s,72℃下延伸1 min,共进行12个循环,每个循环退火温度降低0.7℃,至56℃;然后再在94℃下变性30 s,56℃下退火30 s,72℃下延伸1 min,再进行23个循环。
4)凝胶电泳和银染。电泳:在选择性扩增产物中加入等体积变性缓冲液,混匀,95℃下变性5 min后立刻转移到冰浴中冷却,以备电泳。制备质量分数为6%的聚丙烯酰胺变性凝胶,25 W恒功率下预电泳30 min,50 W恒功率下电泳2 h。
银染:电泳后进行硝酸银染色,染色方法参照文献[10]略加改进。银染结束后,将胶放入Alphaimage凝胶成像系统拍照,然后将其做成干胶,长久保存。
1.2.3 AFLP标记的统计分析 AFLP是显性标记,相应的基因型分别为AA、Aa及aa,统计表型的时候只有“有”和“无”两种,分别记录为“1”和“0”。利用卡方(P<0.05)进行检验,筛选出多态标记在子代的分离中呈现出1∶1孟德尔分离规律,符合规律的分离标记用来构建大口黑鲈的遗传连锁图谱。
1.2.4 标记的命名 连锁群上的标记名称由四个位置的数字或字母组成,如D5f289,具体含义如下:第一位是一个大写字母,表示EcoR I的选扩引物;第二位的数字,表示Mse I的选扩引物;第三位是小写字母f,表示的是fragment;第四位的数值,表示该标记片段长度的估计值,此数值采用软件AlpaEaseFC 4.0预测。
1.2.5 连锁分析 按照软件MAPMAKER/EXP 3.0[11]的计算方法将父母本和子代符合1∶1分离标记分别转化为MAPMAKER/EXP 3.0的数据格式,即0转化为A,1转化为0,设置LOD≥3.0,两个标记间的最大距离为50 cM(厘摩),图距通过Kosambi[12]函数计算,单位为cM。连锁图谱通过作图软件Mapdraw 2.1[13]进行绘制,连锁群按由大到小顺序排列。
1.2.6 遗传图谱的平均间隔、实际长度、预期长度和覆盖率的计算 连锁图谱的标记平均间隔为图谱长度除以总间隔数。间隔数加上1即为每个连锁群上的标记数。遗传连锁图谱的长度有两种计算形式:第一种形式为所有连锁标记的总长度,其中包括三联体和连锁对的长度(Goa)[14];第二种形式为框架图的长度(G),图谱预期长度采用G[15]
ofe1和Ge2[16]两种计算方法,将两种方法的平均值作为图谱的估计长度(Ge)。
框架图覆盖率Cof=Gof/Ge,
总的图谱覆盖率Coa=Goa/Ge。
2 结果与分析
2.1 AFLP扩增结果分析
64种选扩引物组合中共检测到2 135个分子标记,有398个标记在大口黑鲈群体中呈现多态性,平均每对引物产生7.55个标记;多态性片段中有189个母本分离标记和209个父本分离标记,其中有45个偏离孟德尔1∶1分离规律(P<0.05),17个属于母本,28个属于父本,偏分离比例为11.3%。
2.2 遗传连锁图谱
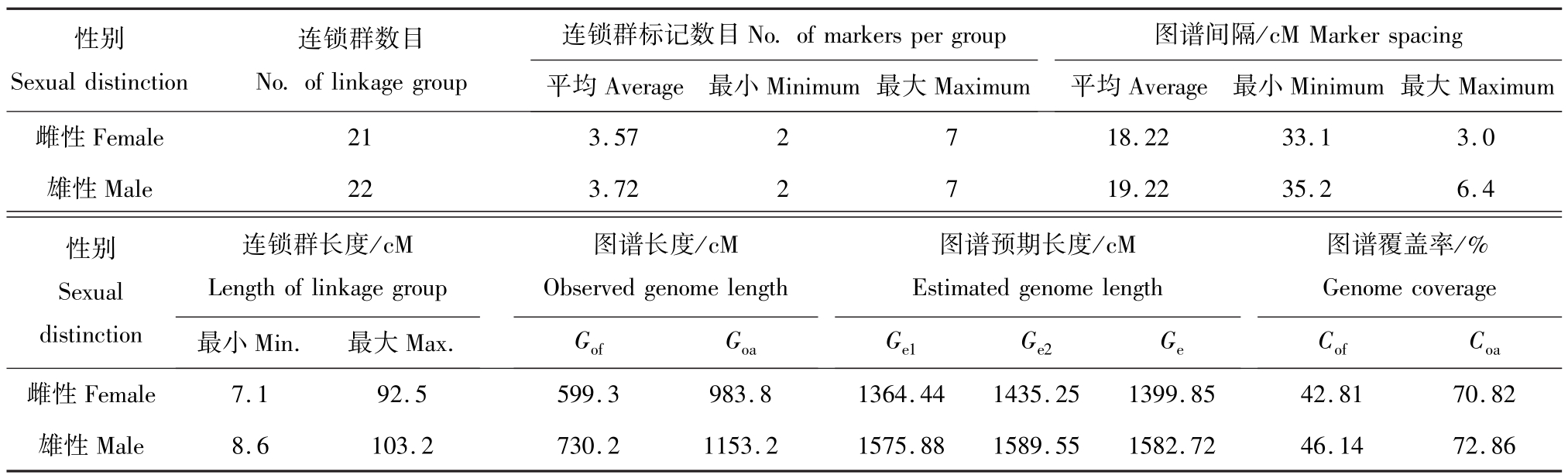
在大口黑鲈雌性连锁图谱中,符合孟德尔分离规律的172个母本分离标记中有75个标记连锁,分布在21个连锁群上(图1)。其中遗传标记数量大于3的连锁群有9个,三联体有6个,连锁对有6个。在雄性连锁图谱中,符合孟德尔分离规律的181个母本分离标记中有82个标记连锁,分布在22个连锁群上(图2)。其中遗传标记数量大于3的连锁群有10个,三联体有7个,连锁对有5个。雌雄连锁图谱的相关数据见表1。
表1 大口黑鲈雌性、雄性连锁图谱相关数据
Tab.1 Summary of female/male linkage maps for largemouth bass Micropterus salmoides
性别Sexual distinction Maximum雌性连锁群数目No.of linkage group No.of markers per group平均Average最小Minimum最大连锁群标记数目图谱间隔Maximum /cM Marker spacing平均Average最小Minimum最大Female213.572718.2233.13.0雄性Male223.722719.2235.26.4性别Sexual distinction连锁群长度/cM Length of linkage group最小Min.最大Max.图谱长度/cM Observed genome length GofGoa图谱预期长度/cM Estimated genome length Ge1Ge2Ge图谱覆盖率/% Genome coverage CofCoa雌性Female7.192.5599.3983.81364.441435.251399.8542.8170.82雄性Male8.6103.2730.21153.21575.881589.551582.7246.1472.86
3 讨论
本研究中采用“拟测交”策略结合AFLP标记技术,构建了大口黑鲈第一个分子标记遗传连锁图谱。雌性图谱包括21个连锁群,框架图总长度为983.8 cM;而雄性图谱包含22个连锁群,总长度为1 153.2 cM。遗传连锁群是生物单倍体染色体数目的一个间接反应,理论上图谱的覆盖率达到一定程度时,连锁群的数目应该与生物的单倍体染色体数目相一致。大口黑鲈染色体数目为2n=48,单倍体染色体数目为24,而雌、雄性图谱可能因标记数目不够导致连锁群少于染色体的数目。这种现象在已构建的多个连锁图谱中都有报道,特别在首次构建一个物种的遗传连锁图谱时常常会出现[17-19]。显然,还需要将更多的标记添加到连锁图谱中,以检测到潜在的连锁信息,达到连锁群数与相应的单倍体染色体数相一致。
本试验中,共产生398个AFLP分离标记,有45个标记偏离孟德尔分离规律,占11.3%。其中,母本标记中偏分离比例为9.0%,父本标记的偏分离比例为13.4%。偏分离现象在作图过程中经常出现。在研究水产动物的遗传连锁图谱时,除日本对虾Penaeus japonicus和鲇以外,其它绝大多数的种类都出现偏分离现象。究竟是什么原因导致偏分离现象,目前尚无答案。笔者推测可能有以下几个原因:第一,不同位点的片段相对分子质量大小相同,因而在一条AFLP电泳条带上可能出现多个位点扩增产物的重叠现象[20-21];第二,AFLP标记依据酶切及两步PCR扩增,低质量基因组DNA不完全的酶切以及PCR扩增过程中的偏差都会导致隐性纯合位点增多,即出现纯合子,另外,酶切位点或选择性碱基结合位点的任何突变都会引起该位点在检测过程中的缺失,同样会造成纯合子过剩;第三,较高的遗传负荷和对有害隐形基因的选择作用,导致了纯合子缺失,从而引起偏分离[22]。这些有害基因很有可能在生物发育阶段就开始起作用。已有研究表明,幼虫期的大量死亡导致太平洋牡蛎Crassostrea gigas出现严重的偏分离现象[23]。
许多研究证明,AFLP标记是非常有效的作图标记。本研究中同样也显示了AFLP标记技术的高效性,在短期内收集了相当数量的标记,并被成功地运用到大口黑鲈初级遗传图谱的构建中。但本研究中每对AFLP引物在大口黑鲈作图群体中仅产生7.55条多态性片段,低于其他很多学者在水产动物中每对引物产生10~20条多态性片段的研究报道[24-26]。分析这种差异,可能是由于研究的物种
不同或统计方法不一致而引起的。另一方面,本研究中采用传统的聚丙烯凝胶电泳和银染法分离检测PCR扩增产物,与毛细管电泳技术和荧光显色技术的测序仪检测方法相比,检测效率和灵敏度相对较低。这也可能是本试验中每对AFLP引物检测到的多态片段少于其它相关研究的主要原因之一。

图1 大口黑鲈雌性遗传连锁图谱
Fig.1 Female linkage map in largemouth bass Micropterus salmoides
注:连锁群按照由大到小依次命名和排序;连锁群右侧为标记名称,左侧为相邻两标记之间的图距(cM),下同。
Note:Linkage groups were named and arranged according to map length;markers were indicated on the right,and distances between markers(in Kosambi cM)on the left,et sequentia.
参考文献:
[1] 梁素娴,孙效文,白俊杰,等.微卫星标记对中国引进加州鲈养殖群体遗传多样性的分析[J].水生生物学报,2008,32(5):80 -86.
[2] Bai J J,Lutz-Carrillo D J,Quan Y C,et al.Taxonomic status and genetic diversity of cultured largemouth bass Micropterus salmoides in China[J].Aquaculture,2008,278(1-4):27-30.
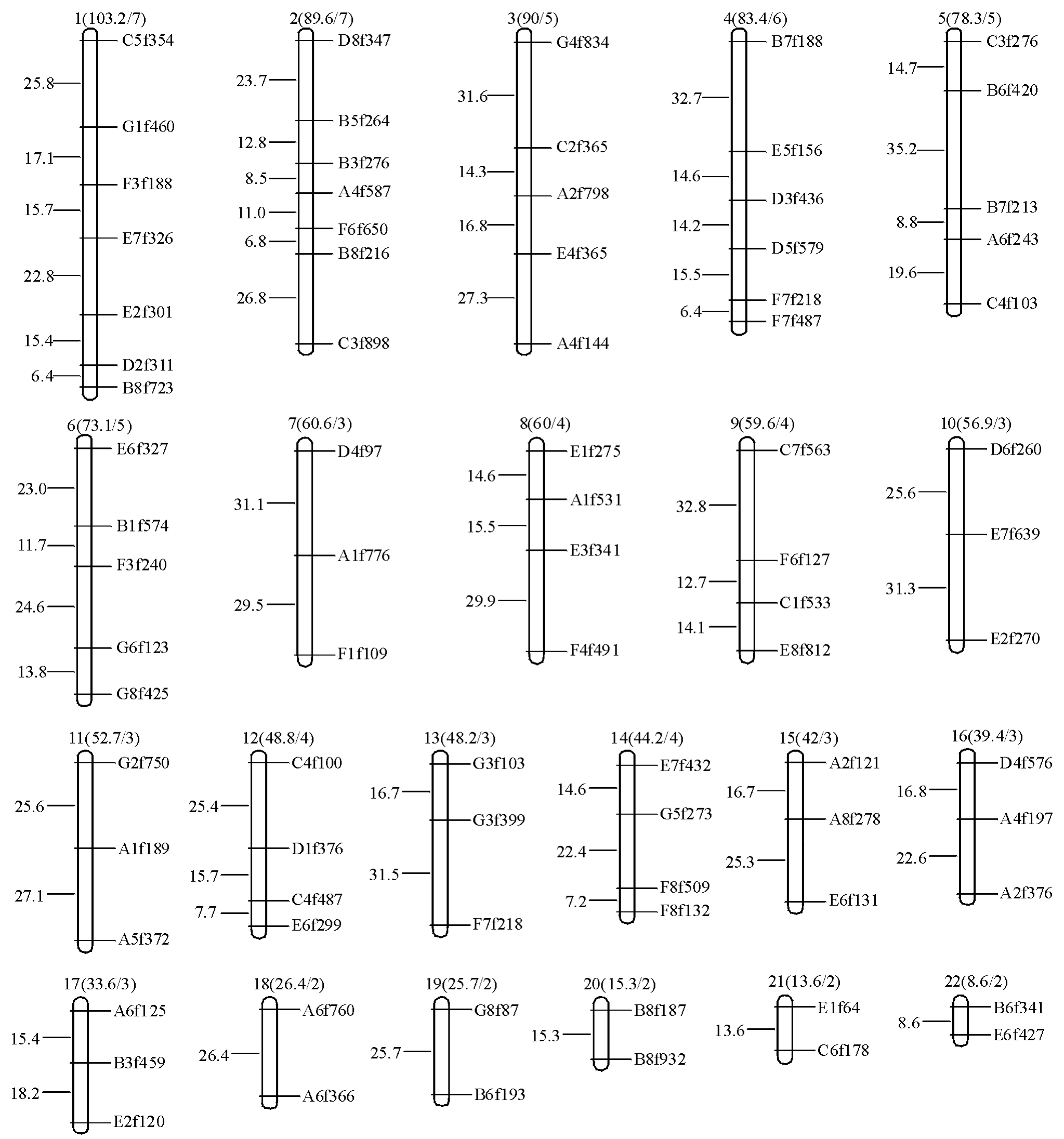
图2 大口黑鲈雄性遗传连锁图谱
Fig.2 Linkage map of largemouth bass Micropterus salmoides male
[3] 李健,刘萍,王清印,等.中国对虾遗传连锁图谱的构建[J].水产学报,2008,32(2):161-173.
[4] 王伟继,孔杰,董世瑞,等.中国明对虾AFLP分子标记遗传连锁图谱的构建[J].动物学报,2006,52(3):575-584.
[5] Kocher T D,Lee W J,Sobolewsha H,et al.A genetic linkage map of a cichlid fish,the Tilapia(Oreochromis niloticus)[J].Genetics, 1998,148(3):1225-1232.
[6] Li L,Xiang J H,Liu X,et al.Construction of AFLP-based genetic linkage map for Zhikong scallop,Chlamys farrere Jones et preston and mapping of sex-linked markers[J].Aquaculture,2005,245 (1-4):63-73.
[7] Grattapaglia D,Sederoff R.Genetic linkage maps of Eucalyptus grandis and Eucalyptus urophylla using a Pseudo-Testcross:mapping strategy and RAPD markers[J].Genetics,1994,137:1121-1137.
[8] Kai W,Kikuchi K,Fujita M.A genetic linkage map for the tiger pufferfish,Takifugu rubripes[J].Genetics,2005,171:227-238.
[9] Vos P,Hogers R,Bleeker M,et al.AFLP:a new technique for DNA
fingerprinting[J].Nucleic Acids Res,1995,23(21):4407-4414.
[10] Bassam B J,Caetano-Anolles G,Gresshoff P M.Fast and sensitive silver staining of DNA in polyacrylamide gels[J].Anal Biochem,1991,196(1):80-83.
[11] Lander E S,Green P,Abrahamson J,et al.MAPMAKER,an interactive computer package for constructing primary genetic linkage map of experimental and natural population[J].Genomics,1987, 1(2):174-180.
[12] Kosambi D D.The estimation of map distances from recombination values[J].Ann Eugen,1944,12(2):172-175.
[13] Voorrips R E.MapChart:software for the graphical prephical presentation of linkage maps and QTLs[J].J Hered,2002,93:77-78.
[14] Cervera M T,Storme V,Ivens B,et al.Dense genetic linkage maps of three Populus species(Populus deltoids,P.nigra and P. trichocarpa)based on AFLP and microsatellite markers[J].Genetics,2001,158(2):787-809.
[15] Postlethwait J H,Johnson S L,Midson C N,et al.A genetic linkage map for the zebrafish[J].Science,1994,264(29):699-703.
[16] Chakravarti A,Lasher L K,Reefer J E.A maximum likelihood method for estimating genome length using genetic linkage data [J].Genetics,1991,128(1):175-182.
[17] Wang L,Song L,Guo X,et al.A preliminary genetic map of Zhikong scallop(Chlamys farreri Jones et preston 1904)[J].Aquaculture Res,2005,36(7):643-653.
[18] Li L,Guo X.A Primary linkage map for the Pacifc oyster Crassostrea gigas with AFLP markers[J].Journal of Shellfish Research,2002,21(1):433-438.
[19] Liu X,Liu X,Zhang G,et al.A preliminary genetic linkage map of the Pacific abalone Haliotis discus hannai Ino[J].Mar Biotechnol,2006,8(4):386-397.
[20] Faris J D,Laddomada B,Gill B S.Molecular mapping of segregation distortion loci in Aegilops tauschii[J].Genetics,1998,149 (1):319-327.
[21] Negi M S,Devic M,Delseny M,et al.Identification of AFLP fragments linked to seed coat colour in Brassica juncea and conversation to a SCAR marker for rapid selection[J].Theor Appl Genet, 2000,101(1):146-152.
[22] Yu Z,Guo X.Genetic linkage map of the eastern oyster Crassostrea virginica Gmelin[J].Biol Bull,2003,204(1):327-338.
[23] Launey S,Hedgecock D.High genetic load in the Pacific oyster Crassostrea gigas[J].Genetics,2001,159(1):255-265.
[24] Liu Z,Nichol A,Li P.et al.Inheritance and usefulness of AFLP markers in channel catfish(Ictalurus punctatus),blue catfish(I. furcatus)and their F1,F2 and backcross hybrid[J].Mol Gen Genet,1998,258(3):260-268.
[25] Moore S S,Whan V,Dacis G,et al.The development and application of genetic markers for Kuruma prawn Penaeus japonicus[J]. Aquaculture,1999,173(1-4):19-32.
[26] Li L,Guo M.AFLP-based genetic linkage maps of the Pacific oyster Crassostrea gigas Thunberg[J].Marine Biotech,2004,6 (1):26-36.
Construction of genetic linkage maps in largemouth bass Micropterus salmoides using AFLP markers
LU Jian-feng1,2,BAI Jun-jie1,LI Sheng-jie1,FAN Jia-jia1,XU Hao1
(1.Pearl River Fisheries Research Institute,Chinese Academy of Fishery Sciences;Key Laboratory of Tropical&Subtropical Fish Breeding&Cultivation,Chinese Academy of Fishery Sciences,Guangzhou 510380,China;2.College of Life Science and Technology,Shanghai Ocean University, Shanghai 201306,China)
Abstract:The linkage maps in the broodstock and F1progeny as segregating population of largemouth bass Micropterus salmoides were primarily constructed using“two way pseude testcross”strategy with AFLP markers.A total of 2 135 markers were generated in the brood stock and 106 progenies of the mapping family by using 64 AFLP primer combinations,in which 398 markers were found polymorphic between the broodstock.Chi square analysis indicated that among the 189 female and the 209 male segregating markers,172(91.0%)female and 181(86.6%)male markers were in agreement with the Mendelian segregating ratio of 1∶1,based on the female and male genetic linkage maps constructed.The female framework map was composed of 75 segregating markers in 21 linkage groups including 7 triplets and 7 doubles,covering a total of 983.8 cM with the average interval 18.22 cM between markers. The male framework map consisted of 82 markers in 22 linkage groups including 7 triplets and 5 doublets,spanning 1 153.2 cM with the average interval of 19.22 cM.The genome length of largemouth bass was estimated o be 1 399.85 cM for the females and 1 582.72 cM for the males,and the coverage of 70.28%for the female map was observed and 72.86%for the male map.The genetic linkage maps revealed that the AFLP markers seemed to be evenly distributed within the linkage groups.
Key words:largemouth bass;genetic linkage map;AFLP
文章编号:2095-1388(2011)02-0138-06
中图分类号:Q75
文献标志码:A
收稿日期:2010-06-03
基金项目:国家科技支撑项目(2006BAD01A1209);农业部公益性行业科研专项(200903045);农业部“948”项目(2010-Z9);广东省科技计划项目(2007B020708008)
作者简介:卢建峰(1985-),男,硕士研究生。E-mail:lujianfeng0122@163.com
通信作者:白俊杰(1957-),男,研究员。E-mail:jjbai@163.net


