表1 上下游简并引物
Tab.1 Upstream and downstream degenerate primers

Block号BlockNo.引物序列(5'-3')primers(5'-3')简并度degreeofdegeneracyBLOCKBCTCAGTACATCTCCTACGTGaartaytggtg4BLOCKETCCGGATCGGCACcanykyttytc64
摘要:为克隆草鱼Ctenopharyngodon idellus多聚免疫球蛋白受体pIgR基因,本研究中借助于NCBI、Blast等数据库及Primer Premier 5.0、DNAMAN等生物软件,利用CODEHOP法设计了简并引物,并对克隆基因进行同源性比较和系统发生分析。结果表明:用CODEHOP法设计的简并引物特异性强,试验成功率高;草鱼pIgR基因属于整个脊椎动物的pIgR基因群,与鲤Cyprinus carpio同源性最高,为84%。
关键词: 草鱼;CODEHOP;多聚免疫球蛋白受体 (pIgR)
黏膜免疫系统作为第一道抵抗外界病毒和细菌入侵的特异性免疫防御屏障,在免疫应答与免疫保护过程中起着极其重要的作用[1-3]。多聚免疫球蛋白受体(polymeric immunoglobulin receptor,pIgR)是由黏膜上皮细胞合成,是重要的免疫因子,能够与分泌型免疫球蛋白(Ig)多聚体结合,介导其跨过上皮细胞进行转运和分泌,进而保证分泌型免疫球蛋白在黏膜防御屏障中发挥局部清除病原体及毒素的作用,因此,pIgR的有效分泌是保证分泌型免疫球蛋白发挥黏膜防御功能的必要条件[4-7]。目前,pIgR基因已在人类、哺乳动物(牛、鼠等)、鸟类和两栖类中克隆较多[5,8-9]。在硬骨鱼中仅有几种鱼类得到克隆,包括对红鳍东方鲀Takifugu rubripes、斑马鱼Danio rerio、大西洋鲑Salmo salar、鲤Cyprinus carpio、牙鲆Paralichthys olivaceus、斜带石斑鱼Epinephelus coioides、大菱鲆Scophthalmus maximus和虹鳟Oncorhynchus mykiss[10-16]等pIgR基因全长cDNA 序列的克隆,对其他鱼类未见有关于pIgR基因克隆的报道。本研究中,首次克隆了草鱼Ctenopharyngodon idellus pIgR 基因,为探索其在鱼类黏膜免疫中的功能及深入研究pIgR的转运机制提供理论支撑。
用传统简并引物设计出的引物简并度一般会比较高,同时因引物长度一般小于25个,导致引物退火温度(Tm)值会偏低,进而造成假阳性较多,不利于RT-PCR扩增[17]。采用CODEHOP法设计引物可降低引物简并度,提高引物Tm值,从而增强反转录PCR(reverse transcription-PCR,RT-PCR)扩增产物的特异性。目前,国内关于应用CODEHOP法克隆水产动物基因仅在对半滑舌鳎Cynoglossus semilaevis CYP17 基因的克隆中报道过[18],本研究中采用CODEHOP(COnsensus-DEgenerate Hybrid Oligonucleotide Primers)在线软件设计草鱼pIgR基因片段的简并引物,并利用生物软件Primer Premier 5.0及DNAMAN对CODEHOP设计的简并引物进行分析,克隆草鱼pIgR基因,该方法较传统设计简并引物的方法有较大改进[17,19],所设计的简并引物能更好地与模板进行匹配,从而更加有利于RT-PCR试验的成功,也为今后克隆水产动物基因提供了新的技术支撑。
1.1 材料
1.1.1 试验鱼 草鱼取自山东省淡水渔业研究院,平均体长为36 cm,平均体质量为531 g,保存于液氮中。
1.1.2 主要数据库和生物软件 NCBI(http://www.ncbi.nlm.nih.gov/)、CODEHOP(http://blocks. fhcrc.org/codehop.html)、Block Maker(http://blocks.fhcrc.org/blockmkr/make_blocks. html)、Primer Premier 5.0、Mega 5.2、DNAMAN。
1.1.3 菌体与质粒 克隆菌体采用大肠杆菌DH5α,质粒采用pMD18-T Vector,均购自TaKaRa公司。
1.1.4 试剂 Trizol试剂购自Invitrogen公司,Gengreen购自北京鼎国昌盛生物技术有限责任公司,凝胶回收试剂盒(Gel Extraction Kit)、反转录试剂盒均购自Omega公司,LA Taq 酶、DNA Marker、克隆载体试剂盒均购自TaKaRa公司。
1.2 方法
1.2.1 简并引物的设计 首先在NCBI 数据库中搜索并从中找出3个与草鱼亲缘关系较近的鱼类pIgR氨基酸序列,分别为鲤Cyprinus carpio (GenBank 登录号:ADB97624) 、斑马鱼Danio rerio(GenBank 登录号:ABQ10652) 、大西洋鲑 (GenBank 登录号:ACX44838),将上述3种鱼类的pIgR氨基酸序列保存为FASTA格式,并提交至BlockMaker 网站,得到保守区后,从BlockMaker Results网页直接登录CODEHOP 数据库在线进行引物设计。设计引物的主要参数:Degeneracy为128,Temperature为60.0 ℃,Genetic code为standard,Codon usage table为Danio rerio。引物密码表中没有草鱼选项,所以选择进化地位较近的模式生物D.rerio。
1.2.2 简并引物的筛选 首先根据简并引物设计的普遍原则和引物退火温度值,筛选出分值较高的引物,尽量选择分数大于75分的引物。然后根据简并引物在氨基酸序列保守区内的位置,筛选出简并度较低的引物。再在3种鱼中选出其中一种鱼(与要克隆的物种亲缘关系尽量相近),采用DNAMAN查看筛选出的简并引物在其mRNA模板中的位置,并分析简并引物与模板mRNA的匹配程度,选取匹配程度较高的引物。最后再利用Primer Premier 5.0进行简并引物自身的检测:(1)分析两条上、下游简并引物自身是否存在发夹结构;(2)两条上、下游简并引物之间是否存在二聚体;(3)两条上、下游简并引物的Tm值不要相差太大,尽量不要超过5 ℃。
1.2.3 简并扩增草鱼pIgR基因 选取草鱼肝组织进行总RNA提取。RNA提取步骤按照Trizol试剂盒说明书进行,操作过程中注意防止RNA酶污染。反转录过程按照cDNA第一链合成试剂盒说明书进行,再用已经筛选好用CODEHOP法设计的简并引物及用传统方法设计的同样保守区的简并引物进行RT-PCR扩增。
筛选出的用CODEHOP法设计的简并引物:
上游引物5′ CTCAGTACATCTCCTACGTGaartaytggtg 3′;
下游引物5′ TCCGGATCGGCACcanykyttytc 3′。
筛选出的用传统方法设计的简并引物:
上游引物5′ CGCARTACRYCAGYHAYGTGAA-GTACTGGTG 3′;
下游引物5′ HCCACTRCGACACCAMYGCTTCT-C 3′。
PCR 反应体系(共25 μL): 10×Taq buffer 2.5 μL,dNTP(2.5 mmol/L) 2 μL,cDNA模板 0.5 μL、上、下游引物各1 μL,Taq DNA Polymerase 0.5 μL,ddH2O 17.5 μL。采用降落PCR的反应程序:94 ℃下预变性5 min;94 ℃下变性30 s, 64~55 ℃下退火30 s(每一循环降1 ℃,之后为55 ℃),72 ℃下延伸90 s,共进行30个循环;最后在72 ℃下延伸10 min。将PCR产物于4 ℃下保存。
1.2.4 PCR 产物分析 反转录PCR产物在含Gengreen的琼脂糖凝胶(10 g/L)上进行检测,经检测含有目的片段后利用Omega琼脂糖凝胶DNA回收试剂盒进行回收。将回收的特异性目的片段采用克隆载体试剂盒连接入pMD18-T Vector,转化至大肠杆菌DH5α中,在氨苄青霉素平板上涂布,挑取平板上的单菌落,以载体通用引物(Primer RV-M和Primer M13-47)进行PCR检测,经检测正确后,摇菌并测序。
1.2.5 序列分析 将生工生物工程(上海)有限公司的测序结果通过NCBI Blast进行序列比对,分析草鱼pIgR基因序列与其他物种的同源性。
1.2.6 系统进化分析 将不同物种的pIgR氨基酸序列通过ClustalX进行多序列比对后,用Mega 4.0软件的邻位相接法(Neighbor-joining,NJ)构建系统进化树。
2.1 用CODEHOP设计简并引物
采用BlockMaker对鲤、斑马鱼、大西洋鲑的pIgR氨基酸序列进行保守区分析比对后,共得到7个非常保守的氨基酸区域。再采用CODEHOP设计的简并引物,经过DNAMAN、Primer Premier 5.0分析,最终筛选出如表1所示的1对引物。
表1 上下游简并引物
Tab.1 Upstream and downstream degenerate primers

Block号BlockNo.引物序列(5'-3')primers(5'-3')简并度degreeofdegeneracyBLOCKBCTCAGTACATCTCCTACGTGaartaytggtg4BLOCKETCCGGATCGGCACcanykyttytc64
注:大写字母表示5′端的非简并夹板区,小写字母表示3′端的核心简并区
Note:Capital letters represent 5′non- degenerate consensus clamp,and letters represente 3′ degenerate core
2.2 简并PCR电泳结果
以草鱼肝脏RNA为模板(图1),用CODEHOP设计的简并引物进行扩增,电泳结果如图2-A所示,在455 bp处出现一条特异性条带,与推断的结果基本一致,并且没有出现非特异性条带。而用传统方法设计的简并引物进行扩增,电泳结果如图2-B所示,在400~500 bp处出现多条带,PCR扩增产物特异性不强。因此,在后续进行PCR产物分析及测序时均采用CODEHOP设计的简并引物及其进行扩增的PCR产物。
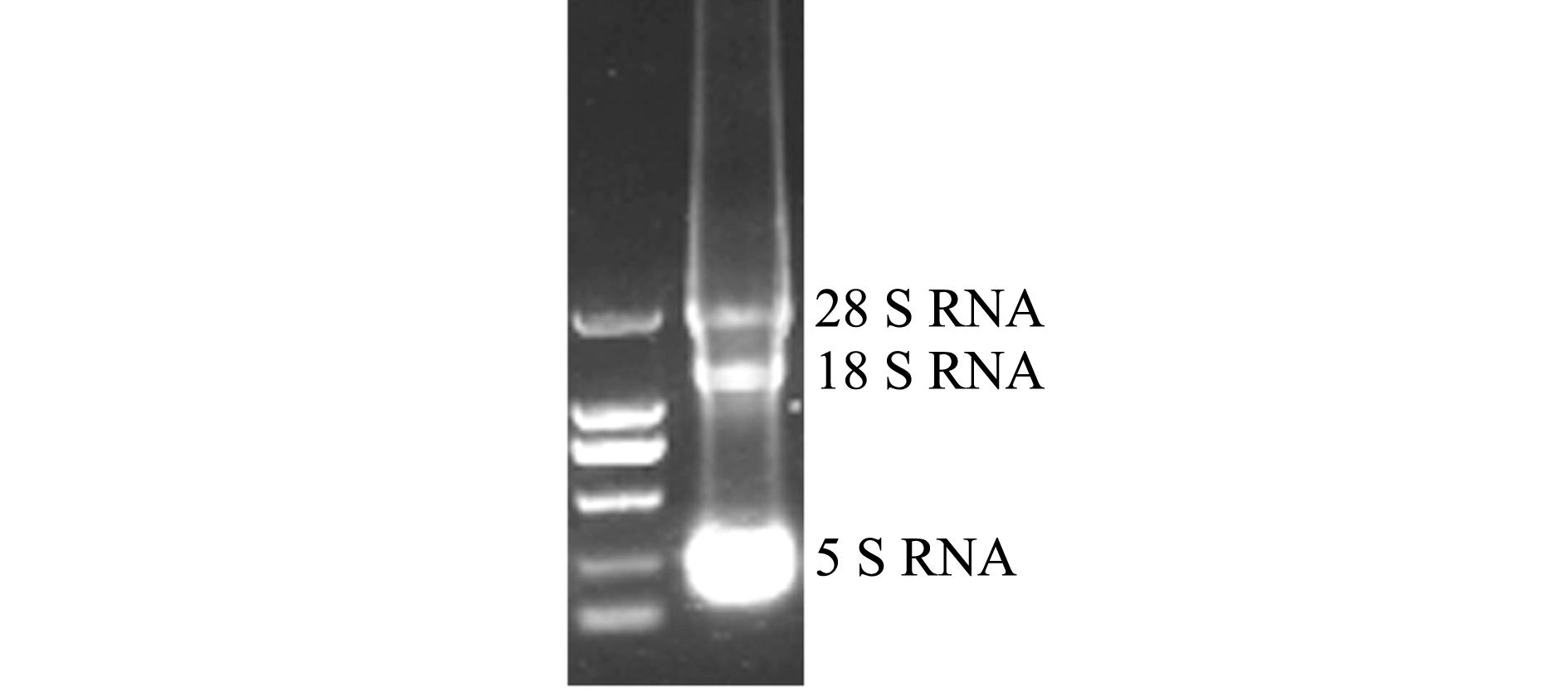
图1 草鱼肝组织总RNA琼脂糖电泳结果
Fig.1 Agarose electrophoresis of total RNA in hepatopancreas of grass carp Ctenopharyngodon idellus
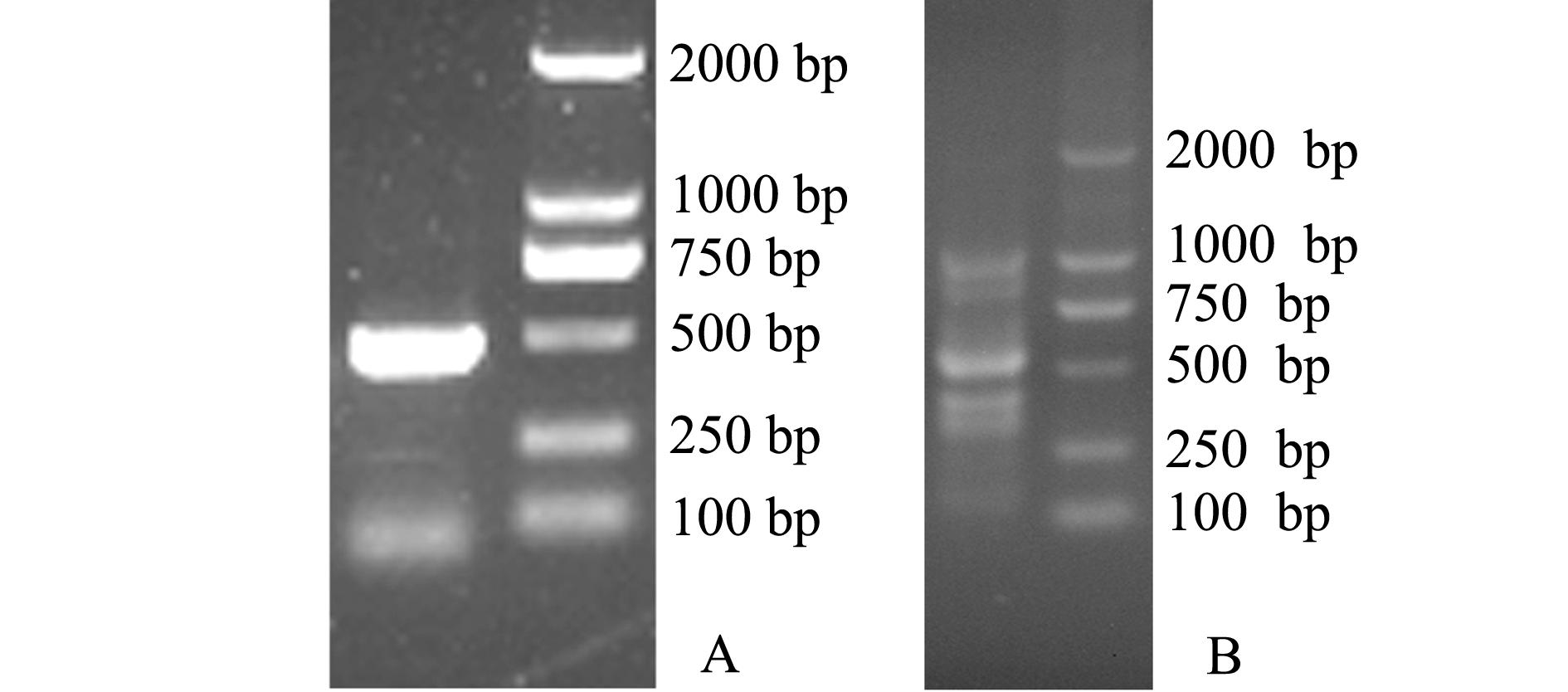
注:A为CODEHOP法;B为传统方法
Note:A,CODEHOP;B,the traditional method
图2 草鱼肝组织RT-PCR电泳图谱
Fig.2 Electrophoresis of RT-PCR in hepatopancreas of grass carp Ctenopharyngodon idellus
2.3 测序结果及序列分析
测序结果如图3所示,将测序结果通过NCBI BlastX进行同源性比较,结果如表2所示。从表2可见:克隆的草鱼pIgR氨基酸序列与鲤的同源性最高(84%),其次为斑马鱼(75%),与虹鳟、大西洋鲑、斜带石斑鱼、大菱鲆、红鳍东方鲀、牙鲆、大西洋鳕pIgR氨基酸序列的同源性为50%~59%。由此可见,草鱼pIgR基因片段属于整个脊椎动物的pIgR基因群。经Sequin提交,得到草鱼pIgR基因序列的GenBank登录号为KP238177,该片段编码151个氨基酸。

图3 草鱼pIgR基因片段
Fig.3 Fragment of pIgR gene from grass carp Ctenopharyngodon idellus
表2 pIgR基因片段的Blast同源性比较结果
Tab.2 Blast homologous comparison of pIgR gene fragment
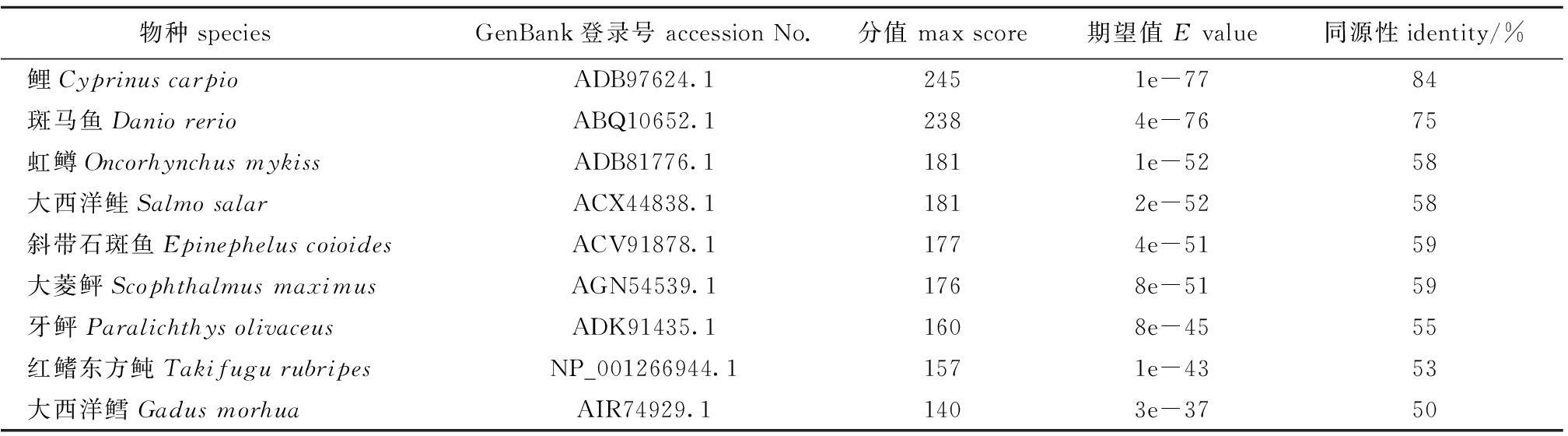
物种species GenBank登录号accessionNo.分值maxscore期望值Evalue同源性identity/%鲤CyprinuscarpioADB97624.12451e-7784斑马鱼DaniorerioABQ10652.12384e-7675虹鳟OncorhynchusmykissADB81776.11811e-5258大西洋鲑SalmosalarACX44838.11812e-5258斜带石斑鱼EpinepheluscoioidesACV91878.11774e-5159大菱鲆ScophthalmusmaximusAGN54539.11768e-5159牙鲆ParalichthysolivaceusADK91435.11608e-4555红鳍东方鲀TakifugurubripesNP_001266944.11571e-4353大西洋鳕GadusmorhuaAIR74929.11403e-3750
2.4 系统进化分析
通过构建18种动物的pIgR 氨基酸序列系统进化树,研究草鱼pIgR在进化上的保守性。其中9种动物pIgR氨基酸序列的GenBank登录号见表2,除鱼草外其余8种动物及pIgR 氨基酸序列的GenBank登录号分别为人Homo sapiens(CAA51532.1)、褐家鼠Rattus norvegicus(EDM09843.1)、牛Bos taurus(AAC41620.1)、小家鼠Mus musculus(AAA67440.1)、马Equus caballus(ACX69975.1)、鸡Gallus gallus(AAP69598.1)、非洲爪蛙Xenopus laevis(ABK62772.1)和眼镜王蛇Ophiophagus hannah(ETE63349.1)。从图4可见:该进化树主要分成3部分,第一部分是哺乳类(包括人、牛、马、小家鼠、褐家鼠),第二部分是鸟类(鸡)、爬行类(眼镜王蛇)和两栖类(非洲爪蛙),第三部分是硬骨鱼类;在硬骨鱼类中,草鱼pIgR氨基酸序列与鲤最接近,其次与斑马鱼较近。
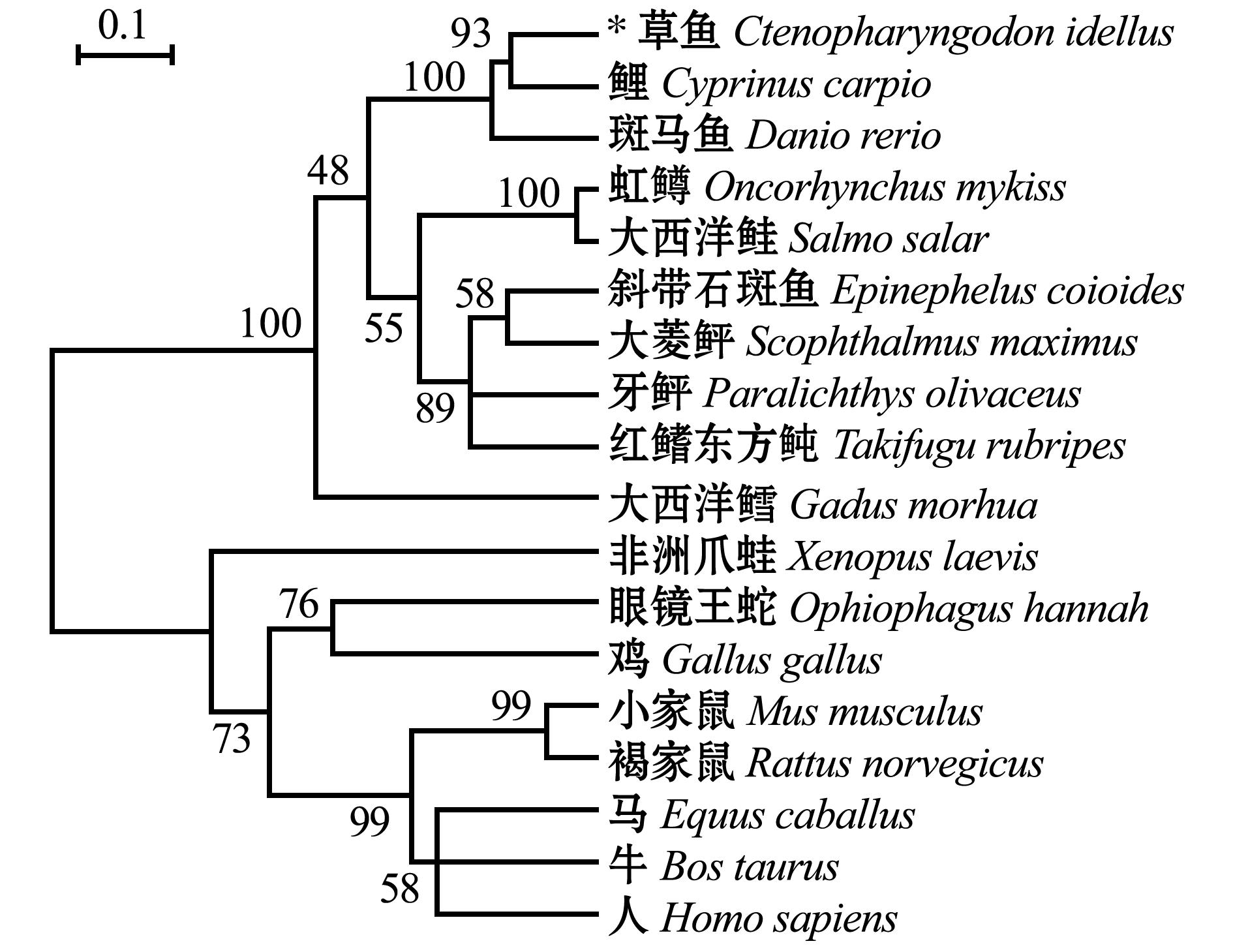
图4 根据不同物种pIgR氨基酸序列构建的系统进化树
Fig.4 Phylogenetic tree constructed for the amino acid sequences of pIgR in different species
用CODEHOP设计的简并引物与用传统方法设计的简并引物不同,分为3′端的核心简并区和5′端的非简并夹板区两部分。3′端核心简并区由小写字母表示,是由9~12个编码高度保守的氨基酸碱基序列组成,在RT-PCR扩增中与最相近的cDNA进行匹配;5′端是非简并夹板区,其长度略长,用大写字母表示。因CODEHOP法设计的简并引物的非简并夹板区最大程度地反推了保守氨基酸的碱基序列,使后续RT-PCR扩增具有较强的特异性,也使得CODEHOP方法优于特异性较差的传统简并PCR方法。传统方法通常不适用于扩增物种间亲缘关系较远或序列拷贝数较低的情况,而CODEHOP法则适用[18,20]。在用CODEHOP设计简并引物时,会检索出大量的引物序列,因此,需要进行仔细筛选。本研究中,先利用CODEHOP设计出简并引物,再用DNAMAN对上下游引物与其中1种鱼模板mRNA进行比对,查看其匹配程度,再用Primer Premier 5.0对简并引物自身进行分析(发夹结构、Tm值、二聚体),从而为后续试验的成功奠定了基础。本试验结果表明,用CODEHOP法设计的简并引物进行RT-PCR扩增,产物特异性强,没有杂带,明显优于传统方法。
在脊椎动物黏膜免疫中,多聚免疫球蛋白的转运和分泌需要一系列免疫分子和免疫细胞的协同作用。其中多聚免疫球蛋白受体pIgR就是起重要作用的免疫分子,它可以很好地与分泌型多聚免疫球蛋白结合,从而介导其向黏膜扩散。在哺乳动物中,pIgR含有5个类似Ig结构功能区(Ig-like domain,ILD1~ILD5),鸟类(鸡)和两栖类(非洲爪蟾)pIgR含有4个Ig样功能区[6,9],而鱼类pIgR被证明仅含有2个Ig样功能区,分别和哺乳动物的ILD1和ILD5相对应[10-16]。对红鳍东方鲀和牙鲆的研究结果表明,鱼类pIgR中2个ILDs能很好地结合分泌型Ig[10,16],说明鱼类pIgR转运分泌型四聚体IgM需要ILDs功能区少,而转运哺乳动物、鸟类或两栖类中分泌型IgA二聚体或IgA三聚体,则需要4~5个ILDs功能区。Norderhaug等[21]研究表明,如果pIgR敲除Ig样功能区2和Ig样功能区3,则不能与分泌型IgA多聚体结合。推测原因,可能是因为IgA二聚体或IgA三聚体中间的亚基距离较远,而IgM四聚体则缩小了亚基之间的空间距离,因此,需要pIgR的ILDs功能区也较少。但真正的结合模式还需要进行深入地研究[22]。本研究中成功地克隆了草鱼pIgR基因,经分析得出,草鱼pIgR基因属于整个脊椎动物的pIgR基因群,尤其是硬骨鱼类亚群。
本研究为后续研究pIgR在健康草鱼不同组织中的差异表达情况及免疫应答特征、蛋白结构特征奠定了基础,不仅丰富了鱼类黏膜免疫方面的基本理论,用于更好地指导生产实践,更为深入地研究草鱼pIgR在黏膜免疫球蛋白转运过程中的作用机制提供了理论支撑。
参考文献:
[1] Santos N M S D,Taveme-Thiele J J,Bames A C,et al.The gill is a major organ for antibody secreting cell production following direct immersion of sea bass (Dicentrarchus labrax L.) in a Photobacterium damselae ssp.piscicida bacterin:an ontogenetic study[J].Fish and Shellfish Immunology,2001,11(1):65-74.
[2] Xu D H,Klesius P H,Shelby R A.Cutaneous antibodies in excised skin from channel catfish,Ictalurus punctatus Rafinesque,immune to Ichthyophthirius multifiliis[J].Journal of Fish Diseases,2002,25(1):45-52.
[3] Cain K D,Jones D,Raison R L.Characterization of mucosal and systemic immune response in rainbow trout (Oncorhynchus mykiss) using surface plasmon resonance[J].Fish and Shellfish Immunology,2000,10(8):651-666.
[4] Gurevich P,Zusman I,Moldavsky M,et al.Secretory immune system in human intrauterine development:immunopathomorphological analysis of the role of secretory component (pIgR/SC) in immunoglobulin transport (review)[J].International Journal of Molecular Medicine,2003,12(3):289-297.
[5] Kaetzel C S.The polymeric immunoglobulin receptor:bridging innate and adaptive immune responses at mucosal surfaces[J].Immunological Reviews,2005,206:83-99.
[6] Braathen R,Hohman V S,Brandzaeg P,et al.Secretory antibody formation:conserved binding interactions between J chain and polymeric Ig receptor from humans and amphibians[J].Journal of Immunology,2007,178(3):1589-1597.
[7] 唐庆娟,戚欣,耿美玉.多聚免疫球蛋白受体(pIgR) 在黏膜免疫中的重要功能[J].中国生物化学与分子生物学报,2007,23(9):724-729.
[8] Asano M,Saito M,Fujita H.Molecular maturation and functional expression of mouse polymeric immunoglobulin receptor[J].Journal of Immunological Methods,1998,214:131-139.
[9] Wieland W H,Orzaez D,Lammers A,et al.A functional polymeric immunoglobulin receptor in chicken (Gallus gallus) indicates ancient role of secretory IgA in mucosal immunity[J].Biochemical Journal,2004,380:669-676.
[10] Hamuro K,Suetake H,Saha N R,et al.A teleost polymeric Ig receptor exhibiting two Ig-like domains transports tetrameric IgM into the skin[J].Journal of Immunology,2007,178:5682-5686.
[11] Rombout J H W M,van der Tuin S J L,Yang G,et al.Expression of the polymeric Immunoglobulin Receptor (pIgR) in mucosal tissues of common carp (Cyprinus carpio L.)[J].Fish and Shellfish Immunology,2008,24:620-628.
[12] Feng L N,Lu D Q,Bei J X,et al.Molecular cloning and functional analysis of polymeric immunoglobulin receptor gene in orange-spotted grouper (Epinephelus coioides)[J].Comparative Biochemistry and Physiology B,2009,154:282-289.
[13] Zhang Y A,Salinas I,Li J,et al.IgT,a primitive immunoglobulin class specialized in mucosal immunity[J].Nature Immunology,2010,11:827-835.
[14] Tadiso T M,Sharma A,Hordvik I.Analysis of polymeric immunoglobulin receptor and CD300-like molecules from Atlantic salmon[J].Molecular Immunology,2011,49(3):462-473.
[15] Xu G J,Zhan W B,Ding B J,et al.Molecular cloning and expression analysis of polymeric immunoglobulin receptor in  ounder (Paralichthys olivaceus)[J].Fish and Shellfish Immunology,2013,35:653-660.
ounder (Paralichthys olivaceus)[J].Fish and Shellfish Immunology,2013,35:653-660.
[16] 丁冰洁,绳秀珍,唐小千.大菱鲆多聚免疫球蛋白受体基因的克隆及表达分析[J].中国水产科学,2013,20(4):792-801.
[17] 黄菁,王少丽,乔传令.程序化设计简并引物与克隆小菜蛾酯酶基因[J].昆虫知识,2002,39(6):458-461.
[18] 陈彩芳,温海深,何峰,等.程序化设计的简并引物克隆半滑舌鳎CYP17基因[J].中国海洋大学学报:自然科学版,2009,39(6):1213-1218.
[19] 夏瑞,陆旺金,李建国.简并引物的程序化设计与荔枝HMGR基因片段的克隆[J].果树学报,2006,23(6):903-906.
[20] 李运合,孙光明.利用CODEHOP和iCODEHOP设计简并引物克隆芒果LAX基因家族片段[J].热带作物学报,2011,32(12):2278-2282.
[21] Norderhaug I N,Johansen F E,Krajci P,et al.Domain deletions in the human polymeric Ig receptor disclose differences between its dimeric IgA and pentameric IgM interaction[J].European Journal of Immunology,1999,29(10):3401-3409.
[22] 王磊.鲤鱼多聚免疫球蛋白受体(cpIgR)功能的研究[D].济南:山东师范大学,2009:27-29.
Abstract: The gene of polymeric immunoglobulin receptor(pIgR)of grass carp Ctenopharyngodon idellus was cloned by a pair of degenerate primers designed by CODEHOP, and by combined bioinformatics including NCBI, Blast and biological software such as Primer Premier 5.0 and DNAMAN, and the homology and phylogenetic tree of the clone of pIgR gene were analyzed in the PCR products. The results showed that degenerate primers designed by CODEHOP were characterized by specificity and benefit for the experiment accomplishing. pIgR gene of grass carp was found to clustered into pIgR gene group in all the other vertebrates, especially into the fish subgroup, with the maximal homology (84%) with common carp Cyprinus carpio.
Key words: Ctenopharyngodon idellus; CODEHOP; polymeric immunoglobulin receptor (pIgR)
基金项目: 国家自然科学基金资助项目(31440090);山东省农业良种工程项目;山东省财政支持农业技术推广项目
收稿日期: 2014-12-29
中图分类号:S965.111;Q512.2
文献标志码:A
文章编号:2095-1388(2015)02-0138-05
DOI:10.3969/J.ISSN.2095-1388.2015.02.005